2. 西南大学药学院, 重庆 400715
2. College of Pharmacy, Southwest University, Chongqing 400715, China
雷公藤属植物 (Tripterygium Hook. F) 是雷公藤类制剂主要原料来源,具有清热解毒、祛风通络、舒筋活血、消肿止痛、杀虫止血等功效; 研究表明,其具有免疫调节、抗炎、抗菌、抗癌等药理作用,临床上广泛用于治疗类风湿性关节炎、红斑狼疮、银屑病等多种自身免疫性疾病,是疗效确切的中药免疫抑制剂[1-4]。
《中国植物志》中文版 (Flora Republicae Popularis Sinicae,2009) 将我国雷公藤属植物分为3个种: 昆明山海棠 (T. hypoglaucum (Lévl.) Hutch)、雷公藤 (T. wilfordii Hook. f.) 和东北雷公藤 (T. regelii Sprague et Takeda); 而《中国植物志》英文修订版 (Flora of China,2013) 将上述3个种合并为雷公藤。该属植物药材主要来源于野生采挖,其自然生长周期为4~5年,更新缓慢; 经过近20余年的开发,原盛产雷公藤属植物的地区,现今资源蕴藏量正在锐减 (浙江、福建、江西、安徽等地雷公藤野生资源已基本枯竭; 四川、云南、贵州等地昆明山海棠野生资源亦在锐减)[5, 6],雷公藤属药用植物野生资源受到严重威胁。
为避免雷公藤属植物种质资源濒临灭绝,有必要对其遗传变异进行系统研究,为其种质资源、良种选育、分类依据和保护策略等提供有效参考。目前,已有的雷公藤属植物分子遗传研究分别在ISSR[7]、SRAP[7]、RAPD[8, 9]、cpDNA[10]、5S rDNA[11]和ITS[11]等序列上进行了该属植物的遗传多样性分析。但由于目前研究报道大多针对昆明山海棠和雷公藤,对同属的东北雷公藤研究甚少,使雷公藤属植物的遗传结构研究缺乏完整性; 同时仅通过对少数地区少数样本的遗传多样性展开研究,无法为雷公藤属植物野生资源的保护和利用提供有效指导。
简单重复序列 (simple sequence repeat,SSR),广泛存在于基因组中,由2~6个核苷酸为单位重复串联组成,不同生物个体在相同的SSR位点上单位片段重复数量可能不完全相同,因而形成多态性,即SSR分子标记。SSR标记变异率高、信息量大、位点数量多、类型丰富,对DNA数量及纯度要求不高,两端序列保守,结果的重复性好; 在兼具RFLP和RAPD优点的同时,SSR弥补了上述二者不能检测基因组串联重复序列,操作繁复、耗时等缺点,具有更好的重复性和稳定性[12, 13]。目前,SSR分子标记已逐步应用于忍冬 (Lonicera japonica Thunb.)[14]、柴胡 (Bupleurum chinense DC.)[15]、苎麻 (Boehmerianivea (L.) Gaud.)[16]和百合属 (Lilium L.)[17]等药用植物遗传多样性分析。本文使用SSR分子标记技术,对全国9个省份28个雷公藤属植物居群进行遗传多样性和遗传 结构研究,进而为该属植物种质资源保护、遗传多样性分析和分子标记辅助育种等方面研究提供理论依据。
材料与方法实验材料 供试野生雷公藤属植物新鲜叶片样本来自28个居群共计396个样本,于2014年6月~9月间,在我国四川、贵州、重庆、湖南、云南、浙江、江西、湖北、吉林等9个省份28个地域内收集所得; 样地和样方设置时,在路线调查中实地选择既反映雷公藤属植物野生资源分布的普遍意义又反映雷公藤属植物集中分布特点的地点设置开展样方调查,样方的选择采取随机抽样方法进行。单个植株样本之间的空间距离为1 km以上,每个居群随机选取5~20个植株样本。所有样本采用装有硅胶的自封袋密封,快速干燥,经重庆市中药研究院生药研究所瞿显友研究员鉴定,保存于重庆市中药研究院中药博物馆,样本来源具体信息见表 1。
| Table 1 Location and sampling size of genus Tripterygium populations |
DNA 提取 用改良的CTAB提取法从30 mg雷公藤属植物新鲜叶片中提取总DNA,通过1% 琼脂糖凝胶电泳检测DNA的完整性和含量,提取的DNA于-20 ℃保存。
SSR 分析方法 本实验前期以昆明山海棠为材料,采用磁珠富集法开发出48对引物; 并对开发的引物进行筛选、验证和多态性检测,筛除条带不清晰或重复性差的标记,最后筛选出19对SSR多态性高、重复性好的引物 (表 2),用于本实验的遗传多样性分析。SSR-PCR扩增反应体系为20 μL,包括ddH2O 14.8 μL、dNTP 0.4 μL (2.5 mmol·L-1)、Buffer 2 μL、F-primer 0.3 μL、R-primer 0.3 μL、DNA模板2 μL (50~100 ng·L-1)、Taq酶0.2 μL (5 U·L-1); 反应在A&B 7900型PCR仪上进行,扩增程序为: 94 ℃预变性5 min; 变性30 s (94 ℃),54 ℃复性35 s,72 ℃延伸40 s,共35个循环; 最终72 ℃延伸3 min。将甲酰胺与分子量内标按100∶1的体积比混匀,加入上样板15 μL,再加入稀释10倍的PCR产物1 μL。用3730XL测序仪进行毛细管电泳,利用Genemarker中的Fragment (Plant) 片段分析软件对测序仪得到的原始数据进行分析,将各泳道内分子量内标的位置与各样品峰值的位置做比较分析,得到片段大小。
数据分析 遗传多样性分析1个引物为1个等位基因,将每个样品在各个等位基因的片段大小按照GenAlEx 6软件要求的格式录入到EXCEL中,转化格式后用POPGENE 3.2软件进行遗传多样性分析,包括计算群体的基因型频率、多态位点百分率 (percent of polymorphic loci,PPL)、等位基因数 (the number of alleles,A)、有效等位基因数 (effective number of allele,Ne)、期望杂合度 (expec ted het erozygosity,He)、观察杂合度 (observed heterozygosity,Ho)、Nei遗传多样度 (H)、固定指数 (F)、F-统计量和基因流 (gene flow,Nm)。用Nei’s的遗传一致度 (genetic identity) 和遗传距离 (genetic distance) 来衡量各种群之间的遗传分化大小。
群体遗传结构分析使用Structure软件检测群体间遗传结构。Structure使用一种基于模型的贝叶斯聚类法将各个个体分配到不同的聚类中去,它不需要群体和相关的地理信息,而是基于对个体基因型的分析,对每个群体样本量的大小没有要求[18]。采用Evanno等统计值ΔK确定个体聚类的数量K值,ΔK是通过相邻K值间L (K) 的第二顺序改变率 (the second order rate of change) 计算得到[19]。Structure运行的相关参数如下: K = 1~28,混合模型 (admixture model),马尔科夫链蒙特卡洛 (Markov Chain Monte Carlo simulation,MCMC) 的统计方法,10次独立运行软件,并将Bum in和Run length分别设为30 000和100 000。使用Structure Harvester计算ΔK值[20]并确定最优K值。根据Structure的聚类结果将28个群体分组; 使用Arlequin对雷公藤属群体进行遗传变异的分子方差 (AMOVA) 分析[21],估测遗传变异在物种间、物种内群体间、群体内个体间的分配情况,以进一步揭示物种和群体的分化程度,分析采用1 000次置换检验。使用Population软件构建基于Nei’s遗传距离的邻接树 (neighbour-joining tree)。
| Table 2 Sequences of SSR primers |
通过19对多态性引物对雷公藤属植物3个物种、28个群体、396个样本进行遗传多样性研究。从19个位点的等位基因来看,观测等位基因数 (A)、有效等位基因数 (Ne) 的范围分别是6~43、2.061 2~26.566 8,平均值分别为13.684 2、5.326 8。A的最高位点是P35,最低是P29和P3; Ne的最高位点是P35,最低是P29,A和Ne的变化规律基本一致。各位点的群体遗传学参数I(Shannon’s信息指数)、Ho、He、H、PIC (多态信息含量) 的平均值分别为1.711 1、0.652 8、0.728 7、0.727 8、0.697 2。I的最大值和最小值分别为3.431 5 (P35) 、0.895 1 (P29) ; Ho的最大值和最小值分别为0.912 3 (P29) 、0.115 5 (P35) ; He的最大值和最小值分别为0.963 6 (P35) 、0.515 5 (P29); H的最大值和最小值分别为0.962 4 (P35) 、0.514 9 (P29) ; PIC的最大值和最小值分别为0.961 0 (P35) 、0.450 2 (P29) (表 3)。根据遗传多样度H的结果,可以看出各个位点的多态性水平较高。
| Table 3 Analysis on genetic diversity of genus Tripterygium at different locus. A: The number of alleles; Ne: Effective number of allele; I: Shannon’s information index; Ho: Observed eterozygosity,He: Expected heterozygosity; H: Nei’s expected heterozygosity; PIC: Polymorphism information content |
群体中,等位基因数 (A)、有效等位基因数 (Ne) 的范围分别是1.473 7~5.052 6、1.349 5~3.064 7,平均值分别为2.842 1、1.964 8。表明雷公藤属植物群体存在等位基因差异,但差异不明显。最大值A在群体吉林省临江 (LJ) 的群体,最小值在群体贵州省印江 (YJ); 最大值Ne在群体江西省南昌 (NC) 的群体,最小值在群体贵州省印江 (YJ); 说明从等位基因的变异来看贵州省印江 (YJ) 群体的变异最差。A和Ne最小值出现在群体湖北通城 (TC),两个结果可以初步看出其群体的数量较小应是引起其变异较差的主要原因。各群体遗传学参数I、Ho、He、H的范围分别为0.233 6 (YJ)~1.174 2 (LJ)、0.466 2 (LQ)~0.817 5 (LL)、0.160 2 (YJ)~0.663 1 (NC)、0.144 2 (YJ)~0.634 2 (NC),平均值分别为0.639 4、0.654 5、0.377 5、0.359 9 (表 4)。
| Table 4 Analysis on genetic diversity of genus Tripterygium populations. A: The number of alleles; Ne: Effective number of allele; I: Shannon’s information index; Ho: Observed eterozygosity; He: Expected heterozygosity; H: Nei’s expected heterozygosity; PPL: Percentage of polymorphic loci |
结果表明,雷公藤属植物不同群体内的遗传多样性水平较高。群体之间存在一定的差异,但差异变化不大。各群体中Ho均大于He (除NC群体中,He和H几乎一致外),说明群体内几乎无杂合子缺失现象。从PPL的结果中可以看出群体贵州雷山 (LS)、贵州剑河 (JH)、江西南昌 (NC)、吉林临江 (LJ) 的多态百分比均为100%,多态性较好; 而贵州印江 (YJ) 的多态百分比为31.58%,多态性最差。贵州印江 (YJ) A、Ne、I、He、H和PPL指标中均为最小,说明其遗传多样性较低。
3 遗传结构研究F-统计量和基因流如表 5所示,Fis有9个位点为负值,其余位点均为正值,正值的范围为0.034 0~0.568 7,平均值为0.038 2,Fis出现了负值,说明杂合子过剩; Fit均为正值,范围为0.077 4~0.844 8,平均值为0.540 5。根据Fis和Fit平均值的统计结果来看,居群间近交程度较高,居群内个体间的遗传近交程度较低。
| Table 5 Analysis on inbreeding coefficient and gene flow. Fis: Mean inbreeding coefficient within individuals relative to sub population; Fit: Mean inbreeding coefficient within individuals relative to the total population; Fst: Mean inbreeding coefficient within subpopulation relative to the total population; Nm: Gene flow |
Fst范围为0.226 2~0.761 1,平均值为0.522 3,根据Fst的结果可以看出,除了位点P35为0.226 2,在0.15~0.25之间,具有较高的遗传分化水平以外,其余范围在0.307 9~0.761 1,均在0.25以上,表明雷公藤属植物居群间的遗传分化水平很高,接近48% 的遗传变异,来自于群体内部; 52% 的遗传变异,来自于群体之间,因此遗传分化存在群体之间、群体内部均存在,且几乎相当。
基因流Nm在0.078 5~0.855 2内,平均值为 0.228 7 < 1。群体间的基因交流相对较低,导致居群内遗传分化较大,因此受限的基因流是雷公藤属植物遗传分化较高的一个原因。
雷公藤属植物分子方差分析 (AMOVA) 如表 6所示,雷公藤属植物的遗传变异主要存在于群体内个体间 (45.22%),其次为物种之间 (30.49%),最后为物种内群体间 (24.29%),其结果与F-statistic的分析结果基本一致,证明雷公藤群体的遗传变异主要是来源于群体内个体间的遗传差异。
| Table 6 Analysis on molecular variance (AMOVA) of genetic diversity in genus Tripterygium. df: Degree of freedom; SS: Sum of squares |
根据Nei’s的计算方法对雷公藤属植物的群体进行遗传距离和遗传一致度的统计分析,可以看出群体间遗传距离的范围是0.026 1~4.553 3,其中遗传距离最近的是四川省米易县普威 (PW) 和四川省米易县横山 (HS) 群体,距离最远的是四川省冕宁县石龙 (SL) 和湖北省通城县 (TC) 群体; 遗传一致度的范围: 0.010 5~0.974 3,遗传一致度最高: PW和HS群体,遗传一致度最低: SL和TC群体。
根据Nei’s遗传距离构建群体聚类树 (图 1),结果显示4个东北雷公藤群体 (TH、JY、LJ、CB) 单独聚在一起; 5个雷公藤群体中的4个 (NC、WY、XS、TC) 聚在一起,浙江龙泉 (LQ) 未与其余4个雷公藤群体聚在一起,而是与昆明山海棠群体湖南南岳 (NY) 的亲缘关系更近。形态上浙江的雷公藤群体LQ是典型的过渡类型,叶片明显大于雷公藤; 四川5个昆明山海棠群体 (PW、HS、LL、SL、DZ) 首先聚在一起,然后依次与云南4个群体 (DL、NH、NJ、BS)、湖南两个群体 (XN、WG)、贵州3个群体 (YJ、LS、JH)、重庆3个群体 (JJ、WL、SZ) 聚在一起,最后与湖北利川 (LC) 和湖南南岳 (NY) 聚在一起。从群体聚类树可以看出有典型的过渡类型, 这些过渡类型在之前的研究[5, 8, 11, 22, 23]中有提过。

|
Figure 1 Neighbour-joining tree of genetic relationships among 28 populations of genus Tripterygium |
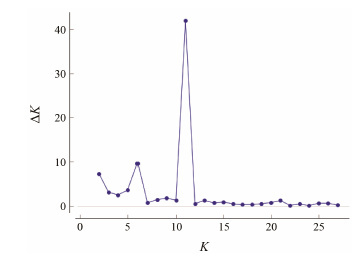
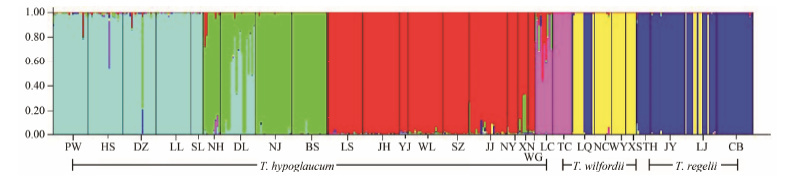
Structure聚类分析根据不同个体等位基因频率的差异对雷公藤属植物群体进行Structure聚类分析。因为群体数量为28,计算中将K值设置为1~28,根据Evanno[24]的评判标准及计算公式得ΔK与K值关系图 (图 2),发现K = 11时,ΔK值最大,但是由于K = 11的数值太大,将类群分类太细无意义,因此选取ΔK次高点对应的K值(K = 6) 。根据Structure结果,将3个物种、28个群体分成遗传组成上6个不同的组 (图 3),昆明山海棠主要被分成四川、贵州和重庆、云南3个组,东北雷公藤分成1组,典型的雷公藤被分成了1组,而过渡类型浙江省龙泉市 (LQ) 和湖南省南岳区 (NY) 则被分成1组。说明雷公藤和昆明山海棠在形态变异上和地理分布上都是连续的,有大量过渡类型存在,区分2个种群存在一定难度。
|
Figure 2 Relational graph between ΔK and K |
|
Figure 3 Structure dendrogram in clustering analysis among 28 populations of genus Tripterygium |
使用19对多态引物对雷公藤属植物3个物种、28个群体、396个样本进行遗传多样性研究。根据各位点遗传学参数I、Ho、He、H、PIC范围(0.895 1~3.431 5、0.115 5~0.912 3、0.515 5~0.963 6、0.514 9~0.962 4、0.450 2~0.961 0) 及平均值 (1.711 1、0.652 8、0.728 7、0.727 8、0.697 2) 、A、Ne及遗传学参数的结果可知,以昆明山海棠开发出的19对引物多态性水平较高,能有效地应用于雷公藤全属植物并对其遗传多样性进行研究。
物种或群体遗传多样性大小是长期进化的产物,一个群体 (或物种) 遗传多样性越高或遗传变异越丰富,对环境变化的适应能力越强,越容易扩展其分布范围和开拓新的环境[25]。本研究以广泛、大量的样本量,真实地反映了雷公藤属内遗传多样性水平: 从不同群体的A和Ne可知,群体的等位基因差异不大; 从A、Ne、I、He、H和PPL可知总体雷公藤属植物各群体存在一定差异,各群体遗传多样性均较高,表明其适应性较强。而群体间基因流水平低导致群体遗传分化程度较高,受限的基因流是雷公藤属植物遗传分化较高的一个原因。
植物群体遗传变异水平主要受到繁育系统、分布范围和生活习性的影响[25-27]; 长寿命、广域分布、远交、动物取食传播的物种具有较高遗传多样性水平[28]。本研究中雷公藤属植物群体遗传多样性较高可归因于其自身生物学特性: 多年生木本,风媒传粉,繁育系统以异交为主,繁殖方式以有性生殖为主等,这些均有利于遗传多样性的积累和保持。
空间遗传结构分析有助于探讨物种进化机制、揭示植物濒危机制; 有助于物种遗传多样性的保护与利用。植物种群的遗传结构反映了各种因素的相互作用,突变、遗传漂变和自然选择会导致群体遗传分化,促进遗传结构的形成; 而基因流则使群体遗传均质化,削弱遗传结构[29]。此外,繁殖系统也会对群体遗传结构产生影响: 具有近亲繁殖系统的物种,群体间遗传分化往往高于混合繁育和远缘杂交的物种; 自交减少了有效群体的大小,从而使遗传漂变和近交水平增加,导致群体间遗传变异增大[30]。群体遗传学中,基因流Nm = 1.0作为临界值,Nm < 1.0时,基因流不足以抵制群体内因遗传漂变而引起的分化而导致遗传结构的产生; Nm >1.0则相反[31]。
雷公藤群体的遗传变异主要来源于群体内个体间遗传差异。根据Nei’s遗传距离构建群体聚类树和Structure聚类分析,昆明山海棠被分成了四川、云南、贵州-重庆3组,东北雷公藤被单独分成1组,雷公藤被分成了2组,其中过渡类型浙江龙泉 (LQ) 和湖南南岳 (NY) 被单独分成1组。这一结果与雷公藤属植物在分类学上一直存在的争议相呼应: 昆明山海棠与雷公藤形态与性状相似,在分子水平上也难以鉴别。本实验研究结果以SSR的角度,支撑了已有研究报道[5, 8, 11, 22, 23]的观点,表明了在形态变异和地理分布上都是连续的雷公藤和昆明山海棠不能有效地被区分,存在大量的过渡类型。本研究所得结果显示雷公藤属植物遗传多样性水平较高,群体间基因流水平较低导致群体遗传分化程度较高。雷公藤属植物繁育系统以异交为主,风媒传粉,本应具有较高的基因流; 而本实验结果显示其基因流低 (Nm = 0.228 7) ,推测导致其基因流受限的原因是人类活动致使生境破碎化,人类的过度采集更加阻碍了群体间的基因交流。
雷公藤属植物是极具开发价值的野生中药材资源,目前该属植物仍具较高的遗传多样性水平和显著的遗传结构。但在本研究资源调查中发现,一些地区的该属植物被恶性杂草紫茎泽兰排挤,适生环境遭到严重破坏; 在华东地区严禁砍伐木材后,山区森林郁闭度高,喜向阳的雷公藤属植物长势渐弱,导致这些地区群体较小; 再加上人类不加节制的药用采集,导致该属植物野生群体资源日渐减少,造成有效群体的减小。
雷公藤属植物各区群和种群均具有较高遗传性,分化较高。主要以自交和种群内部近交方式进行繁殖。人为因素是其野生资源逐步枯竭的主要原因,与自身繁殖和适生能力没有太大关系,但与其他种群竞争的因素也不容忽视。受限的基因流和缩减的有效群体会使该属植物受到遗传漂变的影响,导致其遗传多样性急剧下降; 因此急需保存雷公藤属植物的遗传多样性,减少人为采集、发展人工种植; 可根据Structure结果,对每一个分组进行资源收集,以便尽量多地保存现有的遗传资源。
| [1] | Bao J, Dai SM. A Chinese herb Tripterygium wilfordii Hook. F. in the treatment of rheumatoid arthritis:mechanism, efficacy, and safety[J]. Rheumatol Int, 2011, 31: 1123–1129. DOI:10.1007/s00296-011-1841-y |
| [2] | Brinker MA, Ma J, Lipsky EP, et al. Medicinal chemistry and pharmacology of genus Tripterygium (Celastraceae)[J]. Phytochemistry, 2007, 68: 732–766. DOI:10.1016/j.phytochem.2006.11.029 |
| [3] | Wong KF, Yuan Y, Luk MJ. Tripterygium wilfordii bioactive compounds as anticancer and anti-inflammatory agents[J]. Clin Exp Pharmacol, 2012, 39: 311–320. DOI:10.1111/j.1440-1681.2011.05586.x |
| [4] | Qiu DM, Kao NP. Immunosuppressive and anti-inflammatory mechanisms of triptolide, the principal active diterpenoid from the Chinese medicinal herb Tripterygium wilfordii Hook. F[J]. Drugs R&D, 2003, 4: 1–18. |
| [5] | Si JP, Ruan XC, Guo BL, et al. Research on resources situation and sustainable utilization in Tripterygium wilfordii[J]. J Chin Med Mater (中药材), 2005, 26: 10–11. |
| [6] | Yao Y, Yang QL. Research overview on Tripterygium hypoglaucum (Lévl.) Hutch[J]. Lishizhen Med Mater Med Res (时珍国医国药), 2001, 12: 1129–1131. |
| [7] | Hu KD. ISSR and SRAP Analysis of Genetic Diversity of Tripterygium wilfordii (雷公藤遗传多样性的ISSR和SRAP分析)[D]. Fuzhou:Fujian Agriculture and Forestry University, 2011. |
| [8] | Liu WS, Guo BL, Chen YT, et al. RAPD analysis for genetic relationship and diversity of three species of genus Tripterygium[J]. China J Chin Mater Med (中国中药杂志), 2007, 32: 1615–1622. |
| [9] | Li QF. Study of Provenance Identified of Genuine Medicinal Materials (Liriope muscari and Tripterygium wilfordii) Base on RAPD Molecular Markers (道地药材短葶山麦冬、雷公藤基于RAPD种源鉴别研究)[D]. Fuzhou:Fujian Agriculture and Forestry University, 2011. |
| [10] | Liu C. Study on the Differences between Tripterygium wilfordii Hook. f. and Tripterygium hypoglaucum (Levl) Hutch Based on Genetic and Chemical Methods (基于遗传与化学的雷公藤与昆明山海棠比较研究)[D]. Beijing:Academy of Chinese Medical Sciences, 2015. |
| [11] | Sue KL, Mark PS, Natascha T, et al. Molecular analyses of the Chinese herb Leigongteng (Tripterygium wilfordii Hook. f.)[J]. Phytochemistry, 2011, 72: 21–26. DOI:10.1016/j.phytochem.2010.10.015 |
| [12] | Zhou YQ. The Application of DNA Molecular Markers in Plant Research (DNA分子标记技术在植物研究中的应用)[M]. Beijing: Chemical Industry Press, 2005. |
| [13] | Powell W, Moigante M, Andre C, et al. The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis[J]. Mol Breeding, 1993, 2: 225–238. |
| [14] | Jiang C, Yuan Y, Liu GM, et al. EST-SSR identification of Lonicera japonica Thunb[J]. Acta Pharm Sin (药学学报), 2012, 47: 803–810. |
| [15] | Zhan QQ, Sui C, Wei JH, et al. Construction of genetic linkage map of Bupleurum chinense DC. using ISSR and SSR markers[J]. Acta Pharm Sin (药学学报), 2010, 45: 517–523. |
| [16] | Zou ZZ, Chen JH, Luan MB, et al. Genetic relationship of ramie germplasm revealed by RSAP, SRAP, and SSR markers[J]. Acta Agron Sin (作物学报), 2012, 38: 840–847. |
| [17] | Yang M. SSR Research on Genetic Diversity of Germplasm Resources of Lilium Distributed in the Middle Reach of Yangtze River (长江中游地区百合属种质资源遗传多样性的SSR分析)[D]. Wuhan:Huazhong Agricultural University, 2013. |
| [18] | Pritchard JK, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155: 945–959. |
| [19] | Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Mol Ecol, 2005, 14: 2611–2620. DOI:10.1111/mec.2005.14.issue-8 |
| [20] | Earl DA, Vonholdt BM. STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conserv Genet Resour, 2011, 4: 359–361. |
| [21] | Excoffier L, Smouse PE, Quattro JM. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J]. Genetics, 1992, 131: 479–491. |
| [22] | Si JP, Ruan XC, Guo BL, et al. Study on morphological change of Tripterygium wilfordii and T. hypoglaucum[J]. J Zhejiang Sci-Tech Univ (浙江理工大学学报), 2005, 25: 1–4. |
| [23] | Ma JS, Brach AR, Liu QR. A revision of the genus Tripterygium (Celastraceae)[J]. Edinburgh J Bot, 1999, 56: 33–46. DOI:10.1017/S096042860000233X |
| [24] | Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Mol Ecol, 2005, 14: 2611–2620. DOI:10.1111/mec.2005.14.issue-8 |
| [25] | Hamrick JL, Godt MW, Sherman BL. Factors influencing levels of genetic diversity in woody plant species[J]. New Forest, 1992, 6: 95–124. DOI:10.1007/BF00120641 |
| [26] | Hamrick J, Godt M. Effects of life history traits on genetic diversity in plant species[J]. Philos T R Soc B, 1996, 351: 1291–1298. DOI:10.1098/rstb.1996.0112 |
| [27] | Ge S, Hong DY. Biosystematic studies on Adenophora potaninii korsh complex (campanulaceae) Ⅳ. Allozyme variation and differentiation[J]. Acta Phytotaxon Sin (植物分类学报), 1998, 36: 481–489. |
| [28] | Nybom H, BartishI V. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J]. Perspect Plant Ecol, 2000, 3: 93–114. DOI:10.1078/1433-8319-00006 |
| [29] | Slatkin M. Gene flow and the geographic structure of natural populations[J]. Science, 1987, 236: 787–792. DOI:10.1126/science.3576198 |
| [30] | Igarashi S, Fukuda H. The life Histories of Asian Butterflies:Vol 2[M]. Shizuoka: Tokai University Press, 2000. |
| [31] | Slatkin M. Gene flow in natural populations[J]. Annu Rev Ecol Sys, 1985, 16: 393–430. DOI:10.1146/annurev.es.16.110185.002141 |
 2017, Vol. 52
2017, Vol. 52


