2. 陕西中医药大学药学院, 陕西 西安 712046
2. College of Pharmacy, Shaanxi University of Chinese Medicine, Xi'an 712046, China
植物光合作用产生丰富的糖类,如葡萄糖、蔗糖、麦芽糖、纤维素、淀粉等,是一切生命体赖以生存的物质基础。糖类作为重要的碳源、能源、细胞组分或信号分子,对植物体生长发育、生理适应等生命活动至关重要[1,2]。在大多数植物中,蔗糖是最主要的碳原运输形式,其从源到库器官的高效运输与合理分配,对调节植物生理、形态建成等有重要意义。蔗糖在植物体内的高效运输需要共质体和质外体途径共同介导,即蔗糖在光合细胞合成积累后,靠胞间连丝、沿浓度梯度,经共质体途径运输到韧皮薄壁细胞,输出到质外体,再转运至筛管伴胞复合体 (sieve element/companion cell complex,SE-CC),最后通过维管系统运输到库器官利用或储藏[3]。研究证实,定位在伴胞质膜上的蔗糖转运蛋白 (sucrose carrier/transporter)控制着蔗糖从韧皮薄壁细胞质外体进入SE- CC的主动运输[4]。而蔗糖从韧皮薄壁细胞转运到质外体的过程由一类特殊的糖转运蛋白SWEET (sugars will be eventually exported transporters)介导[4,5]。
SWEET是一类新鉴定的、真核生物中普遍存在的多基因家族糖转运蛋白[6]。自2012年Chen等[7]首次报道拟南芥AtSWEET11、AtSWEET12蛋白的蔗糖输出作用以来,已从拟南芥、水稻、番茄和葡萄等多种植物中鉴定发现众多SWEET蛋白具有糖转运功能[7, 8, 9, 10]。除了转运蔗糖,SWEET蛋白亦可转运单糖或其他二糖,如AtSWEET16负责葡萄糖、果糖和蔗糖的转运[11]。大量证据揭示,SWEET基因家族成员在染色体、转录、翻译及翻译后、亚细胞等水平调控糖转运,参与韧皮部装载、花粉发育、种子灌浆、胁迫生理等多种生命活动过程。AtSWEET1、AtSWEET8在拟南芥花粉管中大量表达,可能为发育中的花粉管提供营养[7]。AtSWEET16参与调节拟南芥种子萌发、生长和逆境胁迫[11],AtSWEET17控制叶片中果糖的累积[12]。OsSWEET11为水稻花粉正常发育所必需[13]。除了参与植物正常生理代谢和生长发育,一些SWEET基因还参与植物-病原物相互作用。AtSWEET4、AtSWEET15和AtSWEET17响应灰霉菌 (Botrytis cinerea)侵染诱导上调[4]。OsSWEET11、OsSWEET13和OsSWEET14受水稻黄单胞杆菌 (Xanthomonas oryzae pv. oryzae)侵染诱导增强表达[13,14]。在葡萄-B. cinerea互作体系中,VvSWEET4受活性氧和细胞坏死诱导表达[10]。这些SWEET基因为病原菌调控的主要靶基因,有利于病原菌从宿主细胞质外体中汲取大量养料从而促进病害发展。
铁皮石斛 (Dendrobium officinale Kimura et Migo)为兰科(Orchidaceae)石斛属 (Dendrobium Sw.)多年生草本植物,药用部位为新鲜或干燥茎,具有益胃生津、滋阴清热、润肺止咳、明目强身等作用,是石斛属药用植物中最为珍稀名贵的物种[15]。石斛属植物主要含有多糖、生物碱、芪类以及酚类等多种活性成分,具有重要的药理活性。石斛多糖作为一类免疫增强剂,具有增强机体体液免疫、细胞免疫以及抗肿瘤、抗氧化、抑制自由基等作用,因而是石斛类药材质量鉴定的主要标准之一[16]。前期,作者利用SSH技术富集铁皮石斛种子受真菌浸染共生萌发的差异表达基因[17],分离得到一条505 bp的EST,BLASTx分析显示其与水稻OsSWEET14 (GenBank注册号B8BKP4)一致性较高。鉴于SWEET基因在植物生理代谢、生长发育及植物-微生物互作中的重要作用,该差异基因可能在铁皮石斛糖转运或种子共生萌发中起一定调控作用。本研究利用RACE技术首次从铁皮石斛中分离到一个SWEET基因cDNA全长DoSWEET1,并进行生物信息学和表达模式分析,为进一步揭示其生物学功能奠定基础。
材料与方法接种与取样 野生铁皮石斛幼苗和成熟蒴果采自浙江金华 (2014年10月),幼嫩石斛小苗处于营养生长阶段,无花蕾,株高约10 ± 2 cm,取根、茎、叶及种子样品。促铁皮石斛种子萌发真菌为胶膜菌属真菌 (Tulasnella sp.)[18]。在OMA培养基上进行种子接菌共生萌发实验[19];参照Zettler等[20]报道的种子萌发级别,取顶端分生组织伸长变粗并弯曲的3级种子作为共生萌发处理组,OMA培养基未接菌的未萌发种子为对照。所有材料液氮速冻后置-80 ℃保存备用。
RNA提取和cDNA合成 按照EASYspin植物RNA快速提取试剂盒 (Aidlab,China)操作说明制备各样品总RNA,NanoDropTM 2000分光光度计 (ThermoFisher,USA)分析RNA质量、纯度,琼脂糖凝胶电泳检测完整性。按照M-MLV Reverse Transcriptase kit (Promega,USA)操作说明,逆转录合成cDNA第一链,-20 ℃保存备用。
3'-RACE和RT-PCR验证 序列分析表明原EST的编码肽段包含N末端119个氨基酸,因此只需进行3'-RACE。设计两条基因特异引物: F1 5'- TCTCACCATCATCAACTCCATCGGCTG-3'; F2 5'- CTTTTGCTCAGAAATGGGTCACAGTCCGT-3'。按照SMARTerTM RACE cDNA Amplification Kit (Clotech,Japan)说明书,分别与试剂盒中提供的UPM引物组合进行两次3'-RACE。
DoSWEET1-F1与UPM引物组合,试剂盒中Program 1程序进行第一轮3'-RACE。反应体系为2.0 μL 10×Advantage® 2 PCR buffer、0.4 μL 10 mmol·L-1 dNTPs、0.4 μL 10 μmol·L-1 DoSWEET1-F1/UPM、 0.5 μL 3'-RACE ready cDNA、0.4 μL 5U·μL-1 50× Advatange® 2 Polymerase Mix,补ddH2O至20 μL。PCR程序为94 ℃ 3 min; 94 ℃ 30 s,68 ℃ 30 s,72 ℃ 90 s,30个循环; 72 ℃ 7 min,4 ℃保温。反应结束后,取1.0 μL产物作为模板,以DoSWEET1-F2与UPM引物组合,进行第二轮巢式3'-RACE。PCR程序为 94 ℃ 3 min; 94 ℃ 30 s,68 ℃ 30 s,72 ℃ 1 min,32个循环; 72 ℃ 7 min,4 ℃保温。PCR产物经1.5%琼脂糖凝胶电泳,TianGen胶回收试剂盒 (TianGen,China)纯化目的条带,连接至pMD18-T vector (TaKaRa,China),转化大肠杆菌 (Escherichia coli) JM109感受态细胞,随机挑选3个克隆送上海生工测序后,与原EST拼接分析,设计跨ORF引物ORF-F 5'-ATGGGTCACAGT CCGTGG-3'和ORF-R 5'-CTAGACGGGCATCATGTT AT-3',进行全长基因的RT-PCR克隆、测序验证分析。
序列分析 使用一系列网络在线工具进行DoSWEET1基因核酸及编码蛋白的生物信息学序列分析。利用NCBI的BLASTx (http://www.ncbi.nlm.nih.gov/blast/)和ORF Finder (http://www.ncbi.nlh.nih.gov/gorf/gorf.html)分析cDNA序列;用ExPASy Proteomics Server的InterProScan (http://www.ebi.ac.uk/cgi-bin/iprscan/)和PROSITE SCAN (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_proscan.html)分析DoSWEET1蛋白质的结构域和基元; Protparam (http://web.expasy.org/protparam/)和SOPMA (http://npsa-pbil.ibcp.fr/cgi-bin/secpred_sopma.pl)分析蛋白质理化性质和二级结构;采用SWISS- MODEL (http://swissmodel.expasy.org/)进行DoSWEET1蛋白质三维建模分析; SignalP 4.0 (http://www.cbs.dtu. dk/services/SignalP/)和TMHMM (
实时定量PCR分析分别用2 μg根、茎、叶、未萌发和共生萌发种子样品总RNA反转录合成cDNA,DoEF1α为内参[21],qPCR分析基因表达模式。引物为qPCR-F (5'-TGCGTCTTCGCTGCTCCT-3')和qPCR-R (5'-TTTGCCCACTCTTCAGTCATTAT-3')的扩增产物长323 bp。用ABI PRISM 7500实时荧光定量PCR仪 (Applied Biosystems,USA)进行扩增。反应体系25 μL包括2×SYBRR® Premix Ex TaqTM Master Mix (TaKaRa,China) 12.5 μL、正反向引物 (10 μmol·L-1) 0.5 μL、ROX 0.5 μL、cDNA 2 μL和ddH2O 9 μL。每个反应重复3次,包括不加模板的对照,实验重复3次。PCR程序为95 ℃ 30 s,95 ℃ 10 s,60 ℃ 45 s,40个循环,反应结束绘制融解曲线。根据ABI PRISM 7500 SDS软件 (Applied Biosystems,USA)生成的循环阈值 (cycle threshold,CT),用2-△△CT法[22]计算基因相对表达量。
结果与分析 1 DoSWEET1基因全长克隆经过两次3'-RACE反应,扩增条带经克隆、测序获得长801 bp的序列,与原EST拼接为一条1 150 bp的cDNA。BLASTx分析表明其与GenBank中已注册的多种植物SWEET基因有很高的相似性 (58%~69%)。该cDNA序列包含的开放阅读框 (open reading frame,ORF)长789 bp,5'-UTR长146 bp,3'-UTR长189 bp,具有真核生物mRNA加尾信号“AATAA”以及polyA尾巴,起始密码子附近碱基序列GAAATGG符合KOZAK规则 (即A/GNNATGG)[23]。用ORF-F/ ORF-R引物进行RT-PCR验证,获得的单一扩增条带 (图 1),克隆、测序分析显示其为完整的ORF,说明已成功获得该基因cDNA全长,定名为DoSWEET1,并提交GenBank获得注册号KT957550。
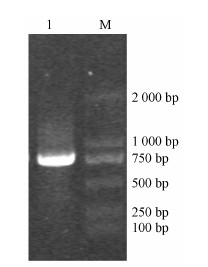 | Figure 1 RT-PCR verification of the full length cDNA of DoSWEET1 gene from Dendrobium officinale. 1: RT-PCR product; M: DL2000 standard molecular marker |
Protparam预测DoSWEET1基因编码蛋白质的分子式为C1374H2164N322O344S15,含262个氨基酸,分子质量29.18 kD,等电点9.49; DoSWEET1带正电残基 (Arg+Lys)为17,负电残基 (Asp+Glu)为16。该蛋白的不稳定系数为31.62,脂肪系数为119.47,亲水性系数为0.697。SOPMA分析表明,DoSWEET1蛋白二级结构主要由α螺旋 (alpha helix,45.80%)、延伸链 (extended strand,24.81%)、随机卷曲 (random coil,21.76%)和少量的β转角(beta turn,7.63%)组成。
3 DoSWEET1蛋白结构域、定位和跨膜区分析InterProScan分析显示,DoSWEET1蛋白含有植物SWEET家族的2个MtN3_slv结构域 (11-94、130-212)。PROSITE SCAN分析表明,DoSWEET1包含有4类数目不等的基序,包括1个N糖基化位点 (165-168)、2个cAMP/cGMP依赖的蛋白激酶磷酸化位点 (34-37、35-38)、1个酪蛋白激酶Ⅱ磷酸化位点 (122-125)和4个N-豆蔻酰化位点 (11-16、14-19、108-113、109-114)。Plant-mPLoc预测蛋白定位在质膜,SignalP 4.0预测无信号肽。图 2 TMHMM分析显示该蛋白具有7个跨膜结构域 (5-27、40-62、67-89、102-120、130-149、162-184、188-210)。
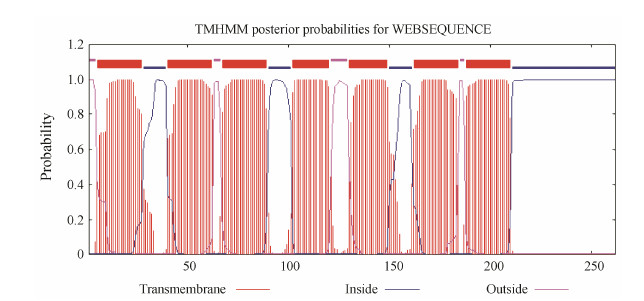 | Figure 2 The transmembrane region of DoSWEET1 protein analyzed by TMHMM (http://www.cbs.dtu.dk/services/TMHMM-2.0/) |
在SWISS-MODEL依据保守结构域作图工具中,以蛋白质数据库 (Protein Data Bank,PDB)中已提交的水稻Bidirectional sugar transporter SWEET2b (PDB No.: 5cth.1) C链晶体分子[24]为模板,对DoSWEET1蛋白进行三维结构建模 (图 3),结果显示DoSWEET1与该蛋白有35.50%的序列相似性,空间结构类似。
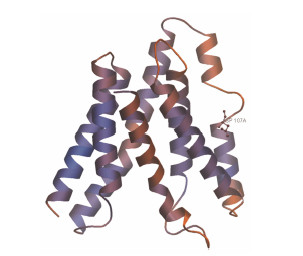 | Figure 3 Three-dimensional structure of DoSWEET1 protein using SWISS-MODEL (http://swissmodel.expasy.org/) |
运用DNASTAR 6.0的MegAlign对DoSWEET1蛋白与6种代表性植物的8个SWEET蛋白进行多序列比对。图 4结果显示,DoSWEET1蛋白与油棕 (Elaeis guineensis) EgSWEET (XP_010929602)、大豆(Glycine soja) GsSWEET14 (KHN23963)、玉米 (Zea mays) ZmSWEET (NP_001149087)和番茄 (Solanum lycopersicum) SlSWEET14 (XP_004235470)的一致性分别为54.6%、51.6%、51.1%、48.5%;与拟南芥(Arabidopsis thaliana) AtSWEET11 (Q9SMM5)、AtSWEET12 (O82587)、水稻 (Oryza sativa) OsSWEET14 (Q2R3P9)和OsSWEET11 (Q6YZF3)的一致性分别为50.8%、50.8%、52.7%、45%。此外,分析还发现,这些SWEET蛋白序列的氨基端保守性高,羧基端变异较大,7个跨膜域高度保守。
 | Figure 4 Multiple sequence alignment of DoSWEET1 and SWEET proteins from other plants. The underlined show the 7 transmembrane regions conserved among SWEET proteins |
为分析DoSWEET1基因编码蛋白的分子进化关系,从GenBank数据库中选取模式植物拟南芥和水稻SWEET家族蛋白序列,利用MEGA 6.0构建系统进化树。图 5结果表明,39个SWEET蛋白组成的无根树,隶属于MtN3-slv蛋白家族的Clade Ⅰ[8],进而分为ClassⅠa、Ⅰb、Ⅰc和Ⅱ四个分支。DoSWEET1与水稻OsSWEET13 (Q2QR07)、OsSWEET14 (Q2R3P9)和OsSWEET15 (Q6K602)亲缘关系最近且聚在一起。
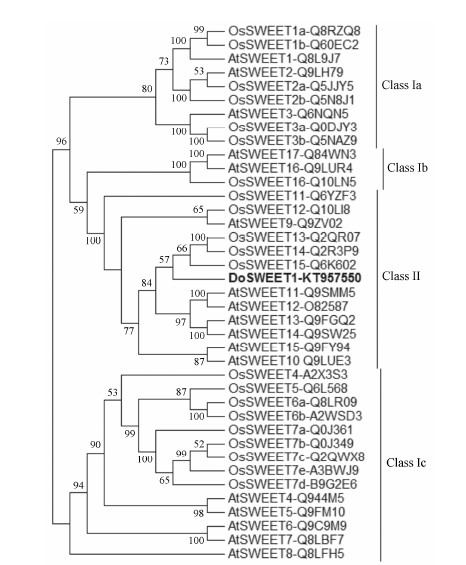 | Figure 5 Phylogenetic tree of DoSWEET1 with Arabidopsis and rice SWEET proteins |
分别提取铁皮石斛根、茎、叶、未萌发和共生萌发种子等样品总RNA并合成cDNA,利用qPCR技术检测基因组织表达模式及在种子共生萌发过程中的转录水平。图 6A结果显示,DoSWEET1在3种器官中均有表达,相对表达量存在差异。以茎为校正样本,基因转录本在石斛根中的表达量最高,叶中次之,分别为茎中的9.88倍和2.85倍;在真菌侵染石斛种子共生萌发3级样本中,DoSWEET1基因显著上调,为对照未萌发种子的1 359.06倍 (图 6B),说明该基因参与石斛种子接菌共生萌发过程。
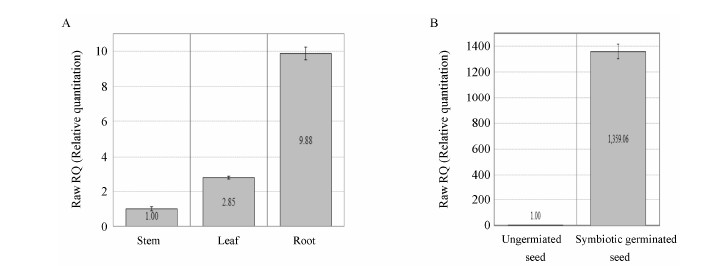 | Figure 6 Expression patterns of DoSWEET1 gene in various tissues (A) and in symbiotic germinated seed infected with fungus (B) using qPCR analysis |
SWEET蛋白介导的糖转运通过调节糖在源-库器官中的有效合理分布及其利用,进而在植物生长发育、逆境生理、植物-微生物互作等方面发挥着极其重要的作用[4, 5, 6, 7]。SWEET基因系统性分析主要来自模式植物和作物,药用植物中尚未见报道。植物SWEET呈多基因家族,拟南芥、水稻、番茄、葡萄和大豆等多种植物中分别鉴定有17、21、29、17、52个成员[7, 8, 9, 10, 25]。本研究利用RACE首次从珍稀药用铁皮石斛中分离到一个DoSWEET1基因,其编码蛋白具有SWEET蛋白的MtN3_slv保守结构域,与多种植物SWEET基因一致性较高,进化上属于MtN3-slv蛋白家族CladeⅠ的ClassⅡ分支,与水稻亲缘关系较近。这些结果说明DoSWEET1是编码铁皮石斛糖转运蛋白新基因。
蛋白质结构域和基元决定其生物学功能。系统性研究发现,SWEET蛋白是MtN3-slv蛋白家族的成员[8],佐证了DoSWEET1蛋白InterProScan预测结果。DoSWEET1蛋白预测存在7个跨膜域,N端与已知植物SWEET蛋白N端高度保守,符合SWEET蛋白典型结构特征[4]。已报道的绝大多数SWEET蛋白定位在细胞质膜上,而AtSWEET16、AtSWEET17均定位于液泡膜[11,12]。DoSWEET1预测定位在质膜上,结合跨膜域分析结果,说明该蛋白系膜蛋白。分子进化研究揭示植物SWEET蛋白包含ClassⅠa、Ⅰb、Ⅰc和Ⅱ四个分支[8],DoSWEET1属于ClassⅡ分支,与水稻聚类在一起符合进化关系。DoSWEET1基因的这些分子特征为其生物学功能研究提供参考。
SWEET基因的分子表达特性差异与其所调控的植物生理代谢关系密切。AtSWEET17在拟南芥根系中表达较其他器官显著,负责果糖的双向转运来维持根部胞质果糖稳态[12]。AtSWEET11、AtSWEET12、OsSWEET11和OsSWEET14在韧皮部薄壁细胞中表达,负责韧皮部质外体蔗糖的输出[7,8]。AtSWEET15主要在衰老组织表达,与衰老有一定关系[26]。此外,部分SWEET基因能响应生物与非生物胁迫诱导表达,参与植物逆境生理适应[27]。本研究qPCR分析表明,DoSWEET1基因在石斛根中高效表达,意味着其可能主要调节根中糖转运与贮存,叶中较高表达丰度说明该基因在光合器官糖转运中发挥一定作用。更有趣的是,共生真菌胶膜菌侵染石斛种子共生萌发三级样本, 种子顶端分生组织可见、伸长变粗并弯曲, 形态学上符合作者前期的报道[20];未萌发的种子生理生化代谢活动较低,DoSWEET1基因表达量很低,而在三级共生萌发种子中的表达量急剧升高。种子萌发生理涉及多种养料分子在细胞内的转化、合成与呼吸代谢,从植物-微生物共生互作角度,推测该基因参与了石斛种子的共生萌发,可能提供给共生真菌生活所需的糖分养料。在水稻、葡萄-病原物等互作体系中,病菌分泌的效应子结合SWEET基因的启动子,诱导SWEET蛋白大量表达,将胞内糖分输出至质外体,以供胞间病菌生长发育,导致病害加速与扩展[8,10]。目前SWEET介导的糖转运在微生物-植物共生中的作用机制尚不清楚。在铁皮石斛种子共生萌发过程中,DoSWEET1基因如何受真菌诱导?该基因编码蛋白如何起作用?如何输出糖分供给真菌利用?这些科学问题仍需要进一步深入的系统研究。
SWEET作为糖转运蛋白参与植物体内糖的积累与转运,在植物体各项生命活动过程中起重要调控作用。然而,由于SWEET基因研究起步较晚,在模式植物或农作物中的有限研究已经证实了SWEET家族成员的多样性及功能的复杂性。药用植物糖分转运与积累和SWEET蛋白密切相关。本研究后续将鉴定铁皮石斛SWEET家族其他成员,并阐明其时空表达特性、亚细胞定位、结构与功能,为揭示石斛品质形成及其与真菌共生等科学问题提供重要理论支撑。
| [1] | Wind J, Smeekens S, Hanson J. Sucrose:metabolite and signaling molecule[J]. Phytochemistry, 2010, 71:1610-1614. |
| [2] | Rolland F, Baena-Gonzalez E, Sheen J. Sugar sensing and signaling in plants:conserved and novel mechanisms[J]. Annu Rev Plant Biol, 2006, 57:675-709. |
| [3] | Slewinski TL, Braun DM. Current perspectives on the regulation of whole-plant carbohydrate partitioning[J]. Plant Sci, 2010, 178:341-349. |
| [4] | Chen LQ, Hou BH, Lalonde S, et al. Sugar transporters for intercellular exchange and nutrition of pathogens[J]. Nature, 2010, 468:527-532. |
| [5] | Braun DM. SWEET! The pathway is complete[J]. Science, 2012, 335, 173-174. |
| [6] | Xuan YH, Zhu YY, Hu YB. Research advances of the SWEET proteins family[J]. Sci Sin Vit (中国科学:生命科学), 2014, 44:676-684. |
| [7] | Chen LQ, Qu XQ, Hou BH, et al. Sucrose efflux mediated by SWEET proteins as a key step for phloem transport[J]. Science, 2012, 335:207-211. |
| [8] | Yuan M, Wang SP. Rice MtN3/Saliva/SWEET family genes and their homologs in cellular organisms[J]. Mol Plant, 2013, 6:665-674. |
| [9] | Feng CY, Han JX, Han XX, et al. Genome-wide identification, phylogeny, and expression analysis of the SWEET gene family in tomato[J]. Gene, 2015, 573:261-272. |
| [10] | Chong J, Piron MC, Meyer S, et al. The SWEET family of sugar transporters in grapevine:VvSWEET4 is involved in the interaction with Botrytis cinerea[J]. J Exp Bot, 2014, 65:6589-65601. |
| [11] | Klemens PA, Patzke K, Deitmer J, et al. Overexpression of the vacuolar sugar carrier AtSWEET16 modifies germination, growth, and stress tolerance in Arabidopsis[J]. Plant Physiol, 2013, 163:1338-1352. |
| [12] | Chardon F, Bedu M, Calenge F, et al. Leaf fructose content is controlled by the vacuolar transporter SWEET17 in Arabidopsis[J]. Curr Biol, 2013, 23:697-702. |
| [13] | Yang B, Sugio A, White FF. Os8N3 is a host disease-susceptibility gene for bacterial blight of rice[J]. Proc Natl Acad Sci USA, 2006, 103:10503-10508. |
| [14] | Liu Q, Yuan M, Zhou Y, et al. A paralog of the MtN3/saliva family recessively confers race-specific resistance to Xanthomonas oryzae in rice[J]. Plant Cell Environ, 2011, 34:1958-1969. |
| [15] | Zhang G, Zhao MM, Song C, et al. Molecular characterization of a mitogen-activated protein kinase gene DoMPK1 in Dendrobium officinale[J]. Acta Pharm Sin (药学学报), 2012, 47:1703-1709. |
| [16] | Chen XM, Wang CL, Yang JS, et al. Research progress on chemical composition and chemical analysis of Dendrobium officinale[J]. Chin Pharm J (中国药学杂志), 2013, 48:1634-1640. |
| [17] | Zhao MM, Zhang G, Zhang DW, et al. ESTs analysis reveals putative genes involved in symbiotic seed germination in Dendrobium officinale[J]. PLoS One, 2013, 8:e72705. |
| [18] | Tan XM, Wang CL, Chen XM, et al. In vitro seed germination and seedling growth of an endangered epiphytic orchid, Dendrobium officinale, endemic to China using mycorrhizal fungi (Tulasnella sp.)[J]. Sci Hort, 2014, 165:62-68. |
| [19] | Wang H, Fang H, Wang Y, et al. In situ seed baiting techniques in Dendrobium officinale Kimura et Migo and Dendrobium nobile Lindl.:the endangered Chinese endemic Dendrobium (Orchidaceae)[J]. World J Microbiol Biotechnol, 2011, 27:2051-2059. |
| [20] | Zettler LW, Hofer CJ. Propagation of the little club-spur orchid (Platanthera clavellata) by symbiotic seed germination and its ecological implications[J]. Environ Exp Bot, 1998, 39:189-195. |
| [21] | Zhang G, Song C, Zhao MM, et al. Characterization of an A-type cyclin-dependent kinase gene from Dendrobium candidum[J]. Biologia, 2012, 67:360-368. |
| [22] | Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Res, 2001, 29:e45. |
| [23] | Kozak M. An analysis of 50-noncoding sequences from 699 vertebrate messenger RNAs[J]. Nucleic Acids Res, 1987, 15:8125-8132. |
| [24] | Tao Y, Cheung LS, Li S. Structure of a eukaryotic SWEET transporter in a homotrimeric complex[J]. Nature, 2015, 527:259-263 |
| [25] | Patil G, Valliyodan B, Deshmukh R, et al. Soybean (Glycine max) SWEET gene family:insights through comparative genomics, transcriptome profiling and whole genome resequence analysis[J]. BMC Genomics, 2015, 16:520. |
| [26] | Zhou Y, Liu L, Huang W, et al. Overexpression of OsSWEET5 in rice causes growth retardation and precocious senescence[J]. PLoS One, 2014, 9:e94210. |
| [27] | Liu C, Jiang J, Han XX, et al. Research advances in SWEET gene family in plants[J]. Plant Physiol J, 2014, 50:1367-1373. |
 2016, Vol. 51
2016, Vol. 51


