2. 南京野生植物综合利用研究院, 江苏 南京 210042
2. Nanjing Institute for Comprehensive Utilization of Wild Plants, Nanjing 210042, China
白芨 [Bletilla striata (Thunb.) Reichb. f.] 为兰科 (Orchidaceae) 白芨属 (Bletilla Reichb. f.) 多年生草本植物,在中国主要产于江苏、浙江、江西、安徽、云南、贵州等地区,在朝鲜半岛和日本也均有分布[1]。白芨基部具膨大假鳞茎,可制成干燥块茎,在中国药典中具有收敛止血、消肿生肌之功效[2]。《神农本草经》中也曾记载,白芨有“主痈肿、恶疮、败疽,伤阴死肌,胃中邪气”等功效。随着对白芨化学成分、药理疗效、日化应用等方面的深入研究和开发,其市场需求量与日俱增[3, 4, 5]。由于人们对白芨野生资源的过度采挖,使其生境受到严重破坏,加上白芨种子自然繁殖率低,其已经濒临灭绝,所以对白芨野生资源的评价和保护已刻不容缓。
目前,白芨的研究主要体现在药用功能和繁育技术两个方面,对白芨遗传多样性及遗传结构研究较少。仅通过SRAP、ISSR等分子标记对少数地区少数白芨属材料的遗传多样性展开研究,未能对白芨野生资源的保护和利用提供有效指导[6, 7]。序列相关扩增多态性 (sequence related amplified polymophism,SRAP) 是由Li和Qurios于2001年开发的一种新型分子标记技术[8],其基于PCR对开放阅读框 (open reading frames,ORFs) 进行扩增,具有独特的引物设计、操作简便、重复性好、高效共显、易于克隆测序等优点[8, 9, 10, 11, 12]。目前,SRAP分子标记已用于粉花石斛 (Dendrobium loddigesii Rolfe)、甜根子草 (Saccharum spontaneum L.) 和桑 (Morus alba L.) 等多种植物的遗传多样性分析[9, 10, 11]。故本文使用SRAP分子标记技术对国内8个省份的12个白芨居群进行遗传多样性和遗传结构开展研究,进而为白芨种质资源的保存提供理论依据。
材料与方法 实验材料供试野生白芨12个居群共计153个样,在2013~2014年间,从我国8个省份12个地区所在行政区域内收集所得,单个植株样本之间的空间距离为5 km以上,每个居群随机选取12~14个植株样本。所收集样本开花后均由丁小余教授鉴定,将样本新鲜叶片装入放有硅胶的自封袋中,保存于南京师范大学生命科学学院植物资源与环境研究所,具体材料编号、来源地及凭证标本号见表 1。
|
|
Table 1 Location and sampling size of twelve B. striata populations used in this study |
通过改良的CTAB提取法[13]从40 mg新鲜叶片中提取总DNA,用1% 琼脂糖凝胶电泳检测DNA的完整性和含量,DNA于-20 ℃保存。
SRAP 标记分析通过参照Li和Quiros[8]的设 计原理建立SRAP标记分析体系。用15份样本对100对SRAP引物组合进行筛选 (表 2),筛除条带不清 晰或者重复性差的标记。最终从100对引物组合中筛选出15对条带清晰且重复性较好的引物组 (表 3)。SRAP-PCR在20 µL反应体系中进行扩增,各成分 及其浓度为: 10×buffer 2 µL,MgCl2 1.5 mmol·L-1,dNTP 250 μmol·L-1,Taq酶1 U (TaKaRa),正、反引物各0.35 μmol·L-1,DNA模板35 ng。反应在PTC-200型PCR仪上进行,扩增程序为: 94 ℃预变性5 min ; 94 ℃变性1 min,36 ℃复性1 min,72 ℃延伸1 min,5个循环 ; 94 ℃变性1 min,52 ℃复性1 min,72 ℃延伸1 min,35个循环 ; 最后72 ℃延伸7 min ; 4 ℃保温。用8% 的非变性聚丙烯酰胺对PCR扩增产物进 行分离,0.1% 的硝酸银染色检测,500 bp DNA Marker为量度标准 (TaKaRa)。
|
|
Table 2 The forward and reverse SRAP primers used in this study |
|
|
Table 3 Polymorphism revealed by 15 SRAP primer combinations |
SRAP-PCR扩增片段有带记为“1”,无带则记为“0”,建立1和0型 数据矩阵。所得矩阵首先用POPGENE 3.2软件进 行分析[14]。在假设哈迪温伯格平衡 (Hardy-Weinberg equilibrium) 的前提下分析遗传多样性,包括: 多态位点百分率 (percentage of polymorphic band,PPB)、观测等位基因数 (observed number of alleles,Na)、有效等位基因数 (effective number of alleles,Ne)、香农信息指数 (I)[15]、Nei基因多样性指数 (H)[16]。居群间的遗传分化指数用Gst来计算,另外基于Gst计算基因流 (gene flow,Nm)[17]。
NTSYS-pc 2.1软件分析为了进一步研究该物种居群间的遗传关系,使用NTSYS-pc 2.1软件[18],根据Nei’s遗传相似系数构建居群非加权配对算术平均(unweighted pair group method of averages,UPGMA) 聚类树。Nei’s遗传距离[19]与实际地理距离之间的相关性通过Mantel test来检测,随机选择计算10 000次。
结果与分析 1 SRAP多态性分析15对引物扩增共得到252条重复性好、清晰的条带,每对引物的条带数为11 (Me5/Em5)~23 (Me5/ Em6) 不等,平均每对引物产生16.8个条带 (表 3)。在252个条带中共有210条多态性条带,其中引物Me5/Em5组合产生最少,为9个多态性条带,Me1/ Em1和Me1/Em10引物组合产生的多态性条带最多,为19个多态性条带,平均每对引物产生14个多态性条带。15对引物的多态位点百分率从72.22% (Me1/ Em8) 到92.86% (Me5/Em8) 不等,每对引物平均多态位点百分率为83.33%。引物扩增长度为100~1 000 bp,其中以统计100~500 bp片段大小为主。
2 居群遗传多样性分析白芨居群水平和物种水平上的基因多样性指数包括: 多态位点百分比、香农信息指数以及基因多态性等 (表 4)。白芨物种多样性指数 (H) 为0.349 4,香农信息指数 (I) 为0.509 6。在每个居群中,多态位点百分率 (PPB) 从66.67% (HZ) 到80.95% (NJ; TC),遗传多样性指数 (H) 从0.217 0 (HZ) 到0.317 7 (NJ),香农信息指数 (I) 也表现出相似趋势,其值从0.331 1 (HZ) 到0.465 1 (NJ)。观测到等位基因数量 (Na) 从1.666 7 (HZ) 到1.809 5 (NJ; TC)。有效等位基因数量 (Ne) 从1.351 8 (XT) 到1.558 7 (NJ)。结果表明,NJ居群的遗传多样性最丰富,HZ居群的遗传多样性较差一些,以上地区遗传多样性都较为丰富。
|
|
Table 4 Genetic diversity of B. Striata populations revealed by SRAP. PPB: Percentage of polymorphism loci; H: Nei’s (1973) gene diversity; I: Shannon’s information index; Na: Observed number of alleles; Ne: Effective number of alleles |
利用 POPGENE 3.2软件对白芨SRAP数据进行分析,得出12个居群总遗传多样性值Ht为0.348 7,居群内遗传多样性值Hs为0.257 8,其遗传分化值Gst为0.260 9,表明在白芨居群间存在26.09% 遗传变异,而在居群内的遗传变异为73.91%,居群内遗传变异大于居群间遗传变异,故居群内的变异占主要部分。其居群间基因交流值Nm = 1.416 7,居群间的基因交流适中。
4居群间遗传距离与地理距离相关性分析12个白芨居群间的遗传距离和地理距离通过Mantel test检测表明遗传距离和地理距离之间有显著的正相关性(r = 0.632 9; P < 0.000 1),即不同居群间遗传距离越小,其地理位置越近,遗传距离越大,地理位置就越远 (图 1)。
 | Figure 1 Relationship between genetic distance and geography distance by Mantel test |
依据Nei’s遗传相似系数构建来自8个省份的12个白芨居群的UPGMA聚类图 (图 2)。聚类图表明,12个居群可分为3个支系,我国东部江苏南京、江苏镇江、安徽宣城和浙江杭州4个居群聚为一支,中部湖北、湖南、江西以及贵州7个居群聚为一大支,西部云南文山单独聚为一支。结果表明,白芨种群间的地理位置越近,其遗传相似系数越大,地理位置相对越远,其遗传相似系数越小,从而被聚在不同的分支上。这与Mantel test检测结果相一致。
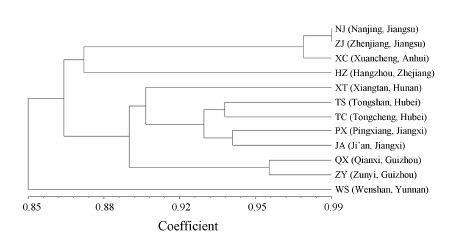 | Figure 2 An unweighted pair-group method with arithmetic averages (UPGMA) dendrogram of genetic relationships among 12 populations of B. striata calculated on the basis of genetic similarity by means of 15 SRAP primer combinations |
总体来说,白芨具有较高的遗传多样性水平。白芨在居群水平上的基因多样性 (H) 是0.257 8,平均多态位点百分比 (PPB) 是74.60%; 在种水平,基因多样性 (H) 是0.349 4,平均多态位点百分比 (PPB) 为90.48%。研究表明,与其生活特性相似的几种兰科植物比较而言[20, 21, 22],白芨同样具有较高的遗传多样性水平。
繁殖体系在物种的生活史特性中,被认为是决定植物遗传多样性高低的最重要的影响因子[23]。自交不亲和现象在兰科植物中很常见,如粉花石斛、铁皮石斛 (Dendrobium officinale Kimura et Migo),及其他兰花 (Octomeria crassifolia Lindl. 和 O. Grandiflora Lindl.) 等都存在自交不亲和现象[9, 24, 25]。白芨的花粉主要依赖昆虫传播,种子则靠风力传播。有文献[26]报道,在人工授粉试验中,白芨属于自交亲和性植物,但是其不发生不完全无配生殖和自发性自交,说明了昆虫传粉对白芨结实的重要性,在维持物种较高遗传多样性水平上起重要作用。另外,Nybom等[23]认为,多年生的特点可能也是白芨在物种水平上遗传多样性较高的原因之一。
物种的地理分布也会对它的遗传多样性产生影响,一般来说,分布广泛的通常比分布范围窄的物种存在更高的遗传变异水平[24],白芨北起河南、江苏南至台湾,东起浙江西至西藏东南部均有分布,具有丰富的遗传多样性[27]。此结果也与Zhang等[28]用SSR标记对15个野生白芨居群聚类结果基本一致。Li等[29]对浙产野生白芨居群遗传多样性研究发现该地区的遗传多样性较低,与本研究杭州居群的结果相一致。相对于其他地区野生白芨居群,浙江杭州居群可能由于受到特殊地域影响,如天目山限制了杭州白芨居群与其他居群间的基因交流,所以杭州野生白芨居群的遗传多样性较低。
2 白芨居群遗传结构物种居群遗传结构评估的重要参数主要包含: 基因分化和基因流。本研究中,白芨居群的平均分化指数是Gst = 0.26,其居群间存在中等水平的遗传分化。在Nybom[30]的结论中,远源杂交物种的平均Gst为0.27,这其中,如果是借助风力传播种子的植物,Gst值则偏为0.25。白芨不仅是虫媒传粉的远源杂交物种,而且白芨的种子细小、轻盈的特性,所以也能借助风力传播。独花兰 (Changnienia amoena S. S. Chien)[31]的分化指数是0.48、兰花 (Himantoglossum hircinum (L.) Spreng.)[32]的分化指数是0.20、粉花石斛[9]的分化指数是0.30,与这些兰科植物相比,白芨居群分化程度处于中等水平。
基因流是指生物个体从其发生地分散出去而导致不同种群之间的基因交流过程,可发生在同种或者不同种的生物种群之间,它与居群的遗传分化成负相关。由于兰科植物的种子细如花粉的特点,所以能够依靠风力长距离传播,这就促进了居群间的基因交流[33]。白芨居群间基因流的大小主要受限于花粉和种子的传播。在自然条件下,白芨的种子一般每年9至10月份开始成熟,成熟的种子会从开裂的朔果中飘散出来,加上白芨的种子很轻、细如花粉,促进了白芨的传播。但是白芨的居群间的基因流还是有限的,可能是因为在自然条件下种子萌发率很低,被传播到远方的种子不能萌发,抵消了风力传播的效果。本研究中,白芨的基因流Nm为1.41,这个水平上的每代迁移率可以阻止持续的居群间分化[34],因此居群间存在一定的遗传变异但居群内的遗传变异仍占主要部分。
Mantel test检测结果表明白芨遗传距离与地理距离呈显著正相关 (r = 0.632 9; P < 0.000 1),白芨居群间种子和花粉的传播受到地理距离的影响,进而形成现有居群结构。由于野生白芨人为的滥采滥挖,其连续分布的生境遭到破坏,被相互隔离的生境所取代,因此这种地理隔离造成不同白芨居群间的基因交流障碍,进而体现出白芨居群间的分化。UPGMA聚类结果支持Mantel test检测结果。地理位置越近的居群间花粉和种子的传播就相对频繁,基因交流较多,遗传相似系数较大; 地理位置相对越远的居群间基因交流相对较少,遗传相似系数较小,从而被聚在不同的分支上。我国东部江苏南京、江苏镇江、安徽宣城和浙江杭州4个居群聚为一支,中部湖北、湖南、江西以及贵州7个居群聚为一大支,西部云南文山单独聚为一支,也可以在一定程度上体现出不同白芨居群间既存在分化,同时也保持基因流动。
3 保护策略考虑白芨是一种珍贵的中药材,但是由于过度采伐导致了该资源的毁灭以及生境的丧失,其野生居群大大减少。白芨的种子极小,结构简单,无胚乳,自然条件下繁殖率极低[35],并且植株生长缓慢。这可能是导致白芨居群以及个体数量下降的生物学特性。
基于中国生物多样性保护策略及行动计划 (2011~2013),近年已经对濒危物种实施了针对性加强的保护和生境修复的措施,并增加了法律的执行力[36]。白芨保护管理的主要目的之一是保护白芨的遗传多样性水平。由于其主要的遗传变异存在居群内,以及高的遗传分化存在于居群间,所以就地保护和迁地保护都要考虑在内。考虑到白芨特殊的生境条件,就地保护是首先要考虑的方法。白芨有着生境偏爱性和传粉者依赖性[37],其生境片段化以及居群退化会增加亲缘关系近的物种繁殖的机会,最终会导致遗传多样性的丢失[22]。由于生境片段化,居群间的基因流也受到种子以及花粉散布的限制,因此保护生境才能保护该物种与其他有机体共存。江苏南京、安徽宣城和湖北通山居群拥有较高遗传变异,在保持该物种遗传多样性水平方面有主要的作用,应该给予优先考虑。在迁地保护中,应该设计和建立一个种质资源库,采集不同居群的种子或者幼苗混栽于合适的保护区,这样可以促进居群间的基因交流,丰富白芨种质资源,进而丰富其野生资源的遗传多样性。
| [1] | Chen XQ, Stephan WG, Phillip JC. Bletilla, In: Wu CY, Raven PH, Hong DY, editors. Flora of China, 25 [DB/OL]. Beijing: Science Press, 2009: 209-210. |
| [2] | Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典) [S]. Part 1. Beijing: China Medical Science Press, 2010: 95. |
| [3] | Dong JW, Cai L, Xiong J, et al. Improving the antioxidant and antibacterial activities of fermented Bletilla striata with Fusarium avenaceum and Fusarium oxysporum [J]. Proc Biochem, 2015, 50: 8-13. |
| [4] | Wang CX, Tian M. Callus-mediated and direct protocorm- like body formation of Bletilla striata and assessment of clonal fidelity using ISSR markers [J]. Acta Physiol Plant, 2014, 36: 2321-2330. |
| [5] | Wu JS, Zhang YS, Liu W, et al. DNA barcoding research and its application on medicinal plants of Bletilla H. G. Reichenbach [J]. Acta Pharm Sin (药学学报), 2014, 49: 1466-1474. |
| [6] | He ZJ, Lv LF, Yang LY, et al. Genetic diversity of Bletilla Rchb.f. germplasms by ISSR analysis [J]. Southwest China J Agric Sci (西南农业学报), 2008, 21: 1081-1085. |
| [7] | Zhu Y, Liu Y, Huang YH, et al. SRAP analysis on Bletilla germplasm resources [J]. Guizhou Agric Sci (贵州农业科学), 2012, 40: 10-13. |
| [8] | Li G, Quiros CF. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica [J]. Theor Appl Genet, 2001, 103: 455-461. |
| [9] | Cai XY, Feng ZY, Zhang XX, et al. Genetic diversity and population structure of an endangered Orchid (Dendrobium loddigesii Rolfe) from China revealed by SRAP markers [J]. Sci Hort, 2011, 129: 877-881. |
| [10] | Hu DC, Zhang P, Hao W. SRAP analysis on the genetic relationships of 24 mulberry (Morus L.) accessions in the lower regions of the Yellow River [J]. Genet Resour Crop Evol, 2015, 62: 13-19. |
| [11] | Chang D, Yang FY, Yan JJ, et al. SRAP analysis of genetic diversity of nine native populations of wild sugarcane, Saccharum spontaneum, from Sichuan, China [J]. Genet Mol Res, 2012, 11: 1245-1253. |
| [12] | Wang Z, Wang JE, Wang XM, et al. Assessment of genetic diversity in Galega officinalis L. using ISSR and SRAP markers [J]. Genet Resour Crop Evol, 2012, 59: 865-873. |
| [13] | Doyle JJ, Doyle JL. Isolation of plant DNA from fresh tissue [J]. Focus, 1990, 12: 13-15. |
| [14] | Yeh FC, Yang RC, Boyle T. POPGENE: Microsoft Windows- based freeware for population genetic analysis [OL]. Release 1.31. Edmonton: University of Alberta, 1999. |
| [15] | Lewontin RC. The apportionment of human diversity [J]. Evol Biol, 1995, 6: 381-398. |
| [16] | Nei M. Analysis of gene diversity in subdivided populations [J]. Proc Natl Acad Sci U S A, 1973, 70: 3321-3323. |
| [17] | Slatkin M, Barton NH. A comparison of three indirect methods for estimating average levels of gene flow [J]. Evolution, 1989, 43: 1349-1368. |
| [18] | Rohlf FJ. NTSYS-pc: Numerical Taxonomy and Multivariate Analysis System Version 2.1. Exeter Software [M]. New York: Applied Biostatistics Inc., 2000. |
| [19] | Nei M. Genetic distances between populations [J]. Am Nat, 1972, 106: 283-292. |
| [20] | Ye MR, Hou BW, Luo J, et al. Genetic diversity and conservation of the endangered herb Dendrobium moniliforme based on amplified fragment length polymorphism markers [J]. Sci Hort, 2015, 189: 51-58. |
| [21] | Wang HZ, Wu ZX, Lu JJ, et al. Molecular diversity and relationships among Cymbidium goeringii cultivars based on inter-simple sequence repeat (ISSR) markers [J]. Genetica, 2009, 136: 391-399. |
| [22] | Li XX, Ding XY, Chu BH, et al. Genetic diversity analysis and conservation of the endangered Chinese endemic herb Dendrobium officinale Kimura et Migo (Orchidaceae) based on AFLP [J]. Genetica, 2008, 133: 159-166. |
| [23] | Nybom H, Bartish IV. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants [J]. Perspect Plant Ecol, 2000, 3: 93-114. |
| [24] | Johansen B. Incompatibility in Dendrobium (Orchidaceae) [J]. Bot J Linnean Soc, 1990, 103: 165-196. |
| [25] | Barbosa AR, Pereira VS, Borba EL. High genetic variability in self-incompatible myophilous Octomeria (Orchidaceae, Pleurothallidinae) species [J]. Braz J Bot, 2013, 36: 179-187. |
| [26] | Eduardo LB, Joaa OS, George JS. Self-incompatibility, inbreeding depression and crossing potential in five Brazilian Pleurothallis (Orchidaceae) Species [J]. Ann Bot, 2001, 88: 89-99. |
| [27] | Yamaki M, Li B, Kato T, et al. Three dihydrophenanthropyrans from Bletilla striata [J]. Phytochemistry, 1993, 32: 427-430. |
| [28] | Zhang YS, Hou BW, Zhang WM. Isolation and characterization of novel microsatellite markers for Bletilla striata and inter- specific amplification in 2 congeneric species [J]. Conserv Genet Resour, 2015, 7: 483-485. |
| [29] | Li JC, Lei CM, Hao SX, et al. Genetic diversity analysis of wild Bletilla striata produced in Zhejiang province by ISSR [J]. Chin Arch Tradit Chin Med (中华中医药学刊), 2013, 31: 294-297. |
| [30] | Nybom H. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants [J]. Mol Ecol, 2004, 13: 1143-1155. |
| [31] | Li A, Ge S. Genetic variation and conservation of Changnienia amoena, an endangered orchid endemic to China [J]. Plant Syst Evol, 2006, 258: 251-260. |
| [32] | Pfeifer M, Jetschke G. Influence of geographical isolation on genetic diversity of Himantoglossum hircinum (Orchidaceae) [J]. Folia Geobot, 2006, 41: 3-20. |
| [33] | Borba EL, Semir J. Pollinator specificity and convergence in fly-pollinated Pleurothallis (Orchidaceae) species: a multiple population approach [J]. Ann Bot, 2001, 88: 75-88. |
| [34] | Wright S. The genetical structure of populations [J]. Ann Eugen, 1951, 15: 323-354. |
| [35] | Wang BQ, Li ZL, Liu GM, et al. Study on seeds aseptic culture and embryo growth routes of several orchid [J]. Acta Bot Yunnan (云南植物研究), 2006, 28: 399-402. |
| [36] | Zeng Y, Xu J, Wang Y, et al. Habitat association and conservation implications of endangered Francois' langur (Trachypithecus francoisi) [J]. PLoS One, 2013, 8: e75661. |
| [37] | Chung MY, Chung MG. Pollination biology and breeding systems in the terrestrial Orchid Bletilla striata [J]. Plant Syst Evol, 2005, 252: 1-9. |
 2016, Vol. 51
2016, Vol. 51


