2. 中日友好医院药学部, 北京 100029
2. Department of Pharmacy, China-Japan Friendship Hospital, Beijing 100029, China
1962年,Keith R. Porter教授及其博士后研究生Thomas Ashford在美国洛克菲勒研究中心首次发现了自噬这一过程; 1963年,比利时生物化学家Christian de Duve将该过程命名为自噬(autophagy)。20世纪90年代,随着自噬酵母模型的建立和基因技术的发展,自噬的研究开始呈爆发式增长。2005年Science期刊将自噬列为科学领域的六大研究方向之一,同年Autophagy杂志创刊。近年来,自噬的研究已广泛并深入至生物学、医学、植物学和微生物学等领域。尤其在生物医学界,许多研究人员正在积极探讨自噬在各种疾病中的作用,研究疾病中自噬水平调控的分子机制,以期利用自噬为临床疾病治疗寻找新的有效靶点。鉴于自噬的广泛生理和病理意义,建立合适的自噬活性筛选系统来获得具有成药潜力的自噬调节剂具有重要的应用价值。本文主要对目 前自噬活性物质筛选系统的研究进展予以综述。
1 自噬概述自噬是细胞在饥饿、能量缺乏等代谢应激状态下的一种分解代谢过程,细胞内的蛋白等大分子、细胞器和胞质通过自噬作用被包裹、消化、降解成核苷、氨基酸和脂肪酸等可被重复循环利用的物质,用作合成新的大分子并提供能量来源ATP,从而维持细胞基本的生命活动。自噬参与维持蛋白代谢平衡及细胞内环境的稳定,其在清除细胞内废物、结构重建以及细胞生长发育中起着重要作用。自噬既是细胞的一种正常生理活动,也可在细胞遭受各种刺激 (缺氧、营养匮乏、化学物质刺激、微生物入侵、细胞器损伤和异常蛋白堆积等) 时作为应激反应而发生[1]。自噬不仅具有维持细胞自我调节和促进细胞生存的作用,过度活跃的自噬活动也可以引起细胞死亡,即“自噬性细胞死亡” (autophagic cell death),又称为II型程序性细胞死亡,是一种非凋亡依赖性的细胞程序性死亡。
1.1 自噬的诱导自噬活化的始动步骤是自噬小体的前体膜包裹胞浆物质形成吞噬泡。自噬可被多种内源性及外源性信号激活,哺乳动物雷帕霉素靶蛋白 (mTOR) 能够感受细胞营养状态,调控蛋白质合成,影响细胞生长,也是饥饿诱导自噬这一经典通路的核心。细胞处于饥饿状态时,可迅速抑制mTOR活性。mTOR活性的下降,一方面可直接抑制蛋白质的合成; 另一方面可诱导自噬的发生,通过自噬来完成细胞中大分子物质的降解,从而缓解细胞内营养匮乏。在正常营养条件下,mTOR与ULK1/Atg1形成复合体,从而抑制ULK1的激酶活性,抑制自噬的发生。饥饿状态下,ULK1复合物与mTOR解离从而启动自噬小体的形成[2]。目前的研究[3]表 明,mTOR-ULK1复合物是细胞对生长与自噬信号进行协调应答的关键核心。除经典的饥饿刺激外,缺氧、内质网应激和活性氧自由基 (reactive oxygen species,ROS) 等均可抑制mTOR活性,激活自噬的发生。
1.2 自噬小体的形成ULK1复合物被上游自噬激活信号刺激后使Beclin1 14位点的丝氨酸发生磷酸化,使Beclin1-Atg14-PI3KC3-Vps15核心复合物的活性增强,进而促进自噬泡的形成[4]。此外,在营养充足条件下,Bcl2通过与Beclin1的BH3结构域结合抑制自噬活性。Bcl2或Beclin1的磷酸化均可干扰二者的相互作用,释放Beclin1。Atg12和Atg8 (哺乳动物中的同源蛋白为LC3) 这两个泛素样蛋白与自噬体膜的延伸有关。Atg12首先被E1泛素样酶Atg7以ATP依赖方式激活,继而在E2泛素样酶Atg10催化下与Atg5共价结合。Atg12-Atg5复合物与Atg16非共价结合形成三聚体; 这个三聚体位于分离膜外侧,参与LC3的募集和分离膜的弯曲及延伸。与此同时,前体LC3经由半胱氨酸蛋白酶Atg4水解形成可溶性LC3 (LC3-I)。LC3-I进一步被Atg7活化并转移给Atg3,经由Atg3催化与磷脂酰乙醇胺 (PE) 共价结合形成自噬体膜型LC3 (LC3-II)。因此将LC3用染料进行标记,可观察到LC3由弥散 (自噬未激活) 到点状聚集 (自噬被激活) 的过程; 同时,由于LC3-I和LC3-II的分子量不同,可通过Western blot电泳进行区分,因此LC3被认为是检测自噬活化的金标准之一 (图 1)。
 | Figure 1 Overview of the autophagy process |
自噬体仅是包裹有待降解物质的双层膜结构,并没有酶活性,因此在形成后不久即向溶酶体运动,二者融合,形成单层膜结构的自噬溶酶体,最终在溶酶体的作用下完成自噬体包裹物质的降解,降解产物在溶酶体表 面转运蛋白的作用下转运出自噬溶酶体,实现降解产物的循环再利用。
2 自噬活性筛选系统近年来,分子生物学手段的进步及特异性工具药的发现使得自噬的检测方法更加成熟及多样化。自噬活性的检测方法主要分为以下三种: ① 形态学检测,如利用电镜观察自噬双层膜结构的形成,或利用 荧光显微镜观察经嗜酸性染料染色的自噬溶酶体; ② 自噬信号通路相关蛋白的检测,如自噬的激活信号蛋白mTOR、ULK1,自噬核心复合物PI3KC3、Beclin1及其他Atg蛋白等; ③ 自噬体标志蛋白的检测,如贯穿于自噬过程的重要标志蛋白LC3和p62,是检测自噬活性的重要标准。
然而,适合用于建立自噬活性物质筛选系统的检测方法应具备以下特征: 定量性强,检测指标应具有数值化特征,便于定量比较; 操作性强,快速简便,具有高通量筛选潜力; 特异性强,应避免与其他信号通路的交叉。
2.1 基于GFP-LC3的筛选系统GFP-LC3是最早用于自噬活性筛选系统的检测指标,结合流式细胞技术可对结果进行定量分析,方便开展高通量筛选。随着对自噬研究的不断深入及自噬流概念的提出,基于GFP-LC3的筛选系统经历了不断改进和完善的过程。早期的筛选系统应用稳定表 达GFP-LC3的细胞系,提出激活自噬会导致LC3的消耗降解,因此GFP的荧光强度应随自噬的激活而降低[5]。然而,随后其他研究者[6, 7]发现,在很多情况下自噬的激活并不伴随GFP-LC3总量的变化,因为GFP-LC3在随着自噬流降解的同时,大量新合成的GFP-LC3随即补充了总量的消耗,因此GFP-LC3的总量并无明显变化。此后的筛选系统开始更加全面地考虑LC3的合成与降解,LC3-I与LC3-II的不同性质及分布等。Eng的研究小组[6]在此基础上进一步改进,建立了具有自噬流活性筛选能力的筛选系统。在该系统中,研究者根据LC3-I与LC3-II溶解性质不同,通过皂素在细胞膜上打孔,将胞浆中可溶性的LC3-I洗去,而膜结合型的LC3-II则被留在细胞中,此时,GFP的荧光强度反映的是结合于自噬体膜上的LC3-II (图 2)。在自噬被激活时,LC3-I向LC3-II的转化增加,因此LC3-II的量更适合表 征自噬的活化状态。同时,联合应用溶酶体抑制剂即可区分自噬激活与晚期自噬抑制,从而判断自噬流的通畅与否。Hancock等[8]发明了另一种方法来定量分析LC3-II,鉴于LC3-II锚定于自噬体膜后其构型发生变化,区别于LC3-I,将可特异识别LC3-II的铽 (terbium,Tb) 标记抗体与GFP-LC3-II组成荧光共振能量转移系统,GFP-LC3-II作为受体发色团,Tb标记抗体作为供体荧光团,即可通过荧光酶标仪快速、高通量地读取数值。该方法操作较为简便,将稳定表 达GFP-LC3的细胞铺于96孔板或384孔板,给予自噬诱导处理一定时间后,加入含有特异性识别LC3-II抗体的裂解液,LC3-II抗原与抗体的结合产生共振能量转移,产生的荧光信号通过荧光酶标仪读取记录。该方法的限制性在于特异性LC3-II抗体的选择,结果的准确性依赖于抗体的高特异性,此外该系统需要GFP-LC3具有较高的表 达才能达到信号的检测限。
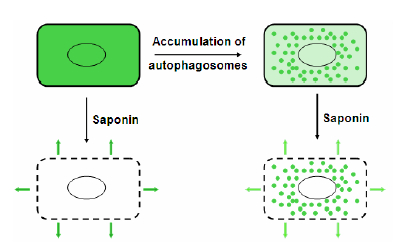 | Figure 2 Separation of LC3-I and LC3-II by saponin extraction |
除了利用GFP-LC3的荧光进行流式定量分析外,将LC3与荧光素酶偶联,应用荧光素酶定量分析也是一种常用手段[9]。Farkas等[10]将荧光素酶 (renilla reniformis luciferase,Rluc) 偶联的LC3成功应用于自噬活性筛选系统。在该系统中作者将荧光素酶与野生型LC3 (LC3wt) 及脂化位点缺陷型LC3 (G120A) 串联表 达,由于缺少磷脂化位点,LC3-Rluc-G120A不能锚定于自噬双层膜,因此对自噬性降解不敏感。根据Rluc-LC3wt与Rluc-LC3-G120A这两种细胞群荧光素酶活性的比值可测定出LC3的自噬依赖性降解速率,进而反映自噬流的活性。利用该系统已成功筛选到正向或反向影响细胞自噬流的小分子化合物、小RNA (microRNA) 及小干扰RNA (siRNA)[11, 12]。
在以LC3为标记的筛选系统中需引起注意的是,溶酶体的多种抑制性状态也均可造成LC3-II的堆积,包括阻碍自噬体与溶酶体的融合 (如抑制微管蛋白聚合的长春碱,增加溶酶体pH值的质子泵抑制剂巴伐洛霉素A1) 及抑制溶酶体内的酶活性 (如半胱氨酸蛋白酶抑制剂E-64D、天冬酰胺蛋白酶抑制剂pepstatin A或氯喹)[13]。为区分自噬的激活与自噬性降解的抑制,提出了自噬流的概念。自噬是一个动态的、多步骤的过程,因此自噬流这一概念更能体现从自噬体形成到自噬底物降解的完整自噬过程。自噬流的检测可通过Western blot观察LC3-II的翻转,即比较加入溶酶体抑制剂前后LC3-II的量是否有变化: 若某一刺激下加入溶酶体抑制剂组LC3-II的量进一步增加,则说明该刺激可活化自噬流 (如给药组联合给予巴伐洛霉素A1后LC3-II的量应高于单独给药组); 若LC3-II的量前后无明显变化则指向自噬流的抑制[14]。这些抑制剂可以是多种阻碍自噬底物降解的抑制剂,如巴伐洛霉素A1、E-64D和pepstatin A及氯喹等[15]。
2.2 基于RFP-GFP-LC3的筛选系统为使自噬流的研究更加方便、直观,研究者进一步开发了LC3偶联双荧光蛋白的检测方法,GFP绿色荧光蛋白对溶酶体内的酸性环境非常敏感,其荧光随着自噬体与溶酶体的融合而淬灭; 而mRFP或mCherry红色荧光蛋白则相对稳定,对溶酶体的酸性环境不敏感,其红色荧光也不会因酸性条件而淬灭。因此,结合双荧光的定位情况可以反映自噬流的不同状态。GFP与RFP荧光的共定位指示双层膜结构还未与溶酶体发生融合的状态,如自噬泡或自噬小体; RFP单荧光则对应于两性体和自噬溶酶体[16]。该方法最大的优点在于可以检测自然状态下的细胞自噬流情况,不需要外源药物的干预。
RFP/mCherry-GFP-LC3串联蛋白的应用使检测自噬流的动态变化更加方便直观,以此为基础建立的筛选系统可以较好地区分作用于自噬不同阶段的化合物。Kimura的研究团队[16]将RFP-GFP-LC3串联蛋白应用于自噬体与自噬溶酶体的区分。Hundeshagen的研究小组[17]将这种双荧光串联蛋白应用于自噬 活性物质的筛选,以稳定表 达mCherry-GFP-LC3的MCF-7细胞为筛选平台,结合流式细胞技术定量分析了Prestwick化合物库中1 120个美国FDA批准的小分子药物,筛选出38个可以促进自噬溶酶体形成的自噬激动剂和36个自噬抑制剂。有报道[18]指出,GFP在pH 4~5的酸性环境中仍有荧光残留,这种不完全淬灭会干扰自噬溶酶体活性的判断,为此,Zhou等[19]对双荧光蛋白进行了改进,建立了mTagRFP- mWasabi-LC3荧光系统,mWasabi比GFP的荧光强度更高同时对酸性环境更加敏感,而mTagRFP也比RFP及mCherry荧光强度更高。双荧光蛋白串联系 统的优势在于免去了加入溶酶体抑制剂来判断自噬流的环节,然而仍存在一些缺陷,如研究者并未考虑荧光蛋白发生自噬性降解的同时其生物合成也在不断进行的矛盾。
2.3 基于GFP-p62的筛选系统除LC3蛋白外,p62或其他货车蛋白如NBR1也可作为自噬的标志蛋白[20]。p62蛋白有440个氨基酸,包含PBl结构域、ZZ型锌指结构域、TB结构域、LIR结构域和UBA结构域等功能域,其中p62通过LIR (LC3 interacting region) 结构域与LC3结合,同时通过UBA结构域与泛素化蛋白结合。因此,p62蛋白介导了泛素化蛋白的自噬性降解,具有指示自噬底物降解程度的意义。通常情况下,p62蛋白的堆积和自噬的抑制相关,而p62水平下降则指向自噬的激活。将p62偶联荧光蛋白标签即可实现对自噬性降解的可视化检测。在以p62作为考察指标的筛选系统中,需要格外注意的是,p62的总量受两方面因素的共同影响: 一是其合成,二是其降解。因此要考察p62通过自噬途径的降解程度就要排除不断新增的合成对其的影响。
Larsen等[21]通过将GFP-p62构建成四环素诱导调控表 达系统排除了蛋白合成的影响。四环素阻遏蛋白 (Tet repressor protein,TetR) 与四环素操纵基因 (Tet operator,TetO) DNA序列有特异的亲和力,当细胞内无四环素存在时,TetR会与TetO结合,从而阻断下游的GFP-p62基因表 达。当细胞培养基中加入四环素后,四环素与TetR结合并使其构象发生改变,导致TetR与TetO分离,从而引起GFP-p62基因的抑制解除,GFP-p62开始转录翻译,大量表 达GFP-p62蛋白。将该调控表 达系统构建入HEK293细胞中,在含有四环素的培养基中待GFP-p62蛋白表 达一定时间后,更换为不含四环素的培养基,这时GFP-p62的表 达停止,通过流式细胞技术或荧光显微镜测定不同时间点GFP-p62的量即可分析其降解程度从而反映自噬流速率。BenYounès等[22]建立了另一种具有区分p62表 达、降解能力的筛选系统。在该系统中,p62与HaloTag蛋白形成嵌合蛋白,作为HaloTag荧光分子的受体。p62-HaloTag嵌合蛋白可与其荧光配基HaloTag形成牢固的共价结合,洗去多余的HaloTag荧光配基,给予一定时间的自噬诱导刺激后,即可通过流式细胞技术等方法测定p62-HaloTag蛋白-HaloTag荧光配基荧光强度的变化,继而分析自噬流的状态。该荧光强度不受新合成的p62-HaloTag蛋白影响,因为嵌合蛋白本身是没有荧光信号的,只有与配基结合后才具有荧光,因此该系统也巧妙地避免了新合成蛋白的影响。
然而,相比自噬标志蛋白LC3,p62作为自噬活性筛选标记的特异性欠佳。一方面,p62处于连接泛素蛋白酶体降解途径和自噬溶酶体降解途径的交叉点,因此,p62水平的变化并不能完全代表 自噬水平的变化,例如当泛素蛋白酶体通路受到抑制也可导致p62的降解受阻[23]。另一方面,p62不仅局限于担当自噬的货车蛋白,在其他信号通路中也发挥着重要作用,如凋亡信号通路和氧化应激信号通路等[24, 25]。
2.4 基于长寿命蛋白降解的筛选系统自噬的本质是一个分解代谢过程,尤其在降解错误折叠蛋白、蛋白聚集体 (protein aggregates) 方面发挥着重要作用。目前研究已经证实几乎所有的神经退行性疾病都存在自噬囊泡的蓄积和胞浆内蛋白质的聚积,指示自噬功能的障碍。反过来,通过蛋白聚集体的降解速率来考察自噬流的活化与否也是一种自噬活性物质筛选策略。Ju等[26]建立了一种荧光素酶定量系统,将 一种长寿命蛋白多聚谷氨酰胺蛋白聚集体polyQ80与荧光素酶 (luciferase) 偶联,同时将可溶性蛋白polyQ19与荧光素酶偶联作为对照,通过测定polyQ80-luciferase与polyQ19-luciferase比值的变化来反映自噬对聚集蛋白的降解能力,进一步反映自噬流的活性。除细胞水平外,作者还在动物水平用饥饿和雷帕霉素刺激验证了该系统的可行性。该系统具有较强的针对性,即更适合于检测聚集体自噬这种选择性自噬的活性,因此对于抗神经退行性疾病药物的筛选具有较强的参考价值。
2.5 基于模式生物的自噬活性检测系统目前细胞模型因其实验条件易于控制,筛选结果便于观察、检测和记录,具有快速、稳定、定量和经济等优点,仍为自噬筛选系统最主要的平台。然而体外的细胞模型在生物学特征上与人体或小鼠仍存在一定差异,单纯使用细胞模型难以反映机体内自噬及其相关疾病的动态关联性变化,因此利用线虫、果蝇、斑马鱼及小鼠等模式生物检测自噬活性受到了广泛关注。因此,通过细胞模型筛选得到的自噬活性物质在动物水平的进一步验证具有重要意义。
线虫中LGG-1是哺乳动物中LC3的同源蛋白,通过GFP标记LGG-1蛋白,在高分辨率的荧光显微镜下即可实现对线虫中自噬的可视化直观研究。以线虫为模型研究自噬在神经退行性疾病中的作用得到了较好的应用,通过在线虫中表 达多聚谷氨酰胺 (polyQ) 可模拟两种多聚谷氨酰胺疾病模型,一种在线虫肌细胞中表 达polyQ形成聚集物,导致线虫运动障碍; 另一种则在线虫感觉神经元中表 达polyQ形成聚集物,导致渐进神经元变性[27]。此外,还可在线虫肌细胞中表 达β淀粉样蛋白 (amyloid β,Aβ),为研究Aβ蛋白聚集引起的毒性机制提供了良好的工具[28, 29]。
果蝇是遗传学上一种经典的模式生物,可利用果蝇研究自噬在神经退行性疾病和先天免疫应答等方面的研究。研究者利用转基因技术建立了表 达GFP-mCherry-atg8a的果蝇品系,便于在果蝇体内观察自噬的激活及自噬流的变化[30]。
斑马鱼因其体外受精、子代数量多、胚胎及幼鱼外观透明、发育迅速及干预方便等特点成为一种很好的模式脊椎动物。为研究斑马鱼中自噬活性,研究者建立了GFP-LC3斑马鱼模型。以斑马鱼为模型,可模拟多种神经变性疾病,如阿尔兹海默病、亨廷顿舞蹈病及肌萎缩侧索硬化症等[31]。此外,研究者还通过在斑马鱼肝脏特异性表 达GFP-LC3来研究自噬在肝脏相关疾病中的作用[32]。通过在斑马鱼尾鳍注入病原体,Hosseini等[33]结合光电显微镜成像技术对病原体与GFP-LC3的共定位进行研究,在体内水平探讨了自噬与感染性疾病的关系。
小鼠因其与人类基因组相似度高、品系多及易 于饲养繁殖等优点,成为研究人类疾病最重要的哺乳动物模型。研究者通过基因打靶技术、基因捕获 技术、组织嵌合和组织特异性的条件性突变建立了各种自噬相关小鼠模型,并且多种自噬相关基因的小鼠模型已可商业化购买。GFP-Lc3及GFP-mCherry- Lc3转基因小鼠模型成为在体研究自噬状态的有利工具[34]。
3 总结与展望自噬是一个多环节的、动态的复杂过程,也是一个具有明显特征的细胞活动,如双层膜结构的自噬体,标志蛋白LC3和p62等,这些特征性标志为自噬的检测提供了多种可行方法。随着自噬在多种疾病中作用机制的日渐阐明,利用高通量、高内涵的自噬筛选平台寻找靶向自噬的调节剂将成为未来药物开发的热点[35, 36]。通过高通量自噬筛选系统对siRNA及小分子化合物进行大规模筛选,一方面可对自噬信号通路的调节网络予以完善,另一方面则为自噬相关疾病的药物开发寻找先导物。然而不同于某一特定靶蛋白激动剂或抑制剂的筛选,自噬激动剂或抑制剂的确定往往不能通过一步高通量筛选完成,需结合多种方法综合判断自噬流的活性,从而对自噬激活与晚期自噬抑制进行严格区分。另外,在选取某一特征蛋白作为高通量筛选的定量指标时应注意该蛋白合成和消耗之间的矛盾关系。
| [1] | Rubinsztein DC, Codogno P, Levine B. Autophagy modulation as a potential therapeutic target for diverse diseases [J]. Nat Rev Drug Discov, 2012, 11:709-730. |
| [2] | Ganley IG, Lam du H, Wang J, et al. ULK1.ATG13.FIP200 complex mediates mTOR signaling and is essential for autophagy [J]. J Biol Chem, 2009, 284: 12297-12305. |
| [3] | Kim J, Kundu M, Viollet B, et al. AMPK and mTOR regulate autophagy through direct phosphorylation of Ulk1 [J]. Nat Cell Biol, 2011, 13: 132-141. |
| [4] | Russell RC, Tian Y, Yuan H, et al. ULK1 induces autophagy by phosphorylating Beclin-1 and activating VPS34 lipid kinase [J]. Nat Cell Biol, 2013, 15: 741-750. |
| [5] | Shvets E, Fass E, Elazar Z. Utilizing flow cytometry to monitor autophagy in living mammalian cells [J]. Autophagy, 2008, 4: 621-628. |
| [6] | Eng KE, Panas MD, Karlsson Hedestam GB, et al. A novel quantitative flow cytometry-based assay for autophagy [J]. Autophagy, 2010, 6: 634-641. |
| [7] | Mizushima N, Yoshimori T, Levine B. Methods in mammalian autophagy research [J]. Cell, 2010, 140: 313-326. |
| [8] | Hancock MK, Hermanson SB, Dolman NJ. A quantitative TR-FRET plate reader immunoassay for measuring autophagy [J]. Autophagy, 2012, 8: 1227-1244. |
| [9] | Ketteler R, Seed B. Quantitation of autophagy by luciferase release assay [J]. Autophagy, 2008, 4: 801-806. |
| [10] | Farkas T, Høyer-Hansen M, Jäättelä M. Identification of novel autophagy regulators by a luciferase-based assay for the kinetics of autophagic flux [J]. Autophagy, 2009, 5: 1018- 1025. |
| [11] | Farkas T, Daugaard M, Jäättelä M. Identification of small molecule inhibitors of phosphatidylinositol 3-kinase and autophagy [J]. J Biol Chem, 2011, 286: 38904-38912. |
| [12] | Frankel LB, Wen J, Lees M, et al. MicroRNA-101 is a potent inhibitor of autophagy [J]. EMBO J, 2011, 30: 4628- 4641. |
| [13] | Ahlberg J, Berkenstam A, Henell F, et al. Degradation of short and long lived proteins in isolated rat liver lysosomes. Effects of pH, temperature, and proteolytic inhibitors [J]. J Biol Chem, 1985, 260: 5847-5854. |
| [14] | Tanida I, Minematsu-Ikeguchi N, Ueno T, et al. Lysosomal turnover, but not a cellular level, of endogenous LC3 is a marker for autophagy [J]. Autophagy, 2005, 1: 84-91. |
| [15] | Ju JS, Varadhachary AS, Miller SE, et al. Quantitation of “autophagic flux” in mature skeletal muscle [J]. Autophagy, 2010, 6: 929-935. |
| [16] | Kimura S, Noda T, Yoshimori T. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3 [J]. Autophagy, 2007, 3: 452-460. |
| [17] | Hundeshagen P, Hamacher-Brady A, Eils R, et al. Concurrent detection of autolysosome formation and lysosomal degradation by flow cytometry in a high-content screen for inducers of autophagy [J]. BMC Biol, 2011, 9: 38. |
| [18] | Bampton ET, Goemans CG, Niranjan D, et al. The dynamics of autophagy visualized in live cells: from autophagosome formation to fusion with endo/lysosomes [J]. Autophagy, 2005, 1: 23-36. |
| [19] | Zhou C, Zhong W, Zhou J, et al. Monitoring autophagic flux by an improved tandem fluorescent-tagged LC3 (mTagRFP- mWasabi-LC3) reveals that high-dose rapamycin impairs autophagic flux in cancer cells [J]. Autophagy, 2012, 8: 1215-1226. |
| [20] | Germain M, Nguyen AP, Le Grand JN, et al. MCL-1 is a stress sensor that regulates autophagy in a developmentally regulated manner [J]. EMBO J, 2011, 30: 395-407. |
| [21] | Larsen KB, Lamark T, Øvervatn A, et al. A reporter cell system to monitor autophagy based on p62/SQSTM1 [J]. Autophagy, 2010, 6: 784-793. |
| [22] | BenYounès A, Tajeddine N, Tailler M, et al. A fluorescence- microscopic and cytofluorometric system for monitoring the turnover of the autophagic substrate p62/SQSTM1 [J]. Autophagy, 2011, 7: 883-891. |
| [23] | Tian Z, Wang C, Hu C, et al. Autophagic-lysosomal inhibittion compromises ubiquitin-proteasome system performance in a p62 dependent manner in cardiomyocytes [J]. PLoS One, 2014, 9: e100715. |
| [24] | Moscat J, Diaz-Meco MT. p62 at the crossroads of autophagy, apoptosis, and cancer [J]. Cell, 2009, 137: 1001-1004. |
| [25] | Duran A, Amanchy R, Linares JF, et al. p62 is a key regulator of nutrient sensing in the mTORC1 pathway [J]. Mol Cell, 2011, 44: 134-146. |
| [26] | Ju JS, Miller SE, Jackson E, et al. Quantitation of selective autophagic protein aggregate degradation in vitro and in vivo using luciferase reporters [J]. Autophagy, 2009, 5: 511-519. |
| [27] | Zhang H, Chang JT, Guo B, et al. Guidelines for monitoring autophagy in Caenorhabditis elegans [J]. Autophagy, 2015, 11: 9-27. |
| [28] | Regitz C, Fitzenberger E, Mahn FL, et al. Resveratrol reduces amyloid-beta (Aβ1-42)-induced paralysis through targeting proteostasis in an Alzheimer model of Caenorhabditis elegans [J]. Eur J Nutr, 2015. DOI: 10.1007/s00394-015- 0894-1. |
| [29] | Regitz C, Dussling LM, Wenzel U. Amyloid-beta (Aβ1-42)- induced paralysis in Caenorhabditis elegans is inhibited by the polyphenol quercetin through activation of protein degradation pathways [J]. Mol Nutr Food Res, 2014, 58: 1931-1940. |
| [30] | DeVorkin L, Gorski SM. Monitoring autophagy in Drosophila using fluorescent reporters in the UAS-GAL4 system [J]. Cold Spring Harb Protoc, 2014, 2014: 967-972. |
| [31] | Fleming A, Rubinsztein DC. Zebrafish as a model to understand autophagy and its role in neurological disease [J]. Biochim Biophys Acta, 2011, 1812: 520-526. |
| [32] | Cui J, Sim TH, Gong Z, et al. Generation of transgenic zebrafish with liver-specific expression of EGFP-Lc3: a new in vivo model for investigation of liver autophagy [J]. Biochem Biophys Res Commun, 2012, 422: 268-273. |
| [33] | Hosseini R, Lamers GE, Hodzic Z, et al. Correlative light and electron microscopy imaging of autophagy in a zebrafish infection model [J]. Autophagy, 2014, 10: 1844-1857. |
| [34] | Terada M, Nobori K, Munehisa Y, et al. Double transgenic mice crossed GFP-LC3 transgenic mice with αMyHC-mCherry- LC3 transgenic mice are a new and useful tool to examine the role of autophagy in the heart [J]. Circ J, 2010, 74: 203- 206. |
| [35] | Joachim J, Jiang M, McKnight NC, et al. High-throughput screening approaches to identify regulators of mammalian autophagy [J]. Methods, 2015, 75: 96-104. |
| [36] | Rossi M, Rotblat B, Ansell K, et al. High throughput screening for inhibitors of the HECT ubiquitin E3 ligase ITCH identifies antidepressant drugs as regulators of autophagy [J]. Cell Death Dis, 2014, 5: e1203. |
 2016, Vol. 51
2016, Vol. 51


