2. 安徽中医药大学, 安徽合肥 230012
2. Anhui University of Chinese Medicine, Hefei 230012, China
高分辨率熔解曲线技术 (high resolution melting,HRM) 是2003年由美国Utah大学Wittwer实验室 和Idaho Technology公司联合开发的一项新技术,因其操作简便快速、成本低、通量高、结果准确且实现了闭管操作而受到普遍关注[1]。新型饱和荧光染料LC Green的同步出现更是为这项新技术的应用打下坚实的基础[2]。与其他分子检测技术相比,HRM技术不受样品DNA双链突变碱基位点与类型的局限,无需序列特异性探针,且闭管操作避免污染,同时实验操作与后期数据分析简单、易懂,因此已广泛应用于突变扫描、基因分型、序列匹配与甲基化检测等基因诊断[3]。
分子鉴定由于具有准确性高、重现性好等特点,且不受样品形态的限制等优势,在中药鉴定领域发挥了越来越重要的作用[4]。建立以满足快速、现场、高通量、低成本鉴定需求的技术体系和工作模式,可使中药分子鉴别技术不仅应用在实验室,还将应用于广泛的实际生产与贸易交流环节[5]。目前常用的分子鉴定技术包括表达序列标签−简单重复序列多态性 (EST-SSR)、聚合酶链式反应−限制性酶切长度多态性 (PCR-RFLP)、位点特异性PCR (AS-PCR)、DNA条形码技术 (DNA barcoding) 和环介导等温扩增技术 (LAMP) 等,它们分别具有各自的优势和劣势。依据检测对象的实际情况,在各个环节上选择最优技术,搭建模块化最适技术体系,是未来中药分子鉴定技术发展的重要方向[6]。
本文重点介绍了HRM技术的原理、仪器和试剂,并分析其在中药分子鉴定模块化最适技术体系建设中的优势和作用,旨在为其应用提供依据。
1 HRM技术基本原理熔解曲线 (melting curve) 是指随温度升高DNA双螺旋结构降解程度的曲线,核苷酸双链的热稳定性由其自身碱基组成、长度和GC含量决定,序列改变会引起升温过程中双链核苷酸解链行为的改变。1997年Wittwer等[7]提出了利用荧光分析来监测DNA的熔解,以实现对PCR产物、DNA序列的快速直接检测,即由于荧光染料可与双链DNA结合,随着加热升温使双链解离,荧光染料会从局部解链的DNA分子上释放,通过实时监测核苷酸双链熔解过程荧光信号的积累,就能直观地看到双链熔解随温度变化曲线的差异 (图 1A),对于野生型和突变型,通过归一化分析 (图 1B) 和差异化分析 (图 1C) 可以使其差异更加显著,从而进行突变扫描、序列匹配及定量检测等分析。与紫外检测技术相比,荧光分析的精确度更高,可以检测至ng级的DNA样品,具有所需样本量小、温度控制好、DNA熔解率快的特点。
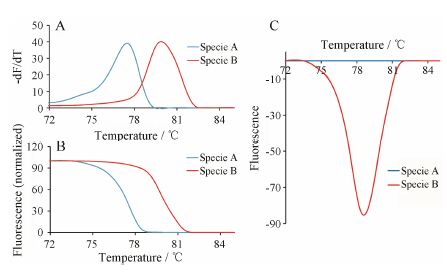
|
图 1熔解曲线差异分析。A: DNA熔解曲线; B: HRM归一化曲线; C: HRM差异曲线 |
2 HRM分析影响因素 2.1 HRM试剂
荧光染料是HRM分析技术的核心试剂,主要分为非饱和荧光染料 (如SYBR Green I等) 和饱和荧光染料 (如LC Green等)。染料的种类对HRM分析结果会产生一定的影响。目前,可用于HRM的商品化荧光染料不断增多,但是各自之间的效果不尽相同[8−11]。
2.1.1 SYBR Green I这类非饱和染料一般只能用于区分片段大小差别为400 bp以上或GC含量差异显著的DNA序列,无法实现单碱基的区分。其主要原因是非饱和性染料对PCR反应有抑制作用,且在DNA双链解链过程中,SYBR Green I染料分子容易发生重排,造成那些从解链DNA片段上脱离下来的染料分子又重新与尚未解链的双链DNA结合,影响了荧光检测的准确性[12−14]。因此,使用SYBR Green I时应注意其浓度要远低于使DNA双螺旋结构小沟饱和的浓度。然而也有研究发现,SYBR Green I 在Rotor-gene 6000系列仪器上用于 HRM分析的效果较好[15]。Pornprasert等[16]在相关研究中应用SYBR Green I染料进行多重PCR扩增和HRM分析,曲线清楚且分型准确。
2.1.2 LC Green和LC Green Plus作为最早的饱和染料,LC Green是由美国IdahoTechnology公司设计的一种专门用于异源双链核酸分子熔解曲线分析的新型核酸荧光染料,其对PCR反应不产生抑制作用,可在高饱和浓度下进行PCR扩增; 当双链DNA局部解链时,游离下来的染料亦不会重新结合到DNA分子上,所以荧光强度的降低可精准反映DNA分子的解链情况[2]。LC Green在单核苷酸多态性(SNP)位点突变检测,尤其是杂合位点分析以及多重PCR分析方面具有检测优势[17],但LC Green的加入会导致DNA解链温度升高1~3 ℃,且加入后反应体系中MgCl2的浓度应调整为2.0~3.0 mmol•L−1,一般只用于HR-1仪器使用[18, 19]。在此基础上,IdahoTechnology公司又推出了HRM分析饱和荧光染料LC Green Plus,该染料具有更强的稳定性,可以配合用于多种仪器使用。
2.1.3 其他染料如Reso Light (RL) 是Roche公司为LC480设计的一种HRM分析饱和荧光染料,由于其具有特殊的结合特性,对目标序列染色的均一性好,可以获得较为清晰的熔解曲线。此外,Biotium公司生产的Eva Green是一种用于实时定量PCR的DNA结合染料,也具有较高的灵敏度,对PCR抑制作用小,同样适用于HRM分析。与SYBR Green系列染料相比,Eva Green稳定性强且无致突变性和细胞毒性,具有良好的应用前景。
2.1.4 染料浓度和加入反应时间适宜的染料浓度 将提高HRM 的灵敏度和准确性,当加入染料量过低 时,PCR 反应信号强度差,无法进行基因分型; 当使用浓度过高时会导致荧光溢出、降低HRM 的分辨率。 因此,合适的染料浓度可以最大限度地确保实验效 果[20]。至于染料加入的时间,蒋立斌等[11]认为染料在 PCR 反应之前或后加入,对HRM 结果无显著影响。
2.2 HRM 仪器HRM 分析对仪器具有较高的要求,常规熔解曲 线升温时每步升高1 ℃,采集荧光4~5 次,而高分辨 率熔解曲线为满足单碱基差异检测的要求,每步升温 0.02~0.1 ℃,每升高1 ℃采集荧光25 次。另一方面,HRM 作为一种高通量的突变筛查和SNP 分型的研究 方法,需要同时比较分析多个样品,且不同样品间Tm 值差异可能极小,因此对不同样品间温度的均一性要 求较高。如果两孔之间温度相差0.1 ℃,就可能导致熔 解温度相差0.1 ℃,影响HRM 分析结果。多数常规的 定量PCR 仪,孔间温度差异多在0.3~0.5 ℃,无法进 行HRM分析,因此涌现出了几款专门进行HRM分析 的仪器[21, 22]。
2.2.1 HR-1作为第一台专门分析HRM 的仪器,是 由Wittwer 实验室和Idaho Technology 公司联合 发明的。HR-1 的出现在HRM 基因分型[1, 2]、突变扫 描[23, 24]、序列比对[25]中开展了较好的研究。该仪器温 度控制精度高,熔解曲线的分析可在1~2 min 内完成,且每小时可分析45 个样本; 随着384 孔板的出现,每 小时内至少可检测4 000 个样本,满足了高通量检测 的需求。然而,HR-1 只能用于高分辨熔解曲线分析,不能进行PCR扩增,可与Roche公司Lightcycler系列 毛细管PCR扩增仪匹配使用,但分析结束后PCR产物 不易取出,无法进行后期测序等工作。
2.2.2 Light Scanner系列 Idaho Technology 公司研 发的高通量的96 孔板式HRM 检测仪,专门用于快速 基因突变扫描和基因分型。该仪器的敏感性和特异性 较好,且整个微孔板可同时进行检测、荧光采集迅速、 温度控制和数据获取优势明显。同时,检测所需时间 短、样品通量高。但该仪器不能进行PCR扩增,且不 能使用独立的PCR 管,易发生样本间污染[26]。Light Scanner 32 将实时荧光定量PCR扩增与HRM分析整 合,扩增结束后无需将PCR产物取出,直接进行HRM 分析,同时引入内标后几乎可以检测任何类型的碱基 突变[27]。
2.2.3 Light Cycler 480 系列德国Roche 公司生产 的具有HRM 分析功能的板式全自动荧光定量PCR 仪。该系列仪器采用革新的Therma-Base 技术,保证 了加热的均一性,并结合独特的散热鳍和高性能风扇 保证散热的快速,从而确保均一的孔间温度,确保了HRM 定量准确性和重复性。同时,在几分钟内可根据 研究需要,完成96 或384 反应模块的更换,不需要繁 琐的校正过程,可实现中高通量的自主选择。此外,该 仪器具有较好的光学检测系统,可以进行多重PCR、 多种探针研究模式分析等[28, 29]。
2.2.4 Rotor-gene 6000 系列澳大利亚Corbett Life Science 公司生产的以离心式为基础的定量PCR 仪。 该系列仪器的离心式设计可使样品始终处于管底,同 时可消除气泡、防止样品凝集,使得PCR反应和荧光 染料结合更充分,具有更好的温度和光学均一性。同 时,该仪器配备36、72、100 孔3 种模块,可选择UV 或红外光源等多个通道,且无需清洁样品孔及更换光 源,具有简单便捷的特点[30]。
2.3 HRM 分析其他影响因素PCR 扩增和HRM 分析是完成高分辨率熔解曲线 技术的两大主要步骤。除仪器、试剂外,模板DNA质 量、PCR 反应体系、PCR产物大小等均对HRM 分辨 能力产生影响,因此不同样品的PCR体系必须保证均 一性,且在原则上PCR产物需小于300 bp时,HRM分 析最为精确[31]。 此外,如果加入内参DNA,需要筛选 获得适合的模板DNA与内参DNA浓度比,否则会影 响熔解曲线的峰形; 在实验中,不同样品中浓度比需 要保持一致,否则可能导致结果误判。由于非特异扩 增条带和引物二聚体的存在也会显著影响样品的熔解 曲线,因此优化PCR的条件以获得单一的扩增片段对 于HRM结果的准确性、可靠性十分重要。
3 HRM 技术优势和限制 3.1 优势与其他突变扫描、基因分型的方法相比,HRM 检 测的优势主要表现在: ① 通量大: 单次分析,在10 分 钟内可同时完成96或384个样本的分析[3]; ② 成本低: 在real-time 仪器上可以运行HRM 分析的情况下,HRM 分析所需的主要试剂是饱和荧光染料,其单次 成本明显低于同类分析 (如变性高效液相色谱分析,DHPLC),同时因其本身闭管反应而拥有极低的出错 率[20]; ③ 灵敏度高: 当PCR产物小于400 bp,敏感度 和特异度是100%; 当PCR产物长度在400~1 000 bp 之间,敏感度是96.1%,特异度是99.4%[32]; ④ 操作简 便: 无需特异性探针,只需要在普通PCR 反应的基础 上加上饱和荧光染料,即可直接进行HRM分析,完成 对样品基因分型; 大部分基于DNA 的分析常规技术,操作过程和分析结果相互分开,需要一个分离步骤,需要PCR产物在凝胶或其他基质,通常需要几个小时 来执行,并增加污染的风险,而HRM技术则是完全一体的,实现了闭管操作,避免了PCR 产物的污染,不 会造成PCR 产物的损伤,可直接用于下游分析[9]; ⑤ 快速: 完成PCR反应后,HRM分析过程可在几分钟内 完成[1]。
3.2 适用限制在HRM分析过程中,仍存在一些技术限制,主要表现为: ① 在PCR产物片段较大的情况下,HRM分析精确度会降低[32]; ② 无法有效区分不同的杂合子样品。研究表明不同纯合子样品的熔解曲线峰形相似,且因熔解温度不同可以区分; 而杂合子样品由于可以产生熔解温度较同源双链低的异源双链,造成同源双链和异源双链形成一条复合的熔解曲线,该曲线在峰形上与纯合体不同,因此杂合体与纯合体样品可以轻易通过曲线峰形差异进行区分[33],但无法用于有效区分不同杂合子; ③ HRM分析技术只能作为一种突变筛查方法,要确定突变的位置和类型,还需进一步测序[34]; ④ HRM分析的准确性受PCR反应质量影响,且难以检测高GC含量DNA片段中的突变或多个位点同时突变。
4 HRM在中药分子鉴定中的应用中药分子鉴定的基础是标记的筛选、获得和检测。HRM分析技术因其高度的敏感性和特异性,在序列差异扫描、单核苷酸多态性、插入/缺失 (Indel)、微卫星 (SSR)、甲基化检测及定量分析中均具有显著的优势。
4.1 序列差异扫描HRM技术可以直接运用于序列差异的扫描。只要待检物种DNA序列存在差异,可直接设计通用引物,通过普通PCR扩增后,加入饱和染料进行HRM分析,利用扩增子的熔解行为的不同,从而对不同物种进行区分。目前已有使用HRM进行序列差异扫描从而进行物种鉴定的研究,如服用铁筷子属黑嚏根草 (Helleborus niger) 过量会产生毒性,Schmiderer等[35]对其与同属易混品的trnL-trnF和matK序列进行HRM分析并加以区分。该方法也可直接运用于多来源中药材混淆品的鉴定,如使用HRM技术对升麻等药材进行DNA分子鉴别,对易混淆药材人参和西洋参原植物及其生药饮品的鉴别等[36, 37]。由于HRM分析可同时对多个样品进行并行检测,当使用96孔板或384孔板时可极大提高检测通量,达到高通量快速鉴别的目的。
4.2 单核苷酸多态性基因分型单核苷酸多态性作为新一代遗传标记,是由单个碱基的转换或颠换引起的点突变,通常以二等位基因的形式出现,具有密度高、遗传稳定性好、二等位性、分析易自动化等特点[38, 39],在中药材真伪鉴别中具有重要意义[40]。熔解曲线具有高分辨率的特点,可成为极好的SNP位点筛选工具。蒋超等[41]将限制性内切酶引入熔解曲线分析中,来检测位于或不位于内切酶识别序列区的SNP位点。2004年,Liew等[42]提出将HRM技术用于DNA小片段的SNP分型,实现致病性SNP突变的筛查。Liew等针对50 bp以下的DNA小片段进行HRM分析,表明纯合子之间可以通过熔解温度 (melting temperature,Tm) 加以区分,但是有接近四分之一的纯合子之间由于自身相近,表现出一定的临近性和对称性,使得Tm值差异较小 (< 0.4 ℃),造成这些纯合子之间无法区分,但通过其自身熔解曲线图形上的变化可进行区分。HRM技术应用于SNP基因分型的方法,简单快速、稳定性好、且可实现准确SNP分型的目的[43, 44]。
4.3 DNA条形码的HRM扫描2003年,加拿大动物学家Hebert等首次提出了DNA条形码,它是利用基因组中一段公认标准短序列来进行物种鉴定的分子诊断新技术[45]。近年来,DNA条形码技术已应用于中药鉴定领域[46−48]。将HRM技术与DNA条形码相结合[49, 50],可充分发挥HRM准确、高通量、快速、可视化的特点,进行中药材基原物种鉴定。Kalivas等[51]针对希腊地区唇形科毒马草属 (Sideritis) 内若干形态相似的植物,通过对其ITS条形码序列进行HRM分析的方法,对毒马草属物种进行区分。陈康等[52]对鹿茸正伪品COI条形码片段熔解曲线峰形和Tm值进行比较分析,建立了基于HRM技术的鹿茸分子鉴定方法,发现梅花鹿、马鹿与常见混淆品基原驯鹿、白唇鹿的熔解曲线均具有区别,见图 2。此外,HRM技术在西红花[53]、羚羊角[54]等药材鉴别中也建立了相应的方法。
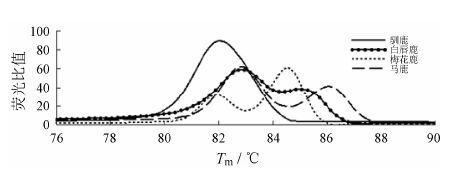
|
图 2 鹿茸熔解曲线差异[52] |
简单序列重复 (Simple sequence repeat,SSR) 是一类一般由1~6个碱基组成的基元串联重复而成DNA序列,具有分布广泛、共显性遗传、多态位点多、信息含量丰富、物种间转移性好、易于检测和重复性好的特点[55],可分为基因组SSR和EST-SSR两大类型。在各种类型的分子标记中,SSR标记需要的DNA量少,对DNA质量要求不高,易于PCR检测,适合进行高通量分析,这些优点使SSR标记在药用植物遗传多样性、物种鉴定等方面广泛应用[56]。HRM技术与SSR标记联合目前主要应用于品种鉴定[56−61]。作为新颖的高通量植物品种的检验方法,与常规聚丙烯酰胺凝胶电泳或毛细管电泳分型相比,不需要PCR后续处理等步骤[62],大大降低了检测时间。该方法将在中药材种质鉴定、遗传图谱构建、基因定位、辅助标记选择、突变检测等方面发挥重要作用。
4.5 甲基化检测DNA甲基化是发生在DNA碱基序列上的一种共价修饰作用,也是表观遗传修饰的重要组成部分。甲基化状态的改变可导致基因结构和功能异常,从而造成生物体表型的差异。HRM技术可用于高通量、高灵敏度、简便快速的DNA甲基化筛选,如甲基化敏感的HRM分析 (methylation-sensitive high resolution melt-ing,MS-HRM) 是一种将不同比例的甲基化和未甲基化DNA混合进行HRM分析,通过标准曲线计算样品中DNA甲基化的发生频率,且同时可通过比较亚硫酸氢盐处理前后DNA序列的改变,确定甲基化位点的技术。目前应用MS-HRM技术检测甲基化已较为广泛[63−67],该技术也将可能用于中药材产地鉴定、质量评价等方面。
4.6 定量分析2009年,Vossen等[68]提出HRM分析技术可以运用于DNA定量检测。Mader等[69]提出HRM在量化检测方面不但可以检测掺伪品,还能结合内参对目标物种在掺伪品中的比例进行定量分析。由于在欧盟市场上常有将较为廉价的果汁掺入较为昂贵的果汁,Faria等[70]筛选出对不同果汁具有相似DNA提取效率的方法,而后对橙子和芒果不同比例 (75∶25,50∶50,25∶75) 进行HRM分析,进而得到两者混掺后,不同比例的图谱,为果汁中混掺现象定量鉴定提供了方法。童宇茹等[71]利用熔解曲线分析技术对三七粉掺伪问题也进行了探讨和研究。可以预见,HRM技术在中药材粉末鉴定以及原药材掺伪鉴定中均将具有很高的应用价值。
5 展望HRM 具有高通量、高灵敏度、特异性好、无污染、重复性好、操作简单、成本低等优势,为中药分子鉴定带来了更为简便、快速的检测方法。目前HRM分析技术仅在金银花、羚羊角、鹿茸、三七等少数药材真伪及掺杂品鉴定中得到应用,且采用的分子标记主要是SNP和DNA条形码片段,其他分子标记报道很少。
随着研究的深入,未来HRM分析技术将在物种鉴定、中成药原料鉴定、掺伪药材的定量鉴定、现场鉴定技术优化等方面发挥重要作用,并逐渐形成中药材HRM数据库,推动多层次中药鉴定体系的构建。
| [1] | Gundry CN, Vandersteen JG, Reed GH, et al. Amplicon melting analysis with labeled primers:a closed-tube method for differentiating homozygotes and heterozygotes[J]. Clin Chem, 2003, 49:396-406. |
| [2] | Wittwer CT, Reed GH, Gundry CN, et al. High resolution genotyping by amplicon melting analysis using LCGreen[J]. Clin Chem, 2003, 49:853-860. |
| [3] | Reed GH, Kent JO, Wittwer CT. High-resolution DNA melting analysis for simple and efficient molecular diagnostics[J]. Future Med, 2007, 8:597-608. |
| [4] | Huang LQ. Prospects for application of molecular biotech-nique to pharmacognosy[J]. China J Chin Mater Med(中国中药杂志), 1995, 20:643-645. |
| [5] | Yuan Y, Jiang C, Huang LQ, et al. Strategy and practice on scene application of molecular identification in Chinese medicine materials[J]. China J Chin Mater Med(中国中药杂志), 2013, 38:2553-2555. |
| [6] | Huang LQ, Yuan Y, Yuan QJ, et al. Key problems in development of molecular identification in traditional Chinese medicine[J]. China J Chin Mater Med(中国中药杂志), 2014, 39:3663-3667. |
| [7] | Ririe KM, Rasmussen RP, Wittwer CT. Product differentia-tion by analysis of DNA melting curves during the polymerase chain reaction[J]. Anal Biochem, 1997, 245:154-160. |
| [8] | Herrmann MG, Durtschi JD, Bromley LK, et al. Amplicon DNA melting analysis for mutation scanning and genotyping:cross-platform comparison of instruments and dyes[J]. Clin Chem, 2006, 52:494-503. |
| [9] | Farrar JS, Reed GH, Wittwer CT. High Resolution Melting Curve Analysis for Molecular Diagnostics[M]. 2nd ed. Amsterdam:Molecular Diagnostics, 2009:229-245. |
| [10] | You CG, Li XJ, Gao LN, et al. Comparison of common fluorescent dyes for SNP genotyping in HRM technology[J]. Chin J Clin Lab Sci(临床检验杂志), 2011, 29:575-577. |
| [11] | Jiang LB, Zhang SY, Yang H, et al. Comparison of com-mon fluorescent dyes for SNP genotyping in FecB gene in sheep[J]. Chin J Anim Sci(中国畜牧杂志), 2014, 50:23-25. |
| [12] | Pirulli D, Boniotto M, Puzzer D, et al. Flexibility of melt-ing temperature assay for rapid detection of insertions, deletions, and single-point mutations of the AGXT gene responsible for type 1 primary hyperoxaluria[J]. Clin Chem, 2000, 46:1842-1844. |
| [13] | Lipsky RH, Mazzanti CM, Rudolph JG, et al. DNA melting analysis for detection of single nucleotide polymorphisms[J]. Clin Chem, 2001, 47:635-644. |
| [14] | Dufresne SD, Belloni DR, Wells WA, et al. BRCA1 and BRCA2 mutation screening using SmartCycler II high-resolution melt curve analysis[J]. Arch Path Lab Med, 2006, 130:185-187. |
| [15] | Pornprasert S, Phusua A, Suanta S, et al. Detection of alpha-thalassemia-1 Southeast Asian type using real-time gap-PCR with SYBR Green1 and high resolution melting analysis[J]. Eur Haematol, 2008, 80:510-514. |
| [16] | Pornprasert S, Wiengkum T, Srithep S, et al. Detection of α-thalassemia-1 Southeast Asian and Thai type deletions and β-thalassemia 3.5-kb deletion by single-tube multiplex real-time PCR with SYBR green1 and high-resolution melting analysis[J]. Korean J Lab Med, 2011, 31:138-142. |
| [17] | Gorringe KL, Choong DYH, Williams LH, et al. Mutation and methylation analysis of the chromo-domain-helicase-DNA binding 5 gene in ovarian cancer[J]. Neoplasia, 2008, 10:1253-1258. |
| [18] | Guo XL, You CG. Instruments and fluorescent dyes generally used in high resolution melting technique[J]. J Mol Diagn Ther(分子诊断与治疗杂志), 2012, 4:50-53. |
| [19] | Zhou L, Myers AN, Vandersteen JG, et al. Closed-tube genotyping with unlabeled oligonucleotide probes and a saturating DNA dye[J]. Clin Chem, 2004, 50:1328-1335. |
| [20] | Montgomery JL, Sanford LN, Wittwer CT. High resolution DNA melting analysis in clinical research and diagnostics[J]. Expert Rev Mol Diagn, 2010, 10:219-240. |
| [21] | Herrmann MG, Durtschi JD, Bromley LK, et al. Instrument comparison for heterozygote scanning of single and double heterozygotes:a correction and extension of herrmann[J]. Clin Chem, 2007, 53:150-152. |
| [22] | Herrmann MG, Durtschi JD, Wittwer CT, et al. Expanded instrument comparison of amplicon DNA melting analysis for mutation scanning and genotyping[J]. Clin Chem, 2007, 53:1544-1548. |
| [23] | McKinney JT, Longo N, Hahn SH, et al. Rapid, comprehensive screening of the human medium chain acyl-CoA dehydrogenase gene[J]. Mol Genet Metab, 2004, 82:112-120. |
| [24] | Willmore C, Holden JA, Zhou L, et al. Detection of c-kit-activating mutations in gastrointestinal stromal tumors by high-resolution amplicon melting analysis[J]. Am J Clin Path, 2004, 122:206-216. |
| [25] | Zhou L, Vandersteen J, Wang L, et al. High-resolution DNA melting curve analysis to establish HLA genotypic identity[J]. Tissue Antigens, 2004, 64:156-164 |
| [26] | van der Stoep N, van Paridon CDM, Janssens T, et al. Diagnostic guidelines for high-resolution melting curve(HRM) analysis:an interlaboratory validation of BRCA1 mutation scanning using the 96-well LightScanner[J]. Hum Mutat, 2009, 30:899-909. |
| [27] | Liew M, Wittwer C, Voelkerding KV. Nucleotide extension genotyping by high-resolution melting[J]. J Mol Diagn, 2010, 12:731-738. |
| [28] | Deback C, Géli J, Aït-Arkoub Z, et al. Use of the Roche LightCycler® 480 system in a routine laboratory setting for molecular diagnosis of opportunistic viral infections:evaluation on whole blood specimens and proficiency panels[J]. J Virol Methods, 2009, 159:291-294. |
| [29] | Derzelle S, Mendy C, Laroche S, et al. Use of high-resolution melting and melting temperature-shift assays for specific detection and identification of Bacillus anthracis based on single nucleotide discrimination[J]. J Microbiol Methods, 2011, 87:195-201. |
| [30] | Tuohy MJ, Reja V, Park S, et al. Use of a high-resolution melt assay to characterize codon 54 of the cyp51A gene of Aspergillus fumigatus on a Rotor-Gene 6000 instrument[J]. Antimicrob Agents Chemther, 2010, 54:2248-2251. |
| [31] | Wu L, Wang Y, Sun MY, et al. Optimization of high resolution melting analysis system for Fad3a gene of soybean fatty acid[J]. Soybean Sci(大豆科学), 2012, 31:882-886, |
| [32] | Reed GH, Wittwer CT. Sensitivity and specificity of sin-gle-nucleotide polymorphism scanning by high-resolution melting analysis[J]. Clin Chem, 2004, 50:1748-1754. |
| [33] | Wittwer CT. High resolution DNA melting analysis:advance-ments and limitations[J]. Hum Mutat, 2009, 30:857-859. |
| [34] | Erali M, Voelkerding KV, Wittwer CT. High resolution melting applications for clinical laboratory medicine[J]. Exp Mol Pathol, 2008, 85:50-58. |
| [35] | Schmiderer C, Mader E, Novak J, et al. DNA-based identification of Helleborus niger by high-resolution melting analysis[J]. Planta Med, 2010, 76:1934-1937. |
| [36] | Li D, Wang Q, Xue C, et al. Application of light cycler polymerase chain reaction and melting curve analysis to the authentication of the traditional Chinese medicinal plant Cimicifuga foetida[J]. Planta Med, 2009, 75:873-875. |
| [37] | Jung J, Kim KH, Yang K, et al. Practical application of DNA markers for high-throughput authentication of Panax ginseng and Panax quinquefolius from commercial ginseng products[J]. J Ginseng Res, 2014, 38:123-129. |
| [38] | Zou YP, Ge S. A novel molecular marker-SNPs and its application[J]. Biodiver Sci(生物多样性), 2003, 11:370-382. |
| [39] | Luo HR, Shi P, Zhang YP. Detection for single nucleotide polymorphisms[J]. Hereditas(遗传), 2001, 23:471-476. |
| [40] | Cui GH, Tang XJ, Huang LQ, et al. Application of multi-plex allele-specific PCR for authentication of Panax ginseng and P. quinquefolius[J]. China J Chin Mater Med(中国中药杂志), 2006, 31:1940-1943. |
| [41] | Jiang C, Huang LQ, Yuan Y, et al. Restriction endonuclease digest-melting curve analysis:a new SNP genotyping and its application in traditional Chinese medicine authentication[J]. Acta Pharm Sin(药学学报), 2014, 49:558-565. |
| [42] | Liew M, Pryor R, Palais R, et al. Genotyping of single-nucleotide polymorphisms by high-resolution melting of small amplicons[J]. Clin Chem, 2004, 50:1156-1164. |
| [43] | Garritano S, Gemignani F, Voegele C, et al. Determining the effectiveness of high resolution melting analysis for SNP genotyping and mutation scanning at the TP53 locus[J]. BMC Genet, 2009, 10:5. |
| [44] | You CG, Li YM, Ma KJ, et al. Evaluation on the methodology of SNP genotyping based on high resolution melting[J]. Chin J Clin(中华临床医师杂志), 2012, 6:1789-1790. |
| [45] | Hebert PDN, Cywinska A, Ball SL. Biological identifica-tions through DNA barcodes[J]. Proc Biol Sci, 2003, 270:313-321. |
| [46] | Jiang C, Huang LQ, Yuan Y, et al. Comparison of several methods and their progresses on molecular authentication of Lonicera japonica Thunb[J]. World Sci Technol-Mod Tradit Chin Med Mater Med(世界科学技术-中药现代化), 2014, 16:1831-1839. |
| [47] | Xu SZ, Li DZ, Li JW, et al. Evaluation of the DNA bar-codes in Dendrobium(Orchidaceae) from mainland Asia[J]. PLoS One, 2015, 10:e0115168. |
| [48] | Chen SL,Yao H, Han JP, et al. Principles for molecular identification of traditional Chinese materia medica using DNA barcoding[J]. China J Chin Mater Med(中国中药杂志), 2013, 38:141-147. |
| [49] | Madesis P, Ganopoulos I, Anagnostis A, et al. The application of Bar-HRM(barcode DNA-high resolution melting) analysis for authenticity testing and quantitative detection of bean crops(Leguminosae) without prior DNA purification[J]. Food Control, 2012, 25:576-582. |
| [50] | Jaakola L, Suokas M, Häggman H. Novel approaches based on DNA barcoding and high-resolution melting of amplicons for authenticity analyses of berry species[J]. Food Chem, 2010, 123:494-500. |
| [51] | Kalivas A, Ganopoulos I, Xanthopoulou A, et al. DNA barcode ITS2 coupled with high resolution melting(HRM) analysis for taxonomic identification of Sideritis species growing in Greece[J]. Mol Biol Rep, 2014, 41:5147-5155. |
| [52] | Chen K, Jiang C, Yuan Y, et al. Molecular identification of hairy antler by analysis of high resolution melting[J]. China J Chin Mater Med(中国中药杂志), 2015, 40:619-623. |
| [53] | Jiang C, Cao L, Yuan Y, et al. Barcoding melting curve analysis for rapid, sensitive, and discriminating authentication of Saffron(Crocus sativus L.) from its adulterants[J]. BioMed Res Int, 2014, 2014:809037. |
| [54] | Tong RU, Jiang C, Yuan Y, et al. Molecular identification of antelope horn by melting curve analysis[J]. Mitochondrial DNA, 2014, 26:1-7. |
| [55] | Zhang LR, Xu DQ, Liu D. SSR marker, ISSR marker and their to plant genetics and breeding[J]. J Agric Univ Hebei(河北农业大学学报), 2002, 25:90-94. |
| [56] | Ganopoulos I, Bosmali I, Madesis P, et al. Microsatellite genotyping with HRM(high resolution melting) analysis for identification of the PGI common bean variety Plake Megalosperma Prespon[J]. Eur Food Res Technol, 2012, 234:501-508. |
| [57] | Jiang C, Yuan Y, Liu GM, et al. EST-SSR identification of Lonicera japonica Thunb[J]. Acta Pharm Sin(药学学报), 2012, 47:803-810. |
| [58] | Mader E, Lukas B, Novak J. A strategy to setup codominant microsatellite analysis for high-resolution-melting-curve-analysis(HRM)[J]. BMC Genet, 2008, 9:69. |
| [59] | Zhao JL, Zhang SH, Liu B. Application of high-resolution melting curve analysis for molecular mark genotying in rice[J]. Sci Agric Sin(中国农业科学), 2011, 44:3701-3708. |
| [60] | Yin H, Wang CH, Tian YT, et al. Differentiation of microsa-tellites in pear by high resolution melting(HRM) analysis[J]. Acta Hort Sin(园艺学报), 2011, 38:1601-1606 |
| [61] | Bai MD, Wang CH. Differentiation of microsatellites in apple by high resolution melting(HRM) analysis[J]. Acta Hort Sin(园艺学报), 2012, 39:2584-2585. |
| [62] | Mackay JF, Wright CD, Bonfiglioli RG. A new approach to varietal identification in plants by microsatellite high resolution melting analysis:application to the verification of grapevine and olive cultivars[J]. Plant Methods, 2008, 4:8. |
| [63] | Wojdacz TK, Dobrovic A. Methylation-sensitive high resolution melting(MS-HRM):a new approach for sensitive and high-throughput assessment of methylation[J]. Nucleic Acids Res, 2007, 35:e41. |
| [64] | White HE, Hall VJ, Cross NC P, et al. Methyla-tion-sensitive high-resolution melting-curve analysis of the SNRPN gene as a diagnostic screen for Prader-Willi and Angelman syndromes[J]. Clin Chem, 2007, 53:1960-1962. |
| [65] | Wojdacz TK, Dobrovic A, Hansen LL. Methyla-tion-sensitive high-resolution melting[J]. Nat Protoc, 2008, 3:1903-1908. |
| [66] | Wojdacz TK, Dobrovic A, Algar EM. Rapid detection of methylation change at H19 in human imprinting disorders using methylation-sensitive high-resolution melting[J]. Hum Mutat, 2008, 29:1255-1260. |
| [67] | Candiloro ILM, Mikeska T, Dobrovic A. Assessing combined methylation-sensitive high resolution melting and pyrosequencing for the analysis of heterogeneous DNA methylation[J]. Epigenetics, 2011, 6:500-507. |
| [68] | Vossen RHAM, Aten E, Roos A, et al. High-resolution melting analysis(HRMA)-more than just sequence variant screening[J]. Hum mutat, 2009, 30:860-866. |
| [69] | Mader E, Ruzicka J, Schmiderer C, et al. Quantitative high-resolution melting analysis for detecting adulterations[J]. Anal Biochem, 2011, 409:153-155. |
| [70] | Faria MA, Magalhães A, Nunes ME, et al. High resolution melting of trnL amplicons in fruit juices authentication[J]. Food control, 2013, 33:136-141. |
| [71] | Tong YR, Jiang C, Huang LQ, et al. Molecular identifica-tion of Radix Notoginseng powder by DNA melt curve analysis[J]. Chin J Pharm Anal(药物分析杂志), 2014, 34:1384-1390. |
 2015, Vol. 50
2015, Vol. 50


