2. 南方医科大学药学院, 广东广州 510515;
3. 美国纽约血液中心Lindsley F. Kimball研究所, 纽约州纽约市 10065
2. School of Pharmaceutical Sciences, Southern Medical University, Guangzhou 510515, China;
3. Lindsley F. Kimball Research Institute, New York Blood Center, New York 10065, USA
2012年,中东地区发现一种新型人冠状病毒,感染人体后可出现严重的呼吸综合征,因此,该病毒被命名为中东呼吸综合征 (MERS) 冠状病毒 (MERS- CoV)[1, 2]。MERS的临床表现和严重急性呼吸综合征 (SARS) 类似,可出现发热、咳嗽和呼吸困难等呼吸系统症状,严重者甚至会出现肺、肝、肾等多器官功能衰竭[3, 4]。据世界卫生组织 (WHO) 报道,截至2015年9月2日,全球范围内已有MERS-CoV感染的确诊病例1 569例,其中死亡554例,致死率高达35%[5],远高于2012年爆发的SARS-CoV的死亡率 (约10%),给人类的生存与健康带来了严重的威胁。
MERS-CoV是单股正链RNA包膜病毒,属冠状病毒科,β属的C系群 (lineage),是继SARS-CoV后第二个对人类高致病性的冠状病毒。MERS-CoV的基因组全长约30 119 bp,包含三个开放阅读框,可编码S、E、M、N四种结构蛋白,其中S蛋白在侵染靶细胞过程中起重要作用。MERS-CoV的受体是二肽基肽酶 (dipeptidyl peptidase 4,DPP4或CD26),该分子在人体细胞中广泛表达。在MERS-CoV传播至人的过程中,蝙蝠可能是其自然宿主[6, 7, 8],而单峰骆驼则是其中间宿主[9]。
MERS-CoV病毒与同属于I型包膜病毒的人类免疫缺陷病毒 (HIV) 或SARS-CoV一样有着类似的膜融合进入靶细胞的机制[10, 11] (图 1A)。早在90年代初,Jiang等[12]发现了第1个衍生于HIV gp41蛋白HR2区的多肽类进入抑制剂SJ-2176,随后其专利转让给美国Trimeris药物公司开发出世界第1个抗HIV的多肽类药物Enfuvirtide (T20)[13]。Liu等[11]在2003年发现了第1个抗SARS-CoV的多肽CP-1。而对于2012年爆发的MERS-CoV,采用了类似的研究策略,多肽类MERS-CoV进入抑制剂很快被设计出来,其体内外的有效性及作用机制的研究也有了快速的发展。本文将对多肽类MERS-CoV进入抑制剂的研究进展进行综述。

|
Figure 1 The process of MERS-CoV fusion with and entry into the target cell (A) and the mechanism of action of the peptidic MERS-CoV entry inhibitors (B). A: After the S1 subunit binds with receptor DPP4,the fusion peptide (FP) inserts into the target cell membrane and HR1 and HR2 interact with other to form 6-HB,bringing the viral and target cell membranes into close proximity for fusion; B: The HR2 peptide binds to the HR1-trimer to form heterologous 6-HB and block homologous 6-HB core formation between the viral HR1 and HR2 domains,resulting in inhibition of MERS-CoV fusion with and entry into the target cell |
MERS-CoV的S蛋白是I型跨膜糖蛋白,含1 353个氨基酸,主要由S1亚单位和S2亚单位构成,并 以三聚体状态呈现在细胞表面。其中S1亚单位负责与受体识别结合,而其受体结合域 (RBD) 分布于C端。S2亚单位主要介导病毒和细胞之间的膜融合,该区域包括融合肽 (FP)、七肽重复区 (HR1和HR2)、跨膜区 (TM) 及胞内区 (CP)。S1蛋白与受体DPP4结合后,S2蛋白构象发生改变,其中FP插入靶细胞膜中,同时诱导HR1和HR2区域相互作用形成六螺旋结构,从而介导病毒融合及病毒基因进入靶细胞进行复制[14] (图 1A)。
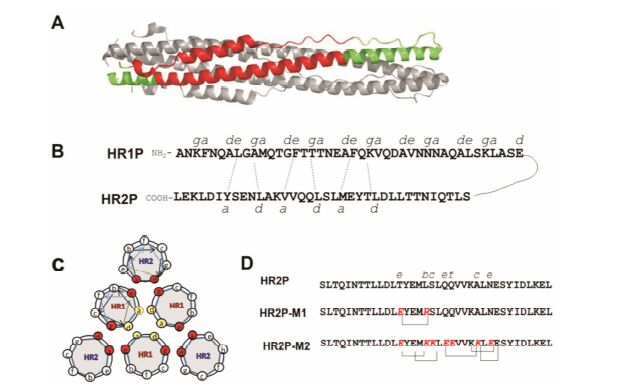
MERS-CoV S蛋白S2亚单位的六螺旋形成过 程是该病毒成功融合进入靶细胞的重要环节,也是多肽类进入抑制剂的重要作用靶点。因此,六螺旋结构成为研发MERS-CoV的进入抑制剂类药物的重要区域,并且其晶体结构也很快得到解析。Lu等[14]将MERS-CoV S蛋白与SARS-CoV的相应功能区域进行序列对比,把HR1和HR2区域分别定位于984~ 1 062和1 245~1 289,据此构建了融合蛋白HR1- L6-HR2 (L6: SGGRGG为HR1和HR2之间的链接序列),进而对该融合蛋白进行晶体解析,成功获得了高分辨的六螺旋结构 (图 2A)。Gao等[15]也开展了相似的工作,通过软件预测,将HR1和HR2区域分别定位于992~1 054和1 252~1 286。与Lu等解析的结构相比,其所解析的六螺旋结构中HR1的α-螺旋长度较短,但HR1和HR2所形成的六螺旋总体构象基本一致。在晶体结构中,HR1形成的α-螺旋结构较长,并以三聚体的状态位于六螺旋的中心,而HR2的α-螺旋较短,在疏水力的作用下,反向平行填补于HR1三聚体的沟槽中 (图 2A)。其中HR2的疏水性氨基酸较多地分布于两端,如N端的I1246、P1247、F1249、L1252、L1255和C端的I1281、L1283和L1286等位点。同时,在六螺旋两端,HR1和HR2存在较多的氢键,称之为N-和C-的帽子结构,它们进一步稳定六螺旋结构[14]。
|
Figure 2 A: The six-helix bundle structure of MERS-CoV S protein S2 subunit. The regions corresponding to the sequences of HR1P and HR2P peptides are in red color. B: The amino acid residues in HR1P and HR2P peptides involve the interaction between HR1 and HR2 domains. C: The amino acid residues located at the a,b,c,d,e,f and g positions in the helical wheels of HR1 and HR2. The residues located at the “a” and “d” positions (yellow) of the HR1 helical wheels interact with each other to form the internal trimer,while the residues of HR1 located at the “e” and “g” positions (red) interact with residues of HR2 at the “a” and “d” positions (red) to form the six-helix bundle structure. D: The optimized peptides with the residues modified to form intramolecular salt-bridge |
根据以前设计多肽类HIV和SARS-CoV进入抑制剂的经验,Jiang及同事们[14]首先根据六螺旋晶体结构确定HR1和HR2相互作用的α-螺旋区域,并以此为中心,设计了系列不同长短的多肽 (图 3),并分别对其生物学活性和抗病毒活性进行检测。在生物学活性的检测中,天然的聚丙烯酰胺凝胶电泳 (N-page或Fn-page) 检测设计的多肽是否可与其作用靶肽相结合,形成新的复合物[16]; 而圆二色谱(CD) 可分析多肽和多肽/作用靶肽复合物的二级结构。一般来说,单一的HR1多肽或HR2多肽的二级结构以无规卷曲为主,α-螺旋度很低; 但复合物中HR1多肽和HR2多肽在相互间疏水力的作用下,可诱导出大量的α-螺旋结构[17]。变温曲线 (Tm),则是通过监测复合物中α-螺旋含量在不同温度下的变化来检测复合物的稳定性。一般HR1多肽/HR2多肽复合物热稳定性越好,多肽的活性越高[14]。另外,凝胶排阻色谱技术可分析多肽/ 作用靶肽复合物分子的大小,从而进一步确定复合物是否以六聚体的状态存在[3]。在检测多肽的抗病毒活性方面,主要包括假病毒抑制实验、活病毒抑制实验以及MERS-CoV S蛋白介导的细胞融合实验[14]。

|
Figure 3 The schematic representation of MERS-CoV S protein and the sequences of the peptidic MERS-CoV entry inhibitors discussed in the article. The interaction regions in the HR1 and HR2 domains of the 6-HB are highlighted in green and red,respectively |
目前,Lu和Gao等[14, 15]设计了5条靶向于MERS- CoV HR1区域的HR2多肽,包括HR2L、HR2P、HR2S、P1及P2 (图 3)。其中HR2S最短,只含有六螺旋的核心区域,预期HR2S/HR1L (HR1L是HR1 α-螺旋区域的衍生肽) 复合物的α-螺旋含量可达100%,但检测后发现仅34.3%[14],提示MERS-CoV HR2 α-螺旋两端的氨基酸对六螺旋的稳定性非常重要。多肽P2也出现类似的现象,多肽P2因缺少α-螺旋N端的氨基酸,而显示完全没有抗病毒活性[15]。由此可推测并非只是HR2 α-螺旋中的氨基酸可影响多肽的抗病毒活性,α-螺旋两端的氨基酸对其抗病毒活性同样存在着重要的影响。相比之下,多肽HR2P和P1都有较好的抗病毒活性,并且多肽HR2P展现出很好的生物学活性,HR2P与其作用靶肽HR1P的结合可以很好地模拟病毒自身的HR1区域和HR2区域的结合 (图 2B),因而可用HR1P/HR2P的模拟体系来筛选或检测可抑制MERS-CoV六螺旋形成的小分子药物或多肽[16, 18]。多肽HR2L的抗病毒活性最好,但其序列也最长 (45个氨基酸)。鉴于对多肽活性的提高与多肽合成成本增加的综合考虑,该多肽没有进一步开发。源自S2亚单位HR2区域的衍生多肽可与病毒的HR1区域相互作用,形成异源的六螺旋,从而竞争性地抑制了病毒自身HR1与HR2六螺旋的形成,有效地抑制了病毒与靶细胞的膜融合及病毒基因进入靶细胞的过程,这是多肽类MERS-CoV进入抑制剂的主要作用机制[2] (图 1B)。另外,目前靶向于MERS- CoV的HR2区域的HR1多肽仅有三条 (HR1P、HR1M和HR1L)。虽然这三条多肽都处于HR1的α-螺旋中,但其中最长多肽HR1L (79个氨基酸) 也仅含有34.5%的α-螺旋,且这三条多肽在MERS-CoV的细胞融合实验中都无抑制活性[15]。HR1多肽的抗病毒活性差,是多肽类I型包膜病毒进入抑制剂中的普遍现象,如多肽类HIV进入抑制剂中,N端的衍生肽DP107和N36等抗病毒活性要明显低于C端的衍生肽 (如C34)[19, 20, 21]; SARS-CoV病毒的HR2多肽CP-1的抗病毒活性要显著高于HR1多肽NP-1[11]。同时还有研究发现,尽管MERS-CoV的六螺旋晶体结构与SARS-CoV相似,但多肽类SARS-CoV融合抑制剂 (如CP1和SARS-pep) 并没有抗MERS-CoV的活性,同样,衍生于MERS-CoV的HR2多肽也没有抑制SARS-CoV的活性[14, 15]。结果表明,衍生于MERS-CoV的HR2多肽HR2P能特异性地作用于MERS-CoV的S蛋白HR1区域,从而抑制了MERS- CoV与靶细胞膜的融合及进入靶细胞。
2.2多肽类MERS-CoV进入抑制剂的优化策略在I型包膜病毒的七肽重复区域中,a、d位点以疏水性氨基酸为主,并且在六螺旋中,HR1通过a、d位点的疏水作用以三聚体的状态位于六螺旋的中心,HR2同样通过a、d位点的疏水性氨基酸与HR1相互作用[14, 15] (图 2C)。同时,关于SARS-CoV的肽类融合抑制剂的研究表明,HR中的疏水性氨基酸对六螺旋的稳定性非常重要,而电荷量较大的氨基酸,如谷氨酸(E)、赖氨酸 (K) 或精氨酸 (R) 对六螺旋的稳定性并没有很大的影响[22]。另外,对HIV-1多肽类融合抑制剂的优化研究表明: 以C34和T20序列为模板,在b、c、f、g位点引入谷氨酸 (E) 和赖氨酸 (K) 突变 —— SC35EK、T20EK后,多肽的抗病毒活性不仅没有减弱,反而提高,并且大大增加多肽的水中溶解度。该多肽优化方案的机制是在多肽内 (非重要位点a、d) 引入E/K突变位点,促使多肽内形成EK盐桥,增加多肽的α-螺旋度和稳定性,从而提高多肽的抗病毒活性,同时也会大幅度地提高多肽的溶解度[23, 24]。在MERS-CoV的多肽类融合抑制剂的优化中,也采用了同样的措施,如在多肽HR2P的b、c、e、f位点,以“i+3”或“i+4”的组合方式引入谷氨酸和赖氨酸或精氨酸,增加多肽的内部盐桥,进而提高多肽 的溶解度及抗病毒活性 (图 2D)。其中,优化的多肽HR2P-M1引入T1263E (e) 和L1267R (b) 突变位点后,α-螺旋度提高1倍,在水中的溶解度提高近70倍,并且其抗病毒活性也有所提高。在优化的多肽HR2P-M2中,引入T1263E (e)、L1267K (b)、S1268K (c)、Q1270E (e)、Q1271E ( f )、A1275K (c) 和N1277E (e) 7个突变位点后,在水中的溶解度提高1 786倍,其抗MERS-CoV假病毒活性的半数抑制剂量 (IC50) 可达0.7 μmol·L-1以下,并且通过增加其浓度,HR2P-M2可100% 竞争性抑制多肽HR2P与HR1P的结合[14]。
3 多肽类MERS-CoV进入抑制剂抗病毒活性的广谱性随着MERS-CoV在世界各地的流行和爆发,越来越多的突变株不断产生,其中MERS-CoV S蛋白的RBD出现较多突变位点 (如482、506、530和534等位点)[25, 26]。另外,研究表明,在靶向于MERS-CoV RBD的抗体作用下,可造成RBD中一些位点的突变 (如T512A、Y540C和R542G等)[27],这些突变位点可能会为相关抗体药物的研制和疫苗的研发带来很多困难。与此相比,MERS-CoV S2亚单位的HR是非常保守的,其中仅HR1的1 020位点出现两种稳定的突变,即Q1020H或Q1020R[25]。Channappanavar等[16]证明,含该突变位点的毒株不仅没有抵抗多肽类MERS-CoV进入抑制剂的活性,反而变得更加敏感,如多肽HR2P-M2抗突变株病毒的活性比野生株提高3~5倍 (表 1)。一系列研究表明,1020Q可与1024D位点形成氢键,有利于增加HR1α-螺旋的稳定性[14]。若该位突变为R/H,可能会进一步增强1020位点和1024位点的氢键作用力。
|
|
Table 1 The biophysical properties and antiviral activity of the HR2P-M2 peptides against MERS-CoV wildtype strain and Q1020H/R mutants[16] |
多肽类MERS-CoV进入抑制剂不仅在细胞水 平可有效地抑制MERS-CoV,在动物体内同样有较好的预防和保护效果。Zhao等[28]通过将人DPP4受体转染入小鼠的呼吸道上皮细胞的方法,快速构建出可被MERS-CoV感染的小鼠模型。在小鼠模型上,Channappanavar等[16]在攻毒前将多肽HR2P-M2 (每只200 μg) 通过鼻道给药,6 h后再用高病毒载量的MERS-CoV感染小鼠的呼吸道,3天后小鼠的肺部病毒载量下降了1 000多倍,甚至使Q1020R/H突变株病毒载量下降到检测水平以下,与阴性对照组之间有显著性差异。在治疗方面,通过鼻道给药,单一多肽HR2P-M2的抗病毒效果甚至比单一的干扰素β (IFN-β)更好,与IFN-β联合用药时,在预防和治疗方面都具有很好的协同抗病毒效果[16]。
5 抗MERS-CoV多肽类融合抑制剂的应用前景目前临床上尚无治疗MERS-CoV的特异性药 物,而主要使用的抗MERS-CoV药物有干扰素和病毒唑等,但这些药物对重症患者的治疗效果还不十分确定[29]。一些科学家在药物库中筛选出可以抑制MERS-CoV感染的药物,其中神经递质抑制剂类药物可以明显抑制MERS病毒进入细胞过程,其作用机制可能主要是通过抑制Clathrin介导的内吞作用[30, 31]。然而也有相关研究发现,这类药物中的盐酸氯丙嗪、盐酸异丙嗪和盐酸氟奋乃静可明显抑制MERS-CoV的细胞融合过程,但Liu等[18]通过六螺旋抑制实验证明,这些药物并不能抑制MERS-CoV S蛋白的六螺旋形成,说明它们的作用靶点并不是六螺旋形成过程。因此,这些药物与多肽类融合抑制剂联合用药,应该有较好的协同保护效果。另外,目前MERS抗体的研究领域也取得较大的发展,其中Ying等[29]筛选出的抗体m336抗病毒效果非常好,IC50可达0.005 μg·mL-1,其作用靶点是S1蛋白上的RBD区域[32],与多肽类融合抑制剂联用,应该会有很好的协同效果,同时可以避免单一抗体药物作用下出现的免疫逃逸现象。动物实验研究同样表明,多肽HR2P-M2与IFN-β联用时,抗病毒效果会有很大的提高,并可明显降低肺部的炎症[16]。
动物实验的结果表明,多肽类MERS-CoV进入抑制剂通过鼻道给药的途径,可达到较好的预防和治疗效果[16],进一步说明这些多肽抑制剂及其鼻道给药的方式,可在疫情严重的地区或人群中广泛推广,以用于预防和早期治疗。另外,在针对MERS- CoV感染晚期患者的治疗方面,多肽类MERS-CoV进入抑制可能还需要进一步被开发成为可注射给药的药物制剂,如抗HIV的多肽类融合抑制剂T20[33]。在抗MERS-CoV多肽药物注射给药方面,由于疗程短,治疗成本低,且不用考虑免疫原性等问题,因而同样会有较好的发展前景。鉴于目前MERS-CoV的疫情仍未被完全控制,且临床上仍然没有针对MERS-CoV的特异性药物和疫苗,多肽类进入抑制剂应进一步被开发为用于紧急预防和治疗的药物。
| [1] | Zaki AM, van Boheemen S, Bestebroer TM, et al. Iso-lation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. N Engl J Med, 2012, 367:1814-1820. |
| [2] | de Groot RJ, Baker SC, Baric RS, et al. Middle East respiratory syndrome coronavirus(MERS-CoV):announcement of the Coronavirus Study Group[J]. J Virol, 2013, 87:7790-7792. |
| [3] | Xia S, Liu Q, Wang Q, et al. Middle East respiratory syndrome coronavirus(MERS-CoV) entry inhibitors targeting spike protein[J]. Virus Res, 2014, 194:200-210. |
| [4] | Assiri A, Al-Tawfiq JA, Al-Rabeeah AA, et al. Epidemiol-ogical, demographic, and clinical characteristics of 47 cases of Middle East respiratory syndrome coronavirus disease from Saudi Arabia:a descriptive study[J]. Lancet Infect Dis, 2013, 13:752-761. |
| [5] | World Health Organization. Middle East respiratory syn-drome coronavirus(MERS-CoV)[EB/OL]. http://www.who.int/emergencies/mers-cov/en. 2015-09-19. |
| [6] | Cui J, Eden JS, Holmes EC, et al. Adaptive evolution of bat dipeptidyl peptidase 4(DPP4):implications for the origin and emergence of Middle East respiratory syndrome coronavirus[J]. Virol J, 2013, 10:304. |
| [7] | Drosten C, Kellam P, Memish ZA. Evidence for camel-to-human transmission of MERS coronavirus[J]. N Engl J Med, 2014, 371:1359-1360. |
| [8] | Memish ZA, Mishra N, Olival KJ, et al. Middle East respiratory syndrome coronavirus in bats, Saudi Arabia[J]. Emerg Infect Dis, 2013, 19:1819-1823. |
| [9] | Lu G, Wang Q, Gao GF. Bat-to-human:spike features determining 'host jump' of coronaviruses SARS-CoV, MERS-CoV, and beyond[J]. Trends Microbiol, 2015, 23:468-478. |
| [10] | Chan DC, Fass D, Berger JM, et al. Core structure of gp41 from the HIV envelope glycoprotein[J]. Cell, 1997, 89:263-273. |
| [11] | Liu S, Xiao G, Chen Y, et al. Interaction between heptad repeat 1 and 2 regions in spike protein of SARS-associated coronavirus:implications for virus fusogenic mechanism and identification of fusion inhibitors[J]. Lancet, 2004, 363:938-947. |
| [12] | Jiang S, Lin K, Strick N, et al. HIV-1 inhibition by a pep-tide[J]. Nature, 1993, 365:113. |
| [13] | Lalezari JP, Henry K, O'Hearn M, et al. Enfuvirtide, an HIV-1 fusion inhibitor, for drug-resistant HIV infection in North and South America[J]. N Engl J Med, 2003, 348:2175-2185. |
| [14] | Lu L, Liu Q, Zhu Y, et al. Structure-based discovery of Middle East respiratory syndrome coronavirus fusion inhibitor[J]. Nat Commun, 2014, 5:3067. |
| [15] | Gao J, Lu GW, Qi JX, et al. Structure of the fusion core and inhibition of fusion by a heptad repeat peptide derived from the S protein of middle east respiratory syndrome coronavirus[J]. J Virol, 2013, 87:13134-13140. |
| [16] | Channappanavar R, Lu L, Xia S, et al. Protective effect of intranasal regimens containing peptidic Middle East respira-tory syndrome coronavirus fusion inhibitor against MERS-CoV infection[J]. J Infect Dis, 2015, doi:10.1093/infdis/jiv325. |
| [17] | Chen YH, Yang JT, Chau KH. Determination of the helix and beta form of proteins in aqueous solution by circular dichroism[J]. Biochemistry, 1974, 13:3350-3359. |
| [18] | Liu Q, Xia S, Sun Z, et al. Testing of Middle East respira-tory syndrome coronavirus replication inhibitors for the ability to block viral entry[J]. Antimicrob Agents Chemother, 2015, 59:742-744. |
| [19] | Lu M, Blacklow SC, Kim PS. A trimeric structural domain of the HIV-1 transmembrane glycoprotein[J]. Nat Struct Biol, 1995, 2:1075-1082. |
| [20] | Wild C, Oas T, McDanal C, et al. A synthetic peptide inhibitor of human immunodeficiency virus replication:correlation between solution structure and viral inhibition[J]. Proc Natl Acad Sci U S A, 1992, 89:10537-10541. |
| [21] | He Y, Xiao Y, Song H, et al. Design and evaluation of sifuvirtide, a novel HIV-1 fusion inhibitor[J]. J Biol Chem, 2008, 283:11126-11134. |
| [22] | Aydin H, Al-Khooly D, Lee JE. Influence of hydrophobic and electrostatic residues on SARS-coronavirus S2 protein stability:insights into mechanisms of general viral fusion and inhibitor design[J]. Protein Sci, 2014, 23:603-617. |
| [23] | Nishikawa H, Oishi S, Fujita M, et al. Identification of minimal sequence for HIV-1 fusion inhibitors[J]. Bioorg Med Chem, 2008, 16:9184-9187. |
| [24] | Oishi S, Ito S, Nishikawa H, et al. Design of a novel HIV-1 fusion inhibitor that displays a minimal interface for binding affinity[J]. J Med Chem, 2008, 51:388-391. |
| [25] | Cotten M, Watson SJ, Zumla AI, et al. Spread, circulation, and evolution of the Middle East respiratory syndrome coronavirus[J]. Mbio, 2014, 5:e01062. |
| [26] | Kim YJ, Cho YJ, Kim DW, et al. Complete genome se-quence of Middle East respiratory syndrome coronavirus KOR/KNIH/002_05_2015, isolated in South Korea[J]. Genome Announc, 2015, 3:e00787. |
| [27] | Tang XC, Agnihothram SS, Jiao Y, et al. Identification of human neutralizing antibodies against MERS-CoV and their role in virus adaptive evolution[J]. Proc Natl Acad Sci U S A, 2014, 111:E2018-E2026. |
| [28] | Zhao J, Li K, Wohlford-Lenane C, et al. Rapid generation of a mouse model for Middle East respiratory syndrome[J]. Proc Natl Acad Sci U S A, 2014, 111:4970-4975. |
| [29] | Ying T, Du L, Ju TW, et al. Exceptionally potent neutralization of Middle East respiratory syndrome coronavirus by human monoclonal antibodies[J]. J Virol, 2014, 88:7796-7805. |
| [30] | de Wilde AH, Jochmans D, Posthuma CC, et al. Screening of an FDA-approved compound library identifies four small-molecule inhibitors of Middle East respiratory syndrome coronavirus replication in cell culture[J]. Antimicrob Agents Chemother, 2014, 58:4875-4884. |
| [31] | Dyall J, Coleman CM, Hert BJ, et al. Repurposing of clinically developed drugs for treatment of Middle East respiratory syndrome coronavirus infection[J]. Antimicrob Agents Chemother, 2014, 58:4885-4893. |
| [32] | Ying T, Prabakaran P, Du L, et al. Junctional and allele-specific residues are critical for MERS-CoV neutralization by an exceptionally potent germline-like antibody[J]. Nat Commun, 2015, 6:8223. |
| [33] | Bellibas SE, Siddique Z, Dorr A, et al. Pharmacokinetics of enfuvirtide in pediatric human immunodeficiency virus 1-infected patients receiving combination therapy[J]. Pediatr Infect Dis J, 2004, 23:1137-1141. |
 2015, Vol. 50
2015, Vol. 50


