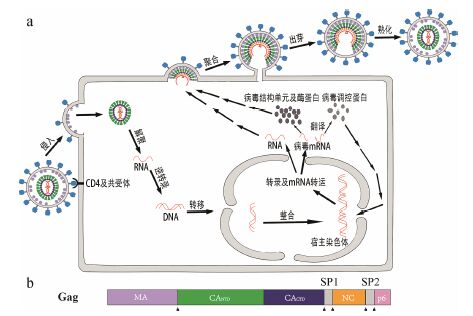
HIV-1是引起AIDS的病原体,据世界卫生组织2013年底统计,全世界3 500万人感染有HIV-1型病毒 (http://www.who.int/hiv/en/),针对该病毒缺乏有效的疫苗或者治愈手段[1]。HIV-1复制感染的机制 (图 1a): 病毒自身的融合蛋白gp120通过宿主细胞CD4受体识别T细胞,gp41 N端的融合肽结合宿主细胞膜,协助病毒所携带的RNA、逆转录酶、整合酶等进入T细胞,逆转录酶逆转录RNA合成得到双链DNA,同时自身降解,在转移至细胞核后通过整合酶整合到宿主细胞染色体,利用宿主细胞的合成机器进行病毒RNA复制和相关蛋白等物质的转录翻译,形成的RNA与Gag蛋白在细胞膜附近融出形成新的未成熟病毒分子,在水解酶作用下,Gag蛋白发生水解、重组装,形成成熟、具有感染能力的病毒分子,完成HIV病毒的感染及复制过程[2]。
|
图 1 a: HIV-1病毒的感染及复制过程; b: HIV-1 Gag蛋白的多个结构域MA、CANTD、CACTD、SP1、NC、SP2以及p6。黑色箭头处为酶切水解位点 |
针对病毒复制感染不同阶段的特异性靶点,目前抑制HIV病毒存在几类不同治疗办法[3, 4],至2009年,美国食品药品管理局 (FDA) 批准的抗HIV病毒药物可以分为以下4种类型[5, 6]: 逆转录酶抑制剂 (核苷类和非核苷类)、蛋白水解酶抑制剂、整合酶抑制剂和进入抑制剂 (entry inhibitor)[7]。绝大多数批准的药物属于逆转录酶抑制剂 (如zidovudine、lamivudine、emtricitabine等) 和蛋白水解酶抑制剂 (如saquinavir、fosamprenavir等),然而目前药物存在着耐药性的问题。针对进入机制的融合蛋白和共受体CCR5以及整合酶等靶点目前也有少量新药上市使用 (如enfuvirtide、maraviroc、raltegravir)[8],这些新型靶点药物开发研究仍在继续。病毒的特点是利用宿主细胞本身功能进行病毒的复制,只通过抑制宿主细胞一些蛋白功能抑制病毒会产生不良反应,而针对病毒自身含有复制生长的功能等靶点,药物则具有更好的选择性。因此病毒成熟机制可以作为一类新的作用靶点用于抗HIV病毒的药物开发。
2 病毒成熟关键步骤HIV病毒融出宿主细胞出芽形成非成熟HIV病毒,需要剪切Gag和Gag-pol聚合蛋白得到小的衣壳蛋白片段单元,衣壳蛋白单元重新组装后病毒才具有感染活性[9],这步组装过程成为病毒成熟关键步骤。在未成熟病毒分子中,Gag分子 (图 1b) N端的基质蛋白 (matrix,MA) 通过肉蔻脂肪链与细胞膜作用,同时MA N端的负电荷区域与磷脂层的酸性部分作用,使Gag蛋白铆定在细胞膜上,同时通过衣壳蛋白 (capsid,CA)-1号小肽 (small peptide 1,SP1) 间的相互作用,在细胞膜上形成多聚并融出宿主T细胞。未成熟病毒分子在水解酶的作用下,剪切成多个片段 (图 1)。剪切后的MA仍与细胞膜作用,CA重新组装,形成锥形体形状的病毒核心[10],核衣壳 (nucleocapsid,NC) 与RNA相互作用,位于病毒核心内部。病毒分子成熟过程中构象发生变化的衣壳蛋白CA[11]的结构信息得到了广大研究者的关注。
相关的基因突变实验和分子生物学研究均证实了Gag蛋白剪切和重组装对于病毒感染均至关重要[12]。大约含有5 000个Gag分子存在于宿主释放的每个未成熟HIV病毒内[13],含量为3 mmol·L-1,经水解酶切后,大约1 500个衣壳蛋白参与病毒的包装,形成平均直径为60~120 nm的锥形体结构[14, 15]。以剪切HIV Gag蛋白的酶为靶点的药物Bevirimat进行了1期临床试验[16, 17, 18],2期试验由于商业等因素暂时搁置。针对衣壳蛋白组装机制的抗HIV病毒药物研究仍然处于初期阶段,目前报道的多为具有活性的小分子和多肽。衣壳蛋白聚集组装的结构和机制直到最近才逐步被了解,导致基于结构的理性药物发现相关的研究并不是特别充分[19]。采取改变衣壳蛋白自组装的动力学过程抑制其他病毒感染力最近也被证实[20],如HBV抑制剂苯基丙烯酰胺类化合物能与衣壳蛋白聚集中间体相互作用,加快衣壳蛋白自组装的完成,对新生病毒的 感染性产生影响[20, 21]。
衣壳蛋白水解前后其聚集态结构发生了变化,通过单晶衍射、核磁共振、冷冻电镜以及动力学计算等方法可阐明Gag蛋白水解前后的结构特点[22, 23, 24, 25, 26, 27, 28],这些结构信息帮助解释成熟机制,有助于开展药物的设计和发现。
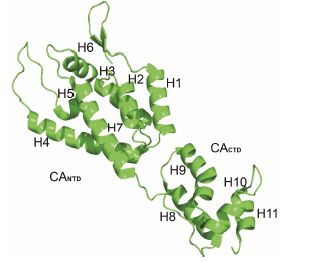
3 衣壳蛋白及其组装聚集体结构特点衣壳蛋白由两个结构域NTD (N-terminal domain) 和CTD (C-terminal domain)[29]连接组成[24, 30, 31]。其中NTD由H1~H7 (7个α-螺旋) 共约150个氨基酸残基组成,CTD有H8~H11 (4个α-螺旋) 共约70个氨基酸残基组成 (图 2)。NTD (residues 1-148; pdb ID: 1gwp) 和CTD (residues 149-219; pdb ID: 1a43) 各自的结构域的晶体结构和完整衣壳的晶体结构已经发表[24]。由于野生型衣壳多聚体不能形成晶体进行测量[32, 33],Pornillos等[25, 26]通过氨基酸残基突变的方 法,新增了蛋白分子间的二硫键得到稳定的六聚体 (A14C/E45C,W184A/M185A) 和五聚体 (P17C/T19C,N21C/A22C) 的晶体结构,依靠这两类多聚体结构单元,HIV衣壳形成类似富勒烯 (fullerene) 锥体结构。这种通过五聚体和六聚体组合形成富勒烯锥体结构的猜想随后也得到了冷冻电镜结构数据的支持[23]。衣壳蛋白的溶液核磁结构得到解析,验证了前面报道的关于衣壳蛋白CTD和NTD的结构信息[34]。
|
图 2 HIV-1衣壳蛋白CA的空间三维结构 |
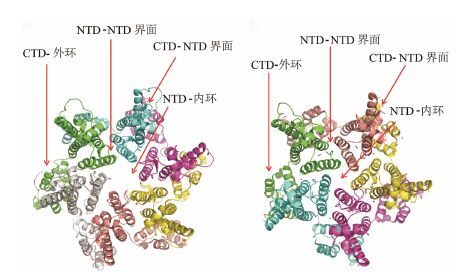
综上,衣壳蛋白五聚体和六聚体的结构单元中的单体,呈现出非常相类似的三级结构,仅在衣壳蛋白的非结构域范围存在细微差别 (主要存在于NTD-NTD相互作用部分),五聚体和六聚体两种聚集体除了依赖H2上的M39、A42,H3上的L20等疏水残基的疏水作用以外,五聚体通过水分子形成氢键连接相邻螺旋上亲水残基T54、T48等,而六聚体可以通过挤出水分子调整蛋白的结合结构,此外五聚体中Arg18可以采取更为紧密的静电相互作用。两种聚集体共同的特征是聚集体中心NTD依靠NTD-NTD相互作用结合组成了内环,聚集体的外环之间存在CTD-NTD相互作用,而CTD圈存在于最外圈,每个多聚体内部CTD圈之间不产生相互作用,但六聚体和五聚体之间靠CTD-CTD相互作用形成富勒烯球面。研究发现存在两类CTD-CTD相互作用模式,恰好反映六聚体与五聚体之间聚集的相互作用情况 (分别为六聚体与六聚体,六聚体与五聚体),它们的结构分别通过晶体和核磁数据得到揭示。五聚体和六聚体的晶体结构如图 3所示。
|
图 3 五聚体 (PDB ID: 3P05) 和六聚体 (3H47,3H4E) 对称结构中NTD内环、CTD外环、CTD-NTD界面、NTD-NTD界面 |
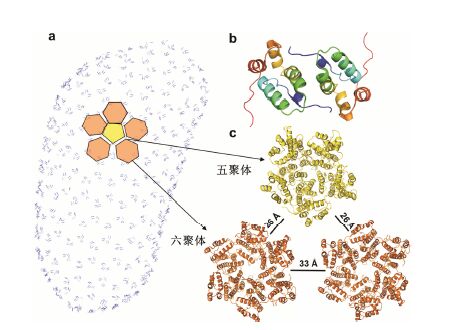
总体来看,CA蛋白聚集状态存在改变[35, 36],在水解前后单体的三维结构并没有变化。通过冷冻电镜断层扫描等技术解析未成熟的Gag蛋白中CA的聚集体结构证实了这一点[27]。大家已经知道CA蛋白在高 盐条件下体外多聚形成管状螺旋结构,在RNA的诱导下形成与真实病毒近似的富勒烯锥体结构。Zhao等[23, 28]通过CA蛋白的单点突变A204C,在体外高盐条件下得到与真实病毒类似的富勒烯的稳定结构,利用冷冻电镜首次报道了成熟病毒的富勒烯锥体结构,分辨率达到8.6 Å。结构表明病毒分子CTD的H9~H11三个螺旋通过疏水作用形成一个三体对称的界面,定点突变证实了这些位点 (I201、K203、A204、E213) 位于三体的疏水核心,该核心对病毒的稳定性和感染性产生重要的影响。在电镜数据的基础上,作者通过MDFF方法 (molecular dynamics flexible fitting) 进行动力学计算建模,在模型中多聚体中71个六聚体中的NTD结构域与单晶衍射数据中的NTD非常相近,不同六聚体中的NTD结构变化很小 [RMSD在 (1.5 ± 1.0) Å]。CA蛋白的CTD结构相对于NTD显现出较大的变化 [RMSD在 (3.0 ± 0.9) Å],说明CTD在病毒分子成熟过程中需要微调自身构象来完成富勒烯锥体结构的搭建。由此结构发现的三体对称中心界面也将成为潜在的药物靶点 (图 4)。
|
图 4 HIV成熟病毒的富勒烯结构. a: 衣壳蛋白多聚体模型; b: 衣壳蛋白CTD-CTD二体; c: 五聚体和六聚体间的作用界面 |
衣壳蛋白的结构、组装动力学过程和锥形衣壳的稳定性均会影响HIV-1病毒分子的自我复制和感染,抗HIV-1的治疗就可以通过影响这些因素来达到目的[37]。CTD基因的缺失、插入或定点突变引发病毒装配严重缺陷将导致病毒丧失感染能力; 此外NTD和CTD涉及的重要位点突变实验可以改变各种相互作用影响衣壳蛋白的组装和病毒的感染能力。例如NTD上H2的M39和A42进行甘氨酸突变实验会直接影响病毒感染性,同样类似的突变实验也证实了NTD-H7上A64、M68、V165、L211等疏水氨基酸位点的作用。这些实验结果均表明关键氨基酸的相互作用会直接影响衣壳蛋白组装,进而抑制病毒感染性。因此衣壳蛋白抑制剂的实质是一类以蛋白-蛋白相互作用为靶点,靶点具有蛋白-蛋白相互作用的特点: 作用的界面面积较大,没有明显已知的口袋或者结合位点等。也正是基于此原因,与酶等其他靶点相比,针对衣壳蛋白的小分子抑制剂研究进展相对缓慢。
早期研究发现,α-羟基甘氨酰胺 (α-hydroxyl glycineamide来自于GPG-NH2的代谢产物G-NH2后氧化得到),具有抗病毒活性[38, 39]。该小分子能够通过内质网相关降解 (endoplasmic reticulum-associated degradation,ERAD) 诱导gp160降解,同时也能减少Env形成HIV-1颗粒。而GPG-NH2、ALG-NH2三肽是最早发现通过作用于衣壳p24而抑制HIV活性的化合物[40]。
首个衣壳蛋白小分子抑制剂1 (CAP-1) 是针对 衣壳蛋白NTD的β-发卡裂口 (β-hairpin cleft) 界面进行虚拟筛选获得 (图 5)[41],该裂口是Gag蛋白水解后被β-发卡的氨基酸残基所占据的位置,通过核磁研究表明该化合物作用于NTD与CTD的界面附近(PDB ID: 2JPR),蛋白形成了一个3-氯-4-甲基苯基基团可以进入的口袋 (图 5 CAP-1方框内基团),化合物并不直接阻止NTD和CTD的相互作用,通过变构 (allosteric effect) 影响NTD与CTD的接触界面从而达到影响两者的相互作用[42]。其中F32产生移动形成了CAP-1所在的口袋,在全长的CA与I3- 的晶体结构中出现了同样类似的口袋[43]。这些研究表明I3- 和CAP-1在全长的CA中调节NTD-CTD取向实现稳定二聚体构象,而此二聚体构象并不利于六聚体的组装形成[43]。
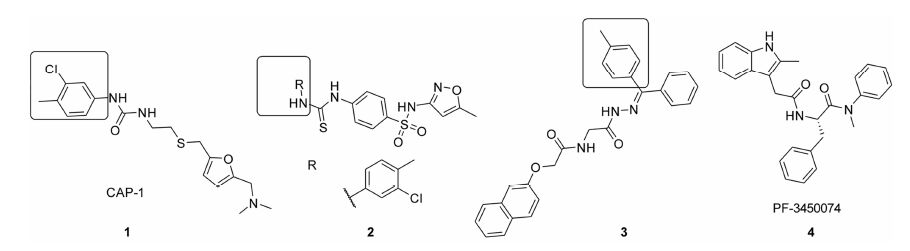
|
图 5 以CA-NTD为靶点的小分子抑制剂 |
CAP-1结合NTD的结合常数比较低 (800 µmol·L-1),Chen等[44]参考CAP-1的结构设计了硫脲系列化合物2,发现化合物对CA和亲环素cyclophilin (CypA) 具有双重的抑制活性。化合物抑制HIV-1具有微摩尔级别的活性,其中与CAP-1具有相同的3-氯- 4-甲基苯基端基的化合物EC50为0.6 µmol·L-1 [44, 45]。化合物同时表现为低毒性,在对未感染的CEM细胞中TC50大于100 mmol·L-1。此外,他们通过计算机进行分子对接和模拟筛选发现腙类化合物3与NTD蛋白具有良好尺寸和相互作用匹配,对分子简单优化后找到了对猿猴免疫缺损病毒 (simian immunodeficiency virus,SIV) 具有较高活性 (EC50 = 0.47 µmol·L-1) 和低毒性 (TC50 > 100 µmol·L-1) 的化合物,该化合物能够降低衣壳蛋白的组装速率[46]。图 5中所有化合物框内的基团被认为占据CAP-1的3-氯-4-甲基苯基口袋。
Pfizer公司通过高通量筛选化合物库得到PF-1385801化合物4能够抑制HIV复制,对其结 构类似物进行构效关系和机制研究表明,这类化 合物抗病毒的靶点为衣壳蛋白[12],化合物通过结合CA干扰病毒的脱壳和形成感染颗粒的过程。类似 物PF-3450074的EC50 = 0.69 µmol·L-1,而 CC50 = 69 µmol·L-1,治疗窗 (therapeutic index) 高达121。高分辨的晶体结构表明类似物PF-3450074小分子作用于NTD表面的口袋,该口袋也是已知宿主CPSF6蛋白 (cleavage and polyadenylation specific factor 6) 的作用位置[47, 48]。最近发现对PF74抗药性的“5Mut”病毒的抗药性来源于PF74分子结合位点附近的残基发生Q67H、K70R和T107N突变[49]。
Boehringer Ingelheim公司研究人员同样通过高通量筛选,发现苯二氮 类5 (BD) 和苯并咪唑类6 (BI) 化合物具有抑制衣壳组装活性从而影响病毒复制[50],研究人员进行结构优化时,尽量避免小分子同时具有逆转录酶抑制活性,发现了仅通过抑制衣壳组装机制产生抗病毒作用的化合物,其中BD化合物的EC50 = 70 nmol·L-1、TC50 = 28 µmol·L-1 (图 6)。他们还发现另一类完全不同结构的BI化合物,EC50 = 62 nmol·L-1和TC50 > 20 µmol·L-1 [51, 52, 53]。两类化合物与CA-NTD的复合物晶体结构都得到了解析[54],结构数据表明,这两类化合物都占据同一个口袋,也是CAP-1所占据的区域,两类化合物所结合NTD蛋 白的α-螺旋区域则有微弱的改变。该公司也报道了BI-1 (7) 和BI-2 (8) 这类化合物结合在CA-NTD的CPSF-6结合位点,证实化合物通过稳定聚集体这类新机制抑制病毒[55]。
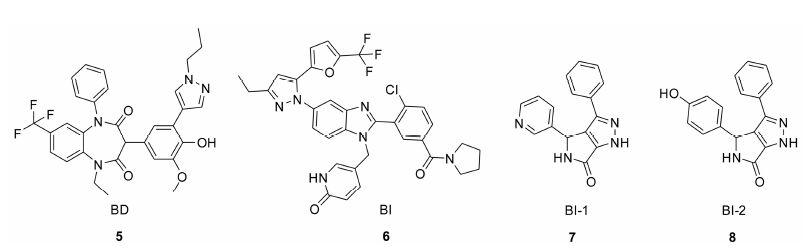
|
图 6 Boehringer Ingelheim公司通过高通量筛选得到的CA抑制剂小分子结构 |
一种12肽 (CAI) 被发现能破坏体外的非成熟和类似成熟衣壳 (immature and mature-like capsid) 的组装[56],CAI作用位点为CTD,阻碍NTD-CTD界面的形成从而破坏衣壳组装。通过比较CTD/CAI复合体结构 (PDB ID: 2buo)[57]和NTD-CTD界面的结构 (PDB ID: 3H4E) 发现,该CAI α-螺旋肽通过依附在CTD阻碍NTD与CTD结合,但CAI肽和NTD与CTD的结合方式和结合区域氨基酸残基性质有很大不同。CAI螺旋肽与CTD主要依靠残基占据疏水空腔,而NTD-H4与CTD相互作用主要依赖于极性残基的作用。
12肽 (CAI) 因不能穿透膜而无法抑制细胞内的HIV-1。根据CAI与衣壳蛋白的CTD复合物结构,通过“碳链装订”的方式改良CAI为可透膜的肽 (NYAD-1),在体外细胞实验中证明了NYAD-1可以破坏HIV病毒颗粒组装[58]。通过对NYAD-1的类似物NYAD-13与CTD在溶液中的核磁结构的解析,发现它们分子间作用主要是通过疏水作用,NYAD-13与疏水口袋的氨基酸残基F3/L8/L9/Y11发生相互作用。
Curreli等[59]针对CAI肽结合的CTD口袋,对ZINC化合物库中100 000类药的结构进行了计算机模拟分子对接研究,从筛选结果中选择8个化合物进行了测试,其中2个 (9和10) 具有抑制病毒感染活性 (图 7)。在MT-2细胞中对HIV-1 IIIB病毒抑制活性 (IC50) 分别为1.06和6.69 µmol·L-1,对这两类骨架进行了相关构效关系研究,未得到更高活性的小分子。通过基于结构的虚拟筛选方法,化合物11 (CK26) 被发现具有抗病毒活性[60],经过对类似物的活性研究,化合物12(I-XW-053) 的活性从164 µmol·L-1提高到化合物13 (CK176) 的14 µmol·L-1,目前只通过分子模拟实验给出此类化合物的结合模式[61]。
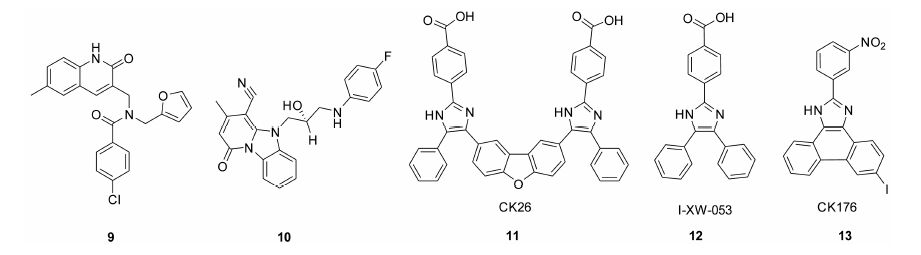
|
图 7 针对CA蛋白进行虚拟筛选得到的活性化合物 |
虽然PF0074和CAI都影响到CTD-NTD相互作用,但是它们分别作用在NTD表面和CTD表面。这些结果揭示阻碍CTD-NTD相互作用存在多种作用界面,可以设计抑制剂能同时结合CTD和NTD,从而破坏衣壳蛋白进行组装,这样抑制剂的效率得到提高,对HIV-1病毒感染的抑制活性可以产生正面的效果。NTD-NTD相互作用处于多聚体的最内环,CA NTD的H2和H3两个螺旋在NTD-NTD相互作用和CA六聚中发挥主要作用,CA NTD的H4和CTD的H8在NTD-CTD相互作用中发挥主要作用。合成具有这些序列的肽段在体外进行活性研究,发现H2、H3、H4均没有活性,H8具有微弱的抑制活性[62, 63]。
本课题组在衣壳蛋白六聚体模型中,通过综合比较分析结构,在一幅模型中构建了各类抑制剂在多聚体的相对位置 (图 8),可以直观展示目前几类抑制剂占据衣壳蛋白不同结合位置,比如CAI和PF74化合物在CTD/NTD界面附近,CAP-1则在该界面附近的发卡凹槽位置。在组装体形成中发挥重要作用的CTD/ CTD界面目前没有抑制剂报道,有待进一步探索。
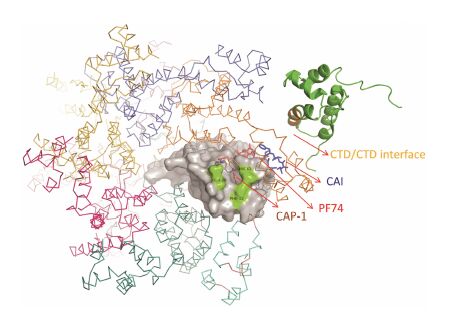
|
图 8 衣壳蛋白六聚体中,CAP-1、PF74 (PF-3450074) 以及CAI肽抑制剂结合位点比较,潜在结合位点CTD/CTD界面的位置 (综合蛋白聚集体,小分子结合结构构建而成)。一个衣 壳蛋白采用灰色表面表示,其余为简单线表示,一个相邻的CTD界面的衣壳蛋白采用卡通表示 |
衣壳蛋白组装聚集主要依赖于衣壳蛋白之间相互作用,这种典型的蛋白-蛋白相互作用与其他靶点相比没有明显的作用位点和口袋,作用比较弱,小分子和多肽均可能通过改变蛋白聚集来抑制病毒的活性。目前针对该靶点为数不多抑制剂的作用位点主要集中在衣壳蛋白两处,一个是NTD与CTD的铰链作用点,另一个是NTD-CTD的作用界面。衣壳蛋白组装体抑制剂的研究现状中值得进一步探索研究的方向分别为: ① 基于这两个位点开发出高效的小分子抑制剂; ② 是否能找到新的作用位点。
此外在对各类病毒衣壳蛋白组装动力学不断研究中发现,仅针对单个蛋白的结构研究抑制蛋白组装,则会忽略蛋白质多聚组装的动力学信息。目前简化的组装模型假设蛋白聚集体的平衡常数热力学行为相同,认为较弱二聚结合常数有利于组装体对错误的组装进行调整,具有容错和纠错的能力。由于HIV-1中衣壳蛋白组装所需数目多 (1 500个),组装过程具有一定容错机制,单纯从抑制衣壳蛋白本身出发进行药物分子开发,病毒自身能采取过表达衣壳蛋白或者依靠衣壳蛋白容错组装方式避免组装失败。从HBV (仅含240个衣壳蛋白单元) 的组装过程研究中发现,少数成核组装体在组装过程中所起作用远大于其他非成核组装体,直接作用于这些成核组装体可以提升消耗衣壳蛋白的水平从而减弱病毒的感染性[21, 64]。HBV已知的抑制剂杂芳环二氢嘧啶干扰组装过程的组装动力学,错误引导中间体或稳定组装的相互作用都有可能破坏组装的过程从而达到降低病毒的感染能力[64]。目前对HIV-1组装反应的动力学还知之甚少,虽然有证据表明无论是加速还是抑制衣壳蛋白组装的聚集,均能产生抑制病毒复制感染能力,但其中具体的动力学组装模型仍不清楚,有待进一步的探讨研究,所以发现新型功能小分子也有利于通过小分子探针进行研究组装反应的动力学过程,加深认识衣壳蛋白组装反应在时空上的历程,使衣壳蛋白药物靶点在治疗各类病毒中得到实际应用。
| [1] | Katlama C, Deeks SG, Autran B, et al. Barriers to a cure for HIV:new ways to target and eradicate HIV-1 reservoirs[J]. Lancet, 2013, 381:2109-2117. |
| [2] | Matthews T, Salgo M, Greenberg M, et al. Enfuvirtide:the first therapy to inhibit the entry of HIV-1 into host CD4 lymphocytes[J]. Nat Rev Drug Discov, 2004, 3:215-225. |
| [3] | De Clercq E. New approaches toward anti-HIV chemotherapy[J]. J Med Chem, 2005, 48:1297-1313. |
| [4] | Mehellou Y, De Clercq E. Twenty-six years of anti-HIV drug discovery:where do we stand and where do we go?[J]. J Med Chem, 2009, 53:521-538. |
| [5] | Lu XF, Chen ZW. The development of anti-HIV-1 drugs[J]. Acta Pharm Sin (药学学报), 2010, 45:165-176. |
| [6] | Zhang Y. The newest developments in anti-HIV-1 drugs[J]. Acta Pharm Sin (药学学报), 2010, 45:194-204. |
| [7] | Shi WG, Jia QY, Liu KL. The current progress in the development of HIV-1 fusion inhibitors[J]. Acta Pharm Sin (药学学报), 2010, 45:184-193. |
| [8] | Lai WH, Huang L, Chen CH. HIV entry inhibitors:progress in development and application[J]. Acta Pharm Sin (药学学报), 2010, 45:165-176. |
| [9] | Wesley I. Sundquist H-GK. HIV-1 assembly, budding, and maturation[J]. Cold Spring Harb Perspect Med, 2012, 2:a006924. |
| [10] | Zandi R, van der Schoot P, Reguera D, et al. Classical nucleation theory of virus capsids[J]. Biophys J, 2006, 90:1939-1948. |
| [11] | Ehrlich LS, Liu T, Scarlata S, et al. HIV-1 capsid protein forms spherical (immature-like) and tubular (mature-like) particles in vitro:structure switching by pH-induced conformational changes[J]. Biophys J, 2001, 81:586-594. |
| [12] | Blair WS, Pickford C, Irving SL, et al. HIV capsid is a tractable target for small molecule therapeutic intervention[J]. PLoS Pathog, 2010, 6:e1001220. |
| [13] | Briggs JAG, Simon MN, Gross I, et al. The stoichiometry of Gag protein in HIV-1[J]. Nat Struct Mol Biol, 2004, 11:672-675. |
| [14] | Ganser BK, Li S, Klishko VY, et al. Assembly and analysis of conical models for the HIV-1 core[J]. Science, 1999, 283:80-83. |
| [15] | Briggs JAG, Wilk T, Welker R, et al. Structural organization of authentic, mature HIV-1 virions and cores[J]. EMBO J, 2003, 22:1707-1715. |
| [16] | Nguyen A, Feasley C, Jackson K, et al. The prototype HIV-1 maturation inhibitor, bevirimat, binds to the CA-SP1 cleavage site in immature Gag particles[J]. Retrovirology, 2011, 8:101. |
| [17] | Martin DE, Salzwedel K, Allaway GP. Bevirimat:a novel maturation inhibitor for the treatment of HIV-1 infection[J]. Antivir Chem Chemother, 2008, 19:107-113. |
| [18] | Smith PF, Ogundele A, Forrest A, et al. Phase I and II study of the safety, virologic effect, and pharmacokinetics/pharmacodynamics of single-dose 3-O-(3',3'-dimethylsuccinyl) betulinic acid (bevirimat) against human immunodeficiency virus infection[J]. Antimicrob Agents Chemother, 2007, 51:3574-3581. |
| [19] | Jiang Y, Liu X. The role of structural protein Gag and related gene (protein) in late stages of the HIV-1 replication cycle and the inhibitors[J]. Acta Pharm Sin (药学学报), 2010, 45:205-214. |
| [20] | Perni RB, Conway SC, Ladner SK, et al. Phenylpropenamide derivatives as inhibitors of hepatitis B virus replication[J]. Bioorg Med Chem Lett, 2000, 10:2687-2690. |
| [21] | Katen SP, Chirapu SR, Finn MG, et al. Trapping of hepatitis B virus capsid assembly intermediates by phenylpropenamide assembly accelerators[J]. ACS Chem Biol, 2010, 5:1125- 1136. |
| [22] | Bharat TAM, Castillo Menendez LR, Hagen WJH, et al. Cryo-electron microscopy of tubular arrays of HIV-1 Gag resolves structures essential for immature virus assembly[J]. Proc Natl Acad Sci USA, 2014, 111:8233-8238. |
| [23] | Byeon IJ, Meng X, Jung J, et al. Structural convergence between cryo-EM and NMR reveals intersubunit interactions critical for HIV-1 capsid function[J]. Cell, 2009, 139:780- 790. |
| [24] | Ganser-Pornillos BK, Cheng A, Yeager M. Structure of fulllength HIV-1 CA:a model for the mature capsid lattice[J]. Cell, 2007, 131:70-79. |
| [25] | Pornillos O, Ganser-Pornillos BK, Kelly BN, et al. X-Ray structures of the hexameric building block of the HIV capsid[J]. Cell, 2009, 137:1282-1292. |
| [26] | Pornillos O, Ganser-Pornillos BK, Yeager M. Atomic-level modelling of the HIV capsid[J]. Nature, 2011, 469:424-427. |
| [27] | Schur FKM, Hagen WJH, Rumlova M, et al. Structure of the immature HIV-1 capsid in intact virus particles at 8.8? resolution[J]. Nature, 2015, 517:505-508. |
| [28] | Zhao G, Perilla JR, Yufenyuy EL, et al. Mature HIV-1 capsid structure by cryo-electron microscopy and all-atom molecular dynamics[J]. Nature, 2013, 497:643-646. |
| [29] | Ivanov D, Tsodikov OV, Kasanov J, et al. Domain-swapped dimerization of the HIV-1 capsid C-terminal domain[J]. Proc Natl Acad Sci USA, 2007, 104:4353-4358. |
| [30] | Alan Engelman PC. The structure biology of HIV-1:mechanistic and therapeutic insights[J]. Nat Rev Microbiol, 2012, 10:279-290. |
| [31] | Gross I, Hohenberg H, Wilk T, et al. A conformational switch controlling HIV-1 morphogenesis[J]. EMBO J, 2000, 19:103-113. |
| [32] | Byeon IJ, Hou G, Han Y, et al. Motions on the millisecond time scale and multiple conformations of HIV-1 capsid protein:implications for structural polymorphism of CA assemblies[J]. J Am Chem Soc, 2012, 134:6455-6466. |
| [33] | Lanman JSJ, Sakalian M, Prevelige PE Jr. Kinetic analysis of the role of intersubunit interactions in human immunodeficiency virus type 1 capsid protein assembly in vitro[J]. J Virol, 2002, 76:6900-6908. |
| [34] | Deshmukh L, Schwieters CD, Grishaev A, et al. Structure and dynamics of full-length HIV-1 capsid protein in solution[J]. J Am Chem Soc, 2013, 135:16133-16147. |
| [35] | Ganser-Pornillos BK, Yeager M, Sundquist WI. The structural biology of HIV assembly[J]. Curr Opin Struct Biol, 2008, 18:203-217. |
| [36] | Jouvenet N, Simon SM, Bieniasz PD. Visualizing HIV-1 Assembly[J]. J Mol Biol, 2011, 410:501-511. |
| [37] | Liu D, Zhan P, Liu X. Recent progress in the development of HIV-1 capsid protein inhibitors[J]. Chin J Med Chem (中国药物化学杂志), 2011, 21:397-404. |
| [38] | Abdurahman S, Vegvari A, Levi M, et al. Isolation and characterization of a small antiretroviral molecule affecting HIV-1 capsid morphology[J]. Retrovirology, 2009, 6:34. |
| [39] | Jejcic A, Hoglund S, Vahlne A. GPG-NH2 acts via the metabolite alpha HGA to target HIV-1 Env to the ER-associated protein degradation pathway[J]. Retrovirology, 2010, 7:20. |
| [40] | Höglund S, Su J, Reneby SS, et al. Tripeptide interference with human immunodeficiency virus type 1 morphogenesis[J]. Antimicrob Agents Chemother, 2002, 46:3597-3605. |
| [41] | Tang C, Loeliger E, Kinde I, et al. Antiviral inhibition of the HIV-1 capsid protein[J]. J Mol Biol, 2003, 327:1013-1020. |
| [42] | Kelly BN, Kyere S, Kinde I, et al. Structure of the antiviral assembly inhibitor CAP-1 complex with the HIV-1 CA protein[J]. J Mol Biol, 2007, 373:355-366. |
| [43] | Du S, Betts L, Yang R, et al. Structure of the HIV-1 full-length capsid protein in a conformationally trapped unassembled state induced by small-molecule binding[J]. J Mol Biol, 2011, 406:371-386. |
| [44] | Chen K, Tan Z, He M, et al. Structure-activity relationships (SAR) research of thiourea derivatives as dual inhibitors targeting both HIV-1 capsid and human cyclophilin A[J]. Chem Biol Drug Des, 2010, 76:25-33. |
| [45] | Li J, Tan Z, Tang S, et al. Discovery of dual inhibitors targeting both HIV-1 capsid and human cyclophilin A to inhibit the assembly and uncoating of the viral capsid[J]. Bioorg Med Chem, 2009, 17:3177-3188. |
| [46] | Tian B, He M, Tan Z, et al. Synthesis and antiviral evaluation of new N-acylhydrazones containing glycine residue[J]. Chem Biol Drug Des, 2011, 77:189-198. |
| [47] | Bhattacharya A, Alam SL, Fricke T, et al. Structural basis of HIV-1 capsid recognition by PF74 and CPSF6[J]. Proc Natl Acad Sci USA, 2014, 111:18625-18630. |
| [48] | Price AJ, Jacques DA, McEwan WA, et al. Host cofactors and pharmacologic ligands share an essential interface in HIV-1 capsid that is lost upon disassembly[J]. PLoS Pathog, 2014, 10:e1004459. |
| [49] | Shi J, Zhou J, Halambage UD, et al. Compensatory substitutions in the HIV-1 capsid reduce the fitness cost associated with resistance to a capsid-targeting small-molecule inhibitor[J]. J Virol, 2015, 89:208-219. |
| [50] | Fader LD, Bethell R, Bonneau P, et al. Discovery of a 1,5-dihydrobenzo[b][1,4] diazepine-2,4-dione series of inhibitors of HIV-1 capsid assembly[J]. Bioorg Med Chem Lett, 2011, 21:398-404. |
| [51] | Yoakim C, Deroy P, Duplessis M, et al. Inhibitors of HIV Replication:WO, 2008067644[P]. 2008-6-12. |
| [52] | Fader LD, Landry S, Goulet S, et al. Optimization of a 1,5-dihydrobenzo[b][1,4] diazepine-2,4-dione series of HIV capsid assembly inhibitors 2:structure-activity relationships (SAR) of the C3-phenyl moiety[J]. Bioorg Med Chem Lett, 2013, 23:3401-3405. |
| [53] | Fader LD, Landry S, Morin S, et al. Optimization of a 1,5-dihydrobenzo[b][1,4] diazepine-2,4-dione series of HIV capsid assembly inhibitors 1:addressing configurational instability through scaffold modification[J]. Bioorg Med Chem Lett, 2013, 23:3396-3400. |
| [54] | Titolo S, Mercier JF, Wardrop E, et al. Discovery of Potent HIV-1 Capsid Assembly Inhibitors[C]. San Francisco:17th Conference on Retroviruses and Opportunistic Infections, 2010. |
| [55] | Lamorte L, Titolo S, Lemke CT, et al. Discovery of novel small-molecule HIV-1 replication inhibitors that stabilize capsid complexes[J]. Antimicrob Agents Chemother, 2013, 57:4622-4631. |
| [56] | Sticht J, Humbert M, Findlow S, et al. A peptide inhibitor of HIV-1 assembly in vitro[J]. Nat Struct Mol Biol, 2005, 12:671-677. |
| [57] | Ternois F, Sticht J, Duquerroy S, et al. The HIV-1 capsid protein C-terminal domain in complex with a virus assembly inhibitor[J]. Nat Struct Mol Biol, 2005, 12:678-682. |
| [58] | Zhang H, Zhao Q, Bhattacharya S, et al. A cell-penetrating helical peptide as a potential HIV-1 inhibitor[J]. J Mol Biol, 2008, 378:565-580. |
| [59] | Curreli F, Zhang H, Zhang X, et al. Virtual screening based identification of novel small-molecule inhibitors targeted to the HIV-1 capsid[J]. Bioorg Med Chem, 2011, 19:77-90. |
| [60] | Kortagere S, Madani N, Mankowski MK, et al. Inhibiting early-stage events in HIV-1 replication by small-molecule targeting of the HIV-1 capsid[J]. J Virol, 2012, 86:8472- 8481. |
| [61] | Kortagere S, Xu JP, Mankowski MK, et al. Structure-activity relationships of a novel capsid targeted inhibitor of HIV-1 replication[J]. J Chem Inf Model, 2014, 54:3080-3090. |
| [62] | Goudreau N, Lemke CT, Faucher AM, et al. Novel inhibitor binding site discovery on HIV-1 capsid N-terminal domain by NMR and X-ray crystallography[J]. ACS Chem Biol, 2013, 8:1074-1082. |
| [63] | Bocanegra R, Nevot M, Doménech R, et al. Rationally designed interfacial peptides are efficient in vitro inhibitors of HIV-1 capsid assembly with antiviral activity[J]. PLoS One, 2011, 6:e23877. |
| [64] | Zlotnick A, Mukhopadhyay S. Virus assembly, allostery and antivirals[J]. Trends Microbiol, 2011, 19:14-23. |
 2015, Vol. 50
2015, Vol. 50


