石斛属 (Dendrobium) 为兰科 (Orchidaceae) 第二大属。在我国, 石斛属植物共有74种和2个变种, 主要分布在华南及西南地区[1]。石斛是一种珍稀名贵中草药, 有生津、止咳、润喉等功效。丁小余等[2]通过对浙江枫斗加工地的实地考察和查证, 发现几乎所有具有柔软茎的石斛均可加工为枫斗。药材的判 断仅凭传统经验, 往往让不法商贩有机可乘。因此, 寻找一种快速、有效的鉴别枫斗类石斛的方法十分必要, 并可作为规范枫斗市场的有力科学依据。
以往对于枫斗类石斛的鉴定主要集中在对其显微结构、生药成分的理化鉴别方面。白音等[3]通过对枫斗类石斛茎材料的角质层厚度、表皮细胞切向直 径和径向直径等17个具有鉴别意义的显微特征研究, 建立了枫斗类石斛的综合鉴定方法, 但其不能快速进行鉴别, 同时枫斗类石斛作药用时通常会加工成石斛粉, 使显微鉴定无法进行。随着现代分子生物学DNA测序技术的迅速发展, 利用DNA barcoding技术对中药材植物的鉴定研究越来越多,DNA barcoding候选基因nrDNA ITS、matK、rbcL以及psbA-trnH等已在人参 (Panax ginseng C. A. Mey)、秦艽 (Gentiana macrophylla Pall)、金银花 (Lonicera japonica Thunb) 等中药材基原植物的鉴定中得到广泛应用[4, 5, 6, 7]。在对石斛属植物的研究方面: 丁小余等[2]建立了21种枫斗类石斛的nrDNA ITS区全序列数据库; 刘静等[8]对12种药用石斛的叶绿体matK基因序列特征及物种 鉴别进行了研究; Yao等[9]发现可以利用psbA-trnH序列对17种石斛及1种混伪品进行鉴定。然而Xiang等[10]在对石斛属植物的DNA条形码序列研究中发现, 单一序列并不能完全鉴定出不同种类的石斛属植物, 同时, 也有研究表明单一片段无法鉴别霍山石斛及细茎石斛[11]。目前, 在利用多片段联合的方法对不同植物进行鉴定的研究过程中发现, 结合叶绿体和核DNA序列可有效提高物种间的分子鉴别率[12, 13, 14]。因此, 可对石斛属植物进行多片段联合鉴定研究, 以达到更好的鉴定目的。
有研究发现, 线粒体基因序列更适于区分亲缘关系较近的物种[15, 16], 而Xiang等[10]利用nrDNA ITS序列及叶绿体基因片段进行研究发现, 在石斛属内存在多个复合种, 如: 细茎石斛 (D. moniliforme (Linn.) Sw) 与霍山石斛 (D. huoshanense C. Z. Tang et S. J. Cheng), 而且nrDNA ITS序列及叶绿体基因片段并不能很好地区分这些物种。目前关于石斛属植物线粒体基因片段的研究还较少, 仅有Zhang等[17]将NADH脱氢酶亚基1编码基因 (nad 1) 内含子2 (intron 2) 序列应用于石斛属植物的鉴定, 其研究结果表明该单一序列并不能直接对枫斗类石斛进行有效鉴别。线粒体nad 5序列多应用于蕨类、藻类植物的物种鉴定, 而在石斛属植物中罕见报道[18, 19]。因此, 本文从线粒体基因片段入手, 利用PCR技术扩增出17种枫斗类石斛39个个体的线粒体基因co I、nad 5部分序列 及NADH脱氢酶亚基1编码基因 (nad 1) 内含子2 (intron 2) 序列、nrDNA ITS序列及叶绿体基因序列rbcL、matK、psbA-trnH, 同时筛选出适合的基因片段来研究多片段联合在枫斗类石斛植物鉴定方面的应用。
材料与方法 材料本实验所用材料主要采自云南、广西、贵州及安徽等地 (表 1), 待检种1、2、3采自安徽霍山。上述实验材料由丁小余教授鉴定, 鲜植株保存于南京师范大学生命科学学院植物资源与环境研究所。
|
|
Table 1 Seventeen species from Dendrobium species of Fengdous used in this study |
取硅胶干燥的材料叶片, 用75% 乙醇擦拭叶片表面后, 用CTAB法[20]提取叶片总DNA。利用紫外吸光法测定所提取的DNA模板质量, 紫外吸光值260/280在1.8~2.0之间,260/230大于1.7, 将模板浓度稀释至30 ng·μL-1, -20 ℃保存。
引物设计根据GenBank公共数据库中可用线粒体基因数据, 对被子植物nad 1外显子部分、nad 5及co I基因序列进行比对, 确定保守区, 利用Primer Premier 5.0软件设计引物, 并用Oligo 6.0软件进行评估。nrDNA ITS序列及叶绿体基因片段psbA-trnH、matK和rbcL则采用通用引物扩增。具体引物见表 2。
|
|
Table 2 Fragment sequence used in our research to get target gene |
PCR反应条件: 10 × PCR Buffer 3 μL、dNTP (10 mmol·L-1) 2.1 μL、Mg2+ (25 mmol·L-1) 2.1 μL、引物 (2 μmol·L-1) 各3 μL、模板DNA (25 ng·μL-1) 11 μL、TaqDNA聚合酶 (5 U·μL-1,TaKaRa) 0.5 μL、加灭菌双蒸水至反应体系为30 μL。全部序列的PCR反应程序: 94 ℃预变性5 min; 94 ℃变性1 min,50~60 ℃退火1 min,72 ℃延伸2 min, 共30个循环后72 ℃延伸10 min, 最后 4 ℃保存。目的片段切胶回收, 经纯化后送华大基因公司进行序列测定。
序列分析 将测序结果用Vector NTI Advance 10.0进行拼接, 提交至NCBI (http://www.ncbi.nlm. nih.gov/), 获得序列号, 见表 3。将整理后的序列利 用MEGA 5.0软件分析[21], 计算各样品DNA序列 之间的差异百分比、简约信息位点及单一多态位点。根据K-2-P (kimura-2-parameter) 模型, 生成距离矩阵, 分析各枫斗类石斛基原植物的种间变异, 同时 利用p-distance对DNA序列进行筛选。使用邻接法 (neighbor-joining) 在MEGA 5.0软件中构建分子系统树, 系统树各分支的置信度用自举检验法 (bootstrap test) 检验, 共进行1 000次循环, 以评价各分支的系统学意义与可靠性。
|
|
Table 3 Plant materials used in this study. The samples of each species have the same GenBank No. |
对17种枫斗类石斛39个个体的7个序列特征 分析结果如表 4所示。由表 4可知,nrDNA ITS变异位点所占百分率最高, 为59.6%, 其次为线粒体基因nad 1-intron 2和叶绿体基因psbA-trnH, 为10.5% 和8.0%。非编码区序列的变异位点远高于基因编码区。
|
|
Table 4 The feature of seven sequences from Dendrobium species of Fengdous used in this study |
2 基于K-2-P模型对枫斗类石斛基原植物的种间变异分析
利用K-2-P模型对17种枫斗类石斛的39个个体的7个片段进行种间变异程度分析。由于种内材料较少, 因此本研究主要针对枫斗类石斛基原植物的种间变异程度进行分析。基于K-2-P模型计算的种间变异值如表 5所示,17种枫斗类石斛基原植物nrDNA ITS序列的平均种间距离最高, 为0.382, 其次为线粒体片段nad 1-intron 2和叶绿体片段psbA-trnH, 分别为0.228和0.046; 非编码区序列的种间变异远高于基因编码区。
|
|
Table 5 Results of interspecific genetic distance analysis for seven candidate DNA barcoding regions |
通过计算17种枫斗类石斛的39个个体的7个片段的p-distance来筛选合适的DNA序列。如表 6所示, 本实验所获得的7个DNA条形码候选序列的长度差异较大, 其中来自于核基因组的nrDNA ITS序列的p-distance最高, 为0.248, 位于线粒体基因组的nad 1-intron 2为0.173, 高于叶绿体片段psbA-trnH,0.043。从表中可以发现,nrDNA ITS和nad 1-intron 2这2个片段的组合比nrDNA ITS和psbA-trnH片段的组合更适合用作枫斗类石斛的DNA条形码, 对其进行分子鉴定。
|
|
Table 6 p-Distance and sequence length of seven candidate DNA barcoding regions |
对本实验室测序获得及NCBI下载的10条细茎石斛、17条霍山石斛的nrDNA ITS序列进行比对后, 共有572个碱基位点, 其中变异位点28个 (表 7)。根据变异位点分析, 霍山石斛共发现2种单倍型: 其中有16条序列为H1型,1条序列为H2型。细茎石斛共发现7种单倍型: 其中4条序列为M1型; M2~M7型各有1条序列。比对发现大多数霍山石斛与细茎石斛共享同一种单倍型: H1、M1, 同时除M3、M6这两种单倍型外, 其他单倍型也不能完全将霍山石斛与细茎石斛区分, 因此有必要采用其他有效方法对细茎石斛与霍山石斛进行区分。
|
|
Table 7 nrDNA ITS sequences variable sites of D. huoshanense and D. moniliforme.*: The same nucleotide in the same position; H: D. huoshanense; M: D. moniliforme. H1: (JN388567.1; KC205197.1; KF143476.1; KC331003.1-KC331012.1; KP264993-KP264995); H2: (EU840696.1); M1: (GU339111.1; HQ114246.1; KP264991-KP264992); M2: (EU003114.1); M3: (AF401489.1); M4 (KC205189.1); M5 (KC346891.1); M6: (AF359254.1); M7: ( AF311777.1) |
利用MEGA 5.0软件分别对nrDNA ITS、psbA- trnH及nrDNA ITS+psbA-trnH联合片段构建UPGMA树进行分析 (图 1)。UPGMA树分析发现在石斛属植物中, 相同的物种聚为一支, 如霍山石斛和铁皮石斛。然而, 单一片段psbA-trnH不能对枫斗类石斛进行 很好的鉴别, 同时, 单一的nrDNA ITS序列或nrDNA ITS+psbA-trnH联合片段构建的UPGMA树中, 霍山石斛与细茎石斛聚为一支, 不能区分, 但利用nrDNA ITS+nad 1-intron 2序列构建的UPGMA树, 可以很 好地区分霍山石斛与细茎石斛, 并能有效地对其进行鉴定。结果表明: 利用nrDNA ITS片段与线粒体 片段nad 1-intron 2的组合优于nrDNA ITS和叶绿体片段psbA-trnH的组合,nrDNA ITS+nad 1-intron 2联合构建UPGMA树可以有效对枫斗类石斛进行分子鉴定。
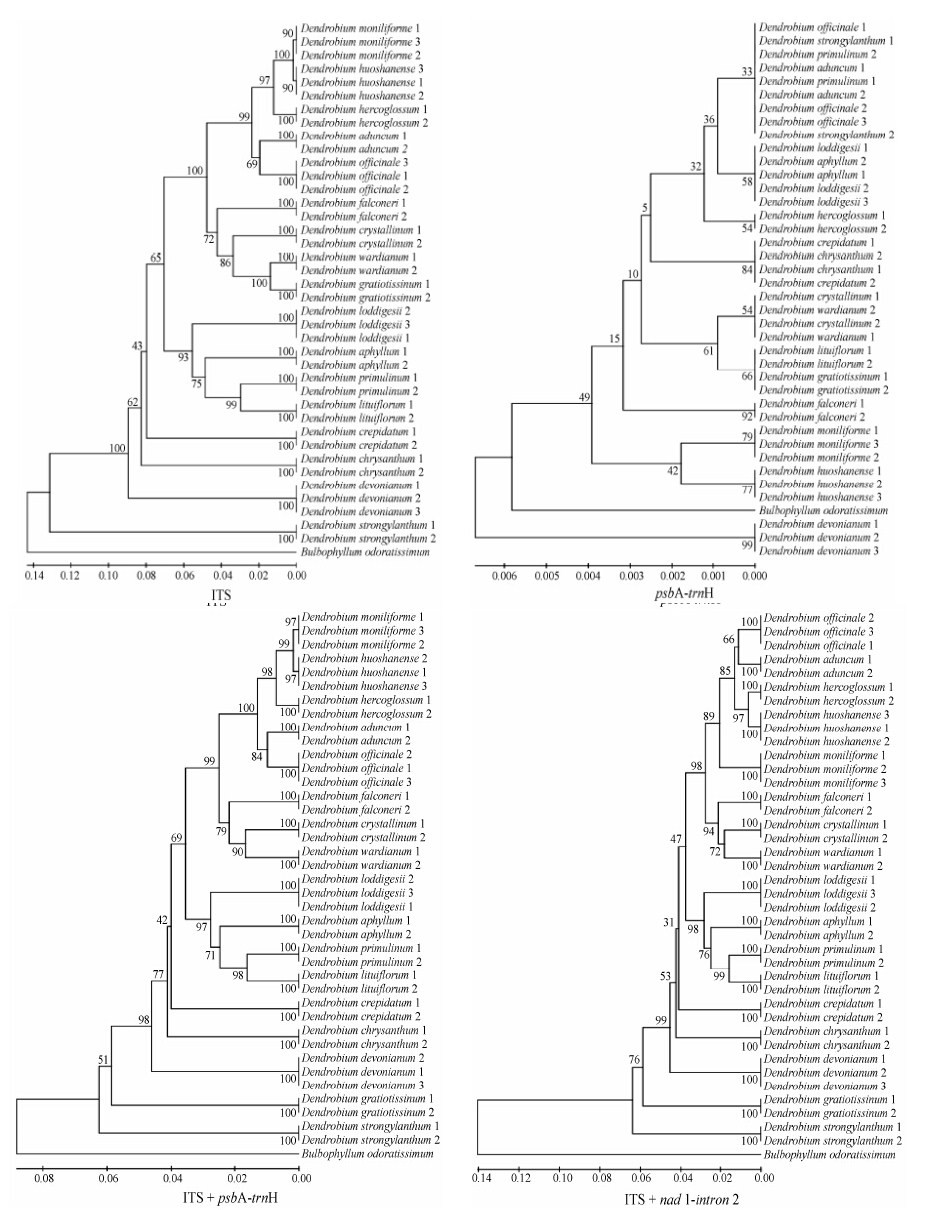 | Figure 1 Phylogenetic tree of Dendroubium species using UPGMA method |
本研究利用建树法, 基于nrDNA ITS+nad 1-intron 2联合片段对采自霍山的3种野生石斛成功地进行了鉴别。结果如图 2所示, 其中待检种1与霍山石斛聚为一支, 待检种2与细茎石斛聚为一支, 待检种3与铁皮石斛聚为一支, 支持率均为100%。这些材料在原植物开花后得到了验证, 即分别为霍山石斛、细茎石斛和铁皮石斛。
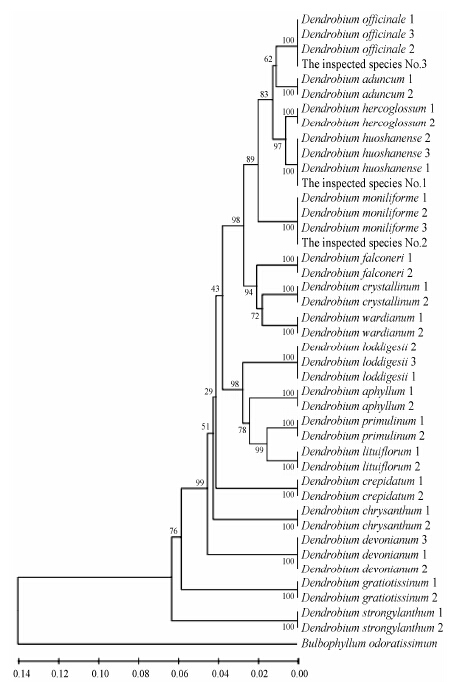 | Figure 2 Phylogenetic tree of Dendroubium species using UPGMA method |
DNA barcoding由Paul Hebert于2003年提出, 即利用一段短的、标准的DNA片段 (400~800 bp) 对物种进行快速、准确的鉴定[22]。自从DNA barcoding提出后便成为了近年来国际生物多样性研究的一个热点, 它对新物种的发现和珍稀濒危物种的保护具有重要的意义。位于核基因组的rDNA ITS区进化速率快, 因而成为植物条形码鉴别的重要候选基因片段, 近年来的研究表明,nrDNA ITS序列在石斛属物种间具有较大的种间变异和较小的种内变异, 因而成为石斛属物种鉴定的有效DNA分子标记[2, 23]。
诸多研究结果表明, 单一个片段不可能对所有的植物物种进行准确鉴定, 许多研究者又相继提出了不同的片段组合方案[9, 14, 24]。国际条形码协会植物工作组在2009年对来自55个物种907个样品的7个序列进行了比较分析, 认为rbcL+matK的组合可以作为植物通用条形码[14]; Chen等[24]研究认为psbA- trnH可以作为nrDNA ITS的互补序列对药用植物进行鉴定; 邵世光等[25]研究发现,psbA-trnH作为叶绿体中进化速率最快的间隔区之一, 可以用来鉴定石斛属物种及其混伪品。在本研究中, 通过对霍山石斛和细茎石斛的nrDNA ITS序列比对后, 发现单一片段无法将其区分开来, 然而nrDNA ITS序列仍具有较高变异位点率, 高达59.6%; 在叶绿体不同片段中,psbA-trnH片段的变异位点率最高, 约占8.0%, 之后依次为matK、rbcL, 分别为6.7% 和2.5%。
线粒体基因序列因具有较好的位点多样性水平, 在动物中有着广泛的应用, 很多序列片段不仅可以应用于谱系地理、系统发育方面的研究, 还可以筛选为DNA条形码序列, 用于物种鉴定研究[26, 27, 28]。一段648 bp的co I基因片段可以适用于多个动物类群鉴 定, 已经成为动物鉴定的基础片段, 然而该基因在植物中非常保守, 并不适合植物的物种鉴定[29]。因此, 筛选出适合的线粒体标记, 可以为植物物种鉴定提供一种新的思路。本文通过对17种枫斗类石斛39份石斛样品的不同区域进行对比研究发现, 线粒体基因nad 1-intron 2变异位点所占百分率远高于叶绿体基因psbA-trnH, 高达10.5%, 而在线粒体基因nad 5和co I片段中,变异位点仅分别有4个和7个。
目前,DNA条形码鉴定分析的方法主要有3种, 即相似性搜索法、距离法和建树法。因nrDNA ITS序列不能对所有枫斗类石斛属植物进行有效鉴别, 因此利用联合片段建树法则是较好的鉴别方法。建树法是应用MEGA或PAUP等生物软件构建NJ、UPGMA、MP等系统发育树, 确定与未知样品的DNA条形码序列聚类在一起的物种, 从而达到鉴定物种的目的。只有当DNA序列的变异率合适的时候才能较好的利用建树法来进行物种鉴定, 目前评价序列变异率的方法主要有两种,PIC[30]和p-distance[31], 其中p-distance方法较为简便、快速。本研究通过计算比较7个DNA序列的p-distance值发现,nrDNA ITS最高, 其次为nad 1-intron 2和psbA-trnH。
在对17种枫斗类石斛构建UPGMA树鉴定的 过程中发现, 利用单一片段或是结合nrDNA ITS和psbA-trnH构建UPGMA树时, 不能对枫斗类石斛中的霍山石斛及细茎石斛进行有效鉴定, 通过对目前已获得的霍山石斛和细茎石斛的nrDNA ITS序列比对发现, 大多数霍山石斛与细茎石斛共享同一种单倍型。而加入了nad 1-intron 2序列片段后可以提供足够的变异位点, 从而使霍山石斛及细茎石斛得以区分。因此, 新型联合片段: nrDNA ITS+nad 1-intron 2的组合明显优于nrDNA ITS和叶绿体片段psbA-trnH的组合, 其构建的UPGMA树则能区分霍山石斛和细茎石斛, 从而对所有枫斗类石斛基原植物进行鉴定。
石斛属植物作为名贵的中药材, 种间鉴定方面的研究仍有待完善。筛选出有效的石斛属DNA条形码序列可以为其物种鉴定及规范石斛市场提供理论依据。本研究首次对线粒体编码及非编码序列进行筛选, 并利用新型联合片段: nrDNA ITS+nad 1-intron 2构建UPGMA树对17种枫斗类石斛进行鉴定, 为石斛属枫斗类药材的鉴定提供了一种新的思路。
| [1] | Lavarack PS, Wayne F, Geoff S, et al. Dendrobium and Its Relatives [M]. Portland: Timber Press, 2002. |
| [2] | Ding XY, Wang ZT, Xu H, et al. Database establishment of the whole rDNA ITS region of Dendrobium species of “Fengdou” and authentication by analysis of their sequences [J]. Acta Pharm Sin (药学学报), 2002, 37: 567-573. |
| [3] | Bai Y, Bao YH, Wang WQ, et al. Characters and its authentication value of stem epidermal cell of medicinal plant in Dendrobium Sw. and their adulterants [J]. Chin Tradit Herb Drugs (中草药), 2011, 42: 593-597. |
| [4] | Hajibabaei M, Singer GA, Hebert PD, et al. DNA barcoding: how it complements taxonomy, molecular phylogenetics and population genetics [J]. Trends Genet, 2007, 23: 167-172. |
| [5] | Zuo Y, Chen Z, Kondo K, et al. DNA barcoding of Panax species [J]. Planta Med, 2011, 77: 182-187. |
| [6] | Luo K, Ma P, Yao H, et al. Identification of Gentianae Macrophyllae Radix using the ITS2 barcodes [J]. Acta Pharm Sin (药学学报), 2012, 47: 1710-1717. |
| [7] | Sun Z, Gao T, Yao H, et al. Identification of Lonicera japonica and its related species using the DNA barcoding method [J]. Planta Med, 2011, 77: 301-306. |
| [8] | Liu J, He T, Chun Z. Analysis and authentication of chloroplast matK gene sequences of Herba dendrobii [J]. Acta Pharm Sin (药学学报), 2009, 44: 1051-1055. |
| [9] | Yao H, Song JY, Ma XY, et al. Identification of Dendrobium species by a candidate DNA barcode sequence: the chloroplast psbA-trnH intergenic region [J]. Planta Med, 2009, 75: 667- 669. |
| [10] | Xiang XG, Schuiteman A, Li DZ, et al. Molecular systematics of Dendrobium (Orchidaceae, Dendrobieae) from mainland Asia based on plastid and nuclear sequences [J]. Mol Phylogenet Evol, 2013, 69: 950-960. |
| [11] | Li GL, Zhang JX, Zeng SJ, et al. Systematic position of Dendrobium huoshanense inferred from ITS, Nad intron 2 and psbA-trnH DNA sequences [J]. Guangdong Agric Sci (广东农业科学), 2013, 13: 145-148. |
| [12] | Chase MW, Cowan RS, Hollingsworth PM, et al. A proposal for a standardised protocol to barcode all land plants [J]. Taxon, 2007, 56: 295-299. |
| [13] | Lahaye R, Bank M, Bogarin D, et al. DNA barcoding the floras of biodiversity hotspots [J]. Proc Natl Acad Sci USA, 2008, 105: 2923-2928. |
| [14] | CBOL Plant Working Group. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 2009, 106: 12794-12797. |
| [15] | Zhang TW, Zhang XW, Hu SN, et al. An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform [J]. Plant Methods, 2011, 7: 38-45. |
| [16] | Liu Y, Forrest LL, Bainard JD, et al. Organellar genome, nuclear ribosomal DNA repeat unit, and microsatellites isolated from a small-scale of 454 GS FLX sequencing on two mosses [J]. Mol Phylogenet Evol, 2013, 66: 1089-1094. |
| [17] | Zhang T, Wang ZT, XU LS, et al. Application of mitochondrial nad 1-intron 2 sequences to molecular identification of some species of Dendrobium Sw. [J]. Chin Tradit Herb Drugs (中草药), 2005, 36: 1059-1062. |
| [18] | Varigerow S, Teerkorn T, Knoop V. Phylogenetic information in the mitochondrial nad 5 gene of Pteridophytes: RNA editing and intron sequences [J]. Plant Biol, 1999, 1: 235-243. |
| [19] | Snyder SW, Karol KG, Delwiche CF. Phylogeny of Klebsormidium (Kelbsormidiales; Charophyta) based on rbcL, atpB, coxIII and nad5 sequence data [J]. J Phycol, 2003, 38: 330- 331. |
| [20] | Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochem Bull, 1987, 19: 11-15. |
| [21] | Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Mol Biol Evol, 2011, 28: 2731-2739. |
| [22] | Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes [J]. Proc Biol Sci, 2003, B 270: 313-321. |
| [23] | Xu H, Li XB, Ding XY, et al. rDNA ITS sequencing Herba Dendrobium (Huang Cao) [J]. Acta Pharm Sin (药学学报), 2001, 36: 777-783. |
| [24] | Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species [J]. PLoS One, 2010, 5: e8613. |
| [25] | Shao SG, Han L, Ma YH, et al. Analysis and authentication of cpDNA psbA-trnH regions of Dendrobium species of Fengdous [J]. Acta Pharm Sin (药学学报), 2009, 44: 1173-1178. |
| [26] | Brykov A, Polyakova N, Skurikhina LA, et al. Geographical and temporal mitochondrial DNA variability in populations of Pink salmon [J]. J Fish Biol, 1996, 48: 899-909. |
| [27] | Hu J, Wang DQ, Wei QW, et al. Molecular identification of Acipenser schrenckii, Huso dauricus and their hybrid offspring [J]. J Fish Sci China (中国水产科学), 2010, 7: 22-31. |
| [28] | Gao YS, Tang XJ, Tu YJ, et al. Studies on the DNA barcoding of fifteen chicken breeds by mtDNA COI gene [J]. Sci Agric Sin (中国农业科学), 2011, 44: 587-594. |
| [29] | Liu J, Li Q, Kong LF, et al. COI-based DNA barcoding in Mytilidae species (Mollusca: Bivalvia) [J]. Acta Hydrobiol Sin (水生生物学报), 2011, 35: 874-881. |
| [30] | Shaw J, Lickey EB, Schilling EE, et al. Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: the tortoise and the hare III [J]. Am J Bot, 2007, 94: 275-288. |
| [31] | Kuang DY, Wu H, Wang YL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics [J]. Genome, 2011, 54: 663-673. |
 2015, Vol. 50
2015, Vol. 50


