2. 中国中医科学院中药研究所, 北京 100700
2. Institute of Chinese Materia Medica, China Academy of Chinese Medical Science, Beijing 100700, China
石斛为我国名贵中药材, 具有益胃生津、滋阴清热等功效, 用于热病津伤、口干烦渴、胃阴不足、食少干呕、病后虚热不退、阴虚火旺、筋骨痿软等症[1]。石斛属 (Dendrobium SW.) 是兰科 (Orchidaceae) 最大属之一, 由于形态特征相似, 分布范围广泛, 该属物种的鉴定是兰科植物里最复杂的问题之一[2]。因资源短缺, 亦有兰科石豆兰属 (Bulbophyllum Thouars.)、石仙桃属 (Pholidota Lind. ex Hook.) 和金石斛属 (Flickingeria Hawkes) 某些植物作为石斛使用[3]。自DNA条形码技术提出以来, 研究人员为寻找通用的DNA条形码序列进行了大量的研究。2007年在台北举行的第二届国际DNA条形码会议上, psbK-psbI序列被韩国Kim Ki-Joong等提出作为植物的候选条形码序列[4], 随后Lahaye等[5]选取南非18个科的31个物种, 针对psbK-psbI及其他序列的鉴定效率进行了研究, CBOL Plant Working Group也对其鉴定能力进行了研究, 结果表明psbK-psbI序列有较高的物种鉴定能力[6]。2010年, Nicolalde-Morejón等[7]对苏铁目泽米属植物DNA条形码分析研究表明, psbK-psbI基因间隔区对该属物种有最佳的物种水平鉴定能力。
本文拟对石斛属药用植物及其混伪品的叶绿体psbK-psbI基因间隔区进行分析比较, 为石斛属药用植物及其混伪品鉴别提供依据。
材料与方法材料 本实验共收集34份样品, 包括28份试 验样本和6份待检样本。所用石斛属植物及其混伪 品采自云南、广西、贵州等地, 具体信息见表 1。材料均经中国医学科学院药用植物研究所林余霖研究员鉴定。
|
|
Table 1 Plant samples and psbK-psbI sequence characters of species used in the present study |
方法 取变色硅胶干燥的叶片, 采用植物基因组DNA提取试剂盒 (北京天根生化有限公司) 提取总DNA。psbK-psbI序列扩增引物psbK-5'-TTAGCC TTTGTTTGGCAAG-3', psbI-5'-AGAGTTTGAGAGT AAGCAT-3'。25 µL PCR反应体系包含10×PCR buffer 2.5 µL、MgCl2 2 µL (25 mmol·L−1)、dNTPs 2 µL (2.5 mmol·L−1)、引物各1.0 µL (2.5 µmol·L−1) (上海生工生物有限公司合成)、聚合酶1.0 U (北京赛百胜生物科技有限公司)、总DNA 1 µL (~30 ng·µL−1)。PCR扩增程序94 ℃ 30 s, 51 ℃ 40 s, 72 ℃ 40 s, 35个循环[4]。PCR扩增产物回收后于中国农业科学院开放实验室进行双向测序。psbK-psbI序列范围参考D. officinale (KC771275, KJ862886) 和D. moniliforme (KC704119), 应用MEGA6.0软件[8]以Kimura-2参数 (K2P) 进行种内种间遗传距离分析, 以密花石豆兰 (Bulbophyllum odoratissimum) 为外类群[9, 10], 基于K2P距离模型构建NJ树, 具体方法参考Chen等[11]已发表文献。
结果 1 psbK-psbI序列特征分析17种石斛属药用植物psbK-psbI序列长度为474~513 bp, 其中细茎石斛序列长度最长, 密花石斛序列最短, 铁皮石斛psbK-psbI序列长度为507 bp, 金钗石斛为493 bp。混伪品密花石豆兰psbK-psbI长度分别为470 bp。17种石斛属药用植物及其混伪品GC含量为25.4%~27.6%, GC含量明显低于AT含量。石斛属药用植物psbK-psbI序列比对后长度562 bp, 变异位点71个, 简约信息位点46个, 分别占序列总长度的12.7% 和8.3%。铁皮石斛、金钗石斛的种内变异位点数均为0。细茎石斛3条序列有2种单倍型, 分别在189~198、358~368、451~452、459~465位点有四处插入/缺失。
2 石斛属及其混伪品种内种间的遗传距离应用MEGA6.0软件, 基于K2P距离模型计算石斛属及其混伪品种内种间的遗传距离, 结果见表 2。铁皮石斛、金钗石斛的种内遗传距离均为0。17种石斛种间平均遗传距离为0.028 4; 石斛属最小种间距离在细茎石斛与金钗石斛之间, 为0.006 1; 最大种间距离在剑叶石斛与铁皮石斛之间, 为0.058 0。混伪品密花石豆兰与17种石斛的遗传距离在0.093 2~0.120 4之间, 平均遗传距离为0.102 6。
3 系统发育树与待检物种鉴定由图 1可见, 17种石斛属药用植物具有单系性, 相同物种的不同样品均各自聚为一支, 外类群密花石豆兰与石斛属药用植物能够很好的区分。根据本研究的石斛属物种psbK-psbI序列, 作者成功鉴定了待检样品No.1~No.6 (inspected species No.1~No.6), 分别为流苏石斛D. fimbriatum (No.1)、美花石斛D. loddigesii (No.2)、金钗石斛D. nobile (No.3、No.4)、铁皮石斛D. officinale (No.5、No.6)。所有6个待检物种均成功与目标物种聚为一支, 种内无变异位点, 种内遗传距离为0, 均达到成功鉴别的效果。6份待检材料在基原植物开花后得到了验证, 待检样品No.1~No.6与所属基原植物准确无误。
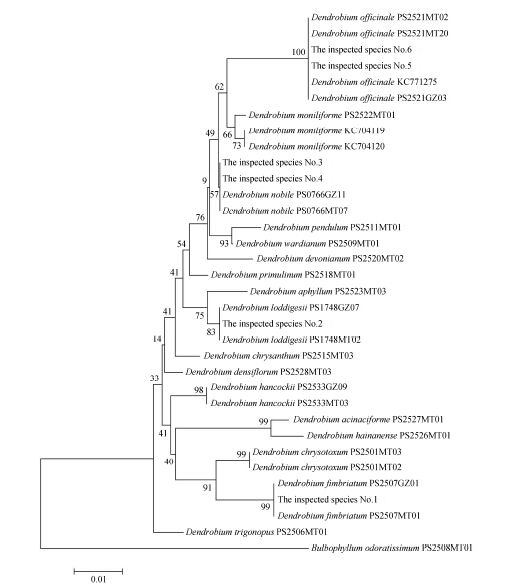
|
Figure 1 NJ tree constructed by MEGA 6.0 based on psbK-psbI of 17 taxa of Dendrobium and B. odoratissimum including 6 inspected species |
目前应用的植物DNA条形码序列主要有核ITS/ ITS2序列及叶绿体rbcL、matK和psbA-trnH。ITS/ ITS2为中国学者提出的DNA条形码序列[11, 12], 在石斛属早期的鉴定研究中一直有应用, 只是PCR扩增引物和条件与DNA条形码通用引物和条件有所不同。叶绿体rbcL+matK为国际DNA条形码协会植 物工作组 (CBOL PWG) 提出作为植物的核心DNA条形码序列[6], 在石斛属鉴定中, Singh等[13]研究表明rbcL+matK对36种石斛的鉴定效率为86.11%, 但在加入GenBank序列后鉴定效率下降至80.77%, 但Xu等[14]研究结果表明rbcL+matK只有24% 的物种分辨率, 得出这种不同结论主要是因为研究样本的不同。psbA-trnH序列被认为是叶绿体DNA基因间隔区中进化速率最快的片段之一, 被推荐作为有花植物鉴定的DNA条形码序列[15], 在石斛属物种鉴定中也有相关应用报道[9, 16]。psbK-psbI序列位于植物叶绿体DNA的LSC (large single-copy) 区, 该基因间隔区从藻类到陆生植物, 甚至寄生植物都相对保守[5]。CBOL PWG研究表明psbK-psbI序列在裸子和隐花植物中扩增效率低, 且由于POLY结构的存在导致测序质量较差, 但其物种鉴定能力在叶绿体DNA序列中仅次于psbA-trnH[6]。
|
|
Table 2 The K2P genetic distances of psbK-psbI sequences of 17 taxa of Dendrobium and B. odoratissimum. 1: D. officinale; 2: D. nobile; 3: D. chrysotoxum; 4: D. fimbriatum; 5: D. loddigesii; 6: D. moniliforme; 7: D. chrysanthum; 8: D. aphyllum; 9: D. densiflorum; 10: D. devonianum; 11: D. hainanense; 12: D. hancockii; 13: D. pendulum; 14: D. primulinum; 15: D. trigonopus; 16: D. acinaciforme; 17: D. wardianum; 18: B. odoratissimum (Bold font value showed the minimum or maximum genetic distances) |
本研究结果显示, psbK-psbI序列在石斛属及其混伪品中的扩增和测序效率均为100%, 通用性较强, 且测序质量高。不同石斛psbK-psbI序列在种间均存在差异, 在石斛属种内保守, 铁皮石斛、金钗石斛等物种的种内均无变异位点, 由实验样本获得的psbK- psbI序列数据, 成功完成了对6份待检样本的鉴定。本实验初步表明psbK-psbI序列可以作为石斛属药用植物及其混伪品鉴定的分子标记。对于能否作为中国石斛属物种鉴定的DNA条形码序列, 尚需扩大样本量进一步研究。对于某些单一序列或序列组合不能成功鉴定的物种, 基于全叶绿体基因组的超级条形码鉴定可能会是解决问题的新途径[17], 目前铁皮石斛和金钗石斛的叶绿体基因组序列已经完成测序[18,19,20], 基于叶绿体全基因组的超级条形码将会给石斛属的物种鉴定和系统进化关系研究提供新的证据。
致谢: 中国医学科学院药用植物研究所云南分所段立胜研究员提供部分研究材料。
| [1] | The State Pharmacopoeia Commission of the People's Republic of China. Pharmacopoeia of the People's Republic of China (中华人民共和国药典, 2010版) [M]. Beijing: China Medical Science Press, 2010: 85, 265. |
| [2] | Adams PB. Systematics of Dendrobiinae (Orchidaceae), with special reference to Australian taxa [J]. Bot J Linn Soc, 2011, 166: 105-126. |
| [3] | Teng YF, Wu XJ, Xu H, et al. A comparison of matK sequences between Herba Dendrobii (Shihu) and its adulterants species [J]. J China Pharm Univ (中国药科大学学报), 2002, 33: 280-283. |
| [4] | Pennisi E. Taxonomy wanted: a barcode for plants [J]. Science, 2007, 318: 190-191. |
| [5] | Lahaye R, Savolainen V, Duthoit S, et al. A test of psbK-psbI and atpF-atpH as potential plant DNA barcodes using the flora of the Kruger National Park (South Africa) as a model system [J]. Nat Precedings, 2008, <http://hdl.handle.net/10101/npre. 2008.1896.1>. |
| [6] | CBOL Plant Working Group. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 2009, 106: 12794-12797. |
| [7] | Nicolalde-Morejón F, Vergara-Silva F, González-Astorga J, et al. Character-based, population-level DNA barcoding in Mexican species of Zamia L. (Zamiaceae: Cycadales) [J]. Mitochondr DNA, 2010, 21 Suppl 1: 51-59. |
| [8] | Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]. Mol Biol Evol,2003, 30: 2725-2729. |
| [9] | Shao SG, Han L, Ma YH, et al. Analysis and authentication of cpDNA psbA-trnH regions of Dendrobium species of fengdous [J]. Acta Pharm Sin (药学学报), 2009, 44: 1173-1178. |
| [10] | Xiang XG, Schuiteman A, Li DZ, et al. Molecular systematics of Dendrobium (Orchidaceae, Dendrobieae) from mainland Asia based on plastid and nuclear sequences [J]. Mol Phylogenet Evol, 2013, 69: 950-960. |
| [11] | Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species [J]. PLoS One, 2010, 5: e8613. |
| [12] | China Plant BOL Group. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants [J]. Proc Natl Acad Sci USA, 2011, 108: 19641-19646. |
| [13] | Singh HK, Parveen I, Raghuvanshi S, et al. The loci recommended as universal barcodes for plants on the basis of floristic studies may not work with congeneric species as exemplified by DNA barcoding of Dendrobium species [J]. BMC Res Notes, 2012, 5: 42. |
| [14] | Xu SZ, Li DZ, Li JW, et al. Evaluation of the DNA barcodes in Dendrobium (Orchidaceae) from mainland Asia [J]. PLoS One, 2015, 10: e0115168. |
| [15] | Kress WJ, Wurdack KJ, Zimmer EA, et al. Use of DNA barcodes to identify flowering plants [J]. Proc Natl Acad Sci USA, 2005, 102: 8369-8374. |
| [16] | Yao H, Song JY, Ma XY, et al. Identification of Dendrobium species by a candidate DNA barcode sequence: the chloroplast psbA-trnH intergenic region [J]. Planta Med, 2009, 75: 667-669. |
| [17] | Li XW, Yang Y, Henry RJ, et al. Plant DNA barcoding: from gene to genome [J]. Biol Rev Camb Philos Soc, 2015, 90: 157-166. |
| [18] | Luo J, Hou BW, Niu ZT, et al. Comparative chloroplast genomes of photosynthetic Orchids: insights into evolution of the Orchidaceae and development of molecular markers for phylogenetic applications [J]. PLoS One, 2014, 9: e99016. |
| [19] | Yang P, Zhou H, Qian J, et al. The complete chloroplast genome sequence of Dendrobium officinale [J]. Mitochondr DNA, 2014, DOI: 10.3109/19401736.2014.945547.Yan WJ, Niu ZT, Zhu SY, et al. The complete chloroplast genome sequence of Dendrobium nobile [J]. Mitochondr DNA, 2015, DOI: 10.3109/19401736.2014.1003858 |
 2015, Vol. 50
2015, Vol. 50


