聚腺苷二磷酸核糖聚合酶 [poly(ADP-ribose) polymerase,PARP] 是一种细胞核酶,广泛存在于真核细胞中。PARP酶家族有17种亚型,其中PARP-1的含量最高,研究最为深入[1]。在炎症、神经性病变或是癌症的发生发展过程中,产生损伤的单链DNA能够激活PARP-1[2],催化ADP-核糖单元从烟酰胺腺嘌呤二核苷酸 (nicotinamide adenine dinucleotide,NAD+) 转移至受体蛋白 (包括PARP-1自身、组蛋白以及其他DNA修复蛋白等),启动DNA损伤修复过程[3]。PARP-1在DNA修复过程中发挥着重要作用,是目前得到广泛关注的抗肿瘤药物靶点。
以PARP-1为靶点的药物研究已有40多年的历史,研究领域涉及抗肿瘤、抗炎及神经损伤修复等,其中PARP-1抑制剂作为抗肿瘤药物,特别是针对BRCA缺陷性肿瘤的研究最为活跃[4, 5, 6, 7]。目前已知的抑制剂主要模拟了PARP-1与其底物NAD+ 的结合模式,作为NAD+ 的竞争性抑制剂发挥作用。到目前为止,已有多个候选药物进入临床研究阶段,包括AZD2281 (Olaparib)、ABT-888、MK-4827和BMN673等[8, 9, 10],结构见图 1。最近,Olaparib已经获得FDA 以及EMA批准,用于治疗至少经历3次化疗或铂敏感复发性的BRCA突变卵巢癌患者的维持治疗。作为第一个上市的PARP-1抑制剂,对该领域的研究意义重大[4, 11]。
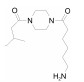
PARP-1蛋白共有3个结构域: N端的DNA结合区、中间的自我修饰区和C端与NAD+ 结合的催化 区[12]。已有的PARP-1与PARP-1抑制剂的共结晶复合物的结构显示,催化域的底物结合腔可分为两个部分: 与NAD+的烟酰胺片断结合的NI区 (nicotinamide-ribose bindingsite); 与腺苷酸结合的AD区(adenine-ribose binding site)[12, 13]。依据PARP-1底物结合腔特征,本文设计了3-(2-氧代-2-取代)乙酰氨基苯甲酰胺类化合物 (5a~5e、10a~10c,图 1)。以3-氨基苯甲酰胺模拟烟酰胺片断,与NI结合腔的氨基酸残基Gly863和Ser904形成氢键作用,苯环与氨基酸残基Tyr907形成π-π相互作用,这也是目前已知 的PARP-1抑制剂共有的药效团特征,对小分子与PARP-1的结合具有较大的贡献。草酰基作为连接基团,引入多种含氮杂环,可与AD结合腔的酸性氨基酸残基结合,增加结合力。本文报道了该类化合物的化学合成及对PARP-1酶的抑制活性。

|
Figure 1 Chemical structures of someknown PARP-1 inhibitors and target compounds |

|
Reagents and conditions: (a) tert-Butyl-2-chloro-2-oxoacetate,TEA,THF,r.t. (b) TFA,DCM,r.t.; (c) EDCI,HOBt,TEA,DMF,r.t.; (d) HCl,1,4-dioxane or ethyl acetate,r.t.; (e) CH3I,K2CO3,DMF; (f) Aldehyde,ammonium formate,Pd/C,IPA/H2O,r.t.; (g) Benzyl-2- chloro-2-oxoacetate,TEA,THF,r.t.; (h) H2,Pd/C,MeOH,r.t. Scheme 1 Synthetic route of3-aminobenzamide derivatives 5a-5e and 10a-10c |
本文设计的目标化合物 (5a~5e、10a~10c) 的合成方法如合成路线1所示。所有目标化合物均未见文献报道,合成中间体及目标化合物结构经1H NMR、LC-MS(ESI) 确证。产物收率、理化性质及波谱数据见表 1。以3-氨基苯甲酰胺为起始原料,Et3N为碱,与草酰氯单叔丁酯反应,再利用三氟乙酸脱除叔丁基得到关键中间体3,两步总收率为88%。3与不同的含氮杂环在EDCI/HOBt/Et3N的条件下发生缩合 反应,得到化合物4a~4e,收率为43%~67%。首先尝试用三氟乙酸脱除4a~4e末端的Boc保护基,但是TLC监测反应较杂。因此本文以2.5 mol·L-1HCl的1,4-二氧六环溶液作溶剂,室温反应得到目标化合物5a~5e。为了考察苯环上3-位氮原子烷基化对反应活性的影响,合成了目标化合物10a~10c。以K2CO3为碱,化合物2与碘甲烷反应引入甲基,在三氟乙酸的作用下脱除叔丁基得到关键中间体8a,总收率为55%。在EDCI/HOBt/Et3N的条件下,8a与单Boc保护的哌嗪发生缩合反应,得到化合物9a。以HCl的乙酸乙酯溶液做溶剂,脱除9a的Boc保护基得到目标物10a,两步收率为56%。采用该方法,引入其他烷基时,没有获得预期化合物。因此采用 还原氨化方法制备N-烷基化产物6b、6c。以甲酸铵为还原剂,Pd/C为催化剂,3-氨基苯甲酰胺与不同的醛发生还原胺化反应得到中间体6b、6c,收率为54%~64%。但是,6b、6c与草酰氯单叔丁酯缩合,所得产物中的叔丁基采用三氟乙酸的条件不易脱除,
|
|
Table 1 Physical and spectral dataof target compounds and intermediates |
且延长反应时间仍没有明显效果。因此,采用草酰氯单苄酯与化合物6b、6c反应,以Et3N作碱,得到7b、7c,常压催化氢化脱除苄基得到关键中间体8b、8c,收率为81%~99%。在EDCI/HOBt/Et3N的条件下8b、8c与单Boc保护的哌嗪发生缩合反应,得到化合物9b、9c。以2 mol·L-1 HCl的1,4-二氧六环溶液或乙酸乙酯溶液作溶剂脱除9b、9c的Boc保护基,得到目标物10b、10c,两步收率为56%~95%。值得一提的是,当苯环3-位氮原子上引入取代基 (R2) 后,产生了阻转异构现象。25 ℃的氢谱表明,化合物9a~9c、10a~10c中存在两个构象异构体,比例约为4∶1。当核磁温度升高至110 ℃时,氢谱峰型简 化,异构体现象消失,证实了上述阻转异构现象的存在。
2 PARP-1酶抑制活性和初步构效关系本文评价了目标化合物5a~5e和10a~10c及其N-Boc衍生物4a~4e、9a~9c对PARP-1酶的抑制活性。化合物对PARP-1抑制活性的IC50值见表 2和表 3。
|
|
Table 2 Chemical structures andinhibitory activities against PARP-1 of compounds 4a-4e and 5a-5e |
|
|
Table 3 Chemical structures andinhibitory activities against PARP-1 of compounds 9a-9c and 10a-10c |
在目标化合物5a~5e中,考察了不同的含氮杂环对PARP-1抑制活性的影响。当R1为哌嗪环时,化合物5a对PARP-1具有较强的抑制活性,IC50值为0.23 µmol·L-1。当R1为3-乙基哌嗪 (5d、5e) 时,化合物对PARP-1的抑制活性丧失,提示哌嗪环上不适于引入较大体积的取代基。当R1为3-氨基吡咯烷时,草酰基与环上氮原子偶联 (5b,IC50 = 2.21µmol·L-1) 或环外氮原子偶联 (5c,IC50 = 2.17µmol·L-1),所得化合物对PARP-1均有抑制活性,IC50值为微摩尔水平。与5a~5e相比,其相应的N-Boc衍生物4a~4e对PARP-1的抑制活性均降低。这些结果表明,杂环末端含有碱性的氮原子对抑制活性较为重要,可能与结合腔中的酸性氨基酸残基形成氢键作用。
本文还考察了苯环上3-位氮原子烷基化对反应活性的影响。与化合物5a比较,在3位氮原子上引入不同体积大小的烷基,如甲基、乙基、丙基后,所得化合物 (10a~10c) 的抑制活性均消失,表明该氮原子上氢原子可能参与了氢键结合,或该位置不宜于引入大体积的取代基。该类化合物的N-Boc衍生物 (9a~9c) 对PARP-1没有抑制活性。
3 分子对接研究本文采用分子对接,探索了目标化合物与PARP-1的结合模式。化合物5a与PARP-1的结合方式如图 2所示。化合物5a苯环上的氨基甲酰基的NH2与Gly863形成氢键; 羰基氧原子分别与Gly863和Ser904形成氢键; 同时苯环与Tyr907形成π-π相互作用,这与已知的PARP-1抑制剂在PARP-1催化域NI腔中的结合模式一致。苯环上3位的NH能够通过一分子水的介导,与Glu988形成氢键作用; 在该位置上引入烷基,不利于形成这种氢键作用,导致抑制活性下降,这与前述构效关系结果一致。哌嗪环末端的碱性氮原子能
|
Figure 2 Computationalprediction of binding mode for compound 5a. A:Predicted binding pose for compound 5a (ball-and-stick,carbon atoms coloredmagenta) within the binding pocket of PARP-1 (tan colored surface); B: Thepredicted interactions between PARP-1 and compound 5a,and hydrogen bonds were presented in orange lines. Molecular image was generated with UCSF Chimera[14, 15] |
够和Asp766形成氢键,表明该位置的碱性氮原子对小分子与PARP-1的结合具有重要作用。哌嗪环末端碳原子与结合腔之间没有较大空间,因此,在该位置引入取代基,由于空间阻碍作用,可导致活性丧失,这与本文获得的构效关系结果一致。
4 小结本文设计合成了新结构的3-(2-氧代-2-取代)乙酰氨基苯甲酰胺类化合物,评价了所有目标化合物 (5a~5e、10a~10c) 及其N-Boc衍生物 (4a~4e、9a~9c) 对PARP-1酶的抑制活性。6个化合物对PARP-1具有抑制活性,其中化合物5a的抑制活性最强,IC50值为0.23 µmol·L-1。本文利用分子对接方法,研究了该类化合物与PARP-1的结合模式,为该类化合物的进一步结构优化提供了启示。
实验部分熔点仪 (MP-J3,熔点未校正); 核磁共振仪 (Varian 300 MHz,Varian400 MHz Plus,内标TMS);质谱仪 (ThermoExactive Plus); 所用试剂均为市售分析纯。
1 化学合成 1.1 3-(2-氧代-2-叔丁氧基)乙酰氨基苯甲酰胺 (2) 的合成将3-氨基苯甲酰胺 (476 mg,3.5 mmol) 溶于THF (15 mL) 中,与三乙胺 (0.97 mL,7 mmol) 的THF (5 mL) 溶液混合。冰浴下,滴加入草酰氯单叔丁酯 (574 mg,3.5 mmol) 的THF (20 mL) 溶液。滴毕,升至室温反应1天,过滤浓缩得白色固体900 mg,
产率98%。mp >270 ℃。1H NMR (300 MHz,DMSO-d6) δ 10.73 (s,1H),8.17 (s,1H),7.94 (s,1H),7.82 (d, J = 8.1 Hz,1H),7.62 (d, J= 8.1 Hz,1H),7.41 (t,J = 7.8 Hz,1H),7.36 (s,1H),1.53 (s,9H). MS (ESI): m/z[M+H]+: 209.04。
1.2 2-(3-氨基甲酰基苯基)氨基-2-氧代乙酸 (3) 的合成将2 (300 mg,1.14 mmol) 溶于二氯甲烷 (5 mL) 中,滴加入TFA(1.5 mL) 的二氯甲烷 (3mL) 溶液,室温反应5 h,停止反应。反应液浓缩后依次加入氯仿 (8mL×3)、甲苯 (8 mL×2) 后减压蒸除溶剂和TFA。最后经无水乙醚/石油醚重结晶得白色固体210 mg,收率89%。mp 232~235 ℃。1H NMR (300 MHz,DMSO- d6) δ 10.83 (s,1H),8.24 (s,1H),7.95 (s,1H),7.82 (d,J = 9.3 Hz,1H),7.60 (d,J = 7.5 Hz,1H),7.41 (t,J = 7.5 Hz,1H),7.38~7.43 (m,2H)。MS (ESI): m/z [M+H]+: 265.24。
1.3 化合物4a~4e的合成以化合物3-[2-氧代-2-(4-N-Boc-哌嗪)乙酰氨基]苯甲酰胺 (4a) 为例: 在Ar气保护下,将3 (208 mg,1 mmol)、EDCI (320 mg,2 mmol)、HOBt (270 mg,2 mmol) 和TEA (300 mg,3 mmol)溶于DMF (10 mL),室温反应1 h,加入N-Boc- 哌嗪 (242 mg,1.3 mmol),室温反应36 h。反应液浓缩,加入乙酸乙酯 (20 mL),有机相经饱和Na2CO3溶液 (5 mL×3)、H2O (5 mL) 洗,无水MgSO4干燥,过滤、浓缩、柱色谱纯化 (DCM-CH3OH = 80∶1),得白色固体179 mg,收率45%。
1.4 化合物5a~5e的合成以化合物3-[2-氧代-2- (1-哌嗪)乙酰氨基]苯甲酰胺盐酸盐 (5a) 为例: 将4a (50 mg,0.133 mmol) 溶于HCl的1,4-二氧六环溶液 (4 mL,2.5 mol·L-1)中,室温反应20 min。反应液减压过滤,滤饼快速转移至梨形瓶中,加少量乙醚,刮壁,有白色固体析出。真空干燥得白色易潮解固体40 mg,收率99%。
1.5 3-N-甲基(2-氧代-2-叔丁氧基)乙酰氨基苯甲酰胺 (7a) 的合成将2 (528 mg,2 mmol)、K2CO3 (1.10 g,4 mmol) 和DMF (15 mL) 加入反应瓶中。冰浴下将CH3I (2.82 g,10 mmol) 用5 mL DMF稀释后滴加入反应液中,室温搅拌6 h。反应液浓缩,加入乙酸乙酯 (50 mL),有机相经H2O(6 mL) 洗,无水MgSO4干燥,过滤浓缩得白色固体443 mg,收率80%。
1.6 2-(3-N-甲基氨基甲酰基苯基)氨基-2-氧代乙酸 (8a) 的合成将7a (300 mg,1.08 mmol) 溶于二 氯甲烷 (5 mL) 中,滴加入TFA (1.5 mL) 的DCM (3mL) 溶液,室温反应5 h。反应液浓缩后依次加入氯仿 (8 mL×3)、甲苯 (8 mL×2) 后减压蒸除溶剂和TFA。最后经无水乙醚/石油醚重结晶得白色固体210 mg,收率88%。
1.7 化合物6b~6c的合成以化合物3-乙基氨基苯甲酰胺 (6b) 为例: 将甲酸铵 (3.15 g,50 mmol) 的H2O (2 mL) 溶液加入装有Pd/C (136 mg,20%)、异丙醇 (20 mL) 的反应瓶中,室温搅拌5 min。依次加入3-氨基苯甲酰胺 (680 mg,5 mmol) 及乙醛 (330 mg,7.5 mmol),室温反应4 h。反应液经硅藻土过滤,滤液浓缩后柱色谱纯化 (DCM-EA = 5∶1) 得白色固体528mg,收率64%。
1.8 化合物7b~7c的合成以化合物3-N-乙基 (2-氧代-2-苄氧基) 乙酰氨基苯甲酰胺 (7b) 为例: 将三乙胺 (404 mg,4 mmol) 的THF (4 mL) 溶液滴入至6b (328 mg,2 mmol) 的THF (5 mL) 溶液中。在冰浴条件下,滴加入草酰氯单苄酯 (594 mg,3 mmol) 的THF (6 mL) 溶液。滴毕,升至室温,反应30 min,停止反应。将反应液浓缩,柱色谱纯化 (PE-EA = 2∶1) 得白色固体658mg,收率100%。
1.9 化合物8b~8c的合成以化合物2-(3-乙基氨基甲酰基苯基)氨基-2-氧代乙酸 (8b) 为例: 将Pd/C (98 mg,20%) 加入7b (489 mg,1.5 mmol) 的甲醇 (30 mL) 溶液。常压氢化反应2 h。反应液经硅藻土过滤,浓缩即得白色固体255 mg,收率72.0%,未做纯化,直接投下一步。
1.10 化合物9a~9c的合成以化合物1-N-甲基-3- [2-氧代-2-(4-N-Boc-哌嗪)乙酰氨基]苯甲酰胺 (9a) 为例: 在氩气保护下,将8a (56 mg,0.25 mmol)、EDCI (78 mg,0.5 mmol)、HOBt (68 mg,0.5 mmol) 和TEA (76 mg,0.75 mmol) 溶于DMF (4 mL),室温反应1 h,加入N-Boc-哌嗪(61 mg,0.325 mmol),继续室温 反应25 h,停止反应。反应液浓缩,加入乙酸乙酯(20 mL),有机相经饱和Na2CO3溶液 (5 mL×2)、H2O (5 mL) 洗,无水MgSO4干燥,过滤、浓缩,柱色谱纯化 (DCM-CH3OH = 80∶1),得白色固体57 mg,收率57%。
1.11 化合物10a~10c的合成以化合物1-N-甲基- 3-[2-氧代-2-(1-哌嗪)乙酰氨基]苯甲酰胺盐酸盐 (10a) 为例: 将9a (100 mg,0.26 mmol) 溶于HCl的乙酸乙酯溶液 (5 mL,2 mol·L-1)中,室温反应过夜。反应液减压蒸除溶剂得白色固体87 mg,收率95%。
2 PARP-1酶抑制活性评价采用ELISA定量法评价化合物对PARP-1酶的抑制活性[16]。96孔板用0.1%Triton 100的PBS和PBS各洗板2次。然后设置空白孔、加酶对照孔、阳性化合物对照孔和测定孔。空白孔加入30 μL缓冲液配制的NAD+ (5 pmol)、10 μL纯化缓冲液、5 μL Sheared DNA (1 μg·mL-1),加酶对照孔加30 μL NAD+ (5pmol)、10 μL PARP-1 (0.005U)、5 μL Sheared DNA(1 μg·mL-1),阳性化合物对照孔加入30 μL NAD+ (5 pmol)、5 μL AZD2281各浓度稀释液、10 μL PARP-1 (0.005 U)、5 μL Sheared DNA (1 μg·mL-1),测定孔加入30 μL NAD+(5 pmol)、5 μL各测定化合物稀释液、10 μL PARP-1 (0.005U)、5 μL Sheared DNA(1 μg·mL-1)。然后各孔补齐50 μL体积,室温反应1 h。利用含0.1% TritonX-100的PBS和PBS各洗板2次。再加入用PBS 1:500稀释的抗PAR的鼠单克隆抗体50 μL,室温孵育1 h,0.1% Triton X-100的PBS和PBS各洗板2次。然后加入PBS 1∶4 000稀释的山羊抗鼠IgG 50 μL,室温孵育30 min,0.1% Triton X-100的PBS和PBS各洗板2次。再加入TMB反应液50 μL,室温避光反应15 min,然后利用含20% 浓盐酸的水溶液50 μL终止反应。450 nm测定吸光度。计算各化合物不同浓度稀释液对PARP-1的抑制率和IC50[17]。
3 分子对接采用DOCK 3.5.54 (UCSF) 程序[14]将化合物分子对接至PARP-1与小分子抑制剂复合物的晶体结构中 (PDB code: 4PJT)。从PDB蛋白质数据库 (protein data bank) 中下载PARP-1复合物的晶体结构 (PDB code: 4PJT),删除受体蛋白中的水分子,并对蛋白进行加氢和氨基酸残基修复,采用AMBER力场优化蛋白。利用ZINC工具为目标化合物分子生成可供使用的db格式文件[15]。以晶体复合物中的小分子为中心,定义其周围12Å以内的氨基酸残基为活性位点,用DMS软件计算生成溶剂可及性表面积,探针半径设为1.4 Å。对接参数设置中,simplex_iterations值设定为25,number_save值设定为10 000,以增加对接结果中配体分子可能的结合方式和对接中随机产生构象的数目。在分子对接结果的基础上,利用预先定义的结构过滤器 (structure filter),删除不合理的结合方式。随后,使用蛋白质局部优化程序 (PLOP 6.0)[18]基于OPLS全原子力场和通用波恩 (GB) 隐式溶剂模型将对接产生的复合物模型进行结构优化[19, 20]。依据计算得到的结合能和结合方式的合理性,选择最优结合模式。
| [1] | Rouleau M, Patel A, Hendzel MJ, et al. PARP inhibition: PARP1 and beyond [J]. Nat Rev Cancer, 2010, 10: 293-301. |
| [2] | Strom CE, Johansson F, Uhlen M, et al. Poly (ADP-ribose) polymerase (PARP) is not involved in base excision repair but PARP inhibition traps a single-strand intermediate [J]. Nucleic Acids Res, 2011, 39: 3166-3175. |
| [3] | Ali AAE, Timinszky G, Arribas-Bosacoma R, et al. The zinc-finger domains of PARP1 cooperate to recognize DNA strand breaks [J]. Nat Struct Mol Biol, 2012, 19: 685-692. |
| [4] | Menear KA, Adcock C, Boulter R, et al. 4-[3-(4-Cyclopropanecarbonylpiperazine-1-carbonyl)-4-fluorobenzyl]-2H-phthalazin-1-one: a novel bioavailable inhibitor of poly(ADP-ribose) polymerase-1 [J]. J Med Chem, 2008, 51: 6581-6591. |
| [5] | McCabe N. Deficiency in the repair of DNA damage by homologous recombination and sensitivity to poly(ADP-ribose) polymerase inhibition [J]. Cancer Res, 2006, 66: 8109-8115. |
| [6] | Ferraris DV. Evolution of poly(ADP-ribose) polymerase-1 (PARP-1) inhibitors. From concept to clinic [J]. J Med Chem, 2010, 53: 4561-4584. |
| [7] | Glendenning J, Tutt A. PARP inhibitors--current status and the walk towards early breast cancer [J]. Breast, 2011, 20: S12-S19. |
| [8] | Shen Y, Rehman FL, Feng Y, et al. BMN 673, a novel and highly potent PARP1/2 inhibitor for the treatment of human cancers with DNA repair deficiency [J]. Clin Cancer Res, 2013, 19: 5003-5015. |
| [9] | Sandhu SK, Schelman WR, Wilding G, et al. The poly(ADP-ribose) polymerase inhibitor niraparib (MK4827) in BRCA mutation carriers and patients with sporadic cancer: a phase 1 dose-escalation trial [J]. Lancet Oncol, 2013, 14: 882-892. |
| [10] | Zhou J, Zhu ZX, Chen XG, et al. Synthesis and activity evaluation of PARP-1 inhibitors with azaindole skeleton [J]. Acta Pharm Sin (药学学报), 2013, 48: 1792-1799. |
| [11] | Liu JF, Tolaney SM, Birrer M, et al. A Phase 1 trial of the poly(ADP-ribose) polymerase inhibitor olaparib (AZD2281) in combination with the anti-angiogenic cediranib (AZD2171) in recurrent epithelial ovarian or triple-negative breast cancer [J]. Eur J Cancer, 2013, 49: 2972-2978. |
| [12] | Langelier MF, Planck JL, Roy S, et al. Structural basis for DNA damage-dependent poly(ADP-ribosyl)ation by human PARP-1 [J]. Science, 2012, 336: 728-732. |
| [13] | Ishida J, Yamamoto H, Kido Y, et al. Discovery of potent and selective PARP-1 and PARP-2 inhibitors: SBDD analysis via a combination of X-ray structural study and homology modeling [J]. Bioorg Med Chem, 2006, 14: 1378-1390. |
| [14] | Meng EC, Shoichet BK, Kuntz ID. Automated docking with Grid-based energy evaluation [J]. J Comput Chem, 1992, 13: 505-524. |
| [15] | Irwin JJ, Shoichet BK. ZINC-a free database of commercially available compounds for virtual screening [J]. J Chem Inf Modeling, 2005, 45: 177-182. |
| [16] | Zhu Z, Jin J, Xue N, et al. Development and validation of high-throughput screening assays for poly(ADP-ribose) polymerase-2 inhibitors [J]. Anal Biochem, 2014, 449: 188-194. |
| [17] | Yao HP, Zhu ZX, Ji M, et al. Design, synthesis and biological evaluation of novel para-substituted 1-benzyl-quinazoline-2, 4 (1H, 3H)-diones as human PARP-1 inhibitors [J]. Acta Pharm Sin (药学学报), 2014, 49: 497-503. |
| [18] | Jacobson MP, Kaminski GA, Friesner RA, et al. Force field validation using protein side chain prediction [J]. J Phys Chem B, 2002, 106: 11673-11680. |
| [19] | Cao R, Liu M, Yin M, et al. Discovery of novel tubulin inhibitors via structure-based hierarchical virtual screening [J]. J Chem Inf Modeling, 2012, 52: 2730-2740. |
| [20] | Cao R, Huang N, Wang Y. Evaluation and application of MD-PB/SA in structure-based hierarchical virtual screening [J]. J Chem Inf Model, 2014, 54: 1987-1996. |
 2015, Vol. 50
2015, Vol. 50


