多肽药物具有生物活性强、免疫原性低、靶向性强和疗效好等优点,但线性多肽药物分子易被蛋
白酶降解,导致其口服生物利用度极低和血浆半衰期较短,这些多肽自身固有的结构特点限制了其作为临床用药的开发应用。具有生物活性的环肽具有明确的限制性构象,由于分子内没有游离的氨基和羧基端,耐受氨肽酶和羧肽酶等代谢酶的降解[1,2]。 目前环肽已在抗肿瘤和抗菌等方面的应用研究中展现出较线性肽更好的受体结合选择性等特点[3, 4, 5, 6]。环肽类药物的发展需要建立行之有效的多肽环化技术。
环肽的化学合成主要采用自然化学连接 (native chemical ligation,NCL) 法实现线性多肽的骨架环化,首先合成N-端为半胱氨酸和C-端含有硫酯 (thioester) 的线性肽,再利用巯基和硫酯之间的特异性反应,经过由N到S的酰基转移反应完成线性肽的环合,该 法受限于线性肽的特殊结构和Fmoc固相合成的肽 链长度 (大于60个氨基酸) 难以规模化合成[7]; 此后,基于生物表达的内含肽介导反式剪接 (intein-mediated protein trans-splicing) 反应及其所衍生的表达蛋白连接 (expressed protein ligation) 半合成方法具有与自然连接法相同的环化机制,也仅限于环肽合成的基础研究[8,9]。最近从G+ 细菌中鉴定的一种分选酶 (sortase),也称为转肽酶,已在蛋白质工程方面展现出巨大的应用潜力。其中从G+ 金葡菌 (Staphylococcus aureus) 鉴定的sortase A (SrtA),能特异地识别蛋白C-端的分选 (sorting) 信号,即保守的LPXTG结构 (Leu-Pro- X-Thr-Gly; X代表任一种氨基酸),使LPXTG结构在苏氨酸和甘氨酸之间的肽键断裂,催化蛋白共价连接到细菌细胞壁肽聚糖 (5个Gly肽) 上[10,11]。该酶已广泛地用于蛋白连接或蛋白标记[12,13]、细胞表面的蛋白标记[14, 15] 和蛋白环化[16, 17, 18]。
目前由分选酶介导的多肽环化已经显现出高效专一的酶促 催化优势[19, 20, 21, 22],但这些研究都采用了相同的液相催化模式,这种均一的水相催化固然有较高的催化效率,但也存在后续分离纯化困难和催化酶难以重复利用的问题。本研究构建了适合于酶蛋 白分离纯化和固相催化的含有2个融合标签的表达纯化系统,以N-端有3个甘氨酸 (GGG) 和C-端有分选酶识别序列 (LPETG) 的线性肽为底物,进行了由分选酶介导的固定化酶催化线性多肽环化的研究。
材料与方法 菌株、质粒和试剂
宿主菌Escherichia coli TG1,E. coli BL21 (DE3) 均为本实验室保存菌种; 大肠杆菌表达载体pET-32a(+)、pET-28a(+) 和pTWIN1分别购自Novagen和北京New England Biolabs公司; 克隆质粒EZ-T购自北京康润诚业生物科技有限公司; 限制性内切酶和T4 DNA连接酶均购自大连宝生物工程公司; KOD-Plus Taq DNA聚合酶购自TOYOBO公司; DNA胶回收试剂盒和质粒纯化试剂盒购自北京百泰克生物技术有限公司; 镍亲和色谱琼脂糖凝胶和几丁质亲和色谱凝胶分别购自北京百泰克生物技术有限公司和北京New England Biolabs公司。5-(2-氨基乙胺)-1-萘磺酸 (1,5-EDANS) 购自美国Sigma- Aldrich公司。其他生化试剂为市售分析纯试剂。 多肽合成
线性多肽及荧光多肽底物由吉尔生化(上海) 有限公司合成,其氨基酸残基序列如表 1。
|
|
Table 1 Amino acid sequences of peptides used in this study |
利用GenBank转肽酶基因碱基序列 (NC_002951) 合成PCR扩增引物如表 2,首先采用引物SORT_9和SORT_10进行DNA片段的补平反应 (95 ℃ 4 min; 95 ℃ 10 s、45 ℃ 30 s、72 ℃ 10 s,7个循环; 72 ℃ 10 min、4 ℃ 10 min); 然后利用补平反应后的DNA产物为模板,利用引物SORT_8和SORT_11进行第一轮PCR扩增 (95 ℃ 4 min; 95 ℃ 30 s、45 ℃ 40 s、72 ℃ 15 s,30个循环; 72 ℃ 10 min; 4 ℃ +∞); 其扩增产物作为下一组引 物进行PCR扩增反应的底物,依次进行PCR扩增的引物为SORT_7和SORT_12、SORT_6和SORT_13、SORT_5和SORT_14、SORT_4和SORT_15、SORT_3和SORT_16; 每一轮PCR扩增反应延伸时间延长 5 s,经过7轮PCR扩增反应,扩增得到570 bp的DNA序列; 然后再利用引物SORT_1和SORT_2及质粒pTWIN1为模板扩增编码几丁质结合结构域的177 bp DNA片段 (50 ng),并与利用引物SORT_3和SORT_16扩增的DNA片段 (150 ng) 进行重叠补平反应 (95 ℃ 4 min; 95 ℃ 10 s、45 ℃ 30 s、72 ℃ 45 s,7个循环; 72 ℃ 10 min、4 ℃ 10 min) 以其补平反 应产物为模板,再用引物SORT_1和SORT_16进行PCR扩增,通过“TA”克隆到EZ-T载体上,得到EZ- SrtA116,测序验证即得到含有转肽酶A (26~206 aa) 编码框的DNA片段; 再利用限制性内切酶NdeI和XhoI亚克隆到表达载体pET-32a(+) 和pET-28a(+),即得到表达载体pET32a-SrtA和pET28a-SrtA,前者表达仅有含有几丁质结合域的融合蛋白,而后者表达含有几丁质结合域和6个组氨酸的两个亲和色谱标签的融合蛋白。
|
|
Table 2 Primers used for the synthesis of sortase A gene |
用氯化钙法制备大肠杆菌E.coli BL21 (DE3) 感受态细胞,将表达载体pET32a-SrtA和pET28a-SrtA分别转化至制备好的感 受态细胞,转化平板挑取单菌落接种于25 mL LB液体培养基 (含100 μg·mL-1氨苄青霉素钠或50 μg·mL-1卡那霉素),利用控温摇床于37 ℃、260 r·min-1振荡培养至菌液OD600值达1.0左右。1/100的接种量转接入100 mL LB液体培养基,于37 ℃、195 r·min-1振荡培养至OD600达0.6~1.0。加入异丙基硫代半乳糖苷 (IPTG) (0.5 mmol·L-1) 在16 ℃诱导融合蛋白表达,振荡 (165r·min-1) 培养18 h。
收集诱导表达的菌体,用反应缓冲液 (0.15 mol·L-1 Tris-HCl、0.15 mol·L-1 NaCl、5 mmol·L-1 CaCl2、2 mmol·L-1 β-巯基乙醇,pH 7.5) 重悬。菌体悬液用超声波破碎细胞使蛋白释放 (超声功率50%,超声时间15 min,超声5 s间歇5 s)。高速离心 (17 000 r·min-1,15 min,4 ℃) 后取上清液,0.22 μm滤膜过滤。由质粒pET32a-SrtA表达的样品经几丁质 (chitin beads) 柱亲和色谱,上样流速为1.0~2.0 mL·min-1。用20倍柱床体积缓冲液以3~4 mL·min-1流速冲洗杂蛋白后,即得到融合表达的分选酶,可直接用于其介导的多肽剪切及环化反应。由质粒pET28a-SrtA表达的样品利用镍亲和色谱进行纯化,用10倍柱床体积的反应缓冲液 (含200~300 mmol·L-1咪唑) 洗脱镍胶上结合的蛋白; 再用20倍柱床体积的灭菌双蒸水漂洗,镍胶浸泡在20% 乙醇溶液中,4 ℃存放。 分选酶酶促动力学分析
酶促动力学分析运用镍亲和色谱凝胶柱纯化得到纯度较高的分选酶,参照文献[23]中分选酶催化蛋白连接反应的条件,本研究采用10 μL底物 (8 mmol·L-1)、4 μmol·L-1分选酶、终体积100 μL、pH 7.5、在37 ℃反应2 h。利用酶标仪EnSpire采用激发波长340 nm记录490 nm波长荧光强度的变化,每隔1 min采集一次分选酶催化底物Dabcyl-QALPETGEE-Edans水解后荧光基团Edans产生的荧光信号。在酶解底物产生的荧光值线性区内,利用Graphpad Prism 5.0软件,以OD340 nm/OD490 nm相对值对时间作图,曲线斜率为反应速度。用线性区段内任一时间点的斜率除以Edans标准曲线的斜率和酶终浓度求得反应初速度。最后,以反应初速度对底物浓度作图,利用“非线性回归拟合—Michaelis- Mente n方程”计算分选酶酶促动力学参数Km和Vmax值。实验重复3次,计算标准差,数据用均值 ± 标准差表示。 分选酶介导的多肽环化反应
取多肽底物各500 μg,用150 μL反应缓冲液溶解,取出50 μL样品再加入反应缓冲液至200 μL作为对照; 取3份经亲和色谱结合分选酶的几丁质凝胶200 μL,利用移液器去除反应缓冲液,再用反应缓冲液漂洗1次,去除反应缓冲液后把100 μL底物样品加入到含有转肽酶的几丁质凝胶上,补加反应缓冲液100 μL,30 ℃反应24 h。高效液相色谱分析酶促反应产物。 环肽分子的MALDI-TOF/MS质谱鉴定
利用ABI 4800 plus MALDI TOF/TOFTM系统,采用正离 子模式进行,利用50% 乙腈(ACN)、0.1% 三氟乙酸 (TFA) 配制8 mg·mL-1 α-氰基-4-羟基肉桂酸 (α-cyano- 4-hydroxycinnamic acid,CHCA) 作为MALDI介质,0.5 μL样品与0.5 μL介质混合点样。质谱仪扫描范 围m/z 500~5 000,采用反射手动模式测定分子离子质荷比。 结果 1 转肽酶基因的克隆和表达载体构建
利用重叠PCR方法首先获得扩增得到约570 bp的DNA序列,再与几丁质结合结构域的177 bp DNA片段进行重叠PCR获得741 bp DNA片段,编码246个氨基酸残基。整个DNA片段的获得见图 1。把获得SrtA基因片段进行克隆测序验证,与预期的编码序列一致,图 2是基因碱基序列及其所编码的氨基酸残基序列。
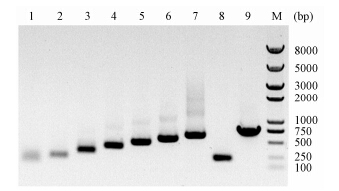
|
Figure 1 The sortase gene fragment amplified by consecutive polymerase chain reaction (PCR). Lane 1 to 9 indicate the amplified DNA fragments using the primers from sort_9/10,sort_8/11,sort_7/12,sort_6/13,sort_5/14,sort_4/15,sort_3/16,sort_1/2 to sort_1/16,separately |

|
Figure 2 The sequence of amino acid residues encoded by sortase A gene form S. aureus. The underline indicates the amino acid residues of his-tag and chitin binding domain,respectively. The arrow shows the start region of sortase A |
表达载体pET32a-SrtA转化大肠杆菌BL21 (DE3) 进行诱导表达,再利用几丁质亲和色谱纯化分选酶融合蛋白,图 3是对诱导表达和几丁质结合样品的SDS- PAGE分析。结果表明分选酶蛋白得到可溶性诱导表达,与理论上蛋白大小相符合,但也可见到该蛋白可能形成小部分二聚体。
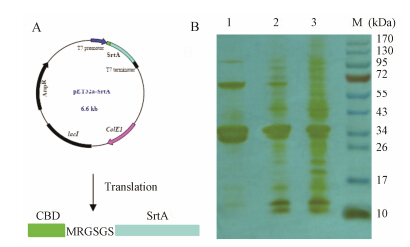
|
Figure 3 The expression and purification of the fusion protein SrtA. A: The construction of recombinant gene. B: SDS- PAGE analysis of SrtA expressed and purified. Lane 1: The purified samples; Lane 2: The inducible expressed samples; Lane 3: The samples not induced |
利用Edans标准品测定标准曲线为y = 1×106x + 350.6 (R² = 0.996 9)。如图 4所示,利用Graphpad Prism 5.0软件计算得到分选酶的相关动力学参数,如Vmax = (105. 4 ± 1.193) nmol·L-1·min-1,Km = (52 ± 2) μmol·L-1,kcat = 4.4 × 10-4 s-1,kcat/Km = 8.51 × 10-6 (μmol·L-1)-1·s-1。文献[23]报道分选酶水解相同的荧光底物,其Km = 10.88 μmol·L-1,kcat = 1.06 × 10-5 s-1,kcat/Km = 9.77 × 10-7 (μmol·L-1)-1·s-1。结果表明本研究表达纯化的分选酶对底物的亲和力与文献报道的基本一致,活性更高,说明两个融合标签引入没有影响其催化活性。
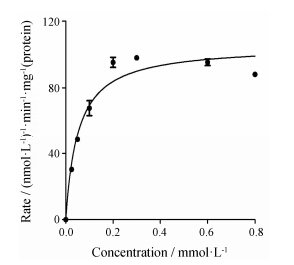
|
Figure 4 Characterization of the purified SrtA using the fluorogenic substrate (Dabcyl-QALPETGEE-Edans). Sortase A recognizes the peptide bond between T and G. Curves are plotted as initial velocity to final concentration of SrtA |
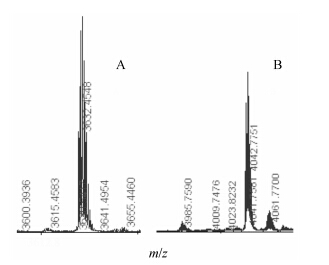
利用经几丁质凝胶纯化的分选酶催化3个线性肽的环化反应,经液相色谱分析表明IMM-W1和IMM-W2两个分子有相应环化的产物合成,其反应效率约在57%、55%左右。图 5A中tR = 41.8 min洗脱样品峰为IMM-W1环化的产物; 图 5B中tR = 37.7 min洗脱样品峰为IMM-W2环化的产物。收集洗脱峰进行质谱分析,图 6A所示,IMM-W1环化的产物的实测m/z为3 632.454 8 Da,与预期的理论值 (3 633.97 Da) 一致; 图 6B所示,IMM-W2环化的产物的实测m/z为4 042.775 1 Da,与预期的理论值 (4 042.01 Da) 一致。
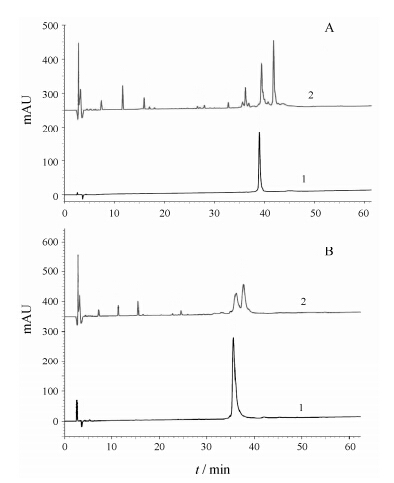
|
Figure 5 The HPLC analysis of the cyclic peptides synthesized by SrtA. A: IMM-W1; B: IMM-W2. 1: Substrates; 2: Reaction products |
|
Figure 6 The analysis of the cyclic peptides by MALDI TOF/ MS. A: Cyclic IMM-W1; B: Cyclic IMM-W2 |
Wu等[22]在2011年报道了一个7肽(VTSAPDT)
的环化,确定了N-端甘氨酸残基对环化效率的影响较大,其中含有5个甘氨酸的线性肽相对于含有3个甘氨酸的线性肽的环化产率提高了12倍,但同时也合成了环化的二聚串联环肽; Bolscher等[21]同期报道了1个N-端含有2个甘氨酸和分选酶识别序列 (LPETGG) 在内的44个氨基酸残基线性肽的环化。富含半胱氨酸的多肽本身具有较强的限制性构象,这些多肽在环化反应前进行了氧化并形成二硫键,使得多肽分子的N-端和C-端在空间结构上靠近,在随后的环化反应中也呈现出较高的环化效率[19,20]。本研究获得了IMM-W1和IMM-W2 2个环肽,环化效率大于50%。分选酶催化是一个可逆反应,其中保守序列中被剪切释放的甘氨酸可能竞争性再次亲核攻击酶和底物形成的硫酯酰中间体 ,去除反应过程中产生的游离甘氨酸,可能会进一步提高环化效率。结合文献报道,推测多肽的空间构象可能是分选酶介导环化反应效率的主要因素。
目前由分选酶介导的蛋白和多肽的连接或环化,依然面临如何快速去除分选酶的技术难题。本研究实现了双亲和色谱标签的融合蛋白酶的高表达,实验结果证实了2个融合标签对其催化反应活性影响很小; 酶促催化反应可以使用镍亲和色谱纯化获得的酶蛋白建立水相催化体系,而后再经过几丁质亲和色谱凝胶直接去除分选酶,有效地实现酶和催化产物的快速分离; 也可以直接利用几丁质凝胶纯化的分选酶建立固定化酶的催化体系,反应结束后移除反应产物,则固定化酶可以再利用。Steinhagen等[24]利用1个linker把表达纯化的SrtAΔ59与PEGA凝胶连接固定化,尽管实现了蛋白与多肽的催化连接,但实验操作过于复杂; 而本研究的固定化酶则可经过几丁质凝胶一步亲和色谱获得,实验操作简便易行。总之,本研究为利用分选酶介导的化学-酶法制备环肽提供了有益的探索,为设计和合成环肽药物提供新的技术手段。
| [1] | Roxin Á, Zheng G. Flexible or fixed: a comparative review of linear and cyclic cancer-targeting peptides [J]. Future Med Chem, 2012, 4: 1601-1618. |
| [2] | Nguyen LT, Chau JK, Perry NA, et al. Serum stabilities of short tryptophan- and arginine-rich antimicrobial peptide analogs [J]. PLoS One, 2010, 5: e12684. |
| [3] | Bogdanowich-Knipp SJ, Jois DS, Siahaan TJ. The effect of conformation on the solution stability of linear vs. cyclic RGD peptides [J]. J Pept Res, 1999, 53: 523-529. |
| [4] | Tiwari RK, Parang K. Conformationally constrained peptides as protein tyrosine kinase inhibitors [J]. Curr Pharm Des, 2012, 18: 2852-2866. |
| [5] | Chan LY, Zhang VM, Huang YH, et al. Cyclization of the antimicrobial peptide gomesin with native chemical ligation: influences on stability and bioactivity [J]. Chembiochem, 2013, 14: 617-624. |
| [6] | Selsted ME. Theta-defensins: cyclic antimicrobial peptides produced by binary ligation of truncated alpha-defensins [J]. Curr Protein Pept Sci, 2004, 5: 365-371. |
| [7] | Dawson PE, Muir TW, Clark-Lewis I, et al. Synthesis of proteins by native chemical ligation [J]. Science, 1994, 266: 776-779. |
| [8] | Scott CP, Abel-Santos E, Jones AD, et al. Structural requirements for the biosynthesis of backbone cyclic peptide libraries [J]. Chem Biol, 2001, 8: 801-815. |
| [9] | Camarero JA, Fushman D, Cowburn D, et al. Peptide chemical ligation inside living cells: in vivo generation of a circular protein domain [J]. Bioorg Med Chem, 2001, 9: 2479-2484. |
| [10] | Perry AM, Ton-That H, Mazmanian SK, et al. Anchoring of surface proteins to the cell wall of Staphylococcus aureus. III. Lipid II is an in vivo peptidoglycan substrate for sortase- catalyzed surface protein anchoring [J]. J Biol Chem, 2002, 277: 16241-16248 |
| [11] | Marraffini LA, Ton-That H, Zong Y, et al. Anchoring of surface proteins to the cell wall of Staphylococcus aureus. A conserved arginine residue is required for efficient catalysis of sortase A [J]. J Biol Chem, 2004, 279: 37763-37770. |
| [12] | Levary DA, Parthasarathy R, Boder ET, et al. Protein-protein fusion catalyzed by sortase A [J]. PLoS One, 2011, 6: e18342. |
| [13] | Matsunaga S, Matsuoka K, Shimizu K, et al. Biotinylated- sortase self-cleavage purification (BISOP) method for cell-free produced proteins [J]. BMC Biotechnol, 2010, 10: 42. |
| [14] | Baer S, Nigro J, Madej MP, et al. Comparison of alternative nucleophiles for sortase A-mediated bioconjugation and application in neuronal cell labelling [J]. Org Biomol Chem, 2014, 12: 2675-2685. |
| [15] | Schneewind O, Missiakas D. Sec-secretion and sortase- mediated anchoring of proteins in Gram-positive bacteria [J]. Biochim Biophys Acta, 2014 1843: 1687-1697. |
| [16] | Popp MW, Dougan SK, Chuang TY, et al. Sortase-catalyzed transformations that improve the properties of cytokines [J]. Proc Natl Acad Sci USA, 2011, 108: 3169-3174. |
| [17] | Zhang J, Yamaguchi S, Hirakawa H, et al. Intracellular protein cyclization catalyzed by exogenously transduced Streptococcus pyogenes sortase A [J]. J Biosci Bioeng, 2013, 116: 298-301. |
| [18] | Antos JM, Popp MW, Ernst R, et al. A straight path to circular proteins [J]. J Biol Chem, 2009, 284: 16028-16036. |
| [19] | Stanger K, Maurer T, Kaluarachchi H, et al. Backbone cyclization of a recombinant cystine-knot peptide by engineered sortase A [J]. FEBS Lett, 2014, 588: 4487-4496. |
| [20] | Arias M, McDonald LJ, Haney EF, et al. Bovine and human lactoferricin peptides: chimeras and new cyclic analogs [J]. Biometals, 2014, 27: 935-948. |
| [21] | Bolscher JG, Oudhoff MJ, Nazmi K, et al. Sortase A as a tool for high-yield histatin cyclization [J]. FASEB J, 2011, 25: 2650-2658. |
| [22] | Wu Z, Guo X, Guo Z. Sortase A-catalyzed peptide cyclization for the synthesis of macrocyclic peptides and glycopeptides [J]. Chem Commun, 2011, 47: 9218-9220. |
| [23] | Ton-That H, Mazmanian SK, Faull KF, et al. Anchoring of surface proteins to the cell wall of Staphylococcus aureus. Sortase catalyzed in vitro transpeptidation reaction using LPXTG peptide and NH(2)-Gly(3) substrates [J]. J Biol Chem, 2000, 275: 9876-9881. |
| [24] | Steinhagen M, Zunker K, Nordsieck K, et al. Large scale modification of biomolecules using immobilized sortase A from Staphylococcus aureus [J]. Bioorg Med Chem, 2013, 21: 3504-3510. |
 2015, Vol. 50
2015, Vol. 50


