2. 武汉轻工大学, 湖北 武汉 430023
2. Wuhan Polytechnic University, Wuhan 430023, China
黄芩为唇形科黄芩属植物黄芩(Scutellaria baicalensis Georgi) 的干燥根,性寒味苦,具有清热燥湿、泻火解毒、安胎等功效[1]。黄芩的主要活性成分为黄芩苷、黄芩素、汉黄芩苷、汉黄芩素等黄酮类化合物,具有抑菌、抗癌、抗氧化、镇静等作用[2]。
野生黄芩药材主要产自于内蒙、河北等省; 随着近年来市场对黄芩药材需求量的日益增加,野生黄芩的种质资源遭到过度采挖和严重破坏,野生资源储量锐减。目前栽培黄芩的面积逐渐增多,但由于生产上药农引种时存在盲目性,导致栽培黄芩个体间性状差异较大、易形成变异类型、种质退化严重,严重影响了药材的品质和产量[3,4]。目前还不明确黄芩野生资源的大量减少,是否影响黄芩某些优良性状及其基因资源的丢失。最大限度的发掘和保护黄芩的遗传多样性,对保持黄芩遗传资源具有重要的作用[5]。
简单重复序列 (simple sequence repeat,SSR) 又称微卫星,是指以2~6个核苷酸为基本单位的串联重复序列组成的DNA片段.它们的长度大多为100~200个碱基对,广泛存在于真核生物基因组中[6]。SSR序列具有分布广泛、共显性遗传、多态性位点多、信息含量丰富、物种间转移性好、易于检测和可重复性好的特点,已被广泛应用于品种鉴定,种质资源保存和利用、遗传多样性分析、分子标记辅助选育等研究领域[7]。
本研究基于黄芩基因组数据 (本课题组保存) 开发SSR分子标记,并以采自10个不同产地的50份黄芩种质为研究对象,对黄芩种质资源的遗传多样性进行了初步研究,揭示不同种群间的亲缘关系,为进一步黄芩种质资源评价、保存和新品种选育等提供物质基础和理论依据。
材料与方法 材料选取10个不同产地的黄芩药材原植物,每个产地取5个植株,每个植株采集新鲜叶片并立即用硅胶干燥保存。样品主要采自河北、内蒙古等地。实验材料详见表 1。
|
|
Table 1 Samples used in this study. n = 5 |
通过ssr.pl程序 (http:// www.gramene.org/db/markers/ssrtool) 从黄芩基因组序列中搜索黄芩SSR序列,识别标准: 二、三、四、五、六核苷酸重复基序的最小重复次数为9、6、5、4、4次。
SSR引物的设计应用Primer 3.0在线软件 (http://frodo.wi.mit.edu/primer3/) 设计引物,如表 2所示。设计原则为: 引物长度18~24 bp,最适长度为20 bp; PCR产物长度为100~400 bp; 最适Tm值为 60 ℃; GC含量为40%~60%。荧光引物为5' 末端添加FAM标记,所有引物均由上海生工生物工程有限公司合成。
|
|
Table 2 Primer sequences used in this study |
参照CTAB法[8]提取供试材料的基因组DNA,1% 琼脂糖凝胶电泳检测DNA质量,用NanoDrop ND-1000分光光度计 (Thermo Fisher Scientific,USA) 测定DNA的浓度和纯度,将其稀释到100 ng·μL-1,-20 ℃冰箱保存备用。PCR反应体系 (25 μL): 2.5 μL 10 × PCR buffer,2 μL 2.5 mmol·L-1 dNTPs,10 μmol·L-1引物各1 μL,0.25 μL 2.5 U rTaq DNA聚合酶 [宝生物工程 (大连) 有限公司],1 μL DNA模板,剩余体系用灭过菌的双蒸水补齐。反应在9700型PCR扩增仪 (ABI公司) 上进行。反应程序为: 94 ℃预变性5 min; 94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,38个循环; 72 ℃延伸10 min。PCR产物送北京睿博兴科生物科技有限公司进行STR分型。
数据统计与分析 利用GeneMarker V1.75 (Soft Genetics LLC,USA) 软件读取毛细管电泳数据,按照引物信息中的目标片段大小以及Marker,在STR分型结果中确定出目标片段,读取时只取目标片段。不同的等位基因用英文字母来表示,按照片段的大小依次标为A、B、C、D等,相同大小的等位基因用相同字母表示。利用Popgene 32软件分析遗传结构和遗传多样性参数。用PIC_CALC软件 (http://dl.getdropbox. com/u/695591/PIC_CALC.rar) 分析多态性含量 (PIC),利用SPSS17.0对分析结果做统计分析,方法为配对t检验。
结果 1 黄芩基因组SSR 频率及其SSR 重复基序类型从黄芩基因组数据库中共获得12 775条SSR序列,从表 3可知,黄芩基因组的SSR重复基序类型 较为丰富,其最主要的重复基序类型为二核苷酸 (68.32%),其次为三核苷酸 (18.63%),五核苷酸 (5.29%),六核苷酸 (4.76%) 及四核苷酸 (3.00%)。二核苷酸占了显著的优势,该结果与芝麻[9]、冷蒿[10]、大白菜[11]、白三叶草[12]等结果一致。另一方面,二、三核苷酸含量比四、五、六核苷酸含量丰富,这可能因为四、五、六核苷酸更易发生移码突变和受到更强的自然选择有关[13]。
|
|
Table 3 Types and distributions of genomic-SSR in Scutellaria baicalensis |
在黄芩各二核苷酸基序中,CT/AG、GA/TC重复次数最多,分别占总二核苷酸基序的22.98% 和22.43%; 其次是TC/GA、AG/CT,分别占总二核苷酸基序的20.31% 和19.39%。三核苷酸基序中出现频率最高的为TTC/GAA、GAA/TTC,分别占总三核苷酸数量的7.85% 和6.81%,其次为AAG/CTT、CTT/AAG各占总三核苷酸基序的5.59% 和5.38%。但未发现GC/CG重复基序,这与菜豆[14]的研究结果相一致。
2 黄芩SSR引物的遗传多态性检测本实验设计并合成了20对SSR荧光标记引物,利用不同产地黄芩DNA进行PCR扩增,选取扩增产物稳定性好、多态性及清晰度高的9对SSR引物用于本实验的研究。STR分型结果如图 1所示。
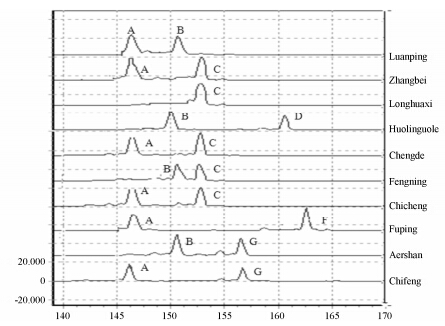
|
Figure 1 Amplification of S.baicalensis from ten populations using primer Sbssr.7. A,B,C,D,et al represent different objective fragment size |
利用9对荧光标记引物对10个不同产地的50个不同植株黄芩基因组DNA进行SSR-PCR扩增,共检测出68个等位基因数,如表 4所示。其中引物Sbssr.1、2的等位基因数最低,为4个; 引物Sbssr.5最高,为12个; 平均每个SSR位点等位基因数为7个,有效等位基因数为3.27个。利用Nei’s[15] (1973) 氏算法计算可知其表观杂合度Ho范围为0.19~0.68,平均值为0.45; 引物Sbssr.7最高,为0.68; Sbssr.2最低为0.19。期望杂合度He值[16]范围是0.23~0.85,平均值为0.60,Sbssr.6最高为0.85,Sbssr.2最低为0.23。
|
|
Table 4 Genetic diversities for fifty S.baicalensis individuals at the loci based on microsatellite data. Na: Number of alleles; Ne: Effective number of alleles; Ho: Observed heterozygosities; He: Expected heterozygosities; PIC: Polymorphism information content. I: Shannon’s information index [Lewontin (1972)]. HWE: Hardy-Weinberg equilibrium probability |
多态性信息含量 (PIC)[17]值是衡量基因变异程度的多态信息含量的指标; 当PIC > 0.5时,该位点为高度多态性位点; 当0.25 < PIC < 0.5时,该位点为中度多态性位点; 当PIC < 0.25时,为低度多态性位点。本实验中9对SSR引物的多态性信息含量平均值为0.72,按照Bostein的理论,9对SSR引物除了Sbssr.2为中度多态性位点 (PIC = 0.39) 外,其余7个均为高度多态性位点,PIC值均大于0.5,其中Sbssr.6最高,PIC值为0.95; Shannon’s信息指数为1.32。PIC和表观杂合度Ho结果一致,说明所筛选出的9对黄芩SSR引物遗传多态性较高。9对引物中,除了引物Sbssr.3、4、5显著偏离哈温平衡外 (P < 0.05),其余6条均符合哈温平衡。以上结果表明,这些位点均可作为有效的遗传标记用于黄芩居群间遗传多样性评估和系统发生关系的研究。
在所选10个野生黄芩居群中,表观杂合度 (Ho) 范围为0.24~0.59,平均为0.45,期望杂合度 (He) 变化范围为0.34~0.54,平均为0.45; Ho和He值最高都是霍林郭勒居群,最低都为承德围场县居群; 等位基因变化范围为2~3,较多的依次为阿尔山、霍林郭勒、丰宁、赤城、张北、滦平等,其次为赤峰、承德围场县、阜平、隆化西等。Shannon’s信息指数 (I) 范围为0.57~0.95,最高为霍林郭勒,最低为承德围场县。综合以上几点,可以看出霍林郭勒居群在10个野生居群中的遗传多样性是最高的,承德围场县居群的遗传多样性则最低 (表 5)。
|
|
Table 5 Genetic diversities in different populations of Scutellaria baicalensis |
在10个不同居群的野生黄芩中,其群体间的基因分化系数 (Fst) 值为0.131; 群体总近交系数 (Fit) 为0.328,群体内的近交系数 (Fis) 为0.226,表明野生黄芩居群内的近交程度高,远交程度低。种间遗传分化系数 (Gst) 为0.131,即13.1% 的遗传变异存在于居群间,86.9% 的遗传变异存在于居群内。基因流Nm为1.654 ( > 1),表明野生黄芩居群间的基因交流在种群分化中比遗传漂变发挥更重要的作用。
3 黄芩种质资源的聚类分析和亲缘关系分析由表 6可知,10个不同产地种质间的遗传一致度 (I) 和遗传距离 (D) 分别为0.56~0.85、0.17~0.58。其中9号种质河北滦平与3号内蒙古霍林郭勒之间 的遗传距离最大 (0.58),遗传相似性最小 (0.56),说明两个种质间的亲缘关系最远、遗传差异最大。9号种质河北滦平与10号河北隆化西阿超满族之间的遗传距离最小 (0.17),遗传相似性最大 (0.85),说明这两种质的亲缘关系最近、遗传差异最小,以上结果可知黄芩不同居群内既存在遗传变异又保持了遗传稳定性。
|
|
Table 6 Genetic similarity coefficient I (above diagonal) and genetic distance D (below diagonal) in ten populations of S.baicalensis |
利用Popegene软件对50份不同产地黄芩种质资源进行聚类分析 (图 2),整体可以分为两大类,其中第一大类又分为两小组,一组为内蒙古赤峰、阿尔山,河北滦平、隆化西阿超满族等; 另一小组为河北围场县、阜平、张北县、丰宁、赤城等。第二大类为内蒙古霍林郭勒单独聚为一类。河北滦平与内蒙古霍林郭勒遗传距离最远,与聚类结果相一致。从整体上看,本文研究的河北省7个黄芩居群聚为一类,但内蒙古赤峰和阿尔山的却与河北滦平等的聚在一起,与霍林郭勒相距较远,可见黄芩种质在该地区的地理界限并不明显,难以判断样品聚类分析结果与地理分布位置有直接关系,这与文献[4,18]的结果相一致。

|
Figure 2 Dendrogram of the ten S. baicalensispolulations based on genomic-SSR markers |
遗传多样性一般是指种内的遗传多样性,即种内个体之间或一个群体内不同个体的遗传变异总和。遗传多样性作为生物多样性的重要组成部分,是物质多样性和生态多样性的基础,其与生物自身的生存能力和竞争能力密切相关[19]。遗传多样性越高,说明生物体在DNA 分子水平上的差异越大,其对环境变化的适应能力也就越强,越容易扩展其分布范围和开辟新的环境,更有利于进一步的选择和保存[20]。
期望杂合度 (He) 被认为是衡量一个物种遗传多样性水平高低的标准[16],本文研究结果表明黄芩的期望杂合度为0.45,高于双子叶植物的平均水平 (He = 0.19); 居群间的遗传一致度 (I) 和遗传距离 (D) 分布范围为0.56~0.85、0.17~0.58; Shannon’s信息指数为1.32。以上结果表明所收集黄芩种质资源具有较高的遗传多样性,为资源保护和进一步评价提供了依据。种间遗传分化系数 (Gst) 为0.131,即13.1% 的遗传变异存在于种群间,86.9% 的遗传变异存在于种群内,可能因为黄芩属于虫媒异花传粉,在同一居群内的个体可充分杂交,后代遗传分化较大; 不同居群间由于地理隔离,降低了杂交的可能性,所以导致居群间的遗传分化较小[4]。
黄芩的野生资源分布极为广泛,河北承德地区是黄芩的道地产区,享有“热河黄芩”之美称。但本文研究结果表明,10个居群中,承德围场县的遗传多样性最小,可能因为在资源开发中,没有处理好保护与利用的关系,一味追求经济利益,过度采挖导致野生资源大量下降,遗传多样性减少; 也有可能因为人类毁林开荒,过度放牧,以及城市建设等破坏了野生黄芩的生态环境和资源分布。由此可见,必须加强野生黄芩种质资源的保护,并且选育高产、高效的优良品种。
聚类结果与种质来源地区分布不一致,推其原因可能为: ① 居群单株取样数量可能导致结果有所差异; ② 虽然不同地区黄芩所受环境不同,但未能使黄芩在DNA分子水平上发生显著差异; ③ 通过自然选择或人工引种导致某些基因在不同地区的渗入; ④ 不同的方法从不同的角度和水平分析,得到的结果就可能不一致,如张红瑞等[21]利用ISSR方法证明黄芩种源间的亲缘关系与地理位置有一定的关系。
本研究成功开发出了9对黄芩基因组SSR分子标记引物,结果表明黄芩具有较高的遗传多样性。同时为黄芩种质资源开发与保护、物种多样性研究、遗传作图、新品种选育等奠定了基础。
| [1] | Song LR, Hong X, Ding XL, et al. Dictionary of Modern Medicine (现代中药学大辞典) [M]. Beijing: People's Medical Publishing House, 2001: 1865-1869. |
| [2] | Chen XQ, Qi XJ, He EM. Latest progress on Scutellaria baicalensis Georgi [J]. Food Drug (食品与药品), 2006, 8: 23-27. |
| [3] | Li X, Huang LQ, Shao AJ, et al. Rescarch progress on genetic diversity of Scutellaria baicalensis Georgi [J]. World Sci Technol (世界科学技术), 2003, 5: 54-58. |
| [4] | Wen MM, Li GS, Zhang LJ, et al. Analysis and evaluation on genetic diversity of Scutellaria baicalensis G. by ISSR markers [J]. Bull Bot Res (植物研究), 2012, 32: 32-37. |
| [5] | Feng XF, Hu SL, Guo BL, et al. A preliminary study on genetic of populations of Scutellaria baicalensis Georgi [J]. World Sci Technol Mod Tradit Chin Med (世界科学技术: 中医药现代化), 2002, 4: 38-43. |
| [6] | Li SS, Sun CY, Jiang SC, et al. SSR molecular markers and its applications in plant genetics and breeding [J]. Jilin Veget (吉林蔬菜), 2014, 5: 33-38. |
| [7] | Ren JW. SSR marker technology and its application to maize [J]. J Anhui Agric Sci (安徽农业科学), 2011, 17: 55-58. |
| [8] | Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochem Bull, 1987, 19: 11-15. |
| [9] | Wei X, Wang LH, Zhang YX, et al. Development of simple sequence repeat (SSR) markers of Sesame (Sesamum indicum) from a genome survey [J]. Molecules, 2014, 19: 5150-5162. |
| [10] | Liu Y, Zhang SX, Huo NX, et al. The development of molecular marker from Mongolian medicinal plant Artemisia frigid [C]. San Diego, CA: International Plant & Animal Genome XXII Conference, 2014. |
| [11] | Yi ZH, Lu YF, Guo XQ, et al. Development of simple sequence repeat (SSR) and insertion/deletion (indel) markers in Chinese cabbage (Brassica rapa spp. Pekinesis) and analysis of their transferability [J]. J Agric Biotechnol, 2012, 20: 1398-1406. |
| [12] | Kölliker R, Jones ES, Drayton MC, et al. Development and characterization of simple sequence repeat (SSR) markers for white clover (Trifolium repens L.) [J]. Theor Appl Genet, 2001, 102: 416-424. |
| [13] | Jiang C, Yuan Y, Liu GM, et al. EST-SSR identification of Lonicera japonica Thunb [J]. Acta Pharm Sin (药学学报), 2012, 47: 803-810. |
| [14] | Blair MW, Hurtado N, Chavarro CM, et al. Gene-based SSR markers for common bean (Phaseolus vulgris L.) derived from root and leaf tissue ESTs: an integration of the BMc series [J]. BMC Plant Biol, 2011, 11: 50. |
| [15] | Nei M. Molecular Evolutionary Genetics [M]. New York: Columbia University Press, 1987. |
| [16] | Hamrick JL, Godt MJW. Allozyme diversity in plant species [M] // Brown AHD, Clegg MT, Kahler AL, et al. Plant Population Genetics, Breeding and Genetic Resource. Sunderland: Sinauer, Associates, 1990: 43-63. |
| [17] | Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkag map in man using restriction fragment length polymorphisms [J]. Am J Hum Genet, 1980, 32: 314-324. |
| [18] | Shao AJ, Li X, Huang LQ, et al. RAPD analysis of Scutellaria baicalensis from different germplasms [J]. China J Chin Mater Med (中国中药杂志), 2006, 31: 452-455. |
| [19] | Liu P, Ma HW, Wang ZJ, et al. Research progress on genetic diversity of germplasm resources of medicinal plants in China [J]. J Agric Sci (农业科学杂志), 2008, 29: 66-70. |
| [20] | Ge SJ, Meng YJ, Li GM, et al. Research progress on genetic diversity in Chinese medicinal plants [J]. Chin Tradit Herb Drugs (中草药), 2006, 37: 1584-1589. |
| [21] | Zhang HR, Ma YQ, Gao ZM, et al. Study on germplasm resources of Scutellaria baicalensis using ISSR makers [J]. J Henan Agric Univ (河南农业大学学报), 2012, 46: 380-384. |
 2015, Vol. 50
2015, Vol. 50


