2. 中国医学科学院、北京协和医学院药用植物研究所, 北京 100193
2. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China
分子遗传连锁图谱的构建是数量性状基因定位、图位基因克隆以及分子标记辅助育种的重要理论基础,利用遗传连锁图谱寻找分子标记与控制优良性状基因之间的连锁关系,对植物基础遗传育种研究具有重要的应用价值[1]。有些植物由于生长周期长、栽培历史短、遗传背景复杂和基因高度杂合等特点[2],很难在短期内构建理想的作图群体,因此制约了这些植物遗传图谱的构建。1994年Hemmat等[3]提出“双假测交(pseudo test cross)”的模型并且将其成功应运用于苹果的遗传作图,高丽霞等[4]认为此方法对于天然杂合性高、主要采取无性繁殖的植物同样适用,这使得这些植物遗传图谱的构建成为可能,近年来利用该模型先后构建了北柴胡[5]、黄花蒿[6]、甘薯[7]、腊梅[8]、菊花[9]、斐济果[10]、紫薇[11]等十几种遗传连锁图,“双假测交”方法的应用为植物的群体构建提供了有力的依据。
丹参(Salvia miltiorrhiza Bge.) 为唇形科鼠尾草属多年生草本植物,并被提出作为药用模式植物研究[1],丹参的干燥根及根茎是我国重要的大宗药材之一,具有重要的药用价值[12]、经济价值,但一直以来关于丹参的遗传基础研究相对滞后。目前,国内外对丹参研究主要集中在相关基因克隆[13,14]、次生代谢 产物[15,16]以及转录组学研究[17,18]等方面,还没有关于丹参遗传连锁图谱的报道。本研究根据“双假测交”作图策略,以配置的F1群体为材料,利用SSR、SRAP和ISSR 3种分子标记技术构建国内外首张丹参分子遗传连锁图谱。研究的开展可为控制丹参重要性状的基因进行定位、作图,进而为基因克隆、分子标记辅助育种和重要数量性状的遗传改良等研究提供坚实的理论基础。 材料与方法 材料
选用山东农业大学农学院中药资源课题组前期筛选得到的两个丹参品系,母本为ZH74品系,其根中脂溶性有效成分含量相对不高,但根冠幅大、根粗壮高产,其叶长形浅绿、花紫色; 父本为BH18品系,其根中脂溶性有效成分含量高、根条多且细,其叶偏圆形、叶色深绿、花白色,开花晚。以两个品系杂交所得到的F1代94株植株作为作图群体,亲本和部分后代叶片和花性状见图 1。所有材料植于山东农业大学药用植物园。
 |
Figure 1 Plant,leaf and flower characters of parents and its F1 progenies for mapping population. A: Male parent BH18 plant; B: Female parent ZH74 plant; C: Leaf of parents and F1 progenies; D: Flower of parents and F1 progenies |
取丹参幼嫩的叶片,采用宋振巧等[19]报道的改良CTAB法提取总DNA,并将DNA浓度稀释至20 ng·μL-1,-20 ℃保存备用。 SSR引物分析方法
研究共选用了517对SSR引物用于遗传图谱构建,其中200对基因组SSR引物为中国医学科学院药用植物研究所提供,编号DSSR+数字; 根据文献报道[20,21,22,23,24,25,26,27]合成另208对丹参EST-SSR引物,引物编号与引用文献编号一致,编号格式为Sm-ES+数字、Smssr+数字、LAB+数字以及SoUZ+数字,用于建图的该类型引物编号及其序列列于表 1; 此外,本课题组通过构建丹参的cDNA文库,经引物设计得到109对EST-SSR引物,编号为DESSR+数字,用于丹参上图的引物编号及其序列见表 1。SSR-PCR扩增总体积15 μL,其中包括Taq PCR Master Mix 7.5 μL、上下游引物各1 μL、模板DNA 1 μL、ddH2O 4.5 μL。扩增反应程序为: 94 ℃预变性4 min; 94 ℃变性40 s,55 ℃~58 ℃退火40 s,72 ℃延伸1 min,35个循环; 72 ℃延伸7 min。扩增产物用6%聚丙烯酰胺凝胶检测,电泳电压120 V,电泳时间2.5 h。
|
|
Table 1 Sequences of SSR primers cited to use for the genetic linkage map |
实验采用的SRAP和ISSR引物序列以及扩增条件参考宋振巧[28]的报道。 数据分析及图谱构建
根据亲本和F1杂交后代的分离类型进行标记分析。标记分离类型包括双亲杂合标记、母本特有标记和父本特有标记,分别包括ab×cd、ef×eg、hk×hk、lm×ll、nn×np类型,根据标记类型对数据进行统计分析。在P < 0.05水平上,对所有分离标记进行卡方检验,检测是否有偏分离存在。使用Joinmap 4.0作图分析软件,选择LOD值为5.0,以“CP”作图模式构建丹参的遗传连锁图谱。框架图谱覆盖率为框架图总长 (Gof) 占图谱预期长度 (Ge) 的百分比。图谱的预期长度Ge = (Ge1 + Ge2) / 2,根据Postlethwait等[29]方法估算连锁图谱长度Ge1 = Gof + 2s×n,s是连锁图谱的标记平均间隔,n是连锁群数目。按照Chakravarti等[30]的方法计算Ge2 = Gof (m+1)/ (m-1),m是连锁群上的总标记数。 结果 1分子标记的多态性分析
本研究首先将517对SSR引物、100对SRAP引物和8条ISSR引物在亲本间进行多态性筛选 (表 2)。其中总共550对引物进行了有效扩增,307对引物具有多态性,包括: 259对SSR引物、47对SRAP引物、1条ISSR引物,分别占所选引物的50.1%、47.0% 和12.5%,其中SRAP引物共产生107个等位基因,平均每对引物产生2.3个等位基因; ISSR引物共产生2个等位基因,平均每对引物产生2个等位基因。据此,本研究选择具有多态性的307对引物用于进一步筛选,以构建丹参的遗传连锁图谱。307个多态性引物在F1群体中共扩增出159个清晰稳定的标记位点,经卡方检验后,共有17个位点表现出偏分离,频率为10.7%。
|
|
Table 2 Amplification results of different primer types |
标记位点的分离类型,是指F1代中亲本的等位基因组成以及子代可能出现的基因型。本研究利用多种分子标记对丹参的F1代群体进行基因检测时,发现在不同遗传位点上电泳检测出不同的分离类型。根据Joinmap4.0软件中的CP作图模式,将亲本及F1后代的基因型划分为5种类型,分别是ab×cd、ef×eg、hk×hk、lm×ll、nn×np五种类型(图 2)。ab×cd型,在亲本中共出现4个等位基因,父本标记为a和b,母本记为c和d,在后代中有4种分离类型,分别是ac、ad、bc和bd,分离比例为1∶1∶1∶1 (图 2-1,引物DSSR69); ef×eg型,在亲本中共出现3个等位基因,其中有一条亲本共有带e,父本标记为e和f,母本 标记为e和g,在后代中有4种分离类型,分别是ef、eg、ee和fg,分离比例为1∶1∶1∶1 (图 2-2,引物DSSR193); hk×hk型,在亲本中共出现2个等位基因,父本标记为k和h,母本也标记为k和h,在后代中只有3种分离类型,分别是hh、hk和kk,分离比例为1∶2∶1 (图 2-3,引物DSSR79); nn×np型和lm×ll型,在亲本中都是出现2个等位基因,其中有一条为亲本共有带,nn×np型特有带出现在母本中 (图 2-4,引物Sm-ES012),lm×ll型特有带出现在父本中,在后代中有2种分离类型,分离比例为1∶1 (图 2-5,引物Sm- ES014)。
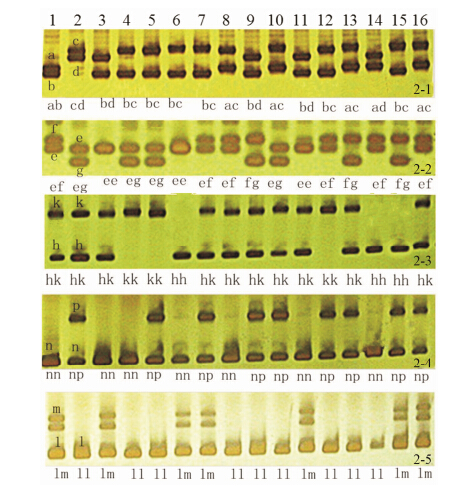 |
Figure 2 Segregation type of heterozygous loci of SSR primer in parents and F1 progenies. 2-1: DSSR69; 2-2: DSSR193; 2-3: DSSR79; 2-4: Sm-ES012; 2-5: Sm-ES014. 1: Male parent BH18; 2: Female parent ZH74; 3-16: F1 progenies |
将数据导入Joinmap 4.0作图软件中,选择LOD = 5.0作为合适的标记连锁群,在去除发生严重偏分离的6个标记后,对丹参F1代植株的94株作图群体的153个标记进行了遗传连锁分析,最终构建了一张93个标记的丹参遗传连锁图谱,其中包含了53个SSR标记、38个SRAP标记和2个ISSR标记,分属8个连锁群 (图 3)。遗传图谱覆盖丹参基因组全长400.1 cm,标记间平均图距4.3 cm,各连锁群长度在3.3~132 cm之间,其中第LG7和LG8连锁群最短,第LG1连锁群最长,不同连锁群上的标记数为2~23个(表 3)。根据遗传学公式计算丹参遗传连锁图谱的预期长度是474 cm,框架图谱覆盖率是84.4%。
 |
Figure 3 Genetic linkage map for Salvia miltiorrhiza Bge based on molecular marker. Numbers to the left are genetic distances in centiMorgans (cm) between adjacent markers |
|
|
Table 3 The distribution of markers and genetic distance of LGs |
Hemmat等[3]提出的“双假测交”构想,为解决树木遗传研究提供了可能。通过“拟测交”在种间或种内杂交所产生的F1代群体就会有很高频率的分离位点[31],这个方法也适用于基因高度杂合的植物,快捷高效的群体构建方法使其已应用于植物遗传研究工作中。本实验以两个性状差异较大的丹参品系为亲本,以其杂交得到的94株F1个体为作图群体构建了丹参首张遗传连锁图谱。多态性分析表明,引物多态性达到49%,表明分子水平上亲本间的差异较大,从F1后代扩增结果来看大多数引物的分离比例符合孟德尔遗传规律,表明群体大小符合构建遗传连锁图谱的基本要求。通常研究者根据研究的需要选择 作图群体的大小,一般用于主基因定位的图谱精度在20 cm即可[8]。本研究使用93个标记构建了丹参 8个连锁群,平均间距4.3 cm,已形成了丹参分子标记连锁框架图,这是目前首张丹参连锁图。该图将进一步用于数量性状的分子标记定位研究,为丹参目标性状基因定位、重要基因的克隆等奠定重要基础。
分子连锁群是植物染色体在分子水平上的反映,分子连锁群的数目应该和相应的配子染色体数目一致。丹参为二倍体,配子染色体数目X = 8,理论上应有8个连锁群,而本研究正好构建了丹参8个连锁群。目前很多植物遗传图谱的连锁群数量未能实现与物种的染色体数相对应,在对石斛 (X = 19) 的研究中,不同研究者构建了不同数目的连锁群,如Xue等[32]构建了铁皮石斛14个连锁群,赵红燕[33]构建了22个连锁群,甚至有研究者还构建了27个连锁群[34],出现这种现象的原因可能与分子标记的数量及其在基因组上的分布有关[35],也与研究者使用的作图软件和作图模式有关[36]。本研究构建了与丹参配子染色体数目一致的连锁群,可能与本研究所用的基于基因组开发的SSR有关,200对基因组SSR中30对上图且在连锁群中的分布相对较均匀,说明这些标记使用效率较高,另外本研究加大标记种类和数量,标记间互相补充,能较好地覆盖丹参的整个基因组。
偏分离是观察到的基因型比例偏离预期的孟德尔频率的分离方式,它不遵循分离规律,无法用传统的遗传理论和方法加以分析,但是偏分离在作图过程中又是普遍存在的。Cervera等[37]比较了14个种间构图群体,发现偏分离率平均有25.3%。本实验中得到的159个标记中有17个在后代出现偏分离,占总标记数的10.7%,属于正常的偏分离范围。偏分离会降低表观的交叉干扰从而导致位点排序错误[38],尽管偏分离标记会影响图谱距离以及标记的顺序,但如果将这些偏分离标记直接删除则可能造成大量遗传信息的缺失,降低基因组的覆盖率和忽略某些重要的QTL[39]。Murigneux等[40]的研究报道指出剔除严重偏分离的标记位点,并使用较高的LOD值可以降低偏分离位点的影响。本实验将6个严重偏分离的标记去除,并将LOD值提高至5.0,最终93个标记定位到遗传连锁图上,这样既提高了图谱的覆盖率,又能保证图谱的准确性。
本研究利用SSR、SRAP、ISSR 3种标记方法构建遗传连锁图谱,探索不同标记在丹参遗传图谱构建中的效果。使用不同的标记方法可以充分利用标记间的互补优势,EST-SSR标记对重复序列的着丝粒区域有较好的覆盖率; SRAP标记通过上下游引物的共同作用,可以与外显子、内含子和启动子区域进行特异性扩增; ISSR标记PCR扩增的是重复序列间DNA片段,正好与SSR引物在扩增区域上可以互补,同时基因组SSR虽开发难度较大,但其在染色体上分布较其他标记均匀,利用价值更大。因此,作者认为多种标记联合使用构建高密度、高饱和性的丹参分子遗传图谱更加有效。
| [1] | Chen SL, Sun YZ, Xu J, et al. Strategies of the study on Herb Genome Program [J]. Acta Pharm Sin (药学学报), 2010, 45: 807-812. |
| [2] | Li FL, Ma XJ. Advances in studies on molecular linkage map in medicinal plants [J]. Chin Tradit Herb Drugs (中草药), 2008, 39: 129-133. |
| [3] | Hemmat M, Weeden NF, Manganaris AG, et al. Molecular marker linkage for apple [J]. J Hered, 1994, 85: 4-11. |
| [4] | Gao LX, Liu N, Huang BH. Linkage maps of the genus Hedychium (Zingiberaceae) based on SRAP [J]. Acta Bot Yunnan (云南植物研究), 2009, 31: 317-325. |
| [5] | Zhan QQ, Sui C, Wei JH, et al. Construction of genetic linkage map of Bupleurum chinese DC. using ISSR and SSR markers. [J]. Acta Pharm Sin (药学学报), 2010, 45: 517-523. |
| [6] | Graham IA, Besser K, Blumer S, et al. The genetic map of Artemisia annua L. identifies loci affecting yield of the antimalarial drug artemisinin [J]. Science, 2010, 327: 328- 331. |
| [7] | Li AX, Liu QC, Wang QM, et al. Construction of molecular linkage maps using SRAP markers in sweet potato [J]. Acta Agron Sin (作物学报), 2010, 36: 1289-1295. |
| [8] | Chen DW, Chen LQ. The first intraspecific genetic linkage maps of wintersweet [Chimonanthus praecox (L.) Link] based on AFLP and ISSR markers [J]. Sci Hort, 2010, 124: 88-94. |
| [9] | Zhang F, Chen SM, Chen FD, et al. SRAP-based mapping and QTL detection for inflorescence-related traits in chrysanthemum (Dendranthema morifolium) [J]. Mol Breeding, 2011, 27: 11-23. |
| [10] | Quezada M, Pastina MM, Ravest G, et al. A first genetic map of Acca sellowiana based on ISSR, AFLP and SSR markers [J]. Sci Hort, 2014, 169: 138-146. |
| [11] | He D, Liu Y, Cai M, et al. The first genetic linkage map of crape myrtle (Lagerstroemia) based on amplification fragment length polymorphisms and simple sequence repeats markers [J]. Plant Breeding, 2014, 133: 138-144. |
| [12] | Chinese Pharmacopoeia Committee. Pharmacopeia of the People's Republic of China (中华人民共和国药典) [S]. Beijing: Chemical Industry Press, 2005: 52. |
| [13] | Cui GH, Wang XY, Feng H, et al. Molecular cloning and SNP analysis of a acetyl-CoA C-acetyltransferase gene (SmAACT) from Salvia miltiorrhiza [J]. Acta Pharm Sin (药学学报), 2010, 45: 785-790. |
| [14] | Cheng QQ, He YF, Li G, et al. Cloning and induced expression analysis of 4-hydroxy-3-methyl-but-2-enyl diphosphate reductase gene (SmHDR) of Salvia miltiorrhiza [J]. Acta Pharm Sin (药学学报), 2013, 48: 236-242. |
| [15] | Zhao SJ, Zhang JJ, Yang L, et al. Determination and biosynthesis of multiple salvianolic acids in hairy roots of Salvia miltiorrhiza [J]. Acta Pharm Sin (药学学报), 2011, 46: 1352-1356. |
| [16] | Yan Y, Zhao X, Zhang SC, et al. Roles of reactive oxygen species in Streptomyces pactum Act12-induced tanshinone production in Salvia miltiorrhiza hairy roots [J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 1985-1991. |
| [17] | Li Y, Sun C, Luo HM, et al. Transcriptome characterization for Salvia miltiorrhiza using 454 GS FLX [J]. Acta Pharm Sin (药学学报), 2010, 45: 524-529. |
| [18] | Gao W, Sun HX, Xiao HB, et al. Combining metabolomics and transcriptomics to characterize tanshinone biosynthesis in Salvia miltiorrhiza [J]. Genomics, 2014, 15: 73. |
| [19] | Song ZQ, Wang MM, Wang JH, et al. Comparison of genomic DNA extraction methods for Salvia miltiorrhiza [J]. Chin Wild Plant Resour (中国野生植物资源), 2008, 27: 54-57. |
| [20] | Song J. Foundation of EST-SSR and cpSSR Technic and Genetic Diversity Investigation of Cultivated Salvia miltiorrhiza Bge. (丹参EST-SSR和cpSSR分子标记的建立及其遗传特征分析) [D]. Chengdu: Chengdu University of Traditional Chinese Medicine. 2006. |
| [21] | Deng KJ. Analysis on Genetic Basis of Salvia miltiorrhiza in ZhongJiang (中江丹参道地性遗传基础分析) [D]. Chengdu: University of Electronic Science and Technology of China. 2011. |
| [22] | Wang XY, Zhou XL, Gao W, et al. New analysis of EST-SSR distribution and development of EST-SSR markers in Salvia miltiorrhiza [J]. China J Chin Mater Med (中国中药杂志), 2011, 36: 289-293. |
| [23] | Karaca M, Ince AG, Aydin A, et al. Cross-genera transferable e-microsatellite markers for 12 genera of the Lamiaceae family [J]. J Sci Food Agric, 2013, 93: 1869-1879. |
| [24] | Mishra RK, Gangadhar BH, Yu JW, et al. Development and characterization of EST based SSR markers in Madagascar periwinkle and their transferability in other medicinal plants [J]. Plant Omics J, 2011, 4: 154-162. |
| [25] | Radosavljević I, Satovic Z, Jakse J, et al. Development of new microsatellite markers for Salvia officinalis L. and its potential use in conservation-genetic studies of narrow endemic Salvia brachyodon Vandas [J]. Int J Mol Sci, 2012, 13, 12082- 12093. |
| [26] | Segarra-Moragues JG, Gleiser G. Isolation and characterization of di and tri nucleotide microsatellite loci in Rosmarinus officinalis (Lamiaceae), using enriched genomic libraries [J]. Conserv Genet, 2009, 10: 571-575. |
| [27] | Radosavljević I, Jakse J, Javornik B, et al. New microsatellite markers for Salvia officinalis (Lamiaceae) and cross-amplification in closely related species [J]. Am J Bot, 2011, 98: e316-e318. |
| [28] | Song ZQ. Genetic Diversity Study of Salvia miltiorrhiza Bge. (丹参种植资源的遗传多样性研究) [D]. Jinan: Shandong Agricultural University, 2008. |
| [29] | Postlethwait JH, Johnson SL, Midson CN, et al. A genetic linkage map for the zebrafish [J]. Science, 1994, 264: 699- 703. |
| [30] | Chakravarti A, Lasher LK, Reefer JE. A maximum likelihood for estimating genome length using genetic linkage data [J]. Genetics, 1991, 128: 175-182. |
| [31] | Gao Y, Han MY, Zhao CP, et al. Construction of AFLP molecular markers linkage map in nectarine [J]. J Fruit Sci (果树学报), 2008, 25: 478-484. |
| [32] | Xue DW, Feng SG, Zhao HY, et al. The linkage maps of Dendrobium species based on RAPD and SRAP markers [J]. J Genet Genomics, 2010, 37: 197-204. |
| [33] | Zhao HY. Construction Genetic Linkage Map of Dendrobium Based on Molecular Markers(石斛分子遗传图谱的构建) [D]. Hangzhou: Hangzhou Normal University, 2011. |
| [34] | Wang S. Genetic Diversity Evaluation and Genetic Linkage Map Construction of Dendrobium officinale (铁皮石斛种质资源遗传多样性评价及其遗传连锁图谱的构建) [D]. Hangzhou: Hangzhou Normal University, 2012. |
| [35] | Song J, Han MY, Zhao CP, et al. Contribution of a general genetic linkage map for peach using a ‘Qinguang2' × ‘Shuguang' F1 progeny by SSR markers [J]. Acta Bot Boreali-Occid Sin (西北植物学报), 2008, 28: 895-900. |
| [36] | Han ML, Liu YL, Zheng XY, et al. Construction of a genetic linkage map and QTL analysis for some fruit traits in pear [J]. J Fruit Sci (果树学报), 2010, 27: 496-503. |
| [37] | Cervera MT, Veronique S, Bart L, et al. Dense genetic linkage maps of three Populus species based on AFLP and microsatellite markers [J]. Geneties, 2001, 158: 787-809. |
| [38] | Yang JP, Rong TZ, Huang LJ, et al. Construction of the frame molecular linkage map in maize [J]. Acta Agron Sin (作物学报), 2004, 30: 82-87. |
| [39] | Luo L, Zhang YM, Xu S. A quantitative genetics model for viability selection [J]. Heredity, 2005, 94: 347-355. |
| [40] | Murigneux A, Baud S, Beckert M. Molecular and morphological evaluation of doubled-haploid lines in maize. 2. Comparison with single-seed descent lines [J]. Theor Appl Genet, 1993, 87: 278-287. |
 2015, Vol. 50
2015, Vol. 50


