2. 福建农林大学中药材GAP研究所, 福建 福州 350002
2. Institute of Chinese Crude Drugs GAP, Fujian Agriculture and Forestry University, Fuzhou 350002, China
地黄(Rehmannia glutinosa L.) 为玄参科多年生草本植物,以块根入药,其道地产区在河南省焦作市的温县、武陟、博爱等地 (古怀庆府)[1],为著名的“四大怀药”之一。然而,地黄生产中存在非常严重的连作障碍问题,连作地黄表现为植株生长不良甚至死亡,地上植株小,块根不能正常膨大[2],生物量降低,品质变差[3]。课题组近期利用Solexa高通量 测序技术,在根和叶部分别筛选出2 817个和1 954个响应连作的差异表达的cDNA片段,其中一个293 bp的片段在连作地黄叶片中呈下调表达,GO注释 属于扩展蛋白基因,与拟南芥 (Arabidopsis thaliana) AtEXPA10的同源性达83%,推测与连作条件下的地黄发育进程严重受阻有关。
扩展蛋白最早从黄瓜 (Cucumic sativus) 中分离出来,能够在酸性环境下诱导细胞壁的伸展[4]。目前已经发现其在双子叶和单子叶植物中广泛存在,能够诱导植物细胞壁长期的、不可逆伸展,提高植物细胞壁的胁迫松弛能力[5]。扩展蛋白调控植物根系生 长[6]、根毛发育[7]、叶片发育[8]、种子萌发[9]、果实成熟[10]、器官脱落[11]、气孔运动[12]、纤维强度[13]、块根发育[14]等不同器官、组织的发育进程。而且,扩展蛋白基因的表达也与植物的抗性有关。如过量表达小麦 (Triticum aestivum) 扩展蛋白基因TaEXPB23能够提高烟草的抗旱性[15]; 异位表达玫瑰 (Rosa hybrida) RhEXPA4能够提高拟南芥的抗旱、耐盐性[16],水稻(Oryza sativa) 对白叶枯病抗性的增强与OsEXPA5、OsEXPA10、OsEXPA7的下调表达有关[17]。
迄今为止,地黄功能基因克隆的报道较少,如 3-酮酯酰CoA-硫解酶基因RghKAT[18]和Aux-IAA家 族基因RgIAA1等[19]。地黃扩展蛋白的研究更少,直到2012年才有报道克隆出其中一个扩展蛋白基因RgEXPA1[20]。本文克隆了一个新的地黄扩展蛋白基因RgEXPA10,并分析了其在地黄不同组织及在连作、盐和渍水胁迫下的表达特性,为地黄连作障碍形成的分子机制及地黄产量形成的物质基础研究奠定基础。
材料与方法 实验材料和培养条件以怀地黄优良品种“温85-5”的叶片为材料,经河南省中医药研究院张留记研究员鉴定为Rehmannia glutinosa L.。3种胁迫处理的地黄植株均在智能光照培养箱 (LRH-300-G,广东医疗器械厂) 里生长,温度为 (25 ± 1) ℃,光照强度为2 000~4 000 lx,每天14 h光照。
RNA提取与反转录从“温85-5”叶片中提 取总RNA。每个样品约50 mg,以Trizol提取液 (Invitrogen,上海) 提取地黄总RNA,提取方法参考说明书进行。反转录采用反转录试剂盒 (Invitrogen,上海),cDNA第一链合成: 约1 μg的RNA、0.5 μg 的oligo(dT)12-18引物、0.5 μL RNase抑制剂、0.5 μL M-MLV反转录酶,终体积20 μL。反应体系为: 37 ℃ 50 min,70 ℃ 15 min。
基因克隆以拟南芥扩展蛋白基因AtEXPA10 (注册号: NM_001084130.1) cDNA为查询序列,在 本课题组前期测定的转录组数据库中进行同源比对,应用DNAMAN和DNASTAR离线软件对获得的 EST序列进行冗余序列剔除和拼接。根据cDNA序列设计特异引物RgEXPA10-F和RgEXPA10-R (表 1),以地黄“温85-5”的cDNA为模板,用PrimeSTAR® HS DNA聚合酶 (TaKaRa,大连) 进行PCR扩增,克隆RgEXPA10的编码序列 (CDS)。
|
|
Table 1 Oligo primer pairs used for cloning and detection of expression of RgEXPA10 |
PCR扩增体系: 5×PrimeSTAR® Buffer (Mg2+ plus) 10 μL、dNTP Mixture (各2.5 mmol·L-1) 4 μL、Forward Primer (10 μmol·L-1) 1 μL、Reverse Primer (10 μmol·L-1) 1 μL、模板cDNA 0.5 μL、PrimeSTAR® HS DNA Polymerase (2.5 U·μL-1) 0.5 μL、灭菌蒸馏水33 μL,总体系50 μL。反应条件: 98 ℃ 10 s,55 ℃ 5 s,72 ℃ 2 min,35个循环; 72 ℃ 10 min。
PCR产物经过加A反应,纯化回收后连接到载体pMD18-T vector (TaKaRa,大连) 上,送深圳华大基因研究院测序。
基因组序列分析以CTAB法提取地黄叶片基因组DNA。应用引物RgEXPA10-F和RgEXPA10-R,以PrimeSTAR HS DNA聚合酶扩增RgEXPA1的基因组序列,PCR产物克隆到pMD18-T vector (TaKaRa,大连) 上,测序确认基因组序列。应用DNAMAN软件进行cDNA序列和基因组序列比对,根据内含子的剪接位点特征,确定RgEXPA10内含子的大小和剪接位点。
氨基酸序列分析同源搜索以NCBI在线软件BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) 进行。氨基酸多序列联配用MAFFT (http://www.ebi.ac.uk/ Tools/msa/mafft/) 在线软件和Genedoc离线软件,进化分析采用MEGA 6.0软件打开并以Neighbor-Joining算法进行Bootstrapping分析,构建系统发生树。
胁迫处理所有实验材料都种植在直径20.8 cm、高21.5 cm的聚乙烯塑料盆里,在光照培养箱里生长。实验设置3种胁迫处理: ① 连作。以至少10年未种植过地黄的土壤为头茬土,以上年种植过地黄的土壤为连作土。在头茬和连作处理的土壤中种植地黄块根,出苗后70天取叶片用于基因表达检测。② 盐胁迫。以2% 的NaCl水溶液浇灌头茬土盆栽种植45天的无菌地黄苗,12 h后取叶片用于基因表达分析。③ 渍水胁迫。自来水浇灌头茬土盆栽种植45天的无菌苗,维持水层高于土Table 2">表 2 cm左右,连续处理3天,取叶片用于表达分析。每个处理重复3次。
基因表达检测时空表达模式分析: 提取地黄“温85-5”膨大初期块根、不定根、茎、幼嫩叶、展开叶、衰老叶和出苗后30、60、90、120和150天块根的总RNA,用PrimeScript™ RT reagent Kit with gDNA Eraser (TaKaRa,大连) 反转录成cDNA模板,根据候选RgEXPA10基因CDS序列信息设计特异引物 (Table 1">表 1),以RgTIP41为内参基因,实时荧光定量PCR (qRT-PCR) 方法检测基因的表达量。胁迫下表达水 平检测: 提取不同胁迫处理叶片的总RNA用于qRT- PCR检测。
所用PCR仪为BIO-RAD iQ5 (伯乐,上海)。实时荧光定量分析试剂盒为SYBR® Premix Ex Taq™ II (Tli RNaseH Plus) (TaKaRa,大连)。PCR扩增体系: SYBR® Premix Ex Taq 12.5 μL、Forward Primer (10 μmol·L-1) 1 μL、Reverse Primer (10 μmol·L-1) 1 μL、cDNA模板2.0 μL、ddH2O 8.5 μL,共计25 μL。反应条件: 95 ℃ 30 s; 95 ℃ 5 s,60 ℃ 30 s,40个循环。根据BIO-RAD iQ5软件生成的Ct (cycle threshold) 值,以2-ΔΔCt计算RgEXPA10基因的相对表达量[21]。
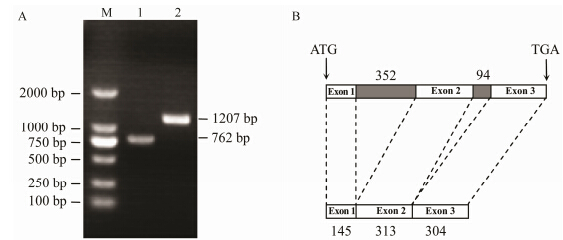
结果 1 RgEXPA10的克隆在转录组EST数据库中,通过同源搜索、拼接延伸,获得一条包含972 bp的cDNA序列,应用NCBI在线分析工具ORF Finder (http://www.ncbi.nlm.nih. gov/gorf/gorf.html) 预测发现其包含一个762 bp的 开放阅读框 (ORF),编码253个氨基酸,5'非编码序列 (UTR) 有1个碱基,3'UTR有209个碱基,命名 为RgEXPA10。cDNA序列已经提交到NCBI,注册 号为KF011918。根据其编码区序列设计特异引物,以地黄叶片cDNA为模板扩增出一条762 bp的条带 (图 1A),测序发现有一个碱基替换,与原拼接序列相似性达到99.87%。为了明确RgEXPA10的剪接位点及内含子大小,以地黄叶片基因组DNA为模板扩增出一条1 200 bp左右的条带 (图 1A),测序确认长度为1 207 bp。比对分析发现RgEXPA10基因组DNA序列包含2个内含子和3个外显子,内含子的长度分别为352 bp和94 bp (图 1B)。基因组扩增序列已经提到NCBI,注册号为KF011919。
|
Figure 1 Electrophoreses of PCR amplified fragment and transcription splicing of RgEXPA10 gene. A: Amplification of RgEXPA10 with cDNA template and DNA template. 1: Amplification product with cDNA template; 2: Amplification product with DNA template; M,DL2000 DNA marker. B: Transcription splicing of RgEXPA10 gene |
应用ExPASy(http://web.expasy.org/protparam/) 计算发现RgEXPA10蛋白的分子量为27.16 kD,等电点pI 9.08。以NCBI在线软件BLASTP对RgEXPA10与以前报道的RgEXPA1 (ACN94264.1) 进行序列相似性比较,并应用SMART分析其结构域,结果(图 2A) 表明两个蛋白氨基酸序列一致性为72%,结构域保守程度很高。从RgEXPA10第66 aa到151 aa有一个DPBB_1结构域,GO分析表明该结构域具有DNA依赖性,具有调控转录的功能; 162 aa到239 aa存在一个Pollen_allerg_1结构域,与植物细胞膨大有关。

|
Figure 2 Characteristics of deduced amino acid sequence of RgEXPA10 protein. A: Alignment of the deduced amino acid sequences of RgEXPA10 and RgEXPA1. Eight conserved Cys residues important in the secondary structure of the catalytic domain are indicated by crosses,and four Trp residues important in the structure of the carbohydrate-binding module are indicated by diamonds. B: Prediction of the transmembrane domain of RgEXPA10 |
在其N端含有8个保守的半胱氨酸 (Cys) 残基,C端含有4个保守的色氨酸 (Trp) 残基。说明RgEXPA10具有典型的扩展蛋白特征。
用SignalP 4.1 (http://www.cbs.dtu.dk/services/ SignalP/) 检测到第1 aa到26 aa的一段信号肽,序列为MAIQGVSMAALALFSFFGFFLRATYG。进一步通过http://expasy.org中的在线工具TMHMM 2.0对RgEXPA10进行跨膜结构域预测,结果 (图 2B) 表明,在RgEXPA10氨基酸中存在1个跨膜结构域,在第 6 aa至第21 aa之间,由19个氨基酸残基组成,是膜中的蛋白质与膜脂结合的主要部位。这与核定位信号的预测结果一致,说明RgEXPA10是一个跨膜蛋白。
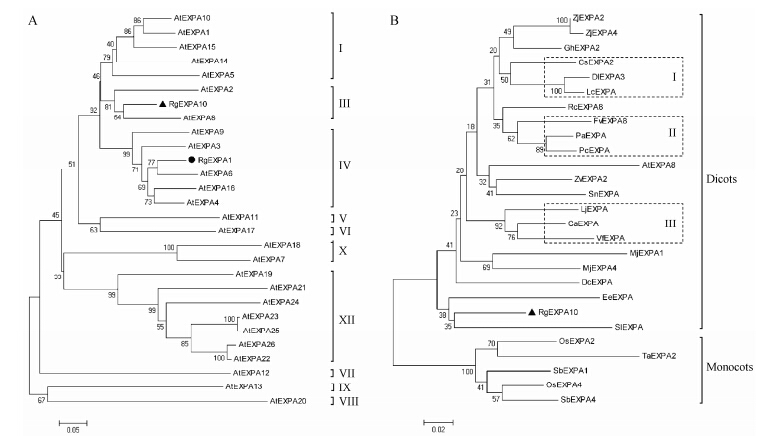
3 系统进化分析通过在拟南芥信息数据库 (http://www.arabidopsis. org/index.jsp) 中检索,获得拟南芥26个α扩展蛋白的氨基酸序列,通过与RgEXPA10和以前报道的RgEXPA1进行多序列联配和进化分析,根据Sampedro等[22]的分类方法,结果 (图 3A) 发现28个蛋白可分为10个类群,RgEXPA10属于第III类群,与AtEXPA8 (NP_181593.1) 亲缘关系最近,序列相似程度最高,一致性达到85.2%,其次为AtEXPA2 (NP_196148.1),一致性为77.6%。
|
Figure 3 Phylogenetic tree of RgEXPA10 and its homologous proteins were generated by the neighbor-joining method using software of MEGA 6.0. Bootstrap values (shown at the corresponding nodes) were obtained from 1000 replicates and are reported as percentages. Rg: Rehmannia glutinosa; At: Arabidopsis thaliana; Zj: Ziziphus jujube; Gh: Gossypium hirsutum; Cs: Citrus sinensis; Dl: Dimocarpus longan; Lc: Litchi chinensis; Rc: Ricinus communis; Fv: Fragaria vesca; Pa: Prunus armeniaca; Pc: Pyrus communis; Zv: Zinnia violacea; Sn: Sambucus nigra; Lj: Lotus japonicus; Ca: Cicer arietinum; Vf: Vicia faba; Mj: Mirabilis jalapa; Dc: Dianthus caryophyllus; Ee: Eustoma exaltatum; Sl: Solanum lycopersicum; Os: Oryza sativa; Ta: Triticum aestivum; Sb: Sorghum bicolor |
利用RgEXPA10蛋白的氨基酸序列在NCBI (http://blast.ncbi.nlm.nih.gov/) 上进行BLASTP同源搜索,获得26个扩展蛋白,分属22个植物物种。通过与RgEXPA10进行多序列联配和进化分析,结果(图 3B) 表明,27个扩展蛋白可初步分为两个大的类群,小的类群包含5个蛋白,均来源于单子叶植物水稻、小麦和高粱 (Sorghum bicolor); 大的类群包含22个蛋白,分属于20个物种,均为双子叶植物。双子 叶植物中,枣 (Ziziphus jujuba) 扩展蛋白ZjEXPA2 (ACK43222.1) 和ZjEXPA4 (AFK24457.1) 属于同一个分支,紫茉莉 (Mirabilis jalapa) 扩展蛋白MjEXPA1 (AAL87025.1) 和MjEXPA4 (AAL87023.1) 属于同 一个分支。而且,还发现一些亲缘关系较近的物种均属于同一小的类群。如甜橙 (Citrus sinensis)、龙眼 (Dimocarpus longan) 和荔枝 (Litchi chinensis) 均属于无患子目植物,其扩展蛋白CsEXPA2 (ACN87961.1)、DlEXPA3 (ACA05166.1) 和LcEXPA(ABO32366.1) 属于同一类群I; 草莓 (Fragaria vesca)、杏树 (Prunus armeniaca) 和西洋梨 (Pyrus communis) 均属于蔷 薇科植物,其扩展蛋白FvEXPA8 (XP_004307732.1)、PaEXPA(AAC33529.1) 和PcEXPA(BAC67190.1) 属于同一类群II; 百脉根 (Lotus japonicus)、鹰嘴豆 (Cicer arietinum) 和蚕豆(Vicia faba) 均属于豆科 植物,其扩展蛋白LjEXPA (AFK41150.1) CaEXPA (CAC19184.1) 和VfEXPA (ABM66452.1) 属于同一类群III。RgEXPA10属于双子叶植物,与番茄 (Solanum lycopersicum) 扩展蛋白SlEXPA (NP_001234165.1) 亲缘关系最近,序列相似程度最高,一致性达到87.1%,其次为洋桔梗 (Eustoma exaltatum) 扩展蛋白EeEXPA (BAN09211.1),一致性为84.0%。
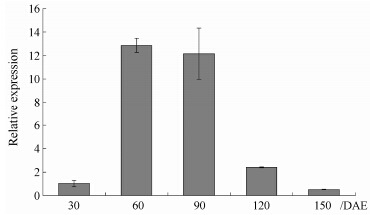
4 RgEXPA10的时空表达特性qRT-PCR结果表明,RgEXPA10在地黄幼嫩叶片里表达强度最大,其次为处于膨大期的块根,在衰老的叶片中表达最弱,几乎检测不到其表达量 (图 4)。说明其在幼嫩的组织和快速生长的组织里表达较强。而且,从图 4中可以看出,随着叶片的伸展,RgEXPA10表达强度迅速降低,说明RgEXPA10可能在叶片伸展中发挥重要的作用。RgEXPA10在块根中的表达量较不定根中的表达量高1倍左右,表明其在块根膨大过程中也很重要。通过检测块根不同发育阶段RgEXPA10的表达量,结果(图 5) 显示在出苗后60天的块根中表达量快速增加,90天略有降低,之后迅速降低,这种表达模式说明RgEXPA10在块根膨大的初期表达量较高。

|
Figure 4 Expression of RgEXPA10 gene in different tissues of R. glutinosa. TR: Tuberous root; AR: Adventitious root: S: Stem; YL: Young leaf; UL: Unfolded leaf; SL: Senescing leaf. n = 3 |
|
Figure 5 Expression of RgEXPA10 gene in different stages of tuberous of R. glutinosa. DAE: Days after emergence. n = 3 |
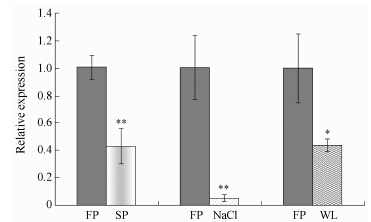
为了探讨RgEXPA10在逆境胁迫下与地黄发育的关系,以qRT-PCR检测了其在连作、2% NaCl处理和渍水处理等3种逆境下的表达量。结果 (图 6) 表明,在连作、盐和渍水胁迫下,地黄叶片内RgEXPA10的表达量均显著降低,且连作和盐胁迫下的表达降低达到了极显著水平。这与3种逆境胁迫下地黄的生长发育进程受阻的表型是一致的。
|
Figure 6 Expression ofRgEXPA10 gene of R. glutinosa under three stresses. P < 0.05,**P < 0.01 vs FP. FP: The first year crop plants; SP: The second year crop plants; WL: Waterlogging |
扩展蛋白基因在植物中具有多个成员,编码的蛋白属于一个大的扩展蛋白家族,目前共发现4个亚家族,分别为α-expansin (EXPA)、β-expansin (EXPB)、expansin-like A (EXLA) 和expansin-like B (EXLB)[22],其中EXPA和EXPB为主要的扩展蛋白。如拟南芥中有26个EXPA基因和6个EXPB基因,毛果杨 (Populus tomentosa) 中有27个EXPA基因和2个EXPB基因,水稻中有34个EXPA基因和19个EXPB基因[22]。扩展蛋白具有相对保守的内含子模式,多数EXPA基因含有两个内含子,个别EXPA基因只含有1个内含子。本研究克隆的地黄RgEXPA10基因含有两个内含子 (图 1B),与RgEXPA1[20]一致。扩展蛋白结构保守性很高,N端一般有1段22~25 aa的信号肽以引导其通过内质网,还包括位于中部的催化区和C端的纤维素绑定区。RgEXPA10含有26个aa的信号肽,中部具有富含8个半胱氨酸 (C) 的DPBB_1结构域,C端具有富含色氨酸 (W) 的Pollen_allerg_1结构域,为典型的扩展蛋白。与RgEXPA1和26个拟南芥扩展蛋白构建进化树,发现RgEXPA10与AtEXPA8亲缘关系最近,属于类群III; 而RgEXPA1与AtEXPA6 (AAM62987.1) 亲缘关系最近,属于类群IV,与臧亚超等[20]的结果一致。与22个植物物种的26个扩展蛋白构建进化树,结果 (图 3B) 双子叶植物扩展蛋白与单子叶植物扩展蛋白分别属于两个大的类群,说明扩展蛋白的进化符合物种进化规律,双子叶植物与单子叶植物扩展蛋白之间的同源性相对较低。
扩展蛋白以酶催化的方式使细胞壁组分松弛、细胞伸展,不同的扩展蛋白基因在植物体内具有不同的表达模式,且与细胞生长和组织分化一致[23]。如调控根毛的分化和生长的AtEXPA7和AtEXPA18在拟南芥根毛细胞中特异表达[7, 23],AtEXPA5、AtEXPA22分别在拟南芥叶片、胚轴中特异表达[8],葡萄 (Vitis vinifera) 扩展蛋白基因VvEXPA8、VvEXPA9、VvEXPA12分别在根、种子、花药中特异表达[24]等。臧亚超等[20]分析发现RgEXPA1、RgEXPA3、RgEXPA7在地黄膨大期的块根中表达较强,而RgEXPA6在地黄花中表达较强。本研究发现RgEXPA10在地黄块根、茎、叶里都有表达,在幼嫩的叶片中表达最强,随着叶片的发育其表达量下降,在膨大期块根中的表达较不定根中的表达量高,说明其在地黄的叶片伸展及块根发育中发挥功能。在地黄块根发育的过程中,RgEXPA10的表达量先增加,之后逐渐降低,这种表达模式与控制矮牵牛 (Petunia hybrida) 花瓣发育的PhEXP1[25]在花瓣发育过程中的表达模式类似。RgEXPA10在出苗后60天和出苗后90天的地黄块根中表达量较其他发育阶段高,而此时为块根拉线期和膨大初期,推测RgEXPA10在块根发育的早期作用较大。
常有一些扩展蛋白基因在响应逆境胁迫时表现差异表达以适应变化了的环境。如在干旱胁迫下,玉米 (Zea mays) 生长发育相关扩展蛋白基因表达量降低[26]。淹水条件下,OsEXPA2、OsEXPA4的上调表达促使水稻植株快速生长到水面以上[27]; 而干旱胁迫下,OsEXPA2表达量上调得以维持水稻根尖细胞壁 的伸展[28]。淹水条件下,耐水植物Rumex palustris 中RpEXPA1表达量上调,而在不耐水植物R. acetosa中并没有发现其表达量增加[29]。高盐浓度下,抗盐 玉米品种SR03茎的继续伸长被认为是ZmEXPB2、ZmEXPB6和ZmEXPB8表达上调的结果,而在盐敏感玉米品种Lector中这3个基因则表达下调[30]。在一定范围内,pH值降低可提高扩展蛋白的活性[4]。长期的连作会导致土壤pH值的小幅降低[31],可能诱导扩展蛋白的积累。但本研究发现连作条件下地黄叶片 的RgEXPA10的表达量显著降低,可能与引起地黄 连作障碍的化感自毒物质的存在有关。地黄为忌连 作和淹水敏感植物,本研究发现叶片内的RgEXPA10在高盐、淹水和连作条件下表达显均著下调,这在一定程度上解释了在3种环境胁迫下地黄生长受抑制的现象,同时有助于盐害、渍害和连作障碍形成机制的研究。本研究结果为进一步研究RgEXPA10在地黄生长发育及逆境胁迫中的分子功能奠定了基础,同时为地黄品种改良提供了新的思路。
| [1] | Wu L, Wang H, Zhang Z, et al. Comparative metaproteo-mic analysis on consecutively Rehmannia gluti-nosa-monocultured rhizosphere soil [J]. PLoS One, 2011, 6: e20611. |
| [2] | Yin WJ, Du JF, Li J, et al. Effects of continuous cropping obstacle on growth of Rehmannia glutinosa [J]. China J Chin Mater Med (中国中药杂志), 2009, 34: 18-21. |
| [3] | Zhang ZY, Li GL, Niu MM, et al. Responses of physio-logical ecology and quality evaluation of Rehmannia gltinosa in continuous cropping [J]. China J Chin Mater Med (中国中药杂志), 2011, 36: 6-9. |
| [4] | McQueen-Mason S, Durachko DM, Cosgrove DJ. Two endogenous proteins that induce cell wall extension in plants [J]. Plant Cell, 1992, 4: 1425-1433. |
| [5] | Zhao MR, Li YC, Wang W. Research progress on relation-ship between expansin and plant resistance [J]. Plant Physiol J (植物生理学报), 2012, 48: 637-642. |
| [6] | Kwasniewski M, Szarejko I. Molecular cloning and characterization of β-expansin gene related to root hair formation in barley [J]. Plant Physiol, 2006, 141: 1149-1158. |
| [7] | Lin C, Choi HS, Cho HT. Root hair-specific expansin A7 is required for root hair elongation in Arabidopsis [J]. Mol Cells, 2011, 31: 393-397. |
| [8] | Goh H, Sloan J, Dorca-Fornell C, et al. Inducible repres-sion of multiple expansin genes leads to growth suppression during leaf development [J]. Plant Physiol, 2012, 159: 1759- 1770. |
| [9] | Arana MV, de Miguel LC, Sánchez RA. A phytochrome-dependent embryonic factor modulates gibberellin re-sponses in the embryo and micropylar endosperm of Datura ferox seeds [J]. Planta, 2006, 223: 847-857. |
| [10] | Yu K, Xu Q, Da X, et al. Transcriptome changes during fruit development and ripening of sweet orange (Citrus sinensis) [J]. BMC Genomics, 2012, 13: 10. |
| [11] | Belfield EJ, Ruperti B, Roberts JA, et al. Changes in expansin activity and gene expression during ethylene-promoted leaflet abscission in Sambucus nigra [J]. J Exp Bot, 2005, 56: 817- 823. |
| [12] | Zhang XQ, Wei PC, Xiong YM, et al. Overexpression of the Arabidopsis α-expansin gene AtEXPA1 accelerates stomatal opening by decreasing the volumetric elastic modulus [J]. Plant Cell Rep, 2011, 30: 27-36. |
| [13] | Bajwa KS, Shahid AA, Rao AQ, et al. Expression of Calotropis procera expansin gene CpEXPA3 enhances cotton fibre strength [J]. Aust J Crop Sci, 2013, 7: 206-212. |
| [14] | Noh SA, Lee HS, Kim YS, et al. Down-regulation of the IbEXP1 gene enhanced storage root development in sweet potato [J]. J Exp Bot, 2013, 64: 129-142. |
| [15] | Li F, Xing S, Guo Q, et al. Drought tolerance through over-expression of the expansin gene TaEXPB23 in transgenic tobacco [J]. J Plant Physiol, 2011, 168: 960-966. |
| [16] | Lü P, Kang M, Jiang X, et al. RhEXPA4, a rose expansin gene, modulates leaf growth and confers drought and salt tolerance to Arabidopsis [J]. Planta, 2013, 237:1547-1559. |
| [17] | Ding X, Cao Y, Huang L, et al. Activation of the indole-3-acetic acid-amido synthetase GH3-8 suppresses expansin expression and promotes salicylate-and jasmonate-independent basal immunity in rice [J]. Plant Cell, 2008, 20: 228-240. |
| [18] | Zhou YQ, Zhang YH, Zhang Y, et al. Gene cloning, fea-tures of sequence, and analysis on temporal and spatial expression of Rehmannia glutinosa f. hueichingensis 3-ketoacyl CoA-thiolase [J]. Chin Tradit Herb Drugs (中草药), 2013, 44: 76-84. |
| [19] | Wang FQ, Tian YH, Li MJ, et al. Molecular cloning and expression analysis of an Aux/IAA gene (RgIAA1) from Rehmannia glutinosa [J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 4033-4039. |
| [20] | Zang YC, Sun P, Yang TX, et al. Cloning and expression analysis of RgExpa1 in Rehmannia glutinosa [J]. Biotechnol Bull (生物技术通报), 2012, 4: 69-73. |
| [21] | Zhao MM, Zhang G, Zhang DW, et al. Molecular cloning and characterization of S-adenosyl-L-methionine decarboxylase gene (DoSAMDC1) in Dendrobium officinale [J]. Acta Pharm Sin (药学学报), 2013, 48: 946-952. |
| [22] | Sampedro J, Cosgrove DJ. The expansin superfamily [J]. Genome Biol, 2005, 6: 242. |
| [23] | Cho HT, Cosgrove DJ. Regulation of root hair initiation and expansin gene expression in Arabidopsis [J]. Plant Cell, 2002, 14: 3237-3253. |
| [24] | Dal Santo S, Vannozzi A, Tornielli GB, et al. Genome-wide analysis of the expansin gene superfamily reveals grape-vine-specific structural and functional characteristics [J]. PLoS One, 2013, 8: e62206. |
| [25] | Zenoni S, Reale L, Tornielli GB, et al. Downregulation of the Petunia hybrida alpha-expansin gene PhEXP1 reduces the amount of crystalline cellulose in cell walls and leads to phenotypic changes in petal limbs [J]. Plant Cell, 2004, 16: 295-308. |
| [26] | Muller B, Bourdais G, Reidy B, et al. Association of spe-cific expansins with growth in maize leaves is maintained under environmental, genetic, and developmental sources of variation [J]. Plant Physiol, 2007, 143: 278-290. |
| [27] | Huang J, Takano T, Akita S. Expression of α-expansin genes in young seedlings of rice (Oryza sativa L.) [J]. Planta, 2000, 211: 467-473. |
| [28] | Yang L, Zheng B, Mao C, et al. Analysis of transcripts that are differentially expressed in three sectors of the rice root system under water deficit [J]. Mol Genet Genomics, 2004, 272: 433-442. |
| [29] | Vriezen WH, De Graaf B, Mariani C, et al. Submergence induces expansin gene expression in flooding-tolerant Rumex palustris and not in flooding-intolerant R. acetosa [J]. Planta, 2000, 210: 956-963. |
| [30] | Geilfus CM, Zörb C, Mühling KH. Salt stress differen-tially affects growth-mediating β-expansins in resistant and sensitive maize (Zea mays L.) [J]. Plant Physiol Biochem, 2010, 48: 993-998. |
| [31] | Gu ZC, Xi XY, Liu HJ, et al. Dynamic influence of continuous cropping of flue-cured tobacco on the amount of microbe and enzyme activities in the rhizosphere soil [J]. J Henan Agric Univ (河南农业大学学报), 2011, 45: 508-513. |
 2015, Vol. 50
2015, Vol. 50


