常用中药百合为百合科植物卷丹 (Lilium Lancifolium)、百合 (Lilium brownie) 或细叶百合 (Lilium pumilum) 的肉质鳞叶。《中国药典》(2010版) 记载百合性甘、味寒,归心、肺经,具有养阴润肺、清心安神的功能。百合是集药用、食用和观赏为一体的经济植物,临床上主要用于治疗燥咳、肺结核、精神分裂症、更年期综合征、糖尿病等,应用广泛[1]。
据《中国植物志》记载,百合属植物共100余 种,在中国主要分布约有39种,包括一些中国特有种,如毛百合 (Lilium dauricum)、新疆百合 (Lilium martagon)、台湾百合 (Lilium formosanum)、野百合 (Crotalaria sessiliflora)、岷江百合 (Lilium regale)、紫花百合 (Lilium souliei) 等。百合属中很多植物均为保健产品的重要原料,但百合属中丰富的种质资源给其鉴定及应用带来困难。由于百合属各物种形态特征相近,且存在很多变种,利用传统的形态、显微等特征很难准确快速的对其进行鉴别。通过性状、形态特征鉴别,应用的范围较小,而且鉴别需要一定的专业知识以及主观判断,没有统一的标准[2, 3]。由于百合属中存在的活性成分的种类较多,运用化学方法进行鉴定的结果不能准确直观的反应药材真伪[4]。早期的一些分子标记技术,如RAPD等,对DNA质量要求较高,结果不稳定,差异性主要表现在种内变异[5, 6]。DNA条形码作为一种新的分子标记技术,操作简单,所需材料少,结果稳定可重复,通用性强,可以迅速、准确鉴定物种,且应用已十分广泛[7, 8, 9, 10, 11, 12]。利用条形码技术对百合属植物的鉴别研究较少,且没有在整个属的水平进行系统的鉴别分析[13]。本文通过实验,并结合GenBank数据库数据,分析ITS2、ITS、psbA-trnH、matK、rbcL五条常用条形码序列对百合属植物的鉴别能力,以筛选能鉴别百合属植物的优选序列及为百合属植物的分子鉴别提供依据。
材料与方法 材料实验所用材料百合、卷丹与细叶百合采于湖南、陕西、北京等地 (详见表 1)。经中国医学科学院药用植物研究所林余霖教授鉴定分别为百合 (Lilium brownie)、卷丹 (Lilium Lancifolium)、细叶百合 (Lilium pumilum),凭证标本保存于中国医学科学院药用植物研究所。实验所用试剂主要有天根植物 基因组DNA提取试剂盒; 2×Taq Mix; ddH2O等。
|
|
Table 1 The source of samples for Lilium brownii and adulterants |
取干燥植物样品30 mg,球磨仪 (Retsch MM400,Germany) 1 800 r·min-1研磨2 min (加入两粒灭菌小钢珠),使用天根植物基因组DNA提取试剂盒 (Tiangen Biotech Co.,China) 提取样 品总DNA。具体步骤如下,研磨后样品加入2.0 EP管中,加入缓冲液GP1 (65 ℃提前预热) 700 μL、巯基乙醇1 μL,65 ℃水浴1 h; 水浴后加入700 μL氯仿-异戊醇 (24∶1) 混合液,混匀后12 000 r·min-1离心 5 min; 取上清液,加等体积异丙醇,混匀15~20 min; 分装过吸附柱,12 000 r·min-1离心40 s; 弃废液,加缓冲液GD 500 μL,12 000 r·min-1离心40 s; 弃废液,加漂洗液PW 700 μL,12 000 r·min-1离心40 s; 弃废液,加漂洗液PW 500 μL,12 000 r·min-1离心40 s; 弃废液,空管离心2 min,室温风干后加100 μL ddH2O洗脱DNA,12000 r·min-1离心2 min,即得样品总DNA模板,-20 ℃保存备用。
PCR扩增、电泳检测PCR体系总体积为25 μL,其中包含: 2×Taq PCR Mix 12.5 μL、DNA模板2 μL、ddH2O 8.5 μL、引物ITS 6F/4R各1 μL。引物序列 及PCR反应条件见表 2。扩增结束后以1% 琼脂糖凝胶电泳检测,凝胶成像分析系统分析。
|
|
Table 2 The primer sequence and PCR reaction condition |
PCR产物直 接使用ABI3730XL测序仪 (Applied Biosystems Co.,USA) 进行双向测序。序列拼接软件CodonCode Aligner (V3.7.1) 对测序峰图文件进行拼接,去除引物区及低质量序列; 使用Clustal (MEGA 5.0) 对拼接后序列进行比对分析; 同时本研究从GenBank数据库中搜集并下载百合属ITS2、ITS、psbA-trnH、rbcL、matK序列717条(表 3),整合处理后结合样品序列,运用MEGA 5.0建立NJ树,分析百合及其伪品亲缘关系; 采用Perl语言统计不同序列种内、种间遗传差异、barcoding gap; 运用相似性搜索算法 (BLAST1) 和最近距离法 (K2P nearest distance) 考察各序列的鉴定成功率[14]。
|
|
Table 3 Accession numbers of GenBank database |
各序列数量、GC含量、遗传距离、鉴定效率等信息见表 4。由数据可以看出,ITS2序列数据库数量较少,只有2条,加上从ITS序列中注释提取出的ITS2序列共计253条。ITS序列数量最多,为275条(包括实验数据),rbcL序列数量最少,为105条。各序列GC含量差异较大,ITS2序列最高,平均含量 达到64.85%,psbA-trnH与matK序列GC含量较低; 平均遗传距离psbA-trnH序列最大,为0.311 8,其次为ITS2,为0.056 3,rbcL序列最小。BLAST1方法分析各序列的鉴定效率,ITS序列比其他4条均较高,为58.91%,rbcL序列最小,为7.41%; 最近距离法分析结果可看出,ITS序列比其他序列均较高,为62.55%,rbcL序列最低,为12.96%。综合分析,ITS序列可作为鉴定百合属的优选序列。
|
|
Table 4 Sequence information and identification efficiency |
种内种间差异是分析条形码序列的重要指标。合适的条形码序列具有较大的种间变异、较小的种内 变异,以便准确鉴别同属不同种的物种。本研究从种间变异、平均种间变异、种间最小变异、种内变异、平均种内变异、种内最大变异分析不同序列的种内 种间差异 (表 5)。5条序列中,种间最小变异ITS2 序列最大 (0.014 2),其次为ITS序列 (0.012 9),最 小为rbcL (0.000 8); 种内最大变异psbA-trnH最大(0.589 2),其次为matK序列(0.139 7),最小为rbcL (0.001 3)。matK序列平均种间变异 (0.086 1) 小于平均种内变异 (0.115 8),其余ITS等4条序列均为平 均种间变异大于平均种内变异; rbcL序列虽然有较小的种内变异,但其种间变异同样较小; psbA-trnH序列虽然与ITS、ITS2序列的种间最小变异均小于种内最大变异,但psbA-trnH序列较ITS与ITS2序列,种间最小变异最小,而种内最大变异最大,且psbA-trnH序列种内变异均大于ITS与ITS2序列; ITS与ITS2序列种内种间差异相差不大,ITS序列较ITS2种内 差异较小,但ITS序列种间变异略小于ITS2。综合 分析,ITS序列是鉴别百合属较理想的条形码序列。
|
|
Table 5 Analysis of intra and inter-specific variation of different barcodes |
Barcoding gap是指序列种内与种间遗传变异由于显著差异而形成的间隔区[15, 16]。如图 1为5条序列的barcoding gap图。5条序列均无明显的间隔区,rbcL序列种内种间变异交叉相对较少,但区分并不明显。相对其他序列,ITS序列虽然无明显间隔区,但种内与种间变异明显分为两大部分,其分布较高的遗传变异可以明显区分,因此在barcoding gap分析方面,ITS序列为鉴别百合属的较理想条形码序列。

|
Figure 1 Relative distribution of inter-specific divergence and intra-specific variation of different barcodes. A: ITS2; B: ITS; C: psbA-trnH; D: matK; E: rbcL |
NJ树是基于K2P遗传距离建立,分析不同物种间的亲缘关系远近,以鉴别物种。该方法可直观展示不同物种的系统关系。如图 2所示,5条序列可以很好的将百合属不同物种分开。ITS序列可以将不同物种分到不同的分支上,与其他序列相比,能更好的区分百合属相近的物种,是鉴别百合属的优选序列。
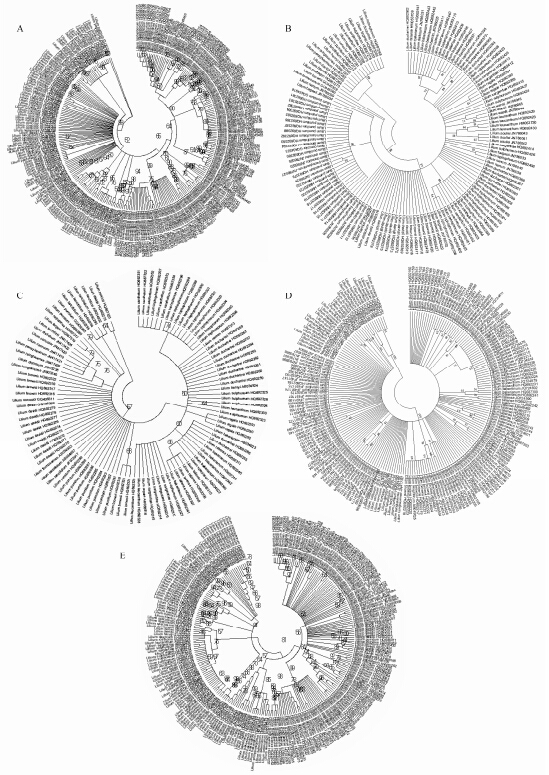
|
Figure 2 Analysis of NJ tree built on different barcodes. A: ITS; B: psbA-trnH; C: rbcL; D: matK; E: ITS2 |
百合属原产于北半球的温带和寒带地区。我国是百合植物自然分布中心,但对于百合属植物资源的系统进化关系及相互间亲缘关系研究较少,导致我国在百合育种方面非常薄弱,大部分的商业栽培品种主要依赖于国外进口。在对百合属种质资源关系的研究中,前期多采用数量性状等对百合属进行分类研究[17],但选用不同的器官性状分类差异可能较大,相对来说生殖器官性状比营养器官性状分类分析结果差异较小; 部分研究利用等位酶系统基因位点的等位基因等对百合属进行分类[18, 19]; RAPD技术是研究百合分类较多的分子标记技术[20, 21, 22],可以根据随机引物扩增出的片段的多态性进行鉴别,灵敏度较高,但结果不稳定,可重复性不强,适合种内不同个体间的鉴别。
DNA条形码是一种较新的分子标记技术,操作简便、技术成熟、通用性强,对于不同物种之间的鉴别能力较强,已有广泛的研究和应用基础。本研究运用ITS2、ITS、psbA-trnH、matK、rbcL 5条常用序列,从种内种间变异、barcoding gap、鉴定成功率3个方面分析DNA条形码对于百合属物种的鉴别能力,目的在于找到能快速、通用地鉴别百合属的DNA条形码序列。研究结果表明matK序列平均中间变异小于平均种内变异,说明matK序列有利于鉴别种内变异较大的物种,不适合在百合属水平上的鉴别; rbcL序列种内与种间变异均较小,可作为鉴别百合属的补充序列; psbA-trnH序列相对于ITS和ITS2序列种间差异小,而种内变异较大,因此不适合鉴别百合属物种; ITS2序列包含于ITS序列中,ITS序列较ITS2种内差异较小,但ITS序列种间变异略小于ITS2,且在实际鉴定工作中,获得ITS序列即可获得ITS2序列。虽然ITS序列在本实验中的鉴定成功率并不高,但考虑到GenBank数据库数据的复杂性及其来源的多样性,且在本实验中ITS序列在扩增效率、鉴定成功率、种内与种间变异、barcoding gap、系统发育树等方面均较其他序列表现良好,因此可以作为鉴别百合属的优选DNA条形码序列。本研究为百合属物种的鉴别提供新的分子鉴别方法,并为亲缘关系较复杂的属筛选理想DNA条形码鉴别序列提供理论 依据。
| [1] | Guo CH, Jiang SX. The study and utilization of lily bulb [J]. Acta Chin Med Pharm (中医药学报), 2004, 32: 27-29. |
| [2] | Sun XL, Xu LC, Zuo X. The source and morphological identification of ten commercial lily [J]. Shandong J Tradit Chin Med (山东中医杂志), 1999, 18: 371-372. |
| [3] | Yang JY, Yang SQ, Han CH. The research of morphology of pollen in three medicinal plant of Lilium [J]. J Inner Mongolia Med Univ (内蒙古医科大学学报), 1999, 21: 202-203. |
| [4] | Qin KM, Shi Y, Sun J, et al. Study on the quality differences of Chinese medicine Bulbus lilii from different places of production (Ⅰ) [J]. J Beijing Union Univ (Nat Sci) (北京联合大学学报自然科学版), 2010, 24: 21-24. |
| [5] | Lin ML, Liu TS, Xiao BM, et al. Identification of commercial crude drug Bulbus lilii with RAPD technique [J]. J Tradit Chin Med Univ Hunan (湖南中医药大学学报), 2006, 26: 30-32. |
| [6] | Tong QZ, Sheng XB, Liu XD, et al. DNA extraction from and optimization of RAPD reaction system for medical lily [J]. J Tradit Chin Med Univ Hunan (湖南中医药大学学报), 2008, 28: 33-36. |
| [7] | Hebert PD, Ratnasingham S, Waard JR. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species [J]. Proc Biol Sci, 2003, 270: S96-S99. |
| [8] | CBOL Plant Working Group. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 2009, 106: 12794-12797. |
| [9] | Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species [J]. PLoS One, 2010, 5: e8613. |
| [10] | Chen SL, Song JY, Yao H, et al. Strategy and key technique of identification of Chinese herbal medicine using DNA barcoding [J]. Chin J Nat Med (中国天然药物), 2009, 7: 322-327. |
| [11] | Zheng SH, Sun ZY, Huang LF. Analysis and identification of Ophiopogon japonicus and its adulterants using ITS2 sequences [J]. Mod Chin Med (中国现代中药), 2012, 14: 21-23. |
| [12] | Zheng SH, Huang LF, Chen SL. Specific PCR molecular identification of Dendrobium chrysotoxum [J]. Chin Tradit Herb Drugs (中草药), 2013, 44: 744-748. |
| [13] | Xiong Y, Chen MH, Xiong HB. Genetic diversity analysis of Lilium based on psbA-trnH gene sequence variation [J]. Biotechnology (生物技术), 2013, 23: 53-56. |
| [14] | Ross HA, Murugan S, Li WL. Testing the reliability of genetic methods of species identification via simulation [J]. System Biol, 2008, 57: 216-230. |
| [15] | Meyer CP, Paulay G. DNA barcoding: error rates based on comprehensive sampling [J]. PLoS Biol, 2005, 3: e422. |
| [16] | Lahaye R, Bank M, Bogarin D, et al. DNA barcoding the floras of biodiversity hotspots [J]. Proc Natl Acad Sci USA, 2008, 105: 2923-2928. |
| [17] | Zhang XL, Zhou HG, Zhou Z, et al. A numerical taxonomic study on Lilium cultivars [J]. Guihaia (广西植物), 2000, 20: 325-328. |
| [18] | Zhou HG, Zhang XL, Wang ZR, et al. Genetic relationship of some lily cultivars by allozyme data [J]. Southwest China J Agric Sci (西南农业学报), 1999, 12: 92-95. |
| [19] | Xin G, Hou DY, Liu LL, et al. Study on peroxide enzyme isozymes in five varieties of oriental lily [J]. J Anshan Norm Univ (鞍山师范学院学报), 2003, 5: 59-61. |
| [20] | Zhao XY, Chen XL, Fang H, et al. Using rapd marker to evaluate the genetic relationship among genus Lilium [J]. J Beijing Agric Coll (北京农学院学报), 1995, 20: 58-63. |
| [21] | Yamagishi M, Abe H, Nakano M, et al. PCR-based molecular markers in Asiatic hybrid lily [J]. Sci Hortic, 2002, 96: 225- 234. |
| [22] | Zhao QF, Ma SR, Zeng XY, et al. RAPD analysis of Lilium cultivar resources [J]. J Lanzhou Univ (Nat Sci) (兰州大学学报 (自然科学版)), 2005, 41: 30-33. |
 2014, Vol. 49
2014, Vol. 49


