2. 南京野生植物综合利用研究院, 江苏 南京 210042
2. Nanjing Institute for Comprehensive Utilization of Wild Plants, Nanjing 210042, China
白及属 (Bletilla H. G. Reichenbach),隶属于兰科 (Orchidaceae) 树兰亚科 (Epidendroideae),中国境内共有4个种,分别是白及 [Bletilla striata (Thunb.) H. G. Reichenbach]、华白及 [Bletilla sinensis (Rolfe) Schltr.]、黄花白及 (Bletilla ochracea Schltr.) 和小白及 [Bletilla formosana (Hayata) Schltr.]; 其分布北起江苏、河南,南至台湾,东起浙江,西至西藏东南部[1]。该属植物不仅具有较高的观赏价值,也是我国重要的中药材。市场流通的中药材白及为白及属白及假鳞茎制成的干燥茎,其味苦、甘、涩,性微寒,具有收敛止血、消肿生肌的功能; 主要用于治疗咳血、吐 血、外伤出血、疮疡肿毒等[2]。近年来,市场上出现一种假鳞茎比普通白及大一倍的“巨茎白及”,它是安徽振扬农林有限公司于2008年首次培育出来并命名,《中国植物志》并未记载,其分类学地位尚未明确。受市场对白及药材需求强劲增长的影响,野生白及属药用植物资源急剧减少,商品药材品种混乱。百合科 (Liliaceae) 黄精属 (Polygonatum Mill.) 黄精 (Polygonatum sibiricum Mill.)、玉竹 (Polygonatum odoratum (Mill.) Druce) 的干燥根或茎形态特征与白及类似,常被作为白及植物以次充好。有关白及属植物的鉴定,已有其形态学、理化性质以及ISSR等DNA分子标记方面的报道[3, 4, 5],这些研究都为白及属药用植物的鉴定提供了依据,但直接用DNA条形码序列进行白及属的鉴定,前人尚未有报道,这也为白及药用植物的鉴定带来了新的思路。
DNA条形码 (DNA barcoding) 是利用短的、标准的DNA序列来鉴定中药材的新技术[6]。该方法由于不受药材发育阶段和药材形态的影响,目前已被广泛应用于蒿属 (Artemisia L.)、重楼属 (Paris L.) 等药用植物及其混伪品的鉴定[7, 8]。在核基因中,nrDNA ITS的长度和序列变化较大,常用于物种鉴定和系统进化研究[9, 10, 11]。nrDNA ITS序列是位于真核生物nrDNA 18S和26S之间的内含子,由于进化中的选择压力小,nrDNA ITS片段在物种水平的变异较快,有更多的突变位点以区分不同的物种。Chen等[12]通过对6 000余份药用植物样本进行DNA条形码序列筛选,发现ITS2序列的物种水平鉴定能力达到92.7%,首次提 出以ITS2为核心的药用植物条形码鉴定体系。另外,LEAFY (LFY) 同源基因处于成花调控网络的关键位置,是开花启动过程的主要调控基因[13]; 近些年,大量的LFY同源基因被克隆出来[14, 15]。邢文瑞等[16]对细茎石斛 (Dendrobium moniliforme (L.) Sw.) LFY同源基因克隆并对比研究,发现该基因内含子2在细茎石斛及铁皮石斛 (Dendrobium officinale Kimura et Migo) 间的长度差异明显,为物种间鉴定提出一种新的方法。在叶绿体DNA方面,国际条形码协会植物工作组提出叶绿体基因matK和rbcL序列可作为植物的核心条形码[17]。而在兰科植物中,Kurt等[18]指出兰科植物的ycf1 (hypothetical chloroplast open reading frame 1) 基因具有高度变异性,并比matK基因更具有可靠的变异信息位点。
本研究利用nrDNA ITS、LFY同源基因内含子2及叶绿体ycf1基因3条候选条形码对白及属药用植物进行DNA条形码研究。依据DNA条形码对其混伪品和待检品进行鉴定,并初步判断“巨茎白及”的分类地位。对白及属DNA条形码的研究将为珍贵药用植物白及属种间的区分、市场药材的判定及混伪品鉴别提供可靠的分子水平依据。
材料与方法 材料试验材料共计41份样品,其中白及属4个物种25份样品,分别来自安徽、江苏、湖南、陕西、江西、甘肃、云南7个省份; “巨茎白及”8份,随机选取4份(JJAH01-JJAH04) 亲代样本,4份 (JJAH05-JJAH08) 子一代样本,均来自安徽; 混伪品样品包括黄精属2个物种6份 (黄精3份,玉竹 3份),待检品为2份粉末状药材,混伪品及待检品 均来自安徽亳州药材市场 (表 1)。实验材料保存于南京师范大学生命科学学院植物资源与环境研究所,所有假鳞茎样品由植物开花后鉴定,所有新鲜叶样品用硅胶干燥保存待用。
|
|
Table 1 Plant samplesused in the present study. * Pseudobulb sample; - Absent data |
所有药材根茎使用75% 乙醇擦洗表面后研磨,无水乙醇浸泡5 min后脱水,植物叶片采用硅胶进行干燥。取干燥叶片0.1 g及药材约20 mg磨碎后,在CTAB法[19]的基础上略做改进以提取植物及药材的总DNA。采用NanoDrop 2000 (Thermo Scientific,USA) 测定A260、A280比值评价其DNA浓度及纯度。
PCR扩增及测序PCR扩增引物见表 2。试验使用ABI Veriti PCR仪 (ABI,USA) 对总DNA利用通用反应条件进行PCR扩增[16, 18, 20]。扩增产物选用 琼脂糖凝胶电泳检测并切胶,用天根胶回收试剂盒 (Tiangen Biotech Co.,China) 对目的片段进行回收,经纯化后送华大公司 (BGI,China) 进行双向测序。
数据处理所得序列用MEGA5.0软件[21]进行查错、比对和分析,基因序列相同视为一个基因型[22]。所有序列上传至GenBank数据库获得登录号。
基于K2P (Kimura-2-Parameter) 模型进行遗传距离分析,对不同序列种间、种内变异大小进行比较。统计不同序列种间、种内遗传距离的分布频度,评价不同DNA条形码候选序列的Barcoding Gap。利用MEGA 5.0构建NJ系统树,利用Bootstrap (1 000次重复) 检验各分支的支持率。最后选用相似性搜索算法 (BLAST1) 考察序列的鉴定成功率[7, 23]。
结果 1 白及属序列分析及种内与种间遗传距离分析在利用DNA条形码序列鉴定物种时,PCR扩增效率和测序成功率是其成功鉴定的基础。3条候选 序列的PCR扩增效率和测序成功率均为100%。白及属25份样品的DNA条形码候选序列的长度、GC 含量、变异、信息位点数等统计结果见表 3。各序列长度从635~1 398 bp不等,其中nrDNA ITS序列 最短为635~636 bp,共12个基因型,变异/信息位点数为235/209个; 其次为LFY同源基因内含子2,其序列长度为1 043~1 398 bp,共12个基因型,变异/信息位点数为438/43个; 最长的为ycf1序列,共有13个基因型,其序列长度为1 039 bp,变异/信息位点数为179/102个。各序列的GC含量也有不同,nrDNA ITS的GC含量最高,达到61.7%,ycf1则最低,GC含量为27.7%。
|
|
Table 3 Sequences characteristics of candidate barcodes |
基于K2P遗传距离,对白及属的3条不同的DNA条形码候选序列的种内变异及种间变异结果进行分析,结果表明 (表 4),白及属植物的nrDNA ITS序 列种间遗传变异 (0.022~0.106) 大于种内遗传变异(0~0.012); ycf1序列种间遗传变异 (0.017~0.106) 亦大于种内遗传变异 (0~0.015),因此,nrDNA ITS序列和ycf1序列可以作为白及属植物的分子鉴定。LFY同源基因内含子2序列种间 (0.005~0.036) 和种内 (0~0.008) 遗传距离之间有重叠区,故LFY同源基因内含子2序列不适用于白及属植物的鉴定。
|
|
Table 4 The intraspecific and interspecific K2P genetic distance of candidate barcodes |
理想的条形码种间遗传变异应明显大于种内 遗传变异,并在两者之间形成显著差异从而在坐标轴上呈现出明显的间隔区,即Barcoding Gap。依据 K2P遗传距离,不同的DNA条形码候选序列构建的Barcoding Gap图 (图 1) 显示: nrDNA ITS序列种内遗传变异和种间遗传变异能够很好的区分,形成了明显的 Barcoding Gap区。ycf1序列虽然没有形成明显的Barcoding Gap区,但种内和种间的平均遗传距离没有相互交叉。相比之下,LFY同源基因内含子2序列的种内和种间遗传变异重叠区比例大,无明显的Barcoding Gap,不利于区分物种。
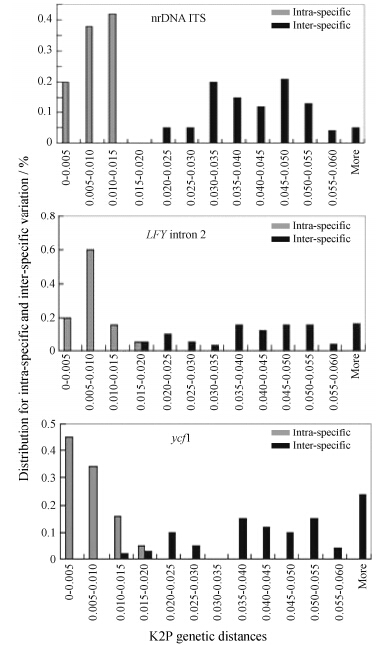
|
Figure 1 Distribution of intra- and inter-specific Kimura 2-parameter (K2P) distances for three candidate barcodes |
为了直观的评价白及属不同DNA条形码候选序列的合适度,本文利用笋兰 [Thunia alba (Lindley) H. G. Reichenbach] 作为外类群,应用MEGA 5.0软件对白及属样品基因型数据分别构建nrDNA ITS、LFY同源基因内含子2、ycf1的NJ树 (图 2、3、4)。从图 3和图 4可以看出,nrDNA ITS序列和ycf1序列的 NJ树都将白及属4个物种聚在各自的同一进化枝上,鉴定效率为100%,能有效的区分白及属4个物种。LFY同源基因内含子2序列构建的NJ树 (图 2),白及属种内植物样品均有聚在不同的进化枝上,鉴定效率为84.6%,因此LFY同源基因内含子2序列不能有效的鉴别白及属种间植物样品。
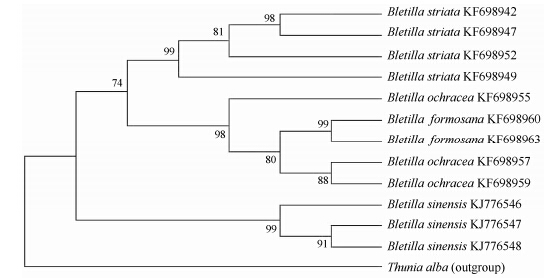
|
Figure 2 LEAFY intron 2 phylogenetic tree constructed using NJ method of Bletilla. Bootstrap values ( > 50%) are shown above the relevant branches |

|
Figure 3 nrDNA ITS phylogenetic tree constructed using NJ method of Bletilla and its adulterants. Bootstrap values ( > 50%) are shown above the relevant branches |
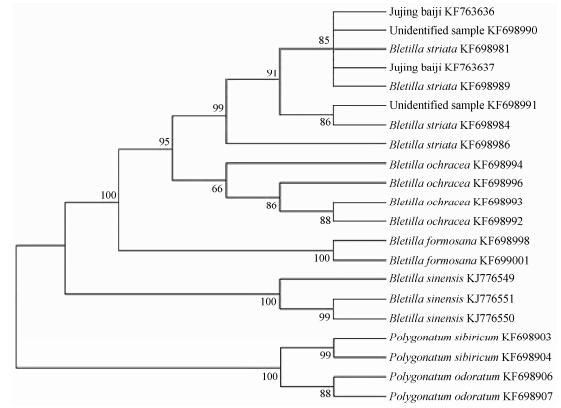
|
Figure 4 ycf1 phylogenetic tree constructed using NJ method of Bletilla and its adulterants. Bootstrap values ( > 50%) are shown above the relevant branches |
为验证nrDNA ITS序列和ycf1序列在更多样本中的鉴定能力,加入从GenBank下载兰科40个物种65条nrDNA ITS序列和40个物种的42条ycf1序列,经BLAST1算法计算其鉴定成功率均为100%。基于白及属植物DNA条形码的Barcoding Gap、NJ树及BLAST1算法鉴定效率分析,本实验将nrDNA ITS和ycf1序列确定为鉴定白及属药用植物和混伪品的最适潜在条形码。
3 白及属药用植物待检品及混伪品的鉴定为了有效的鉴定白及属药用植物药材及混伪品,本文利用白及属nrDNA ITS和ycf1条形码分别构建NJ树 (图 3,图 4)。其中6种黄精属混伪品,nrDNA ITS片段共检测到了4个基因型,ycf1基因共检测到了4个基因型。结果显示,NJ树均分为两大支,白及属植物聚为一支,黄精属两个物种聚为另一支,两个属植物样品得到了准确的区分。白及属2份待检品的nrDNA ITS和ycf1基因序列构建的NJ树与白及属白及聚为同一大枝,同属白及植物; 粉末状待检品药材为白及属白及加工制成的。
另外,8份“巨茎白及”核基因及叶绿体基因序列均与安徽六安白及最为接近。在核基因方面,“巨茎白及”的nrDNA ITS拥有3个基因型,其中,亲代有2个基因型,子一代有3个基因型。序列号为KF763633的基因型与白及样品 (KF680621) 的序列共有3个碱基的替换 (60 bp、121 bp、481 bp位点),序列号为KF763634的基因型与白及样品 (KF680621) 的序列有2个碱基的替换 (60 bp、481 bp位点),序列号为KF763635的基因型与白及样品 (KF680621) 的序列共有4个碱基的替换 (60 bp、120 bp、121 bp、481 bp位点),均是G-C之间的转换。在叶绿体基因方面,“巨茎白及”的ycf1序列 (KF763636) 与白及样品(KF698981) 序列相同,序列号为KF763637的基因型与白及样品 (KF698981) 的序列存在两个碱基的替换,分别是在121 bp处TA转换与898 bp处GC的转换,变异发生在子一代。nrDNA ITS和ycf1基因序列构建的NJ树均与白及属白及聚为一支 (图 3,图 4)。
讨论 1 DNA条形码候选序列对白及属鉴定准确性评价理想的DNA条形码通常需要满足以下3个标 准: ① 在物种水平具有显著的遗传变异和分化,能够鉴别不同物种; ② 合适的长度,便于DNA扩增、测序、拼接及排序时不需要手动调整; ③ 目的片段两 端具有保守位点,可用于通用引物设计[6]。对于单拷贝核基因—— LEAFY同源基因,在植物魔芋属 (Amorphophallus Blume ex Decaisne)、绣线梅属(Neillia D.) 植物类群的系统发生分析研究中,LFY基因序列相比ITS和cpDNA能提供更多的信息位
点[24, 25]。本研究对白及属植物LFY同源基因内含子2序列分析结果表明,白及属植物LFY同源基因内含子2序列种内和种间遗传距离有重叠区,且种间遗传距离较小,种内植物NJ树有聚在不同的进化枝上,LFY同源基因内含子2序列不适合作为白及属植物鉴定的有效条形码。故LFY同源基因在植物条形码中的应用还有待进一步研究。
nrDNA ITS序列在长期进化中,选择压力小、变异位点多,并与保守的5.8S和26S区段形成特定的颈环二级结构[26, 27]。近些年,陈士林等先后利用ITS序列对羌活 (Notopterygium incisum C. C. Ting ex H. T. Chang)[28]、洋金花[29]、重楼属[7]等药用植物以及混伪品鉴别,也证明了ITS/ITS2条形码鉴定的稳定性与准确性。另外,在翅茎草属 (Pterygiella Oliver) 和石斛属的DNA条形码研究中,也证明了ITS序列比matK和rbcL等叶绿体基因有更强的鉴别能力[30, 31]。Chang等[32]发现兰科植物台湾蝴蝶兰 (Phalaenopsis aphrodite Reichb. f.) 叶绿体基因ycf1存在于小单拷贝区 (SSC),并具有高度变异,可用于兰科植物系统发生研究。本研究对白及属4个物种的3条候选条形码进行鉴定,种内和种间遗传距离比较和NJ树分析表明,nrDNA ITS序列和叶绿体基因ycf1序列种间遗传变异大于种内遗传变异,其构建NJ树能有效地区分白及属的4个物种。结果表明核基因nrDNA ITS序列以及叶绿体基因ycf1序列的鉴定效率均为100%,因此推荐采用核基因nrDNA ITS序列并辅助选用叶绿体基因ycf1序列作为鉴定白及属的DNA条形码。
2 DNA条形码对白及属药材及其混伪品的鉴定一个成功的DNA条形码需要足够的序列变化位点来区分物种间的区别,单片段的低识别率很大程度上局限了条形码的识别度[33]。Singh等[31]在对石斛属DNA条形码研究中发现3个或两个基因位点联合比单基因位点鉴定效率高,而超过4个基因位点联 合的鉴定效率并没有显著提升。在兰科植物中,Kim等[34]用5个cpDNA片段共26种联合片段组合对兰科植物进行DNA条形码研究,结果表明atpF-atpH、psbK-psbI和trnH-psbA 3个组合片段是鉴定韩国兰 科物种的最适DNA条形码。Kress等[33]对陆生植物48个属进行了单片段及其联合鉴定的对比分析,推荐rbcL和trnH-psbA组合片段作为陆生植物的DNA条形码。本研究核基因nrDNA ITS和叶绿体基因ycf1序列均可作为白及属鉴定的有效DNA条形码,但由于中国境内白及属仅有4个物种,采用单片段nrDNA ITS或ycf1基因序列就能成功鉴定出白及属物种及其混伪品。
本研究采用适合于白及属鉴定的nrDNA ITS和ycf1基因序列分别对白及属药用植物及其混伪品黄精属植物进行鉴定,结果表明白及属样品单独聚为一支,表现出了单系性,明显的与黄精属的黄精、玉竹混伪品区分开。针对市场上粉末状药材难以鉴定 的难题,陈士林等[35]为药用植物未知样品的鉴定提供了可参考的标准化流程: 通过采用BLAST分析、距离法、建树法、成对序列比对法等方法确保未知样品鉴定的准确性。本研究的2份粉末状待检品经序列比对、NJ树分析后归于白及植物; 这也再次证明了nrDNA ITS和ycf1条形码鉴定中药材的能力。nrDNA ITS和ycf1条形码技术对中药材原植物及其药材与混伪品准确快速鉴定,将为市场上对白及药用植物鉴定提供科学依据。
3 运用DNA条形码探讨“巨茎白及”的基源传统的植物物种界定是依据植物的形态特征 (如叶片、花等) 与近缘种进行匹配[36]。安徽“巨茎白及”,是近年来出现在市场上的商业名称,《中国植物志》并未记载,其分类地位尚未有文献报道。安徽“巨茎白及”样品开花后呈粉红色,花大,萼片和花瓣近等长,这与《中国植物志》记载的白及属植物的白及花形态最为相似; 但其又具有假鳞茎大、产量高等特性。此外,Pons等[37]认为物种界定依据形态学特征界定含糊不清,应使用物种最原始的数据即DNA的序列信息开展物种界定。如Vanhaecke等[38]利用DNA条形码和微卫星标记技术把智利海域的杂种鱼 (Aplochiton spp.) 归为A. zebra物种。本研究利用白及的DNA条形码对“巨茎白及”进行序列分析,结果显示,“巨茎白及”样品与安徽六安的白及样品的nrDNA ITS序列和ycf1序列相似度最高; 基于NJ 树也将“巨茎白及”和白及属白及聚为一支。因此,本研究在形态学以及分子水平上均支持将安徽“巨茎白及”归为白及属白及植物。
| [1] | The Chinese Academy of Sciences. Flora of China (China version): Vol 18 (中国植物志 中文版: 第18卷) [M]. Bei-jing: Science Press, 1999: 46. |
| [2] | Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典) [S]. Beijing: China Medical Science Press, 2010: 95. |
| [3] | Liu R, Teng XJ, He JF, et al. Partial splenic embolization using Bletilla striata particles for hypersplenism in cirrhosis: a prospective study [J]. Am J Chin Med, 2011, 39: 261-269. |
| [4] | Zhai M, Ma YY, Zheng GY, et al. Comparative identifica-tion between commercial and Bletilla striata three kinds of counterfeits [J]. J Chengdu Univ Tradit Chin Med (成都中医药大学学报), 2012, 3: 53-56. |
| [5] | He ZJ, Lv LF, Yang LY, et al. Genetic diversity of Bletilla Rchb. f. germplasms by ISSR analysis [J]. Southwest China J Agric Sci (西南农业学报), 2008, 4: 1081-1085. |
| [6] | Chen SL, Pang XH, Yao H, et al. Identification system and perspective for DNA barcoding of traditional Chinese materia medica [J]. World Sci Technol (世界科学技术), 2011, 13: 747-754. |
| [7] | Zhu YJ, Chen SL, Yao H, et al. DNA barcoding the medicinal plants of the genus Paris [J]. Acta Pharm Sin (药学学报), 2010, 45: 376-382. |
| [8] | Liu MZ, Song JY, Luo K, et al. Identification of nine common medicinal plants from Artemisia L. by DNA barcoding sequences [J]. Chin Tradit Herb Drugs (中草药), 2012, 43: 1393-1398. |
| [9] | Avarez I, Wendel JF. Ribosomal ITS sequences and plant phylogenetic inference [J]. Mol Phylogenet Evol, 2003, 29: 417-434. |
| [10] | Giuseppe E, Cocuzza MC, Cavalieri V. Identification of aphids of Aphis frangulae-group living on Lamiaceae species through DNA barcode [J]. Mol Ecol Resour, 2014, 14: 447-457. |
| [11] | Schoch CL, Seifert KA, Huhndorf S, et al. Nuclear ribo-somal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi [J]. Proc Natl Acad Sci USA, 2012, 109: 6241-6246. |
| [12] | Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species [J]. PLoS ONE, 2010, 5: e8613. |
| [13] | Blazquez MA, Soowal LN, Lee I, et al. LFY expression and flower inition in Arabidopsis [J]. Development, 1997, 124: 3835-3844. |
| [14] | Liu YX, Hu GB, Lin SQ, et al. Cloning and sequence analysis of LEAFY homologous gene from loquat [J]. South China Agric Univ (华南农业大学学报), 2005, 26: 66-68. |
| [15] | Schlüter PM, Kohl G, Stuessy TF, et al. A screen of low-copy nuclear genes reveals the LFY gene as phylogenetically informative in closely related species of orchids (Ophrys) [J]. Taxon, 2007, 56: 493-504. |
| [16] | Xing WR, Hou BW, Guan JJ, et al. Sequence analysis of LEAFY homologous gene from Dendrobium moniliforme and application for identification of medicinal Dendrobium [J]. Acta Pharm Sin (药学学报), 2013, 48: 597-603. |
| [17] | CBOL Plant Working Group. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 2009, 106: 12794-12797. |
| [18] | Kurt M, Neubig W, Mark W, et al. Phylogenetic utility of ycf1 in orchids: a plastid gene more variable than matK [J]. Plant Syst Evol, 2009, 277: 75-84. |
| [19] | Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochem Bull, 1987, 19: 11-15. |
| [20] | Ding XY, Wang ZT, Xu H, et al. Datebase establishment of the whole rDNA ITS region of Dendrobium species of “FengDou” and authentication by analysis of their sequences [J]. Acta Pharma Sin (药学学报), 2002, 37: 567-573. |
| [21] | Tamura K, Peterson D, Peterson N, et al. MEGA5: Mo-lecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Mol Biol Evol, 2011, 28: 2731-2739. |
| [22] | Gabriel SB, Schaffner SF, Nguyen H, et al. The structure of haplotype blocks in the human genome [J]. Science, 2002, 296: 2225-2229. |
| [23] | Ross HA, Murugan S, Li WL. Testing the reliability of genetic methods of species identification via simulation [J]. Syst Biol, 2008, 57: 216-230. |
| [24] | Oh SH, Potter D. Molecular phylogenetic systematic and biogeography of tribe Neillieae (Rosaceae) using DNA sequences of cpDNA, rDNA, and LEAFY [J]. Am J Bot, 2005, 92: 179-192. |
| [25] | Grob GB, Gravendeel B, Eurlings MC, et al. Potential phylogenetic utility of the nuclear FLORICAULA/LEAFY second intron: comparison with three chloroplast DNA regions in Amorphophallus (Araceae) [J]. Mol Phylogenet Evol, 2004, 30: 13-23. |
| [26] | Selig C, Wolf M, Müller T, et al. The ITS2 database II: homology modeling RNA structure for molecular systematic [J]. Nucleic Acids Res, 2007, 36: D377-D380. |
| [27] | Dassanayake RS, Gunawardene YI, Silva BD. ITS-2 secondary structures and phylogeny of Anopheles culicifacies species [J]. Bioinformation, 2008, 2: 456-460. |
| [28] | Xin TY, Yao H, Luo K, et al. Stability and accuracy of the identification of Notopterygii Rhizoma et Radix using the ITS/ITS2 barcodes [J]. Acta Pharm Sin (药学学报), 2012, 47: 1098-1105. |
| [29] | Han JP, Li MN, Luo K, et al. Identification of Daturae flos and its adulterants based on DNA barcoding technique [J]. Acta Pharm Sin (药学学报), 2011, 46: 1408-1412. |
| [30] | Dong LN, Wortley AH, Wang H, et al. Efficiency of DNA barcodes for species delimitation: a case in Pterygiella Oliv (Orobanchaceae) [J]. J Syst Evol, 2011, 49: 189-202. |
| [31] | Singh HK, Parveen I, Raghuvanshi S, et al. The loci recommended as universal barcodes for plants on the basis of floristic studies may notwork with congeneric species as exemplified by DNA barcoding of Dendrobium species [J]. BMC Res Notes, 2012, 5: 42-53. |
| [32] | Chang CC, Lin HC, Lin P, et al. The chloroplast genome of Phalaenopsis aphrodite (Orchidaceae): comparative analysis of evolutionary rate with that of grasses and its phylogenetic implications [J]. Mol Biol Evol, 2006, 23: 279-291. |
| [33] | Kress WJ, Erickson DL. A two-locus global DNA barcode for land plants: the coding rbcL gene complements the non-coding trnHpsbA spacer region [J]. PLoS One, 2007, 6: e508. |
| [34] | Kim HM, Hunoh S, Bhandari G, et al. DNA barcoding of Orchidaceae in Korea [J]. Mol Ecol Resour, 2014, 14: 499-507. |
| [35] | Chen SL, Song JY, Yao H, et al. Strategy and key tech-nique of identification of Chinese herbal medicine using DNA barcoding [J]. Chin J Nat Med (中国天然药物), 2009, 7: 322-327. |
| [36] | Duminil J, Michele M. Plant species delimitation: a comparison of morphological and molecular markers [J]. Plant Biosystems, 2009, 7: 528-542. |
| [37] | Pons J, Tomothy GB, Jesus GZ, et al. Sequence-based species delimitation for the DNA taxonomy of undescribed insects [J]. Syst Biol, 2006, 55: 595-609. |
| [38] | Vanhaecke D, Leaniz CG, Gajardo G, et al. DNA barcoding and microsatellites help species delimitation and hybrid identification in endangered Galaxiid Fishes [J]. PLoS One, 2012, 7: e32939. |
 2014, Vol. 49
2014, Vol. 49


