2. 中国医学科学院、北京协和医学院药用植物研究所, 北京 100193
2. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China
近年随着高通量测序技术的迅猛发展,生物系统发育研究中开始采用基因组数据,因此出现一些新术语,如系统发育基因组学 (phylogenomics,基因组系统学/基因组亲缘学)、药用基因组亲缘学 (pharmacophylogenomics)、转录组亲缘学 (phylotranscriptomics) 等。系统学 (亲缘学) 是生命科学各分支和交叉学科的根基所在,在生药学和药用生物学等药学相关领域起到提纲挈领的作用。在此对与药用亲缘学 (pharmaphylogeny) 密切相关的若干术语做一简要辨析。 1 系统发育基因组学
系统发育基因组学是进化和基因组学的交叉学科,是将基因组数据用于进化关系重建的综合分析,因此需要系统发育 (phylogenetics) 研究技术和基因组学技术的有机整合。系统发育研究是比较分析单 个基因或几个基因序列[1,2],也常结合其他类型数据,例如形态学和植物化学[3,4]数据。系统发育基因组学基于基因组时代之前的分子系统学研究,通过比较全基因组序列或至少大部分基因组序列来获取对进化关系重建有用的信息。目前该领域研究包括: 1.1 基因功能预测和进化推演
现存种子植物超 过26万种,在进化史上均经历多次全基因组倍增 (whole genome duplication,WGD),倍增基因拷贝在基因组中通常以保守的同线块 (syntenic block) 形式存在。在植物进化过程中,基因组大小变化是一种相对频繁的事件,这些变化一般并不与基因多少及顺序变化相关联。基因数量及顺序的保守性称为同线 性 (synteny或colinearity)。倍增影响新性状起源,近年来植物次生代谢路径多样化与WGD有关的例子 越来越多。硫代葡糖苷 (GS) 及其降解产物异硫氰酸盐的抗癌活性已引起关注。Hofberger等[5]发现WGD和串联倍增 (tandem duplication,TD) 加速十字花科 (Brassicaceae) 硫苷次生代谢路径的遗传多样性 (图 1)。在模式植物拟南芥 (At) 中硫苷生物合成需要 至少52个生物合成和调控基因。发现位于十字花目 (Brassicales) 系统树基部的阿拉伯岩芥菜 (Aethionema arabicum,Aab) 有多达67个硫苷生物合成基因,大部分拟南芥硫苷生物合成基因家族都能在阿拉伯岩芥菜找到直系同源基因,且大多表现出同线型。拟南芥45% 蛋白编码基因有多于一个拷贝,而拟南芥和岩芥菜硫苷路径多达95% 和97% 基因有多拷贝,提示硫苷类化合物在植物防御中的重要性。对倍增基因拷贝的序列联配表明在最近一次WGD发生了显著的硫苷路径基因倍增。硫苷路径基因的串联倍增及随后发生的亚功能化 (subfunctionalization) 和新基因化 (neofunctionalization) 过程进一步增加了硫苷次生代谢产物的遗传多样性和化学多样性,增强了植物适应环境变化的能力,显示了植物次生代谢产物化学空间在药物发现方面的巨大潜力。 倍增基因拷贝同样可以解释萜类等其他多基因路径合成的次生代谢产物的多样化过程。被子植物中已发现次生代谢产物超过20万种,可能大部分源自复杂性状的快速创新。
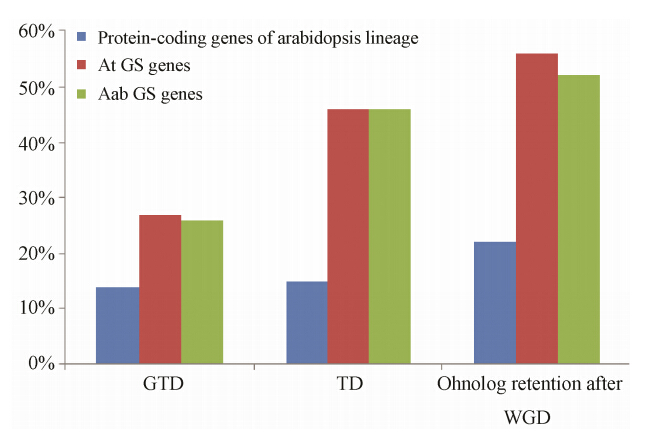 |
Figure 1 Duplicate distribution among Arabidopsis protein- coding genes compared with AtGS and Aethionema GS loci,redrawn from ref[5]. Percentage of genes with retained ohnolog (clusters of dose-sensitive genes organized in functional modules),tandem duplicate,and gene transposition duplicate (GTD) are shown. GS metabolic flexibility during lineage evolution arose from a permutation of increased ohnolog retention and TD rates |
甾体糖生物碱 (steroidal glycoalkaloid,SGA) 具细胞毒活性,能抑制乙酰胆碱酯酶,破坏细胞膜功能。Manrique-Carpintero等[6]研究了茄属 (Solanum) 6个野生种和种植马铃薯SGA代谢路径5个基因的自 然变异和全基因组SNP基因分型。5个基因包括属 于初级代谢的3-羟基-3-甲基戊二酰辅酶A还原酶1和2 (HMG1、HMG2) 以及2.3-鲨烯环氧酶 (SQE),属于次级代谢的茄碱半乳糖基转移酶 (SGT1) 和葡糖基转移酶 (SGT2)。测序3.7 kb DNA检测到354个变异。内含子中发现的变异多于外显子,次生代谢途径的关键酶编码基因中的变异多于初级代谢基因。dN/dS < 1以及Tajima’s D检验为负值提示存在纯化选择和遗传搭车 (hitchhiking,指看似对生物体适应性及进化没有贡献的搭车客突变)。比较核苷酸多样性估计值和dN/dS,提示初级代谢基因经受的选择限制强于次生代谢途径的关键酶编码基因,可解释次生代谢多样性。发现SGA含量低的马铃薯和SGA含量高的S.chacoense的HMG2、SQE、SGT1和SGT2各有特定的SNP基因型。Illumina SNP芯片分型发 现8个有信息SNPs,其不同组合可区分SGA含量高中低的不同代谢表型。这类研究有助于评价SGA在分离或关联作图群体中的差异分布,对道地药材研究具参考价值。
具抗癌抗菌活性的苯并嗪类防御化合物存在于禾本科 (Gramineae)、毛茛目 (Ranunculales) 和唇形目 (Lamiales) 多种植物中,其生物合成涉及9个酶[7],最终形成糖基化产物存储。其中7个基因 (Bx1-Bx6和Bx8) 在玉米四号染色体短臂末端形成一簇 (cluster),4个P450基因 (Bx2-5) 均属于CYP71C亚家族。推测在禾本科辐射进化之前TSA (色氨酸合成酶α亚单位) 基因和1个与Bx2相似的CYP71C祖先基因数次倍增,并且Bx8征募到近旁,从而形成次生代谢途径的关键酶编码基因簇。但是Bx6和Bx7的起源需要进一步研究。与Bx2相似的CYP71C祖先基因的功能与苯并嗪合成无关,基因倍增后Bx2-5基因的几个位点经受正选择,发生功能歧异,形成了目前酶特定的生化性质。已测序的禾本科Bx基因区域缺乏同线性,而TSA基因区具保守的同线性,说明基因倍增后发生重排,导致Bx1和Bx2的新拷贝在禾本科共同祖先的1个染色体末端成簇。成簇有利于相关基因共分离,末端染色体的定位则便于基因重排,也便于有关合成基因的进一步征募。这些奠基事件 (founding event) 和延伸 (elongation) 事件对于后续的苯并嗪生合基因簇的进化至关重要。双子叶植物尚未发现CYP71C,很可能双子叶和单子叶植物的苯并嗪生物合成途径彼此独立进化,即属于趋同进化。生氰糖苷的生物合成途径也存在类似的进化现象[8]。对次生代谢产物生物合成途径的深入研究有助于育种方案的理性设计,优化药用化合物的生产,实现基于生物技术的生产方式改进。
研究多基因家族的进化时,基因树比物种树更有助于了解成员基因的进化历史和基因倍增过程。通过对基因树和物种树冲突进行解释,可推测进化机制,包括快速辐射分化、杂交/渐渗、不完全谱系分 选、水平基因转移、旁系基因、基因倍增/丢失以及基因重组等。这些进化机制也可部分地解释近缘物 种的化学表型多样性,有助于推测药用化合物的来源。对于次生代谢产物生物合成基因家族和转录调 控基因家族均可在系统发育框架内挖掘分析全基因组有关序列。 1.2 构建和厘清物种进化关系
桔梗科 (Campanulaceae) 中一些植物的叶绿体基因组与烟草 (茄科Solanaceae) 基因组相比经历了大量的基因重排。Cosner等[9]对桔梗科18个种叶绿体基因组的基因 排列顺序进行分析,并据此构建系统树,从全新的角度阐述了桔梗科内部18个属间的系统发育关系。天南星科 (Araceae) 系统发育树根部结构不清楚,提示需要用更高分辨率的技术研究该科的早期进化。Henriquez等[10]采用Illumina高通量测序平台获得 该科32属线粒体基因组序列,结合GenBank数据 构建系统树。发现线粒体系统树支持率低且与叶绿 体系统树不一致。基于叶绿体全基因组序列的系统 树表明水芋属 (Calla) 和落檐属 (Schismatoglottis) 在一个主枝基部聚在一起,形态学和细胞学证据支持此发现。植物中线粒体DNA的基因顺序可能进 化较快,但是核苷酸序列的进化速率仅为动物的1/100。叶绿体DNA核苷酸序列的进化速率比线粒 体快3~4倍,目前在种间进化关系研究中应用最多,例如对银杏[11]、金壳果科 (Chrysobalanaceae)[12]和金虎尾目 (Malpighiales)[13]的研究。但是叶绿体全基因组数据不足以解决经历快速分化的类群,例如姜目 (Zingiberales)[14]。结合大量核基因组数据全面分析十分必要。
药用植物多样性是药用植物与环境形成的生态复合体以及与此相关的各种生态过程的总和,有遗传多样性、药用物种多样性和生态系统多样性3个 层次。对于物种不均匀分化程度较强的地区,在解释气候生态因子与药用植物多样性之间的关联时,要充分考虑进化过程的影响。如中国东喜马拉雅和横断山地区在1 000万年内迅速抬升,导致物种的快速分化,对生物多样性格局产生了显著影响,形成中国西南地区的“天空之岛”[15],亦由此形成了丰富的药用植物资源。全叶绿体基因组数据,可视为细胞器尺度的条形码,可用其研究分布于不同地理位置的同一物种 (例如道地药材) 的种内变异[16]。叶绿体基因组在居群水平的应用可为研究种内分化时间和分化强度提供线索。种内谱系关系的确立可视为更细致的系统发育重建。结合核基因组数据进行分析还可更 全面了解药用植物的花粉传播、迁移途径及物种进 化动力等。 1.3 预测和追溯侧向基因转移 (lateral gene transfer)
侧向 (水平) 基因转移在微生物进化中广泛存在的事实从根本上动摇了生命之树的假定形态[17]。已发现很多原核和真核生物间的侧向基因转移,一个有趣的例子是红豆杉 (Taxus) 和其内生真菌均有紫杉烷生物合成路径,其隐含的系统发育基因组学规律有待揭示,这将有助于药用植物和其内生菌互作的研究,为开发植物药资源提供参考。
全基因组测序花费高,对于重复序列占的比例大、杂合度高和非二倍体基因组,序列正确拼接组装十分困难。显然大规模比较转录组研究更具可行性。转录组数据基于表达的mRNA,不包括内含子信息,没有重复序列和倍性的干扰。Kohonen等[18]注意到基因组测序的快速发展,提出了转录组亲缘学概念,意指多物种的基因共表达分析,物种间进化距离分析可基于转录组信息。显然转录组亲缘学也可应用于药用植物研究,例如从19种植物 (包括虎杖、红豆杉、银杏和人参等药用植物) 转录组数据中提 取50个单拷贝直系同源基因[19],可用于系统树构建和进化分析。Chan等[20]分析482个编码红藻植物门头发菜纲紫菜属 (Porphyra) 膜转运蛋白的EST序列,其来自紫菜属P. umbilicalis和P. purpurea的454焦磷酸测序。红藻和绿藻序列在系统树上显示了预期的单系形态 (符合植物界假说),19个EST提示存在内共生和与原生藻菌有关的水平基因转移。77% 转运蛋白的系统发育位置不明确,需要积累更多序列信息。此研究提供了海洋藻类钠偶联的转运系统的分子特征,可能存在转运蛋白基因与脂肪酸生物合成及胞内脂质运输的共调控机制。
类似地,基于蛋白质组数据进行系统发育分析,有蛋白质组亲缘学 (phyloproteomics)[21],但尚未用于植物研究。表观基因组亲缘学 (phyloepigenomics) 在表观遗传修饰层面考查物种亲缘关系[22],用于药用植物研究将有新发现。宏基因组亲缘学 (phylometagenomics)[23]将可用于研究药用植物根际微生物和植物内生菌等。系统发育基因组学对计算能力和分析方法提出了新的挑战,故有计算基因组系统学。 2 药用基因组亲缘学
直系同源基因是在物种形成过程中由共同祖先基因演变来的分布于不同物种的基因,在药物发现过程中有助于动物模型的选择和分析流程的建立。葛兰素公司的遗传学家David B. Searls认为充分利用基因组数据有助于挖掘直系同源基因,更好地预测基因功能[24]。比较实验动物和人的基因组,不仅可找到保守功能元件,包括蛋白编码基因和非编码序列,而且能发现基因功能的迁移,从而使研究者充分认识物种间遗传差异,避免选用不适当的动物模型和药物筛选方案。
构建直系同源关系依赖于可靠的系统发育重建。系统发育分析也提供一个估算选择压力和基因功能迁移的框架。可基于计算非同义核苷酸替换与同义核苷酸替换的比值 (dN/dS) 来估计自然选择的特征和范围[25,26]。选择性清除 (selective sweep) 是由于近期较强的正向自然选择,一个突变位点相邻DNA上的核苷酸之间的差异下降或消除,引起核苷酸多态性模式的显著改变。在群体水平,可用选择性清除评价自然选择的特征和范围。
旁系同源基因是由基因倍增产生的同一物种内的同源基因,其对药物发现也有重要作用。旁系同源基因的产物可作为候选的药物靶点[27],通过结构预测、分子对接、虚拟筛选和实验手段从中获得依从性好的药靶,也有助于充分考虑候选先导化合物的选择性问题。多效性 (pleiotropy) 即多功能性,指基因或其产物具有多种表型效应或有多种功能[28]。在药物发现过程中需要充分了解旁系同源基因与药靶基因的多效性和功能冗余之间的进化关系,因为多效性和功能冗余是评价蛋白质成药性 (druggability) 时必须考虑的因素。
与多效性和功能冗余相关的概念是基因对话 (crosstalk) 和异价同作用基因 (heteromery)。对话是指不同信号路径或调控路径组成成分的相互作用使得一条路径的输入信号影响到另一路径的输出信号。异价同作用是指相似但是不同的大分子形成了物理联系[29],例如一对蛋白质亚单位形成异二聚体行使特定功能,此概念相对于同价同作用 (homomery) 概念而言。对话和异价同作用在药物研发中已受到关注,它们和基因转录的可变剪接都被用来解释源于基因组的功能的组合多样性。以上多种基因到功能的投射都能以路径和网络的进化观念来解读,系统发育基因组学方法可检测路径和网络的组成成员的共进化 (协同进化) 模式,从而有助于功能解析。
系统发育基因组学方法可用于研究可能药靶的进化性质,包括进化速率、选择压力、旁系同源的程度和特征,以及反映基因多效性的因素,包括蛋白质尺寸 (高保守蛋白质通常较大)、基因表达的广度以及互作潜力等。基于上述思考,Searls[24]提出了药用 基因组亲缘学 (pharmacophylogenomics) 概念。Wang等[30]研究了与亚细胞定位有关的药靶蛋白的进化特征。比较人、猕猴、小鼠和大鼠可成药的基因组,找到1 362个直系同源基因,发现位于细胞核的8.4% 在进化上比位于细胞膜 (33%) 和细胞外 (10.5%) 的药靶基因更保守。对于进化上不太保守的候选靶点,可利用人化的动物模型开发药物或疫苗。严重的毒性反应或副作用会导致药物撤出市场,意味着药物开发失败,原因主要是脱靶 (off-target) 和多靶点等。值得注意的是,撤出市场药物的靶点多位于细胞膜和细胞外。与靶点位于细胞核的药物相比,开发这类药物的难度稍小。今后应加强药物透膜机制研究,发展新的药物传递策略。Hutchins等[31]预测了50个进化水平迥异的生物基因组中包含的泛素酶 (UBE) 和去泛素酶 (DUB),找到91个UBE/DUB直系同源基因。由于所涉及物种数量较大,直系/旁系同源基因的预测较可靠。UBE/DUB抑制剂组成的化合物库可以用靶蛋白进行筛选,故该研究预测的各物种UBE/DUB可成为药用基因组亲缘学分析的工具箱。预测结果能帮助识别直系同源基因的功能迁移,帮助深入分析互作分子的共进化、基因多效性和功能冗余,这些问题在药物开发流程中推断物种差异、选择适合的药靶和疾病动物模型时必须考虑到。药用基因组亲缘学认为分析进化速率可帮助识别候选药靶。Gladki等[32]认为理想的抗生素药靶在进化上受限,经受严格的纯化选择,即药靶蛋白的突变通常因为有害而被选择作用移除,这样便不易产生细菌耐药性。此点已被药靶进化速度的分析所证实。pN代表物种内某基因的非同义 (碱基变化引起相应氨基酸替换) 多态性,pS代表物种内同义 (碱基变化未导致氨基酸替换) 多态性,pN/pS是通过种内多样性比较考查纯化选择的指标。发现8种致病菌药靶基因的pN/pS显著低于各自基因组平均值,也低于由实验方法识别的基本基因 (essential gene,与非基本基因比较,进化速度明显较慢; 图 2) 的pN/pS。用dN/dS可比较来自不同种菌的直系同源基因,结果有类似pN/pS的发现,提示药靶进化较慢,而且进化速率比基本性 (essentiality) 能更好地预测蛋白质成药性(drugability)。慢进化基因中不仅包含熟知的抗生素药靶,例如核糖体蛋白、拓扑异构酶、RNA聚合酶和旋转酶,也包括尚未作为药靶的蛋白,例如琥珀酸脱氢酶和金属结合蛋白等,值得深入研究。
 |
Figure 2 Distribution of antibiotic drug target genes in genomes of eight pathogenic bacteria. Antibacterial drugs include cycloserine, triclosan, trimethoprim,sulphonamides,fusidicacid,mupirocin,quinolones,rifampin, macrolides,tetracyclines,glacycyline,retapamulin,aminoglycosides,β-lactam antibiotics and fosfomycin[32]. ω = dN/dS |
Searls[24]提出的药用基因组亲缘学是针对药靶的研究,实际上药用基因组亲缘学在药用植物领域可以有多方面的应用,例如: ① 基于基因组信息构建不同尺度的生命之树,明确药用植物类群间的系统发育关系; ② 利用基因组数据进行时间估算和地理分布区重建,推测现存药用植物/道地药材的起源和空间分布格局及其成因; ③ 基于时间树,结合生态、环境因素及进化创新性状,探讨药用植物的多样化进程和成因; ④ 揭示药用植物多样性的来源和格局,基于生物多样性探讨药用化合物 (例如次级代谢产物) 多样性,实现创新药物发现; ⑤ 预测药用植物多样性动态变化,提出相应的保护性开发策略。由于针对药用植物的基因组亲缘研究的内容完全不同于Searls原初的概念,似乎可用pharmaphylogenomics区别于药用基因组亲缘学 (pharmacophylogenomics)。
本文作者整合形态分类数据、化学分类数据、分子生物学数据、生物活性数据和传统药物学知识,探索以毛茛科Ranunculaceae等药用植物科属为重点的药用亲缘学。毛莨科乌头属 (Aconitum L.) 全世界约有300余种,其中超过半数分布在中国。发现牛扁亚属subgen. Lycoctonum是以牛扁碱和C18-二萜生物碱为主的类群,由于其毒性中等,因而是寻找镇痛、抗炎等新药的一个对象[33]。乌头亚属subgen. Aconitum下唐古特乌头系ser. Tangutica和圆叶乌头系set. Rotund ifolia是以内酯型二萜生物碱为主的类群,毒性较小,是新药寻找的重点研究类群。褐紫乌头系ser. Brunnea则以C20-二萜生物碱如光翠雀碱和宋果灵为主,杂有高度进化的乌头碱型二萜生物碱如乌头碱等成分。化学分类上不支持其独立成为一个分支。显柱乌头系set. Stylosa是以含大茴香酸酯基的乌头碱型二萜生物碱以及塔拉萨敏和查斯曼宁胺醇类为主的类群,是块根较大的“大乌头”的主要来源,具大毒。乌头系ser. Inflata以含15-羟基的单酯、双酯或多酯以及胺醇类乌头碱型二萜生物碱为主,且酯基中无大茴香酸酯基,此系是草乌的主要植物来源,具大毒,应十分慎用。显柱乌头系、乌头系、兴安乌头系ser. Ambigua和蔓乌头系ser. Volubilia可能代表乌头亚属进化的类群。从二萜生物碱化学成分来看,露蕊乌头亚属subgen. Gymnaconitum与乌头属另外两亚属差别很大,结合分子系统发育研究、形态学和细胞学结果,支持其为一单独属,介于翠雀属和乌头属之间 (图 3)[34]。基于细胞核和叶绿体DNA序列的分子系统树将形态极相近的九系分为两群[35],一为甘青乌头系、圆叶乌头系、保山乌头系和短柄乌头系; 另一为乌头系、兴安乌头系、显柱乌头系、蔓乌头系和准噶尔乌头系,化学分类数据支持此分群。为了乌头资源的可持续利用和发现高效低毒的新化合物,有必要将近年涌现的新技术用于乌头研究。系统生物学和组学技术将在促进乌头生物活性化合物研究中发挥关键作用。
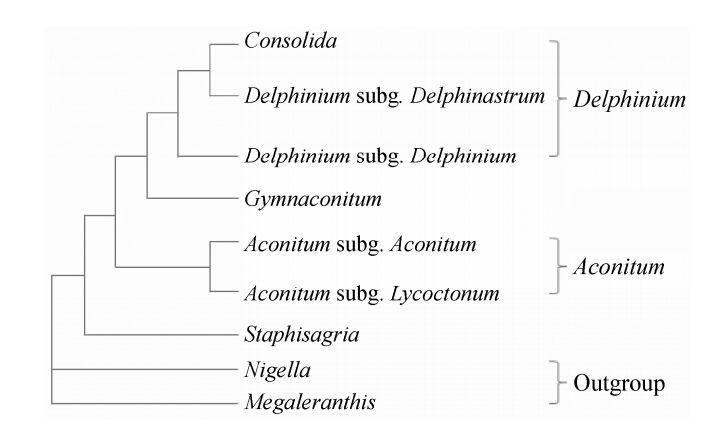 |
Figure 3 Cladogram of the Ranunculaceae tribe Delphinieae,modified from ref. [34]. Gymnaconitum and Staphisagria were regarded as the subgenus of Aconitum and Delphinium respectively. Consolida,usually treated as an independent genus,could belong to the genus Delphinium |
毛茛科是一个比较原始的植物类群,已知代表性药用化合物包括苄基异喹啉生物碱、毛茛甙、三萜皂苷和二萜生物碱等。结合近年植物化学研究进展,对毛茛科所含主要化学成分类型和分布进行了系统归纳总结[36]。毛茛甙和木兰花碱在一些属 (例如毛茛属、铁线莲属和驴蹄草属等) 共存,而非交替出现[37]。结合了疗效数据[38]的药用亲缘分析[36,39,40],支持Wang等[41]基于分子标记和形态数据提出的分类系统。毛茛科可分为5个亚科: 毛茛亚科Ranunculoideae、唐松草亚科Thalictroideae、黄连亚科Coptidoideae、黄毛茛亚科Hydrastidoideae和白根葵亚科Glaucidioideae。毛茛亚科可分为十族。铁线莲属与白头翁属和银莲 花属均属于银莲花族,均含较多五环三萜皂苷; 铁线莲属还含有黄酮、花青素、木质素、香豆素和生物碱等[42],其中五环三萜皂苷已用于化学分类研究。升麻族中,类叶升麻属Actaea和升麻属Cimicifuga的疗效和化学成分相近,因此两属的亲缘关系也近[43]。由于它们果实的形态差异,以及细胞学特征不同,考虑这两属为升麻族植物的一个分支,且类叶升麻属较升麻属更为进化。从化学分类学的角度来看,铁破锣属Beesia含有特殊铁破锣皂苷可以成为一个独立的分支。基于细胞核ITS序列的系统发育树支持以上分 析[44]。黄三七属Souliea既和铁破锣属一样含有五环三萜和铁破锣型环阿尔廷烷 (环菠萝蜜烷) 四环三萜类,又和升麻属、类叶升麻属一样含有吲哚生物碱,因此认为它是铁破锣属和升麻属、类叶升麻属之间的一个过渡类型[36,43]。近年药用亲缘研究还涉及百合科贝母属[45]、冬青科冬青属[46]、五味子科[47]和唇形科鼠尾草属[48]等。
药用植物亲缘学研究药用植物的植物亲缘关系、化学成分和疗效 (传统疗效和药理活性) 间的相关性[49],交叉性和涉及多学科是其特点,故这门学科的成长需要开放地吸收有关领域的新知识和新技术。Pharmaphylogenomics扩展了药用亲缘学研究的内涵,可视为药用亲缘学的升级版,将有力推动药用植物资源的开发和可持续利用。
致谢: 中国中医科学院中药研究所陈士林研究员给予指导和支持。
| [1] | Hao DC, Xiao PG, Huang BL, et al. Interspecific relation-ships and origins of Taxaceae and Cephalotaxaceae revealed by partitioned Bayesian analyses of chloroplast and nuclear DNA sequences [J]. Plant Syst Evol, 2008, 276: 89-104. |
| [2] | Hao DC, Huang BL, Yang L. Phylogenetic relationship within the genus Taxus inferred from protein-coding genes and non-protein-coding DNA sequences [J]. Biol Pharm Bull, 2008, 31: 260-265. |
| [3] | Hao DC, Xiao PG, Peng Y, et al. Research progress and trend analysis of biology and chemistry of Taxus medicinal resources [J]. Acta Pharm Sin (药学学报), 2012, 47: 827-835. |
| [4] | Zhu M, Xiao PG. Chemosystematic studies on Thalictrum L. in China [J]. Acta Phytotaxo Sin (植物分类学报), 1991, 29: 358-369. |
| [5] | Hofberger JA, Lyons E, Edger PP, et al. Whole genome and tandem duplicate retention facilitated glucosinolate pathway diversification in the mustard family [J]. Genome Biol Evol, 2013, 5: 2155-2173. |
| [6] | Manrique-Carpintero NC, Tokuhisa JG, Ginzberg I, et al. Sequence diversity in coding regions of candidate genes in the glycoalkaloid biosynthetic pathway of wild potato species [J]. G3 (Bethesda), 2013, 3: 1467-1479. |
| [7] | Dutartre L, Hilliou F, Feyereisen R. Phylogenomics of the benzoxazinoid biosynthetic pathway of Poaceae: gene duplications and origin of the Bx cluster [J]. BMC Evol Biol, 2012, 12: 64. |
| [8] | Takos AM, Knudsen C, Lai D, et al. Genomic clustering of cyanogenic glucoside biosynthetic genes aids their identification in Lotus japonicus and suggests the repeated evolution of this chemical defence pathway [J]. Plant J, 2011, 68: 273-286. |
| [9] | Cosner ME, Raubeson LA, Jansen RK. Chloroplast DNA rearrangements in Campanulaceae: phylogenetic utility of highly rearranged genomes [J]. BMC Evol Biol, 2004, 4: 27. |
| [10] | Henriquez CL, Arias T, Pires JC, et al. Phylogenomics of the plant family Araceae [J]. Mol Phylogenet Evol, 2014, 75: 91-102. |
| [11] | Wu CS, Chaw SM, Huang YY. Chloroplast phylogenomics indicates that Ginkgo biloba is sister to cycads [J]. Genome Biol Evol, 2013, 5: 243-254. |
| [12] | Malé PJ, Bardon L, Besnard G, et al. Genome skimming by shotgun sequencing helps resolve the phylogeny of a pantropical tree family [J]. Mol Ecol Resour, 2014, doi: 10.1111/1755-0998.12246. |
| [13] | Xi Z, Ruhfel BR, Schaefer H, et al. Phylogenomics and a posteriori data partitioning resolve the Cretaceous angiosperm radiation Malpighiales [J]. Proc Natl Acad Sci USA, 2012, 109: 17519-17524. |
| [14] | Barrett CF, Specht CD, Leebens-Mack J, et al. Resolving ancient radiations: can complete plastid gene sets elucidate deep relationships among the tropical gingers (Zingiberales)? [J]. Ann Bot, 2014, 113: 119-133. |
| [15] | He K, Jiang XL. Sky islands of southwest China. I: an overview of phylogeographic patterns [J]. Chin Sci Bull, 2014, 59: 585-597. |
| [16] | Whittall JB, Syring J, Parks M, et al. Finding a (pine) needle in a haystack: chloroplast genome sequence divergence in rare and widespread pines [J]. Mol Ecol, 2010, 19 (S1): 100-114. |
| [17] | Koonin EV, Wolf YI. Evolution of microbes and viruses: a paradigm shift in evolutionary biology? [J]. Front Cell Infect Microbiol, 2012, 2: 119. |
| [18] | Kohonen P, Nera KP, Lassila O. Avian model for B-cell immunology -new genomes and phylotranscriptomics [J]. Scand J Immunol, 2007, 66: 113-121. |
| [19] | Hao DC, Ma P, Mu J, et al. De novo characterization of the root transcriptome of a traditional Chinese medicinal plant Polygonum cuspidatum [J]. Sci China Life Sci, 2012, 55: 452-466. |
| [20] | Chan CX, Zäuner S, Wheeler G, et al. Analysis of Por-phyra membrane transporters demonstrates gene transfer among photosynthetic eukaryotes and numerous sodium-coupled transport systems [J]. Plant Physiol, 2012, 158: 2001-2012. |
| [21] | Villar M, Popara M, Mangold AJ, et al. Comparative proteomics for the characterization of the most relevant Amblyomma tick species as vectors of zoonotic pathogens worldwide [J]. J Proteomics, 2013, 105: 204-216. |
| [22] | Martin DI, Singer M, Dhahbi J, et al. Phyloepigenomic comparison of great apes reveals a correlation between somatic and germline methylation states [J]. Genome Res, 2011, 21: 2049-2057. |
| [23] | Brindefalk B, Ettema TJ, Viklund J, et al. A phylometage-nomic exploration of oceanic alphaproteobacteria reveals mitochondrial relatives unrelated to the SAR11 clade [J]. PLoS One, 2011, 6: e24457. |
| [24] | Searls DB. Pharmacophylogenomics: genes, evolution and drug targets [J]. Nat Rev Drug Discov, 2003, 2: 613-623. |
| [25] | Hao DC, Yang L, Huang BL. Molecular evolution of paclitaxel biosynthetic genes TS and DBAT of Taxus species [J]. Genetica, 2009, 135: 123-135. |
| [26] | Hao DC, Xiao PG. Prediction of sites under adaptive evolution in flavin-containing monooxygenases: selection pattern revisited [J]. Chin Sci Bull, 2011, 56: 1246-1255. |
| [27] | Manhas R, Tripathi P, Khan S, et al. Identification and functional characterization of a novel bacterial type asparagine synthetase A: a tRNA synthetase paralog from Leishmania donovani [J]. J Biol Chem, 2014, 289: 12096-12108. |
| [28] | Guillaume F, Otto SP. Gene functional trade-offs and the evolution of pleiotropy [J]. Genetics, 2012, 192: 1389-1409. |
| [29] | Gomes I, Fujita W, Gupta A, et al. Identification of a μ-δ opioid receptor heteromer-biased agonist with antinociceptive activity [J]. Proc Natl Acad Sci USA, 2013, 110: 12072-12077. |
| [30] | Wang X, Wang R, Zhang Y, et al. Evolutionary survey of druggable protein targets with respect to their subcellular localizations [J]. Genome Biol Evol, 2013, 5: 1291-1297. |
| [31] | Hutchins AP, Liu S, Diez D, et al. The repertoires of ubiquitinating and deubiquitinating enzymes in eukaryotic genomes [J]. Mol Biol Evol, 2013, 30: 1172-1187. |
| [32] | Gladki A, Kaczanowski S, Szczesny P, et al. The evolutionary rate of antibacterial drug targets [J]. BMC Bioinformatics, 2013, 14: 36. |
| [33] | Xiao PG, Wang FP, Gao F, et al. A pharmacophylogenetic study of Aconitum L. (Ranunculaceae) from China [J]. Acta Phytotaxo Sin (植物分类学报), 2006, 44: 1-46. |
| [34] | Wang W, Liu Y, Yu SX, et al. Gymnaconitum, a new genus of Ranunculaceae endemic to the Qinghai-Tibetan Plateau [J]. Taxon, 2013, 62: 713-722. |
| [35] | Hao DC, Gu XJ, Xiao PG, et al. Recent advances in the chemical and biological studies of Aconitum pharmaceutical resources [J]. J Chin Pharm Sci, 2013, 22: 209-221. |
| [36] | Hao DC, Xiao PG, Ma HY, et al. Mining chemodiversity from biodiversity: pharmacophylogeny of medicinal plants of the Ranunculaceae [J]. Chin J Nat Med (中国天然药物), 2014, In press. |
| [37] | Xiao PG. A preliminary study of the correlation between phylogeny, chemical constituents and pharmaceutical aspects in the taxa of Chinese Ranunculaceae [J]. Acta Phytotaxo Sin (植物分类学报), 1980, 18: 142-153. |
| [38] | Xiao PG, Wang LW, Lü SJ, et al. Statistical analysis of the ethnopharmacologic data based on Chinese medicinal plants by electronic computer I. Magnoliidae [J]. Chin J Integr Tradit West Med (中西医结合杂志), 1986, 6: 253-256. |
| [39] | Peng Y, Chen SB, Chen SL, et al. Preliminary pharmaphy-logenetic study on Ranunculaceae [J]. China J Chin Mater Med (中国中药杂志), 2006, 31: 1124-1128. |
| [40] | Peng Y, Chen SB, Liu Y, et al. Pharmaphylogenetic study on Isopyroideae (Ranunculaceae) [J]. China J Chin Mater Med (中国中药杂志), 2006, 31: 1210-1214. |
| [41] | Wang W, Lu AM, Ren Y, et al. Phylogeny and classifica-tion of Ranunculales evidence from four molecular loci and morphological data [J]. Persp Plant Ecol Evol Syst, 2009, 11: 81-110. |
| [42] | Hao DC, Gu XJ, Xiao PG, et al. Chemical and biological research of Clematis medicinal resources [J]. Chin Sci Bull, 2013, 58: 1120-1129. |
| [43] | Gao JC, Peng Y, Yang MS, et al. A preliminary pharmaco-phylogenetic study of tribe Cimicifugeae (Ranunculaceae) [J]. J Syst Evol, 2008, 46: 516-536. |
| [44] | Hao DC, Gu XJ, Xiao PG, et al. Recent advances in chemical and biological studies on Cimicifugeae pharmaceutical resources [J]. Chin Herb Med, 2013, 5: 81-95. |
| [45] | Hao DC, Gu XJ, Xiao PG, et al. Phytochemical and biological research of Fritillaria medicinal resources [J]. Chin J Nat Med (中国天然药物), 2013, 11: 330-344. |
| [46] | Hao DC, Gu XJ, Xiao PG, et al. Research progress in the phytochemistry and biology of Ilex pharmaceutical resources [J]. Acta Pharm Sin B, 2013, 3: 8-19. |
| [47] | Xu LJ, Liu HT, Peng Y, et al. A preliminary pharmacophy-logenetic investigation in Schisandraceae [J]. J Syst Evol, 2008, 46: 692-723. |
| [48] | Li MH, Li Q, Liu Y, et al. Pharmacophylogenetic study on plants of genus Salvia L. from China [J]. Chin Herb Med, 2013, 5: 164-181. |
| [49] | Chen SB, Peng Y, Chen SL, et al. Introduction of pharmaphylogeny [J]. World Sci Technol/Modern Tradi Chin Med Mater Med (世界科学技术-中医药现代化), 2005, 7: 97-103. |
 2014, Vol. 49
2014, Vol. 49


