2. 山西大学 化学化工学院, 太原 030006;
3. 地产中药功效物质研究与利用山西省重点实验室, 太原 030006
2. College of Chemistry and Chemical Engineering of Shanxi University, Taiyuan 030006, China;
3. Key Laboratory of Effective Substances Research and Utilization in TCM of Shanxi Province, Taiyuan 030006, China
代谢组学是继基因组学和蛋白质组学之后新近发展起来的一门科学,是系统生物学的重要组成部分,是以生物体液、细胞、组织或器官中内源性代谢物在时空上的动态变化为研究目的,并探索代谢物与生理病理变化相对关系的新兴科学[1]。其中,非靶标代谢组学旨在从全局角度分析生物样本中所有的代谢物质的轮廓,系统辨识能特征表征机体时空变化规律的差异代谢物。
能够全面正确地辨识出差异代谢物是非靶标代谢组学的目的,也是进一步分析代谢通路和阐明组间差异生物学意义的关键。但是由于内源性代谢物种类繁多,浓度动态范围大,代谢物识别的准确性不高。另外,对于代谢物组这样复杂的研究体系,理想的检测分析方法应具备无偏向性、良好的分辨率和重现性、高灵敏度和系统性、样本制备的简易性和高通量分析可操作性等优点,而现有的检测手段很难达到。另外,代谢组学研究产生的海量数据及其生物学信息挖掘,也是代谢组学研究现存的难点和热点。本文从代谢组学的研究流程出发,从样本的前处理到代谢通路分析综述了非靶标代谢组学各个步骤现阶段研究中的问题以及应对策略。
1 样本的前处理生物样本的前处理是代谢组学的第1步,包括样本的收集、储存和制备3个过程。
1.1 样本的收集血样是临床检测及病理学研究最常用的生物样品。根据是否加入抗凝剂分为血浆与血清2种。有研究[2]表明,血清和血浆在中药药理学研究中差异颇大。Yu等[3]通过比较血浆与血清样本中共有的122个代谢物的含量,发现血清中104种代谢物的浓度显著高于血浆,血浆样本的重现性优于血清样本。同时该研究还指出,只有严格按照相同的标准程序收集制备样品,才能得到相似的临床与生物研究结果。此外,抗凝剂种类的选择对血浆的代谢轮廓影响也颇大。Zhou等[4]以135个代谢产物评价不同抗凝剂对血浆的影响,发现肝素钠所制血浆样本代谢物种类最多,是最佳的非靶标代谢组学研究样本。
相对于血样而言,尿样具有易采集,非破坏性,可采集量大,前处理过程简单等优势,且尿样一般不需要去蛋白处理。因尿样采集时间较长,为保证样品质量,可以通过加入防腐剂或者控制采集样本的环境温度以保证样本质量。Roux等[5]研究发现,叠氮化钠能有效抑制尿液样品中的细菌增长,但不能避免代谢物的化学降解,而采集样品温度的控制则对细菌的繁殖和代谢物的稳定都有效果。
1.2 样本的储存样本的储存对其代谢轮廓的影响较收集小,林璋[6]发现不同保存时间的血浆样品的峰面积的RSD仅为3%。而凝结时间和抗凝剂等差异造成的RSD均超过40%。但是存储时间过长也会影响样本质量,Yang等[7]研究发现,-80 ℃下保存了5年的样本中36种代谢物的含量发生了显著改变,其中3种未知代谢物的含量变化甚至过百倍。
1.3 样本的制备样本制备过程中,不规范的制备过程容易造成信息的丢失,进而影响实验的重复性。此外,评价指标的不同也会影响样本制备方法的优选。如代谢轮廓的峰容量、强度和峰面积总和等评价方法。
血样的制备有沉淀蛋白、液液萃取、固相萃取等方法。液液萃取及固相萃取过程较烦琐,不适用于大批量备样,因此非靶标代谢组学研究过程中,为尽量多获取信息,血样一般采用沉淀蛋白法处理。徐婧[8]利用RPLC-ESI-MS平台,以血浆中常见的16个代谢物提取率为评价指标,考察了溶剂种类、体积和温度等因素对血样沉淀蛋白的影响。结果表明,以3倍量的冷藏乙腈提取效果最佳。
尿液样本个体体积差异大,其代谢物浓度易受饮水量和尿液量的影响。针对该问题,已有多种解决办法:(1)肌酐测试;该方法简单,但是备样方法复杂。(2)同位素标记;该方法操作简单,缺点是成本较高。(3)冻干后粉末称量;该方法操作简单,成本低。基于这些情况,尿液样本在制备中建议先测定尿肌酐确认取样量后,再进行冻干处理,以消除饮水量和尿液量造成的差异[9]。
与体液样本相比,组织样本能更直观地说明病理状态的改变,然而组织样本种类多,成分复杂,不同的前处理过程获取的信息也有所差异。组织器官的前处理方法一般包括组织均匀化和代谢物提取2步。均匀化可以人工研磨液氮处理的组织样本,也可解冻后用组织匀浆机处理[10-11];代谢物提取一般采用有机溶剂萃取。Wang等[12]利用大鼠食道癌组织系统考察了不同的前处理方法,结果表明,就低水平代谢物的提取效率以及重复性而言,逐步加入二氯甲烷、甲醇、水萃取方法优于其他萃取方法。组织萃取法目前有一步萃取法和两步萃取法,但是有研究[13]表明,用两步萃取法处理得到的样品,无论是水相还是有机相,所含的信息量都较多。因此,两步萃取法目前使用较普遍。
近年来,代谢组学研究全面发展,已从宏观延伸至微观,从体液、组织到细胞乃至亚细胞器,通过研究细胞中发生的动态代谢过程,揭示细胞器与胞浆之间的代谢物传递规律。细胞样本的制备过程包括细胞淬灭及细胞内代谢物的提取。常用的细胞淬灭剂有60%预冷甲醇或者是外加不同缓冲溶剂,然而Dietmair等[14]研究表明,60%预冷甲醇会导致细胞内容物外泄,而用冷等渗盐水(0.9%氯化钠,0.5 ℃)淬灭可以有效地阻止ATP转化为ADP和AMP,且不破坏细胞。此外,50%的冷乙腈所提取的胞内代谢物的效果最佳。梁琼麟[15]从细胞中提取线粒体,研究线粒体中代谢物组的变化规律,有助于进一步阐释细胞内的物质传递及其对生物机制的作用。
2 分析方法学的确证代谢组学样本复杂,成分之间相互干扰,必须构建客观的方法学考察其前处理和分析方法的可靠性。使用高通量检测技术进行代谢组学研究时,随着时间的变化,仪器性能的改变会造成分析结果的偏差。为了监测实验过程中仪器的性能,有研究者提出随机排列样本检测的顺序并制备质控样本(QC样本)[16]。QC样本可以是代表性物质的混合对照品,也可以是等量待分析样品均匀混合的pooled QC样本[17]。使用QC样本进行质控时,正式开始实验前需要进5~10针,稳定仪器状态;在分析序列中间也要随机地插入QC样本,用来监测整个分析序列的稳定性。
选择合适的QC样本后,如何用QC样本来保证方法的稳定性呢?FDA提出可以通过计算QC样本中特征分子的RSD来判断分析序列的稳定性[18]。另外,也有通过稀释QC样本,检测其线性关系、基质效应及精密度以验证其可靠性,但是这种方法不适合样本中低含量的物质。除此之外,Spagou等[19]建议可以集合QC样本中特征代谢物的保留时间和质荷比的变异度来监控分析方法的稳定性。非监督的主成分分析(PCA)也可用来评价分析方法的可靠性,QC样本在得分图空间中的聚集程度可以体现其一致性。但若待测样品的差异性较大,会掩盖仪器系统不稳定及色谱柱老化等原因造成的QC样本的微小差异。因此,Spagou等[19]建议,采用PCA的得分图考察QC样本的稳定性时,应同时结合第一主成分时间序列进行相关性分析。
除此之外,在分析过程中还应考虑分析的规模。Zelena等[20]通过比较同一批样本分批和不分批测试的结果,发现不分批测试前后存在明显的分界线,而分批测试结果样本间无明显差异。结合本实验室以往的经验,建议样本量大于90时,应多批次进行分析。
3 数据处理代谢组学研究所产生的海量原始数据形成一个庞大的数据集,常规的数据处理方法无法实现全面的信息挖掘,容易造成信息丢失。质谱数据的预处理包括峰识别、去卷积、峰对齐、归一化、标尺化等,核磁数据预处理包括基线校正、相位调整、归一化和标尺化等。预处理时应根据需要选择相应的处理过程,本文就数据预处理中常见的峰识别、峰对齐、归一化和标尺化4个问题进行探讨(图 1)。
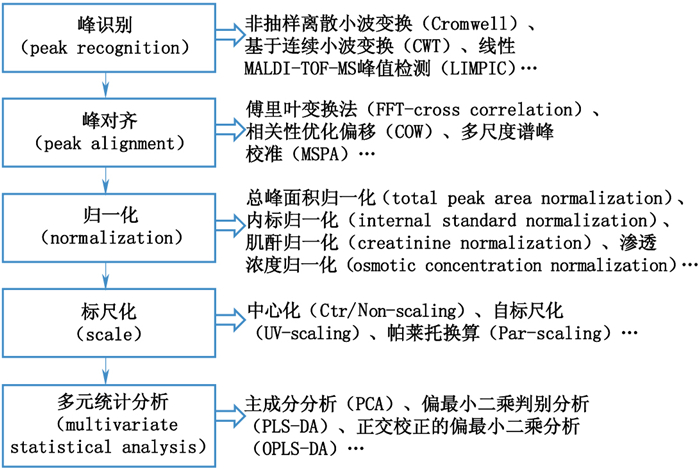
|
图 1 代谢组学数据分析流程图 Figure 1 The flow of metabolomics data analysis |
数据预处理首先需要将原始图谱转换成数据矩阵,也就是色谱峰识别过程。色谱峰识别的算法很多,主要涉及色谱峰平滑、基线校正以及峰提取3步,不同的算法有不同的原理和应用。Yang等[21]利用MALDI MS数据对现有的峰值检测算法进行分类研究,显示现有算法之间存在差异和相似之处。研究还选择了Cromwell、CWT、LMS、LIMPIC和PROcess 5种典型的峰值检测算法比较仿真数据和真实的实验研究数据,其中CWT(基于连续小波变换的算法)[22]处理效果最好。
核磁数据则需先进行分段积分,以降低原始数据维度并进行归一化和标准化。分段积分有等步长与不等步长之分,等步长积分操作简单,但积分区段需要谨慎选择,太宽会造成多峰落入1个区间,太窄会使得到的高维数据难以运算。相比而言,不等步长积分可避免化合物特征峰落入不同区段,但操作较烦琐[23]。可以使用Metlab编程进行不等步长积分,避免化合物的特征峰落入不同的积分区段内。也有研究提出可根据数据特点自行划分积分区间[24]和自适应分段积分方法等[25]。
由于仪器、pH、温度、盐浓度、样本稀释度等因素均可造成谱峰的化学位移或保留时间漂移现象,因此峰对齐是代谢组学数据预处理中的另一必要步骤。现有的谱峰对齐算法很多,包括快速傅里叶变换法(FFT-cross correlation)、COW(correlation optimized warping)[26]和MSPA(multiscale peak alignment)等[27],但是由于其运行速度慢,不适用于代谢组学数据处理。黄赫等[28]在分析了多种峰对齐的方法的特点后,提出了一种基于高斯平滑的谱峰对齐算法(peak alignment based on gaussian smoothing,GPA),与COW和MSPA 2种常用对齐算法相比,GPA对齐效果更好,时间复杂度适中,且对齐后数据的平均相关系数更高,有利于后续的多变量建模。
数据归一化可以消除或减轻由于采集过程中的明显差异(如尿液体积)所致的代谢物浓度的不均一性。常用的归一化方法有总峰面积归一化、内标归一化、肌酐归一化、渗透浓度归一化及质量归一化等,各方法特点见表 1。王丹丹等[29]提出实验过程中应选择3种或以上的归一化方法,比较模型参数,选择合适的方法,确保结果的准确性。另外,也有研究优化传统的归一化方法,以取得更好的归一化效果,如董继扬等[30]提出了一种基于问题相关性的迭代归一化法,降低无关谱峰及基线部分的权重计算归一化系数;黄赫等[31]提出一种基于代谢物统计的归一化算法,通过排除差异性较显著的代谢物,将剩余代谢物进行归一化,提高了数据归一化精度,促进后期的模式识别及标志物寻找。
|
|
表 1 归一化方法分类及特点 Table 1 Classification and characteristics of normalization methods |
经过归一化以后的代谢组学数据需进行数据标准化,将变量的强度统一标准。数据标准化的方法一般有中心化(Ctr/Non-scaling)、自标尺化(UV-scaling)[32]以及帕莱托换算(Par-scaling)[33]等,但是现有的这些尺度缩放方法只是从数据本身出发,并未考虑对后续多元统计分析的影响,因此,其多元统计分析的结果往往并不理想。基于此,刘新卓等[34]提出一种模型自适应的标准化方法(model adaptive scaling,MAS),即针对所采用的多元统计分析建模方法,给出适合该多元统计模型的最优标准化方法。
随着代谢组学及多元统计分析方法的深入发展,各仪器公司及课题研究组也相继开发出辅助处理代谢组学数据的软件,如可用于处理质谱联用数据的Progenesis QI、XCMS、MZmine、Metalign、Metaboanalysist、R packages等,可处理NMR数据的MestreNova、Topsin、Nuts、Mest Re-C等。这些软件特点不同,一定程度上均可实现海量数据的批量处理。但各种软件算法不同,且各种算法特点不同。Koh等[35]发现用不同软件预处理后得到的数据矩阵所构建的OPLS-DA模型的预测能力存在差异。Lu等[36]比较了AMDIS(NIST)、chmmaTOF和AnalyzerPro软件,发现它们在谱峰识别和鉴定上存在很大不同。因此,尚需研发更有效、更自动化、更灵活可靠的软件来分析代谢组学的海量数据。针对此问题,王敏等[37]建议在实际工作中选择数据处理软件时应慎重,可运用多种处理软件进行交互验证,另外也可手动核对信息,以确保数据预处理的准确度。
4 代谢物的鉴定在非靶标代谢组学研究中,代谢物鉴定是非常重要的一步,也是现阶段技术瓶颈之一。代谢物鉴定的问题主要表现在代谢物数量庞大,而标准物质非常有限(仅5 000种左右)[38]。根据Metabolomics Standards Initiative发布的代谢物定性层次,鉴定的成分至少需要2个相互独立且正交的数据相互验证。
代谢物鉴定可以分为数据库依赖的鉴定方法及非数据库依赖的鉴定方法。GC-MS和NMR可以通过标准图库可供谱图检索定性,而LC-MS则没有通用的数据库,仅通过精确相对分子质量以及可能产生的碎片离子会给鉴定带来极大的不确定性。近年来快速发展的超高分辨率质谱,为化合物确定分子式带来极大的便利。另外,数据库的完善对于化合物的鉴定也非常重要。目前可用于LC-MS代谢物鉴定的数据库有HMDB、Massbank、METLIN等,各数据库特点不同,如HMDB在使用过程中需人为确定分子离子峰,METLIN[39]可自动确定。其中,HMDB因其综合了大量的文献数据,包括内源性代谢物及多种蛋白的详细信息,因此使用最为广泛。在设定好的误差范围内筛选可能的化合物,但是由于碎片信息及保留时间在不同的仪器间难以重现,容易出现漏解、误解的情况。非数据依赖的鉴定方法根据其精确相对分子质量、同位素比例及保留时间等推测其结构,Fiehn研究小组提出的Seven Golden Rules,是预测代谢物分子式比较经典的方法,研究者[40]发现Pub Chem数据库中仅0.6%的化合物不符合该规则。另外,在非数据库依赖的鉴定过程中,也开发了一些预测离子碎裂规律的软件,如Mass Frontier、MetFrag等,但是所能鉴定的代谢物也非常有限。
为准确鉴定尽可能多的代谢物,需要技术和数据库两方面的共同发展。一方面,在保证分辨率的情况下提高仪器的稳定性;另一方面,完善标准物数据库,建立更为稳健的质谱智能解析和谱图预测算法,更为全面的谱图树和数据库,真正实现自动化和半自动化的化合物结构鉴定。
5 代谢通路分析非靶标代谢组学通路分析目前面临的最大问题是每个代谢物参与多个代谢途径,无法准确将代谢物含量的变化定位至目标代谢通路中。现有方法主要从3个方面解决该问题。
首先,以传统的生物数据库如KEGG通路为基础,将差异代谢物在通路中进行富集分析[41]。目前常用的代谢通路数据库有KEGG、Reactome、SMPDB等。其中KEGG应用最广泛,可以从基因、蛋白、代谢物不同层次进行关联分析;Reactome汇集了20多种模式生物的通路数据库,在整体水平上为研究者提供了对代谢通路进行探索研究的工具;SMPDB中大多数的通路是其独有的,且每个小分子都可以链接到DrugBank或HMDB等数据库[42]。现有多种算法和软件实现了代谢通路的富集分析和拓扑分析(MetaboAnalyst、reporter reactions和MetaboNetworks等),但仍不能辨识其在代谢通路的定位。
其次,将差异代谢物置于生物体的代谢网络中,以代谢产物和反应为节点,试图找到代谢物的子链接清单[41]。
最后,近年来稳定同位素示踪技术(stable isotope assisted metabolomics,SIAM)[43]的引入为问题的解决提供了新的思路,将有助于内源性代谢物在机体内的识别、定量及其代谢途径的定位分析。稳定同位素示踪技术可利用同位素作为示踪剂标记1种或多种类型的内源性代谢物,通过质谱仪、核磁共振仪等分析仪器测定其反应后的位置、数量等变化,从而定位其代谢途径。
Salabei等[44]利用蛋白质印记法以及同位素示踪技术发现了2型糖尿病糖代谢途径与正常代谢的不同,同时结合相关通路上所涉及酶或基因的表达差异,精准辨识该分子所参与的生化途径。除葡萄糖以外,代谢组学结合SIAM还可鉴定其他重要的代谢物。Jain等[45]利用代谢组学结合C-13标记甘氨酸,发现其对促进肿瘤细胞株的生长起到非常重要的作用。由此可见,代谢组学结合SIAM这一分析方法在判别生物体系的代谢变化规律上有着独特的优势。
6 展望非靶标代谢组学的发展逐渐成熟,但仍存在诸多不足。深入地研究解决代谢组学中的技术问题,建立1套完整、规范、合理的研究规范,保证数据的可靠性以及重现性是目前需要解决的问题。本文结合本实验室的经验及近年来的文章,综述分析了非靶标代谢组学各个研究过程中的影响因素并给出规范化操作建议。
按照规范化操作建议系统全面找出差异代谢物,其准确鉴定还需建立全面的标准化数据库。此外,新技术的应用也有利于代谢组学的发展,如SIAM的引入实现了代谢通路的准确定位。
随着科学技术的发展,大数据时代的到来,将各种组学技术、分析测试技术以及数据处理方法相整合,互相取长补短,在得到的海量数据中挖掘有用的信息,并且针对某特定种类或特定代谢物进行研究,发展靶标代谢组学是未来代谢组学发展的趋势。
| [1] |
NICHOLSON JK, LINDON JC, HOLMESE. 'Metabonomics':understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data[J]. Xenobiotica, 1999, 29(11): 1181. DOI:10.1080/004982599238047 |
| [2] |
刘林.中药含药血浆与血清有效成分比较及血浆药理学方法研究[D].长沙: 湖南中医药大学, 2016 LIU L.Comparison Active Ingredients of Drug-containing Plasm and Serum and Research for Chinese Medicine Plasma Pharmacology Method[D].Changsha: Hunan University of Chinese Medicine, 2016 |
| [3] |
YU Z, KASTENMULLER G, HE Y, et al. Differences between human plasma and serum metabolite profiles[J]. PloS One, 2011, 6(7): e21230. DOI:10.1371/journal.pone.0021230 |
| [4] |
ZHOU Z, CHEN Y, HE J, et al. Systematic evaluation of serum and plasma collection on the endogenous metabolome[J]. Bioanalysis, 2017, 9(3): 239. DOI:10.4155/bio-2016-0078 |
| [5] |
ROUX A, THEVENOT E A, SEGUIN F, et al. Impact of collection conditions on the metabolite content of human urine samples as analyzed by liquid chromatography coupled to mass spectrometry and nuclear magnetic resonance spectroscopy[J]. Metabolomics, 2015, 11(5): 1095. DOI:10.1007/s11306-014-0764-5 |
| [6] |
林璋.基于色谱质谱联用的代谢组学样本收集处理程序标准化和代谢综合征研究[D].长沙: 中南大学, 2014 LIN Z.Standardization of Collection and Pretreatment for Metabolomics Samples and Metabolic Syndrome Study Based on Chromatography-Mass Spectrometry[D].Changsha: Central South University, 2014 |
| [7] |
YANG W, CHEN Y, XI C, et al. Liquid chromatography-tandem mass spectrometry-based plasma metabonomics delineate the effect of metabolites stability on reliability of potential biomarkers[J]. Anal Chem, 2013, 85(5): 2606. DOI:10.1021/ac303576b |
| [8] |
徐婧.基于RRLC-MS/MS技术的食管癌代谢组学研究[D].北京: 北京协和医学院, 2013 XU J.RRLC-MS/MS-Based Metabolomics of Esophageal Cancer[D].Beijing: Peking Union Medical College, 2013 |
| [9] |
彭国茳.基于尿液代谢组学的逍遥散抗抑郁临床疗效分析[D].太原: 山西大学, 2015 PENG GJ.Clinical Antidepressant Effect of Xiaoyaosan in Depressed Patients Using Urinary Metabolomics[D].Taiyuan: Shanxi University, 2015 |
| [10] |
LY-VERDU S, SCHAEFER A, KAHLE M, et al. The impact of blood on liver metabolite profiling-a combined metabolomic and proteomic approach[J]. Biomed Chromatogr, 2014, 28(2): 231. DOI:10.1002/bmc.3010 |
| [11] |
CHINTAPALLI VR, BRATTY MA, KORZEKWA D, et al. Mapping an atlas of tissue-specific Drosophila melanogaster metabolomes by high resolution mass spectrometry[J]. PLoS One, 2013, 8(10): e78066. DOI:10.1371/journal.pone.0078066 |
| [12] |
WANG H, XU J, CHEN Y, et al. An optimization and evaluation strategy of esophageal tissue preparation protocols for metabolomics by LC-MS[J]. Anal Chem, 2016, 88(7): 3459. DOI:10.1021/acs.analchem.5b04709 |
| [13] |
贾红梅.基于代谢组学方法的柴胡疏肝散配伍规律及抗抑郁药效成分研究[D].北京: 中国医学科学院、北京协和医科大学药用植物研究所, 2013 JIA HM.Metabonomics Based Study on the Compatibility Rule and Medicinal Composition Antidepressant Effect of Chaihu Shugan[D].Beijing: Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences & Peking Union Medical College, 2013 |
| [14] |
DIETMAIR S, TIMMINS NE, GRAY PP, et al. Towards quantitative metabolomics of mammalian cells:development of a metabolite extraction protocol[J]. Anal Biochem, 2010, 404(2): 155. DOI:10.1016/j.ab.2010.04.031 |
| [15] |
梁琼麟.亚细胞代谢组学分析方法及其在线粒体能量代谢研宄中的应用[C]//中国化学会第30届学术年会摘要集-第四十三分会: 质谱分析.大连: 中国化学会, 2016: 1 LIANG QL.Analysis of subcellular metabolomics and its application in mitochondrial energy metabolism[C]//Proceedings of the 30 Chinese Chemical Society Annual Symposium.Dalian: Chinese Chemistry Society, 2016: 1 |
| [16] |
GIKA HG, THEODORIDIS GA, WINGATE JE, et al. Within-day reproducibility of an HPLC-MS-based method for metabonomic analysis:application to human urine[J]. J Proteome Res, 2007, 6(8): 3291. DOI:10.1021/pr070183p |
| [17] |
SANGSTER T, MAJOR H, PLUMB R, et al. A pragmatic and readily implemented quality control strategy for HPLC-MS and GC-MS-based metabonomic analysis[J]. Analyst, 2006, 131(10): 1075. DOI:10.1039/b604498k |
| [18] |
VISWANATHAN CT, BANSAL S, BOOTH B, et al. Quantitative bioanalytical methods validation and implementation:best practices for chromatographic and ligand binding assays[J]. Pharm Res, 2007, 24(10): 1962. DOI:10.1007/s11095-007-9291-7 |
| [19] |
SPAGOU K, WILSON ID, MASSON P, et al. HILIC-UPLC-MS for exploratory urinary metabolic profiling in toxicological studies[J]. Anal Chem, 2010, 83(1): 382. |
| [20] |
ZELENA E, DUNN WB, BROADHURST D, et al. Development of a robust and repeatable UPLC-MS method for the long-term metabolomic study of human serum[J]. Anal Chem, 2009, 81(4): 1357. DOI:10.1021/ac8019366 |
| [21] |
YANG C, HE Z, YU W. Comparison of public peak detection algorithms for MALDI mass spectrometry data analysis[J]. BMC Bioinformatics, 2009, 10(1): 4. |
| [22] |
熊智新.基于小波变换的化学谱图数据处理[D].杭州: 浙江大学, 2004 XIONG ZX.Chemograms Data Processing Through Wavelet Transform[D].Hangzhou: Zhejiang University, 2004 |
| [23] |
SMOLINSKA A, BLANCHET L, BUYDENS LM, et al. NMR and pattern recognition methods in metabolomics:from data acquisition to biomarker discovery:a review[J]. Anal Chim Acta, 2012, 750: 82. DOI:10.1016/j.aca.2012.05.049 |
| [24] |
ANDERSON PE, REO NV, DELRASO NJ, et al. Gaussian binning:a new kernel-based method for processing NMR spectroscopic data for metabolomics[J]. Metabolomics, 2008, 4(3): 261. DOI:10.1007/s11306-008-0117-3 |
| [25] |
MEYER TD, SINNAEVE D, GASSE BV, et al. NMR-based characterization of metabolic alterations in hypertension using an adaptive, intelligent binning algorithm[J]. Anal Chem, 2008, 80(10): 3783. DOI:10.1021/ac7025964 |
| [26] |
CHRISTIN C, SMILDE AK, HOEFSLOOT HC, et al. Optimized time alignment algorithm for LC-MS data:correlation optimized warping using component detection algorithm selected mass chromatograms[J]. Anal Chem, 2008, 80(18): 7012. DOI:10.1021/ac800920h |
| [27] |
ZHANG ZM, LIANG YZ, LU HM, et al. Multiscale peak alignment for chromatographic datasets[J]. J Chromatogr A, 2012, 1223: 93. |
| [28] |
黄赫, 邓伶莉, 周玲, 等. 一种新的核磁共振谱峰对齐算法[J]. 光电技术应用, 2013, 28(2): 51. HUANG H, DENG LL, ZHOU L, et al. New algorithm for peak alignment of nuclear magnetic resonance[J]. Electro-Optic Technol Appl, 2013, 28(2): 51. DOI:10.3969/j.issn.1673-1255.2013.02.014 |
| [29] |
王丹丹, 李萍, 刘丽芳, 等. 基于色谱质谱联用的尿液代谢组学归一化方法[J]. 中国新药杂志, 2016, 25(20): 2336. WANG DD, LI P, LIU LF, et al. Normalization strategies for urine metabolomic analysis using chromatography-mass spectrometry technology[J]. Chin J New Drugs, 2016, 25(20): 2336. |
| [30] |
董继扬, 李伟, 邓伶莉, 等. 核磁共振代谢组学数据的尺度归一化新方法[J]. 高等学校化学学报, 2011, 32(2): 262. DONG JY, LI W, DENG LL, et al. A new method of scale normalization for NMR metabonomics data[J]. Chem J Chin Univ, 2011, 32(2): 262. |
| [31] |
黄赫.核磁共振代谢组学谱峰对齐及数据归一化方法研究[D].厦门: 厦门大学, 2013 HUANG H.Study on Peak Alignment and Data Normalization of NMR Metabonomics[D].Xiamen: Xiamen University, 2013 |
| [32] |
van den BERG RA, HOEFSLOOT HC, WESTERHUIS JA, et al. Centering, scaling, and transformations:improving the biological information content of metabolomics data[J]. BMC Genomics, 2006, 7(1): 142. DOI:10.1186/1471-2164-7-142 |
| [33] |
ODUNSI K, WOLLMAN RM, AMBROSONE C B, et al. Detection of epithelial ovarian cancer using 1H-NMR-based metabonomics[J]. Int J Cancer, 2005, 113(5): 782. DOI:10.1002/(ISSN)1097-0215 |
| [34] |
刘新卓, 邓伶莉, 王婉兰, 等. 核磁共振波谱信号分析中的尺度缩放新算法[J]. 厦门大学学报(自然科学版), 2016, 55(4): 564. LIU XZ, DENG LL, WANG WL, et al. A new scaling algorithm for NMR signal analysis[J]. J Xiamen Univ(Nat Sci), 2016, 55(4): 564. |
| [35] |
KOH Y, PASIKANTI KK, YAP CW, et al. Comparative evaluation of software for retention time alignment of gas chromatography/time-of-flight mass spectrometry-based metabonomic data[J]. J Chromatogr A, 2010, 1217(52): 8308. DOI:10.1016/j.chroma.2010.10.101 |
| [36] |
LU H, LIANG Y, DUNN WB, et al. Comparative evaluation of software for deconvolution of metabolomics data based on GC-TOF-MS[J]. Trends Anal Chem, 2008, 27(3): 215. DOI:10.1016/j.trac.2007.11.004 |
| [37] |
王敏, 黄寅, 张伟, 等. 代谢组学信息获取与数据预处理瓶颈问题探讨[J]. 药学进展, 2014, 38(2): 81. WANG M, HUANG Y, ZHANG W, et al. Discussion on bottleneck problems in data acquisition and pre-processing of metabolomics[J]. Prog Pharm Sci, 2014, 38(2): 81. |
| [38] |
孔宏伟, 戴伟东, 许国旺. 基于液相色谱-质谱联用的代谢组学研究中代谢物的结构鉴定进展[J]. 色谱, 2014, 32(10): 1052. KONG HW, DAI WD, XU GW. Advances of metabolite identification in liquid chromatography-mass spectrometry based metabolomics[J]. Chin J Chromatogr, 2014, 32(10): 1052. |
| [39] |
SMITH CA, O'MAILLE G, WANT EJ, et al. Metlin:a metabolite mass spectral database[J]. Ther Drug Monit, 2005, 27(6): 747. DOI:10.1097/01.ftd.0000179845.53213.39 |
| [40] |
KIND T, FIEHN O. Seven golden rules for heuristic filtering of molecular formulas obtained by accurate mass spectrometry[J]. BMC Bioinformatics, 2007, 8(1): 105. |
| [41] |
XIA J, MANDAL R, SINELNIKOV Ⅳ, et al. MetaboAnalyst 2.0- a comprehensive server for metabolomic data analysis[J]. Nucl Acid Res, 2012, 40(W1): 27. |
| [42] |
FROLKIS A, KNOX C, LIM E, et al. SMPDB:The small molecule pathway database[J]. Nucl Acid Res, 2010, 38(Database issue): D480. |
| [43] |
FAN WM, LANE AN. NMR-based stable isotope resolved metabolomics in systems biochemistry[J]. J Biomol NMR, 2011, 49(3-4): 325. DOI:10.1007/s10858-011-9503-7 |
| [44] |
SALABEI JK, LORKIEWICZ PK, MEHRA P, et al. Type 2 diabetes dysregulates glucose metabolism in cardiac progenitor cells[J]. J Biol Chem, 2016, 291(26): 13634. DOI:10.1074/jbc.M116.722496 |
| [45] |
JAIN M, NILSSON R, SHARMA S, et al. Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation[J]. Science, 2012, 336(6084): 1040. DOI:10.1126/science.1218595 |
 2019, Vol. 39
2019, Vol. 39 
