2. 南京农业大学 动物医学院,南京 210095;
3. 江苏大学生命科学学院/食品与生物工程学院,镇江 212013;
4. 贵州省种畜禽种质测定中心,贵阳 550018;
5. 黔西市动物疫病预防控制中心,黔西 551500;
6. 西北农林科技大学动物医学院,杨凌 712100
2. College of Veterinary Medicine, Nanjing Agricultural University, Nanjing 210095, China;
3. School of Food and Biological Engineering/School of Life Sciences, Jiangsu University, Zhenjiang 212013, China;
4. Guizhou Testing Center for Livestock and Poultry Germplasm, Guiyang 550018, China;
5. Qianxi Animal Disease Control Center, Qianxi 551500, China;
6. College of Veterinary Medicine, Northwest A&F University, Yangling 712100, China
牛冠状病毒(bovine coronavirus, BCoV)属于冠状病毒科(Coronaviridae),β冠状病毒属(beta coronavirus),可感染肠道和呼吸道,临床上主要引起犊牛腹泻、成年牛出血性冬痢和牛呼吸道症状[1-2]。自1970年Mebus等首次在美国发现BCoV以来,BCoV在世界范围内广泛存在[3]。BCoV的高传染性及隐性带毒情况的存在,是导致BCoV广泛存在的重要原因[4-5]。据相关研究报道,BCoV在中国的感染率为69.05%,巴西为68.60%,日本为57.70%,韩国为39.80%,土耳其21.90%,伊朗7.2%[6-11]。BCoV主要通过粪便和鼻分泌物水平传播[12]。BCoV除了感染牛以外,已证实可以感染多种动物[9, 13]。Abi等[14]和Vijgen等[15]的研究表明,BCoV的HE蛋白RBD关键位点突变将会改变受体、宿主范围和趋向性,提示BCoV病可能具有人畜共患的潜力。近年来,随着我国畜牧业的迅速发展,BCoV的流行情况呈现逐年递增的趋势。此外,BCoV也常与其它病原(如牛星状病毒等)混合感染,给养牛业造成重大损失[16-17]。目前,国内暂无可用商业化疫苗及特效治疗药物,因此建立一种特异性强、灵敏快速的检测方法对于BCoV病的防控尤为重要。
BCoV为单股正链RNA病毒,分子大小为32 kb,主要编码5种蛋白:N蛋白、S蛋白、HE蛋白、M蛋白、E蛋白[18]。其中,N蛋白相对保守,是诊断的常用靶标[19]。同一种属的S蛋白相对保守,且S1蛋白中包含受体结合域(RBD)和N端结合域(NTD),是主要的抗原结合位点,可以诱导强烈的免疫反应,产生中和抗体[20-21]。因此S1蛋白可以作为检测BCoV抗体的理想抗原。
临床上常用的检测BCoV抗体的方法有血凝抑制试验、中和试验和酶联免疫吸附试验(ELISA)。中和试验耗时长、对试验人员要求较高,且需要操作病毒,但BCoV极难分离,国内仅西南大学有分离株报道,因此对BCoV感染/免疫牛的中和抗体水平尚不明确[14, 22]。ELISA检测方法以其特异性强、灵敏度高,操作简便等优点在临床上广泛使用[23-24]。目前报道的BCoV抗体ELISA检测方法都是利用原核表达N蛋白作为包被抗原[25]。原核表达的蛋白缺乏真核生物翻译后修饰和加工,可能产生无功能和错误折叠的蛋白质,无法与待检血清中抗体发生特异性反应,易造成假阴性结果[26]。昆虫杆状病毒作为真核表达系统,能对表达蛋白进行修饰,保持原有生物学活性[27]。因此,本研究以昆虫杆状病毒表达系统表达并纯化的S1蛋白作为包被抗原,建立了一种间接ELISA抗体检测方法,该方法与中和抗体检测结果高度一致,有望能成为替代中和试验的便捷方法。
1 材料与方法 1.1 材料1.1.1 病毒、细胞、菌株、临床样品 BCoV分离株由本实验室分离鉴定并保存;昆虫Sf9细胞、粉纹夜蛾High5细胞系、HRT-18细胞、BCoV阳性血清、BCoV阴性血清、标准牛病毒性腹泻病毒(bovine viral diarrhea virus, BVDV)、牛轮状病毒(bovine rotavirus, BRV)、牛星状病毒(bovine astroviruses, BAstV)、牛副流感病毒(bovine parainfluenza, BPIV)、传染性牛鼻气管炎病毒(infectious bovine rhinotracheitis virus, IBRV)、牛腺病毒(bovine adenovirus, BAV)阳性血清、鼠源抗BCoV多克隆抗体由江苏省农业科学院兽医研究所制作并保存,DH10Bac感受态细胞从上海唯地生物技术有限公司订购。303份牛血清从我国各省的牛场采集。
1.1.2 主要实验试剂 HindⅢ限制性核酸内切酶、BamHⅠ限制性核酸内切酶购自Thermo Fisher公司;FITC标记山羊抗小鼠IgG(H+L)、Bradford蛋白浓度测定试剂盒购自上海碧云天生物技术有限公司;FITC兔抗牛IgG(H+L)购自北京博奥森公司;兔抗牛IgG-HRP抗体购自Abcam公司;His-Tag Monoclonal Antibody、HRP-conjugated Affinipure Goat Anti-Mouse IgG(H+L) 购自Proteintech公司;转染试剂X-tremeGENETM HP DNA Transfection Reagent购自瑞士罗氏公司;5 mL His Trap HP柱购自Cytiva公司。
1.2 方法1.2.1 引物的合成 合成鉴定重组杆粒DH10Bac-pFastBacTM1-BCoV-S1的通用引物M13,引物序列见表 1。
|
|
表 1 M13F和M13R引物序列 Table 1 The primer sequences of M13-F and M13-R |
1.2.2 重组质粒pFastBacTM1-S1的鉴定(BamHⅠ—HindⅢ) 将BCoV的S1基因序列送至南京金斯瑞生物科技股份有限公司进行优化,S1基因序列的C端加了6个组氨酸(His)标签利于后期蛋白鉴定,N端加了蜂素信号肽(honeybee melittin signal peptide, Mels)利于蛋白分泌到胞外,获得高纯度蛋白。优化后的基因序列连接至pFastBacTM1载体上,获得pFastBacTM1-S1质粒。将质粒用BamHⅠ与HindⅢ酶进行双酶切验证。双酶切体系:10×Buffer 2 μL、核酸内切酶各1 μL、质粒1 μg,加ddH2O补至总体积20 μL,37 ℃金属浴酶切30 min。酶切后的产物进行琼脂糖凝胶电泳分析。
1.2.3 重组杆粒S1-Bacmid的构建 将质粒pFastBacTM1-S1转化入DH10Bac感受态细胞中,37 ℃、220 r·min-1复苏4 h。用无抗LB培养液以10、100、1 000倍稀释转化液,每个稀释度取100 μL涂于蓝白斑固体LB平板(平板含50 μg·mL-1卡那霉素、7 μg·mL-1庆大霉素、7 μg·mL-1四环素、40 μg·mL-1 X-gal、40 μg·mL-1 IPTG)。倒置于37 ℃恒温培养48 h。挑选白色单菌落重新划线于蓝白斑LB平板,37 ℃恒温培养24 h。经过2次蓝白斑纯化后,挑取白色单菌落于含有上述三种抗生素的LB培养基,37 ℃ 220 r·min-1培养箱培养。用M13引物进行菌液PCR扩增,扩增产物进行凝胶电泳分析。将符合大小的片段切胶送测序。提取质粒,获得重组杆粒S1-Bacmid。
1.2.4 S1蛋白的表达 用转染试剂X-tremeGENETM HP DNA Transfection Reagent将重组杆粒S1-Bacmid转染于Sf9细胞细胞,培养并观察细胞72 h,收集细胞上清液,即为P1代重组杆状病毒。重复传代至出现明显细胞病变,获得的重组杆状病毒避光于4 ℃短期保存或者-80 ℃长期保存。
1.2.5 间接免疫荧光鉴定S1蛋白 将Sf9细胞铺于24孔板中,待细胞汇合度到80%时,按0.1 MOI接毒,培养72 h。用预冷的无水乙醇固定细胞,4 ℃作用45 min,PBS洗3次。50 g·L-1脱脂乳37 ℃封闭2 h。PBS洗3次。用1 000倍稀释的6×His标签抗体37 ℃孵育1 h。PBS洗3次。500倍稀释的FITC标记山羊抗小鼠IgG 37 ℃孵育45 min。PBS洗3次。荧光显微镜观察。
1.2.6 Western blot鉴定S1蛋白 取上清培养物及细胞裂解液80 μL加上20 μL的5×loading buffer混匀,100 ℃水浴10 min制得蛋白样品。进行蛋白凝胶电泳。结束后,用干式转印仪将蛋白转移到NC膜上。50 g·L-1脱脂乳室温摇床封闭2 h。PBST洗3次。10 000倍稀释6×His标签抗体,室温摇床孵育2 h。PBST洗3次。20 000倍稀释HRP-羊抗鼠IgG抗体室温摇床孵育1 h。PBST洗3次。用滤纸将NC膜吸干,加化学发光试剂避光显影,通过化学发光成像仪器曝光。
1.2.7 S1蛋白的纯化及鉴定 准备300 mL的细胞活率90%以上、密度在2×106 cells·mL-1的High5细胞悬浮液,按0.1 MOI接毒P4代重组杆状病毒,于27 ℃、110 r·min-1摇床培养。当细胞活率降至60%~70%时,收集培养基上清备用。用5 mL His镍柱亲和层析进行蛋白纯化。蛋白纯化的步骤:10倍柱体积(CV)的ddH2O清洗镍柱;10倍CV的Binding buffer平衡镍柱;蛋白溶液经过镍柱;5倍CV的Binding buffer洗脱未结合或者结合不牢的蛋白;10倍CV的Elution buffer洗脱目的蛋白;收集各组分流出液制成蛋白样品,Western blot和SDS-PAGE鉴定纯化效果。
1.2.8 S1蛋白的超滤及鉴定 将S1蛋白加入50 ku超滤管中,补加5%甘油的PBS溶液至4 mL,3 000 r·min-1离心10 min;弃去外管内溶液;补加5%甘油的PBS溶液至原位置,离心后继续补加。经过反复多次换液,最终延长离心时间使溶液浓缩,用移液枪吸出经过超滤后蛋白。将蛋白样品用SDS-PAGE验证蛋白超滤效果。超滤后的蛋白用Bradford蛋白浓度测定试剂盒测定蛋白浓度,分装,每管5 μL冻于-80 ℃。
1.2.9 最佳抗原包被浓度及血清稀释度的确定 使用方正滴定法确定S1蛋白抗原最佳包被浓度和血清稀释度。使用碳酸盐包被液将S1蛋白进行倍比稀释,将其包被浓度稀释为2、1、0.5、0.25、0.125、0.062 5 μg·mL-1,每孔100 μL,4 ℃包被过夜;使用2%脱脂乳将BCoV阳性血清和阴性血清进行倍比稀释,稀释比例为1∶50、1∶100、1∶200、1∶400,每孔100 μL。每组3个重复。选择P/N较大,阴性血清OD450 nm(N)<0.2,阳性血清OD450 nm(P)趋近于1的条件作为最佳条件。
1.2.10 ELISA反应条件的优化 在“1.2.9”的基础上,对其余条件依次进行单一变量的筛选,逐步确定最佳反应条件。用5% 脱脂乳、2.5% 脱脂乳、1% 明胶、1% BSA、5% BSA作为封闭剂;封闭温度37 ℃,封闭时间分别设定为30、60、90、120、180 min;血清样品置于37 ℃孵育,血清作用时间分别设定为30、45、60、75、90 min;二抗稀释度分别设定为1∶5 000、1∶10 000、1∶15 000、1∶20 000、1∶25 000;酶标二抗置于37 ℃孵育,二抗作用时间分别设定为30、45、60、75 min;加入TMB显色液置于37 ℃,作用时间分别设定为5、8、10、13、15 min。每组3个重复,P/N最大值对应条件即为最佳条件。
1.2.11 临界值分析 根据最优反应条件,对75份BCoV阴性血清进行检测,测定其OD450 nm值,计算平均值(x)和标准差(s),利用正态分布法确定阴阳性临界值。
1.2.12 特异性试验 根据最优反应条件,对BVDV、BRV、BAstV、BPIV、IBRV、BAV的阳性血清进行检测,同时设置BCoV阳性与阴性对照。根据OD450 nm值,确定该方法的交叉情况。
1.2.13 敏感性试验 根据最优反应条件,将BCoV阳性血清分别进行分别进行1∶100、1∶200、1∶400、1∶800、1∶1 600、1∶3 200、1∶6 400、1∶12 800和1∶25 600倍稀释,同时设置BCoV阴性对照。根据OD450 nm值,判断该方法的敏感性。
1.2.14 重复性试验 批内重复性试验:根据最优反应条件,选取BCoV阳性、阴性血清各3份进行检测,测定OD450 nm值,计算批内变异系数。批间重复性试验:根据最优反应条件,采用不同批次制备的S1蛋白包被酶标板,选取BCoV阳性、阴性血清各3份进行检测,测定OD450 nm值,计算批间变异系数。
1.2.15 ELISA方法与间接免疫荧光试验(IFA)的一致性比较 将200 TCID50·100 μL-1的BCoV接于细胞密度为90%的HRT-18细胞。96 h后固定细胞板,按照“1.2.5”步骤进行IFA,检测40份牛血清样品。其中,本试验将2倍比稀释的牛血清作为一抗,100倍稀释的FITC兔抗牛IgG(H+L)作为二抗(能检出荧光的细胞孔对应牛血清最大稀释度≤1∶20的血清判定为阴性)。将能检出荧光的细胞孔对应牛血清最大稀释度检测结果作为IFA的结果,同步进行ELISA方法的检测。
1.2.16 ELISA方法与中和试验的一致性比较 根据ELISA结果,选择40份牛血清样品进行BCoV的血清中和试验。将牛血清进行56 ℃ 30 min灭活;用DMEM分别将血清从2-1进行2倍比稀释至2-9,BCoV病毒液稀释至200 TCID50·100 μL-1,取等量不同稀释度的血清与等量稀释好的病毒液混合,置于37 ℃ 5% CO2的培养箱中进行中和1 h;将密度为90%,培养于96孔板的HRT-18细胞用PBS溶液洗3遍后,加入中和后的溶液,吸附3 h;PBS洗3遍,加入150 μL·孔-1的维持液,观察5~7 d。由于BCoV感染HRT-18细胞无明显病变,因此采用IFA进行血清中和效价的判定。在IFA试验中,以BCoV鼠源抗BCoV多克隆抗体作为一抗,FITC标记山羊抗小鼠IgG(H+L)为二抗。将无荧光的细胞孔对应牛血清最大稀释度的检测结果作为中和试验结果(其中中和效价≤1∶4为阴性血清),同步进行ELISA方法的检测。
1.2.17 临床样品检测 采用本试验建立的ELISA方法,对303份临床牛血清样品进行检测,评价临床应用效果。
2 结果 2.1 重组质粒pFastBacTM1-S1的双酶切鉴定将重组质粒pFastBacTM1-S1用BamHⅠ酶与HindⅢ酶进行双酶切鉴定,S1的片段大小应为2 391 bp,载体大小为4 775 bp。结果如图 1,表明成功构建重组质粒pFastBacTM1-S1。
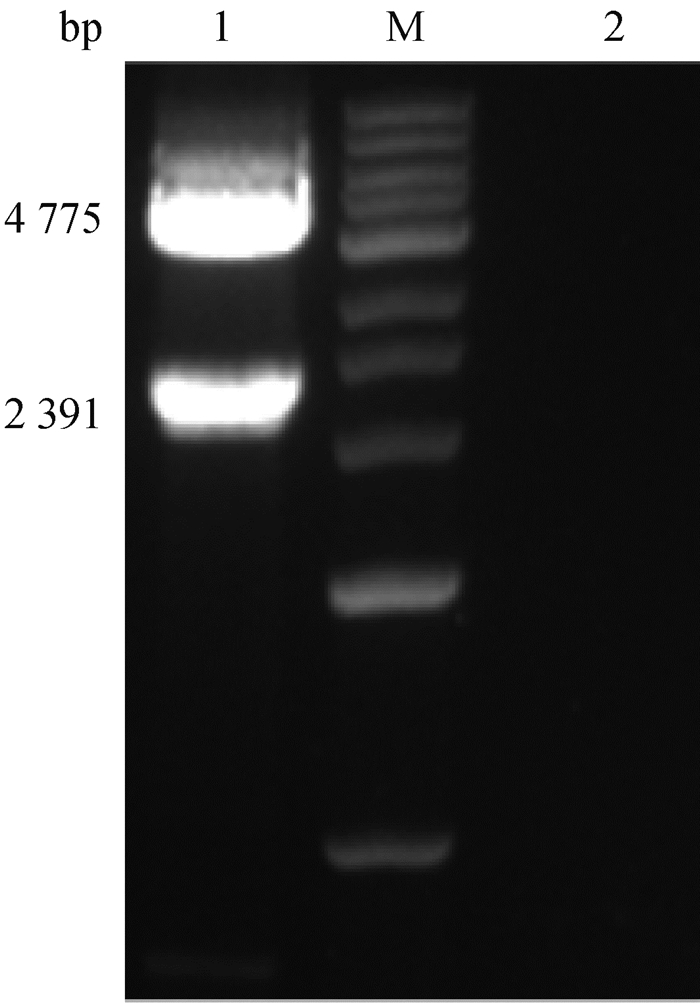
|
1. 双酶切质粒pFastBacTM1-S1;M. 1 kb DNA Ladder相对分子质量标准;2. 阴性对照 1. The pFastBacTM1-S1 plasmid was digested with enzyme; M. 1 kb DNA Ladder; 2: Negative control 图 1 重组质粒pFastBacTM1-S1双酶切鉴定 Fig. 1 Identification of recombinant plasmid pFastBacTM1-S1 |
用M13引物进行菌液PCR扩增鉴定,结果如图 2,1~8菌株中PCR扩增获得片段大小与预期大小相符(4 691 bp),成功构建重组杆粒;切胶块送测序,结果显示:3、4序列比对符合率99.8%,确定3、4菌株可用于后续试验。
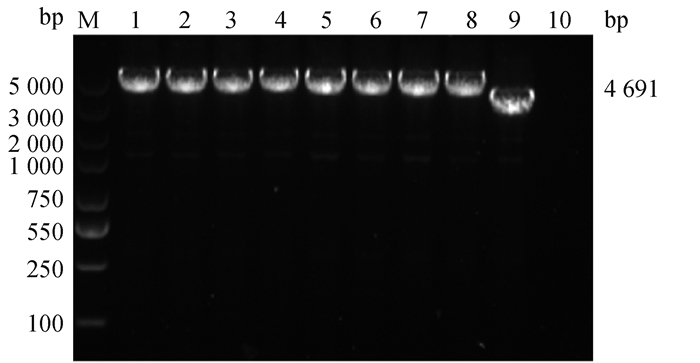
|
M. 1 kb DNA相对分子质量标准;1~9. 重组杆粒DH10Bac-pFastBacTM1-S1;10. 阴性对照 M. 1 kb DNA marker; 1-9. Recombinant bacmid DH10Bac-pFastBacTM1-S1; 10. Negative control 图 2 M13引物菌液PCR鉴定结果图 Fig. 2 Identification of recombinant bacterial by PCR |
2.3.1 重组杆状病毒的构建 重组杆粒DH10Bac-pFastBacTM1-S1转染Sf9细胞,收上清命名为P1代重组杆状病毒。传代接种,当用P3病毒液接毒Sf9细胞培养72 h后,显微镜下可见细胞出现变大、变圆,细胞脱落等病变情况,对照孔细胞均正常。
2.3.2 IFA鉴定S1蛋白在Sf9细胞的表达 用6×His标签抗体作为一抗,FITC标记的山羊抗小鼠IgG作为二抗进行IFA。如图 3,结果表明,接毒重组杆状病毒的Sf9细胞可见明显绿色荧光,正常Sf9细胞无绿色荧光,证明S1蛋白在Sf9细胞中表达。

|
A. 接毒的Sf9细胞;B. 正常Sf9细胞 A. Sf9 cells infected with Bacmid-S1; B. Sf9 cells 图 3 S1蛋白的间接免疫荧光鉴定(200×) Fig. 3 Identification of S1 protein by IFA (200×) |
2.3.3 Western blot鉴定S1蛋白在Sf9细胞的表达 用6×His标签抗体进行Western blot鉴定。如图 4,结果表明,目的蛋白大小约为100 ku,正常细胞无对应条带;表明S1蛋白在细胞和上清均可表达。
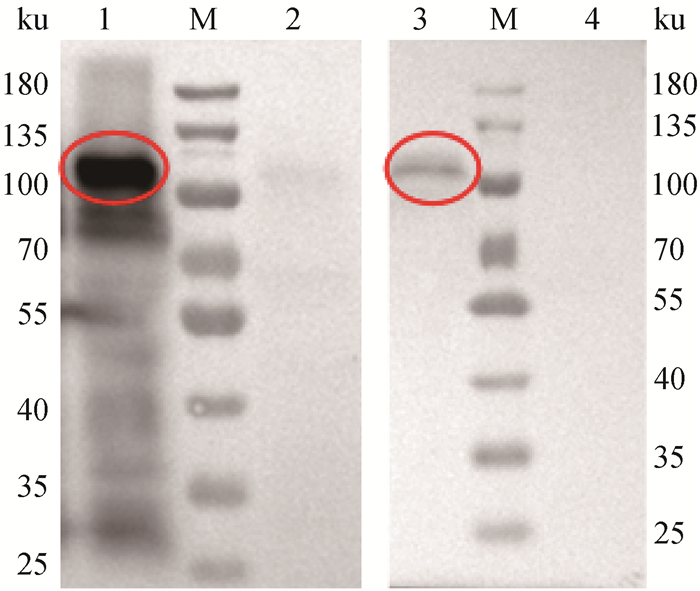
|
M. 蛋白质相对分子质量标准;1. 接毒Sf9细胞;2. 正常Sf9细胞;3. 接毒Sf9细胞培养上清;4. 正常Sf9细胞培养上清 M. Protein ladder; 1. Infected Sf9 cells; 2. Sf9 cells; 3. Supernatant of infected cells; 4. Supernatant of Sf9 cells 图 4 S1蛋白的Western blot鉴定 Fig. 4 Identification of S1 proteins by Western blot |
为了获得纯度高的蛋白,采用镍柱对培养上清液中的蛋白进行了纯化。经SDS-PAGE和Western blot检测,如图 5所示,结果表明,洗杂过程能除去部分杂蛋白和结合不牢的目的蛋白,洗脱过程能洗下结合于镍柱上的全部目的蛋白以及少量结合顽固的杂蛋白;目的蛋白主要洗脱在9、10和11管中。

|
A. S1蛋白的SDS-PAGE结果;B. 重组S1蛋白的Western blot结果;M. 蛋白质相对分子质量标准;1. 原液;2. 流穿液;3~6. 洗杂过程;7~12. 洗脱过程 A. Identification of protein S1 by SDS-PAGE; B. Identification of recombinant protein S1 by Western blot; M. Protein Ladder; 1. Protein solution; 2. Sample loading effluent; 3-6. Binding buffer effluent; 7-12. Elution buffer effluent 图 5 纯化蛋白S1的SDS-PAGE和Western blot鉴定 Fig. 5 Identification of the purification of the protein S1 |
超滤管浓缩后用SDS-PAGE验证蛋白的浓缩效果,如图 6。使用Image J软件对图像进行处理,计算对应条带灰度值,见表 2。结果表明,蛋白浓度明显增加。用Bradford蛋白浓度测定试剂盒测定蛋白浓度,最终获得500 μL蛋白浓度为0.46 mg·mL-1的S1蛋白。5 μL·管-1分装,冻存于-80 ℃。

|
M. 蛋白质相对分子质量标准;1. 纯化后蛋白;2. 浓缩后蛋白 M. Protein Ladder; 1. Purified protein; 2. Concentrated protein 图 6 SDS-PAGE验证浓缩效果图 Fig. 6 SDS-PAGE to verify enrichment effect |
|
|
表 2 条带灰度值比较表 Table 2 Comparison of gray values of the bands |
2.6.1 最佳抗原包被浓度和血清稀释度的确定 最佳抗原包被浓度和血清稀释度结果表明,本方法的最佳抗原包被浓度为0.125 μg·mL-1, 最佳血清稀释度为1∶200。
2.6.2 最佳封闭剂和封闭时间的确定 最佳封闭剂结果表明,最佳封闭剂为1%的明胶,最佳封闭时间为37 ℃条件下封闭3 h。
2.6.3 血清最佳反应时间的确定 血清最佳反应时间结果表明,血清最佳反应时间为37 ℃条件下孵育30 min。
2.6.4 酶标二抗最佳稀释度和最佳最佳反应时间的确定 酶标二抗最佳稀释度结果表明,酶标二抗最佳稀释度为1∶25 000,最佳反应时间为37 ℃条件下孵育45 min。
2.6.5 底物最佳反应时间的确定 底物最佳反应时间结果表明,底物最佳反应时间为37 ℃条件下避光显色13 min。
2.7 临界值分析根据最优反应条件,选取75份牛BCoV阴性血清进行间接ELISA检测OD450 nm,结果见图 7,计算得到OD450 nm平均值(x)为0.156,标准差(s)为0.047,根据正态分布法,确定x+3s为阴阳性临界值,即所测样品OD450 nm≥ x+3s=0.297为阳性;OD450 nm≤ x+2s=0.25为阴性,介于二者之间的为可疑。

|
图 7 间接ELISA临界值确定 Fig. 7 Definition of the critical value of the indirect ELISA method |
根据最优反应条件,对BVDV、BRV、BAstV、BPIV、IBRV、BAV的阳性血清进行检测,同时设置BCoV阳性与阴性对照,结果表明,其他病原的阳性血清OD450 nm均为阴性,本方法不与其他病原血清产生交叉反应,特异性良好。
2.9 敏感性试验根据最优反应条件,将BCoV阳性血清从1∶100倍进行2倍比稀释至1∶25 600倍,测OD450 nm。结果表明,BCoV阳性血清稀释3 200倍时,仍为阳性(OD450 nm>0.297),本方法的敏感性良好。
2.10 重复性试验根据最优反应条件,用3份BCoV阳性血清和3份BCoV阴性血清进行批内和批间重复试验。结果表明,批内变异系数在0.05%~6.67%之间,批间变异系数在1.20%~7.12%之间,均小于10%,本方法重复性良好。
2.11 ELISA方法与IFA的一致性比较对40份临床牛血清进行IFA和ELISA方法的检测。以IFA的结果作为参照,牛血清的ELISA检测结果见表 3。结果表明,当IFA检出荧光的牛阳性血清最大稀释度≥1∶160时,两者符合率为100.00%(24/24);当IFA的牛血清最大稀释度≤1∶40时,两者符合率为87.50%(7/8)。当临界值为OD450 nm为0.297时,ELISA的敏感性为96.88%(31/32),特异性为87.50%(7/8),与IFA具有很强的一致性(κ=0.844,P<0.01)。
|
|
表 3 不同血清的IFA最大稀释度与ELISA的一致性结果 Table 3 Results of agreement between IFA maximum dilutions and ELISA of different sera |
对40份牛血清进行中和是验和ELISA方法的检测。以中和试验结果为参照,牛血清的ELISA检测结果见表 4。结果表明:当血清中和效价≥1:16时,两者符合率为100.00%;当血清中和效价为1∶8时,两者符合率为87.50%;当血清中和效价为≤1∶4时,两者符合率为100.00%。当临界值为OD450 nm为0.297时,ELISA的敏感性为100.00%(31/31),特异性为88.89%(8/9),与血清中和试验具有很强的一致性(κ=0.925,P<0.01)。
|
|
表 4 不同血清中和效价与ELISA的一致性结果 Table 4 Results of agreement between neutralization titer of serum and ELISA of different sera |
采用本试验建立的方法对采集自4家牛场的303份牛血清进行临床检测。结果见表 5。其中227份血清为阳性,阳性率为74.91%。
|
|
表 5 样品检测结果 Table 5 Sample test results |
BCoV是一种高传染性且具有种间传播潜力的冠状病毒,对养牛业的持续发展和公共卫生造成威胁[28]。临床上无可用的疫苗和特效治疗药物,因此便捷、特异性强的检测方法对于疾病的防控具有重大意义。基于临床诊断过程的及时性和准确性原则,间接ELISA具有特异性强、可批量检测等优点,常被作为检测抗体的首选方法[29]。
ELISA的原理是抗原抗体的特异性结合,因此抗原的反应原性对于实验结果至关重要。研究表明,BCoV的S1亚基有主要的中和抗体表位,可以诱导强烈的免疫反应产生中和抗体[30]。因此S1基因可作为血清学诊断的候选基因。除此之外在ELISA试验中,血清的非特异性吸附会使得背景过高,影响试验的准确性,是特别需要注意的一点。在本次研究中发现,使用脱脂奶粉会使得本底背景过高,推测脱脂奶粉中的蛋白会与抗体产生交叉反应;更换1%的明胶后,背景值在正常范围内,因此本试验的最佳封闭剂确定为1%明胶。
基于以上,本研究首次采用昆虫杆状病毒表达系统表达S1蛋白,经过镍柱纯化和超滤浓缩,获得高纯度蛋白,后续以重组S1蛋白作为包被抗原进行ELISA方法构建。在构建方法的研究中,利用阳性血清OD450 nm读值P和阴性血清OD450 nm读值N比值作为标准,选取P/N最大值作为最佳条件。利用方正滴定法确定抗原最佳包被浓度和血清最佳稀释度,而后通过单一变量逐步进行优化。采用利用正态分布确定临界值,而后进行ELISA的特异性、敏感性、重复性、一致性试验,验证该方法的可靠性。特异性试验表明,S1蛋白抗原不与常见腹泻病原BVDV、BRV、BAstV、BAV的阳性血清发生非特异性结合;敏感性试验表明,血清的稀释比例为1∶3 200时,检测结果仍为阳性,表明该方法的敏感性较高;批间及批内重复性试验最大变异系数均小于10%,表明所建立的ELISA方法具有较好的重复性。在一致性试验中,本研究首次同时采用IFA和中和试验与ELISA进行一致性比较,在与IFA的一致性试验中,当临界值为OD450 nm为0.297时,ELISA的敏感性为96.88% (31/32),特异性为87.50%(7/8),与IFA具有很强的一致性(κ=0.844,P<0.01);在中和试验与ELISA的一致性比较中,当临界值为OD450 nm为0.297时,ELISA的敏感性为100.00%(31/31),特异性为88.89%(8/9),与血清中和试验具有很强的一致性(κ=0.925,P<0.01)。在两次一致性比较中,ELISA的特异性均低于敏感性,分析原因可能是在一致性比较中阴性样本总数过少导致。最后采用本试验建立的ELISA方法对4个牛场的303份临床牛血清进行检测,阳性率为74.91%,低于胡林杰的血清BCoV抗体的检出率98.84%,分析原因可能是不同地区BCoV的感染情况不同以及本次血清采样过于集中导致[31]。
综上所述,本研究通过杆状病毒表达系统成功表达BCoV的S1蛋白,以S1蛋白为包被蛋白成功建立BCoV的间接ELISA检测方法,该方法特异性、敏感性、重复性、一致性良好。有望用于临床牛感染情况调查,检测BCoV感染/免疫后中和抗体水平。
4 结论本研究首次用昆虫杆状病毒表达BCoV的S1蛋白,并成功建立间接ELISA检测方法;建立的ELISA方法特异性、敏感性、重复性、一致性良好;对临床牛血清样品进行检测,BCoV的抗体阳性率为74.91%。
| [1] |
SAIF L J. Bovine respiratory coronavirus[J]. Vet Clin North Am Food Anim Pract, 2010, 26(2): 349-364. DOI:10.1016/j.cvfa.2010.04.005 |
| [2] |
FEHR A R, PERLMAN S. Coronaviruses: an overview of their replication and pathogenesis[M]//MAIER H J, BICKERTON E, BRITTON P. Coronaviruses: Methods and Protocols. New York: Springer, 2015: 1-23.
|
| [3] |
TORRES-MEDINA A, SCHLAFER D H, MEBUS C A. Rotaviral and coronaviral diarrhea[J]. Vet Clin North Am Food Anim Pract, 1985, 1(3): 471-493. DOI:10.1016/S0749-0720(15)31297-4 |
| [4] |
TOFTAKER I, HOLMOY I, NODTVEDT A, et al. A cohort study of the effect of winter dysentery on herd-level milk production[J]. J Dairy Sci, 2017, 100(8): 6483-6493. DOI:10.3168/jds.2017-12605 |
| [5] |
COLLINS J K, RIEGEL C A, OLSON J D, et al. Shedding of enteric coronavirus in adult cattle[J]. Am J Vet Res, 1987, 48(3): 361-365. |
| [6] |
COURA F M, FREITAS M D, RIBEIRO J, et al. Longitudinal study of Salmonella spp., diarrheagenic Escherichia coli, Rotavirus, and Coronavirus isolated from healthy and diarrheic calves in a Brazilian dairy herd[J]. Trop Anim Health Prod, 2015, 47(1): 3-11. DOI:10.1007/s11250-014-0675-5 |
| [7] |
HE Q F, GUO Z J, ZHANG B, et al. First detection of bovine coronavirus in yak (Bos grunniens) and a bovine coronavirus genome with a recombinant HE gene[J]. J Gen Virol, 2019, 100(5): 793-803. DOI:10.1099/jgv.0.001254 |
| [8] |
KIM E M, CHO H C, SHIN S U, et al. Prevalence and genetic characterization of bovine coronavirus identified from diarrheic pre-weaned native Korean calves from 2019 to 2021[J]. Infect Genet Evol, 2022, 100: 105263. DOI:10.1016/j.meegid.2022.105263 |
| [9] |
LOTFOLLAHZADEH S, MADADGAR O, REZA MOHEBBI M, et al. Bovine coronavirus in neonatal calf diarrhoea in Iran[J]. Vet Med Sci, 2020, 6(4): 686-694. DOI:10.1002/vms3.277 |
| [10] |
MAWATARI T, HIRANO K, IKEDA H, et al. Surveillance of diarrhea-causing pathogens in dairy and beef cows in Yamagata Prefecture, Japan from 2002 to 2011[J]. Microbiol Immunol, 2014, 58(9): 530-535. DOI:10.1111/1348-0421.12174 |
| [11] |
SEVINC TEMIZKAN S, ALKAN F. Bovine coronavirus infections in Turkey: molecular analysis of the full-length spike gene sequences of viruses from digestive and respiratory infections[J]. Arch Virol, 2021, 166(9): 2461-2468. DOI:10.1007/s00705-021-05147-2 |
| [12] |
SAIF L J, REDMAN D R, MOORHEAD P D, et al. Experimentally induced coronavirus infections in calves: viral replication in the respiratory and intestinal tracts[J]. Am J Vet Res, 1986, 47(7): 1426-1432. |
| [13] |
ALEKSEEV K P, VLASOVA A N, JUNG K, et al. Bovine-like coronaviruses isolated from four species of captive wild ruminants are homologous to bovine coronaviruses, based on complete genomic sequences[J]. J Virol, 2008, 82(24): 12422-12431. DOI:10.1128/JVI.01586-08 |
| [14] |
ABI K M, ZHANG Q, ZHANG B, et al. An emerging novel bovine coronavirus with a 4-amino-acid insertion in the receptor-binding domain of the hemagglutinin-esterase gene[J]. Arch Virol, 2020, 165(12): 3011-3015. DOI:10.1007/s00705-020-04840-y |
| [15] |
VIJGEN L, KEYAERTS E, LEMEY P, et al. Evolutionary history of the closely related group 2 coronaviruses: porcine hemagglutinating encephalomyelitis virus, bovine coronavirus, and human coronavirus OC43[J]. J Virol, 2006, 80(14): 7270-7274. DOI:10.1128/JVI.02675-05 |
| [16] |
ZHU J, QI M P, JIANG C W, et al. Prevalence of bovine astroviruses and their genotypes in sampled Chinese calves with and without diarrhoea[J]. J Gen Virol, 2021, 102(8): 001640. |
| [17] |
ALFRED N, LIU H, LI M L, et al. Molecular epidemiology and phylogenetic analysis of diverse bovine astroviruses associated with diarrhea in cattle and water buffalo calves in China[J]. J Vet Med Sci, 2015, 77(6): 643-651. DOI:10.1292/jvms.14-0252 |
| [18] |
YOO D, PEI Y. Full-length genomic sequence of bovine coronavirus (31 kb). Completion of the open reading frame 1a/1b sequences[J]. Adv Exp Med Biol, 2001, 494: 73-76. |
| [19] |
YE Q Z, WEST A M V, SILLETTI S, et al. Architecture and self-assembly of the SARS-CoV-2 nucleocapsid protein[J]. Protein Sci, 2020, 29(9): 1890-1901. DOI:10.1002/pro.3909 |
| [20] |
WALLS A C, TORTORICI M A, SNIJDER J, et al. Tectonic conformational changes of a coronavirus spike glycoprotein promote membrane fusion[J]. Proc Natl Acad Sci U S A, 2017, 114(42): 11157-11162. DOI:10.1073/pnas.1708727114 |
| [21] |
LI F. Structure, function, and evolution of coronavirus spike proteins[J]. Annu Rev Virol, 2016, 3: 237-261. DOI:10.1146/annurev-virology-110615-042301 |
| [22] |
HASOKSUZ M, ALEKSEEV K, VLASOVA A, et al. Biologic, antigenic, and full-length genomic characterization of a bovine-like coronavirus isolated from a giraffe[J]. J Virol, 2007, 81(10): 4981-4990. DOI:10.1128/JVI.02361-06 |
| [23] |
TOFTAKER I, SANCHEZ J, STOKSTAD M, et al. Bovine respiratory syncytial virus and bovine coronavirus antibodies in bulk tank milk-risk factors and spatial analysis[J]. Prev Vet Med, 2016, 133: 73-83. DOI:10.1016/j.prevetmed.2016.09.003 |
| [24] |
TOFTAKER I, TOFT N, STOKSTAD M, et al. Evaluation of a multiplex immunoassay for bovine respiratory syncytial virus and bovine coronavirus antibodies in bulk tank milk against two indirect ELISAs using latent class analysis[J]. Prev Vet Med, 2018, 154: 1-8. DOI:10.1016/j.prevetmed.2018.03.008 |
| [25] |
刘合义, 孙留霞, 王进轶, 等. 牛冠状病毒重组N蛋白间接ELISA检测方法的建立[J]. 中国预防兽医学报, 2009, 31(8): 618-622. LIU H Y, SUN L X, WANG J Y, et al. Development of an indirect ELISA for the detection of Bovine coronavirus using recombinant N protein[J]. Chinese Journal of Preventive Veterinary Medicine, 2009, 31(8): 618-622. (in Chinese) |
| [26] |
KESIDIS A, DEPPING P, LODÉ A, et al. Expression of eukaryotic membrane proteins in eukaryotic and prokaryotic hosts[J]. Methods, 2020, 180: 3-18. DOI:10.1016/j.ymeth.2020.06.006 |
| [27] |
CHAMBERS A C, AKSULAR M, GRAVES L P, et al. Overview of the baculovirus expression system[J]. Curr Protoc Protein Sci, 2018, 91: 5. 4. 1-5. 4. 6. |
| [28] |
OMA V S, KLEM T, TRÅVÉN M, et al. Temporary carriage of bovine coronavirus and bovine respiratory syncytial virus by fomites and human nasal mucosa after exposure to infected calves[J]. BMC Vet Res, 2018, 14(1): 22. DOI:10.1186/s12917-018-1335-1 |
| [29] |
OHLSON A, BLANCO-PENEDO I, FALL N. Comparison of Bovine coronavirus-specific and Bovine respiratory syncytial virus-specific antibodies in serum versus milk samples detected by enzyme-linked immunosorbent assay[J]. J Vet Diagn Invest, 2014, 26(1): 113-116. DOI:10.1177/1040638713509377 |
| [30] |
VAUTHEROT J F, MADELAINE M F, BOIREAU P, et al. Bovine coronavirus peplomer glycoproteins: detailed antigenic analyses of S1, S2 and HE[J]. J Gen Virol, 1992, 73(Pt 7): 1725-1737. |
| [31] |
胡林杰, 孟野, 周玉龙, 等. 检测牛冠状病毒抗体间接ELISA方法的建立与应用[J]. 微生物学通报, 2020, 47(1): 330-338. HU L J, MENG Y, ZHOU Y L, et al. Establishment and application of indirect ELISA for detection of bovine coronavirus antibody[J]. Microbiology China, 2020, 47(1): 330-338. (in Chinese) |
(编辑 白永平)



