2. 河南省农业科学院畜牧兽医研究所, 郑州 450000
2. Institute of Animal Husbandry and Veterinary Medicine, Henan Academy of Agricultural Sciences, Zhengzhou 450000, China
从1977年Walter Gilbert和Frederick Sanger发明了第一台测序仪,并应用其测定了第一个基因组序列,到2001年人类基因组项目完成,由此开始,人类获得了探索生命遗传本质的能力,生命科学的研究进入了基因组学的时代[1]。至今四十余年,测序技术取得了相当大的发展,已从第一代测序技术发展到了第三代。在此过程中,出现了基因组、表观基因组、转录组、蛋白质组、代谢组、微生物组等新的组学测序技术[2]。这些单一层面的组学研究通过单一技术对单一分子的单一功能(遗传信息、蛋白功能或代谢通路)进行阐释。但仅通过单一组学数据很难对复杂的生物网络调控进行系统全面地解释,且不足以解释遗传信息表达调控的传递链条[3-4]。而多组学技术是一种全新的系统研究生物学的方法和技术,以无偏差的方式去整合基因组学、表观基因组学、转录组学、蛋白质组学、代谢组学及微生物组学等,从而系统解析畜禽重要经济性状的机理和表型,进而深度挖掘影响其性状的关键候选基因[5]。本文综述了多组学技术的发展、方法、优势及其在畜禽重要经济性状研究中的应用,以期为畜禽重要经济性状的研究提供参考。
1 测序技术的发展在过去的几十年,测序技术飞速发展(图 1)。1953年沃森和克里克发现DNA双螺旋结构,为测序技术的出现奠定了基础。1977年,Walter Gilbert和Frederick Sanger发明双脱氧DNA测序方法,被称为第一代测序技术。直到人类基因组计划的提出及实施,测序技术发展愈发迅猛[6]。发展到今天,除了以Illumina为代表的高通量测序技术外,还有蓄势待发的Pac Bio高通量测序技术[2]。
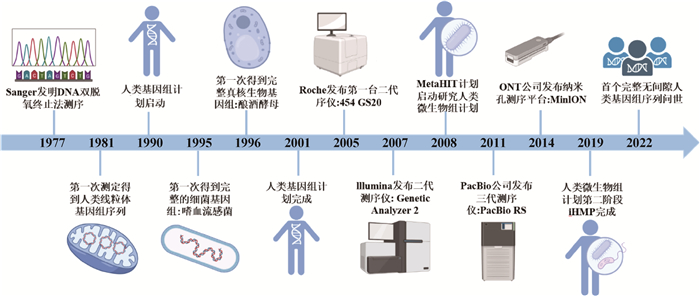
|
图 1 测序技术的发展历程 Fig. 1 Development history of sequencing technology |
第一代测序技术主要可以分成双脱氧链终止法(又称Sanger法)和化学降解法。这代测序技术准确率高、费用高、测序序列短[7]。主要应用于PCR产物测序、重测序等。下一代测序技术也称为新一代测序技术NGS,基于大规模平行测序技术(massive parallel analysis,MPS),主要以Roche公司的454、Illumina公司的Sloexa和ABI公司的SOLiD等平台为代表,它能同时完成测序模板互补链的合成和序列数据的获取。与第一代测序技术相比,下一代测序技术具有成本低、通量大、测序速度快等优点,是目前全基因组测序中应用最广泛的技术[8],主要应用于基因组测序、转录组测序、群体测序、扩增子测序、宏基因组测序、重测序等。但它的序列读长较短,Illumina平台最长为250~300 bp,454平台也只有500 bp左右;且由于在建库中利用了PCR富集序列,因此有一些含量较少的序列可能无法被大量扩增,造成一些信息的丢失。而第三代测序技术是指单分子测序技术,在测序时,不需要经过PCR扩增,实现了对每一条DNA分子的单独测序,也叫单分子实时DNA测序。它以Pacific Biosciences(PacBio)的Single Molecule Real-time(SMRT)、Illumina的Tru-Seq和Oxdord的Nanopore测序平台为代表,以单分子测序、纳米孔测序为特点,解决了测序读长短、对GC含量敏感、无法测出特殊插入序列和重复序列等问题,此方法可用于基因组组装、全长转录组测序、DNA甲基化分析等[9]。
2 单一组学技术随着技术的进步,各种组学技术不断出现,围绕中心法则(图 2),基因组学、表观基因组学、转录组学、蛋白质组学、代谢组学和微生物组学已被广泛应用于分析研究中。根据研究对象不同,组学技术研究策略不同,所依赖的技术手段也不尽相同(表 1)。
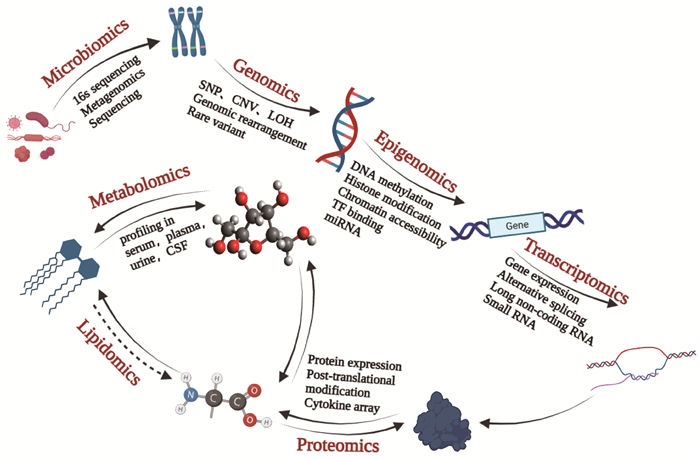
|
图 2 传统单一层面不同组学的中心法则应用 Fig. 2 The central law of different omics at the traditional single level |
|
|
表 1 单一组学技术及其研究方法 Table 1 Single-omics techniques and their research methods |
基因组学最早于1986年由美国遗传学家Thomas H. Roderick提出。基因组学主要研究基因组的结构、功能、进化、定位和编辑等,通过对个体及群体的所有基因进行定性定量分析,并进一步对不同个体及群体的全基因组信息进行比较分析,挖掘基因型与表型之间的关系[10],旨在阐明生物体完整DNA序列而不是单个基因的结构、进化和功能[11]。目前基因组学的研究方法主要包括基因组从头测序、重测序和简化基因组测序。2004年,发表在Nature上的一篇文章[12]利用从头测序方法对雌性红色原鸡进行全基因组测序,获得红色原鸡的基因组草图,为脊椎动物基因组进化提供了一个新的视角,同时也改进了哺乳动物基因组的注释。Groenen等[13]对雌性杜洛克猪的基因组序列进行组装和分析,得到了猪的第一个基因组序列图谱,为进一步改进这一重要牲畜物种提供了重要资源,使猪在农业生产和生物医学研究中得到有效利用。
2.2 表观基因组学表观基因组学是一门在基因组水平上研究表观遗传修饰的学科。表观遗传修饰作用于细胞内的DNA和其包装蛋白、组蛋白,用来调节基因组功能,表现为染色质可及性、DNA甲基化和组蛋白修饰,这些分子标志影响了染色体的架构、完整性和装配,同时也影响了DNA接近它的调控元件,以及染色质与功能型核复合物的相互作用能力[14]。虽然一个多细胞个体只有一个基因组,但是它具有多种表观基因组,表现为在生命的不同时期、健康或者受损的情况下,个体的细胞类型及其属性的多样性。例如,Li等[15]通过对猪肌肉组织和脂肪组织的miRNA表达进行比较,发现在猪皮下脂肪发育中存在一种复杂的表观遗传调控网络,为猪物种骨骼肌和脂肪组织的比较miRNA谱图添加了新的有价值的信息;Liu等[16]对牛骨骼肌中的lncRNA进行测序分析,发现牛骨骼肌中的lncRNAs的特征在很多方面与已知的其他哺乳动物中的lncRNAs类似,也为后续利用表观遗传技术进行牛育种改良提供了良好的基础。
2.3 转录组学转录组学是功能基因组研究的重要手段[17],是细胞的第一个在基因组尺度上可访问的“功能性”分子层[10],在整体水平上研究细胞中基因转录情况及转录调控规律,揭示基因转录、转录调控规律以及疾病发生过程中的分子机理[17]。基于高通量测序的RNA-seq技术是当前转录组学研究的主要手段,可用于研究单细胞基因的表达、翻译组和RNA结构[18],其具有灵敏度高、噪音低、检测范围广的优点[19],被广泛运用于畜禽功能基因的挖掘和分子遗传网络调控机制的研究中[20]。例如,Jin等[21]利用空间转录组学深入解析猪的肉质性状形成的分子机理,绘制猪不同组织基因转录调控图谱,揭示了组织特异性及转录进化动态;Zhao等[22]通过绘制猪基因组启动子、增强子、开放染色质区域及三维基因组精细图谱,鉴定了顺式调控元件及调控区突变位点,揭示了影响猪表型变异的潜在调控机理。通过转录组学研究可以促进采用猪作为人类生物学和疾病的生物医学模型,并揭示有价值性状的分子基础。
2.4 蛋白质组学蛋白质组学是细胞桥接基因表达到表型的主要“功能”层[23-24],是用来研究蛋白质的特性、生化特性和功能作用,以及它们的数量、修饰和结构如何在发育过程中以及对内外部刺激的反应中发生变化。2001年,人类蛋白质组组织以更精确的方式提出了蛋白质组学的目标,即鉴定人类基因组编码的所有蛋白质,将蛋白质组学的发展转向功能蛋白质组的研究[25]。目前最常使用的2种定量蛋白质组学分析技术是利用同位素标记质谱分析的定量蛋白质组学技术i TRAQ、TMT[26]。质谱(MS)和其他技术的进步推动了蛋白质组学领域的发展,这些技术使得快速、低成本地分析大量生物样品中的蛋白质成为可能。例如,Wang等[27]对2个不同品种的6月龄中国本土迷你型猪背最长肌进行蛋白质组学分析,并与2个西方引进品种进行比较,发现了与猪肌肉生长、脂肪沉积相关的关键蛋白和调控肌纤维生长、脂质沉积能力的关键基因,为参与猪肌肉生长和脂质沉积的关键蛋白质提供了新的见解。
2.5 代谢组学代谢组是第一个不直接编码在基因组中的细胞层,而是蛋白质组功能谱的产物,与细胞环境接触。因此,代谢组构成了细胞的“表型”[28]。代谢组学可更直观地展示生物体内真实发生的物质代谢过程,可以解释转录组和蛋白质组无法解释的细胞内的复杂调控活动,可以直接影响表观遗传调控和酶活性,影响细胞功能,实时定量地反映细胞或组织器官内正在发生的代谢过程[29]。最常见的代谢组学类型包括靶向代谢组学、非靶向代谢组学和脂质组学,可根据试验目的来选择所需要的技术。例如,Welzenbach等[30]利用代谢组学数据解析猪肉滴水损失的潜在功能途径和候选基因,为直接涉及性能特征代谢的基因的遗传变异提供全面的见解;Bovo等[31]靶向检测180种代谢物,发现乙酰鸟氨酸和鞘磷脂等代谢物在大白猪和杜洛克猪2个品种间存在差异,为研究两个品种猪之间的生物学差异提供了重要的生物标志物,可进一步评估这些生物标志物在猪育种和营养中的实际应用的相关性。
2.6 微生物组学微生物组(microbiome)是指一个特定环境或生态系统中全部微生物及其遗传信息的集合,其内涵包括了微生物与其环境和宿主的相互作用。2006年,高通量测序和质谱技术的革命性突破以及生物信息学的快速发展极大推动了微生物组研究。由于微生物之间以及微生物与其所处环境间的相互作用极为复杂,通过宏基因组学结合宏转录组学以及新一代质谱技术催生下的宏蛋白质组学和宏代谢组学,人们可以更全面、系统地解析微生物组的结构和功能。例如,Xie等[32]通过宏基因组测序揭示高谷物饲喂改变了盲肠微生物群的组成和代谢并导致绵羊盲肠黏膜损伤;Bi等[33]通过宏基因组和宏转录组揭示了绵羊胎羔肠道中存在微生物群,为胎儿肠道微生物定植始于子宫提供了依据。宏基因组和宏转录组技术打破了微生物培养的局限性,解决了之前无法解决的生物学问题,并加速了基于基因组的新微生物基因的发现,现已被应用于畜禽各个领域中。
畜禽生命活动是一个复杂的调控过程,即通过基因组学、表观基因组、转录组学、蛋白质组学、代谢组学和微生物组学等单一组学无法解释某种生物学的变化,只能对单一分子的单一功能(遗传信息、蛋白功能或代谢通路)进行阐释。
3 多组学技术多组学技术是指结合两种或者两种以上组学,包括基因组学、表观基因组学、转录组学、蛋白质组学、代谢组学、微生物组学,对生物样本进行系统研究,从而探究生物系统中多种物质之间相互作用[40-41]。多组学技术可以补充任何单一组学中缺失或不可靠的信息,通过探究遗传物质在不同层面的共有通路和差异表达及其在系统层面的整体动态变化规律,可以获得更丰富、更全面的生命系统相关信息,从而实现不同组学不同层面的相互印证、相互补充、相互解释。
多组学联合分析利用不同组学分析方法分别检测不同组学层面遗传物质的表达量变化。当一个基因在不同组学层面都有表达量时,则认为该基因在不同层面被关联上[42]。而针对不同的研究背景和目的,可以综合比较选择不同的组合方式来进行联合分析(表 2),主要包括基因组和转录组联合分析,表观基因组和转录组联合分析,转录组和蛋白质组联合分析,转录组和代谢组联合分析,代谢组与微生物组联合分析等两种组学联合分析;另外还有三种组学联合分析,如基因组、表观基因组和转录组联合分析,转录组、代谢组和蛋白质组联合分析,代谢组、转录组和微生物组联合分析等。
|
|
表 2 多组学联合分析的方法和特点 Table 2 Methods and characteristics of multi-omics joint analysis |
不同的组学数据大多是异质的,具有不同的类型和格式,因而难以整合。因此运用多组学将各个水平的数据整合为一个类似于生物复杂调控网络的模型,从更高维度解析生物过程,才符合生物系统的复杂性,进而做出更全面更准确的机制解释。探究多组学数据整合的方法,有助于研究生命科学问题,挖掘其中的重要信息。而近年来,出现了许多方法来整合多组学,主要包括基于共定位分析整合多组学、孟德尔随机化整合多组学、基于网络整合多组学以及基于机器学习整合多组学等(表 3)。
|
|
表 3 多组学整合技术的方法及其优缺点 Table 3 Methods of multi-omics integration technology and their advantages and disadvantages |
畜禽生长性状是过去我国育种的主要目标之一,为了适应社会、市场的需求,我国以提高生长速度和提高产量为目标,加大对畜禽生长性状的研究。畜禽的生长性状包括生长速度、饲料转化率、活体背膘厚等,而目前运用较多的是用分子生物学方法分析影响生长性状的原因,提高选育效果和效率。但影响生长性状的分子遗传机制尚不明确。而在系统层面整合多组学分析可以更加精确地定位与生长性状相关的候选基因,解释影响生长性状的复杂调控机制,提高育种的准确性。
肌肉生长在猪生产中是一个重要的经济性状,是一个受多基因调控的数量性状,对猪肉产量和肉品质的研究一直是我国畜牧学者研究的重点。商鹏[42]利用转录组RNA-seq和蛋白质组iTRAQ技术对猪(藏猪、乌金猪、大约克猪)胚胎60日龄的背最长肌进行分析,筛选出13个与猪胚胎时期肌肉生长发育相关的候选基因,为出生后猪生长性状候选基因的筛选与鉴定提供了宝贵数据。Shang等[55]分析了来自产前肌肉组织的转录组和蛋白质组数据,发现209个基因在小型猪和其他两个品种猪之间在信使RNA和蛋白质水平上均一致表达,为猪的肌肉生长特性提供了新的候选基因和分子机制的见解。
目前尽管在牛表观基因组研究取得了重大进展,但对于胎儿骨骼肌发育的表观遗传基础仍然知之甚少,为了更精确地注释牛生长性状的遗传变异,Li等[48]整合基因组、表观组及转录组数据,发现牛成肌细胞体外增殖和分化过程中染色质可及性和基因表达呈现动态变化,并鉴定出阶段特异性基因和染色质可及性区域,从多个层面探究了牛重要经济性状基因组调控位点与基因组染色质开放区的关系,为了解骨骼肌发育的调节机制和开展肉牛遗传改良计划提供了有价值的信息。
4.2 多组学技术在畜禽繁殖性状研究中的应用繁殖性状作为低遗传力性状,低繁殖力限制了母畜的生产力。作为一个复杂的数量性状,繁殖性能受到多种因素(遗传、表观修饰和激素)的调控。近年来,研究者通过不同的组学方法筛选出了一些与繁殖性能相关的候选基因,但对于高繁殖性能的调控机制尚不清晰,而通过多组学技术可以挖掘其潜在的关键候选基因,明确影响畜禽繁殖性能的分子调控机制。
产仔数不仅是繁殖性状的一个重要经济性状,还是受微效多基因控制的数量性状,很难通过传统的育种手段来快速提高。而卵巢、子宫、输卵管作为畜禽最重要的繁殖器官,在每个发情周期都会经历一系列的生物学过程。Huang等[56]对高产和低产蛋鸡卵巢的转录组学和代谢组学进行分析,预测与产蛋相关的基因和途径,更好地了解低产和高产母鸡卵巢之间的分子差异,并为进一步研究家禽产蛋机制提供理论基础。喇永富[57]以FecB++基因型(无FecB突变)小尾寒羊母羊为研究对象,通过子宫RNA-Seq、子宫蛋白质组学和MassARRAY技术筛选出SDS、SDSL等基因可以作为绵羊产羔数性状相关的候选基因,为解析绵羊多羔性状的分子机制提供理论基础。Sun等[58]通过对20只雌性山羊的输卵管进行蛋白质组学和转录组学分析,揭示了输卵管基因调控繁殖力的新方面。这些研究通过卵巢、子宫和输卵管等繁殖器官的多组学测序分析,筛选出与繁殖性状相关的候选基因,以期解析畜禽繁殖性能的分子调控机制。
下丘脑-垂体-性腺(Hypothalamic-Pituitary-Gonadal, HPG)轴在畜禽繁殖系统发育调控中扮演重要角色。目前关于繁殖性状的研究主要集中在下丘脑、垂体中,寻找与繁殖性状相关的基因和分子标记一直都是育种学家关注的热点。张壮彪[59]利用转录组学和蛋白质组学分析探究小尾寒羊下丘脑影响产羔数的分子机制,发现LGALS3、ASPA和TTR可能是影响无FecB突变小尾寒羊产羔数差异的候选基因,为揭示绵羊多羔分子机理以及培育高繁殖力绵羊新品种提供了依据。Chang等[60]通过对开放染色质图谱和转录组的综合分析来确定参与调节鹅垂体的孵化行为的顺式调控元件及其潜在的转录因子,发现了5个与DAR相关的DEG与维持鹅的孵化行为密切相关,揭示了垂体水平转录因子参与调节马岗鹅的孵化行为。这些研究也为HPG轴及其在动物繁殖过程的作用机理提供参考。
4.3 多组学技术在畜禽肉质性状研究中的应用随着人们对肉质要求的不断提升,畜禽肉质性状的改良已经成为畜牧业关注的热点。单一层面的组学在畜禽肉质性状中广泛应用,但这些研究仅仅停留在单一层面,并不能系统地解释影响畜禽肉质性状的复杂调控机制,而多组学技术则能很好地解决这一问题。
基因表达程序与顺式调控元件和染色质相关RNA(caRNA)的相互作用密切相关。然而,由于缺乏染色质构象信息,以及缺少针对caRNA的研究手段,对这些遗传变异进行功能解释成为了猪功能基因组研究的瓶颈。Li等[61]通过染色质-染色质互作图谱和RNA-染色质互作图谱的联合分析,揭示有助于复杂骨骼肌特征的基因组变异。同时,运用整合多组学策略,对猪15个产肉相关性状的GWAS信号进行了解析,为猪产肉性状遗传机理解析奠定了重要基础,也为猪产肉性状改良提供了重要支撑。
随着高通量测序技术的发展,转录组和蛋白质组技术的整合已成为分析动物复杂性状分子机制的重要手段。Wang等[62]通过对南阳黑猪肌内脂肪含量有差异的的背最长肌进行了基于转录组和蛋白质组分析,确定了25个决定脂质沉积遗传差异的候选基因,为未来脂质沉积性状的分析提供了宝贵资源。本团队Yu等[49]通过转录组学、代谢组学和蛋白质组学探究中国地方猪品种与商业瘦肉猪品种之间脂肪沉积和肉质差异的分子机制,并通过构建加权基因共表达网络(WGCNA),鉴定出RapGEF1是与IMF含量相关的关键基因,为高肉品质的遗传选择提供了一种新策略。
转录组分析可以提供遗传调控机制的全面证据,进一步的代谢组学分析可以提供转录后调节引起的辅助代谢变化,代谢物的变化最终会影响肉品质的变化。Zhan等[63]采用转录组学与代谢组学发现,功能基因PNPLA3、PLIN1和PRKG1等与花生酸和甘油三酯等与脂肪相关代谢物显著相关,为检测恩施黑猪肌内脂肪沉积和肉色变化提供了新见解。Li等[64]通过代谢组和转录组谱的整合分析揭示了鸡肉的年龄依赖性动态变化,有助于了解肉质发展的生物学过程,并探索特定代谢物积累的有价值的生物标志物。
有研究报告表示,微生物群的变化可能会影响猪的生长性能、免疫性能和肉质[65]。因此,改善肠道健康和减少抗生素使用的调控手段能够在养猪生产中发挥巨大作用[66]。本团队Tian等[50]通过靶向代谢组学分析、转录组测序和宏基因组测序联合分析,发现葡萄渣可以改善肉质,缓解炎症,减少氧化应激,为提高育肥猪的肉质提供了一种新的策略。
4.4 多组学技术在畜禽抗病性状研究中的应用随着我国经济的快速发展,疾病是目前影响畜禽产量和质量的主要原因之一。随着畜禽养殖规模不断扩大,强化疾病防控、深化畜禽常见疾病研究至关重要。过去单一组学技术在畜禽抗病性状中已有了一定的研究进展,而多组学技术可以更加精准、有效地揭示疾病发生的整体机制,并为快速寻找靶向药物提供更加科学的方法和手段。
病毒及细菌感染伴随着畜禽体内多种代谢途径的变化,已经成为世界范围内严重的疾病问题。Saelao等[67]通过对热应激下两个高度近交和基因不同的鸡肺组织差异表达响应新城疫病毒(NDV) 感染的蛋白质组学和转录组学进行综合分析,为两个遗传系的全局蛋白质和表达谱提供新的见解,并提供了在家禽热应激期间与NDV抗性相关的潜在遗传目标。Yu等[68]通过对鸡感染E. tenella期间的肠道微生物组和宿主转录组联合分析,为鸡感染E. tenella后的菌群和关键免疫基因提供了有价值的信息。
目前营养性疾病在畜禽疾病发生中也越来越常见,例如奶牛酮症是围产期高产奶牛的主要营养代谢紊乱性疾病。许秋实[69]通过转录组学和蛋白质组学分析,发现TGFβ1在酮病奶牛脂肪组织炎症反应中起到关键调控作用,为理解酮病奶牛脂肪组织的能量代谢机制和免疫应答作用提供了新的线索。
高海拔低气压、低氧分压严重影响人类和畜禽的生存发育。由于自然选择,青藏高原的本土动物对这种极端环境表现出可遗传的适应能力,藏猪则是研究缺氧相关分子生态学和病理学的理想动物模型。Zhang等[70]组联合分析,筛选出与缺氧适应性相关的HIF-1通路,以及通路中的关键对藏猪和约克夏猪的心组织进行转录组和蛋白质基因CRYAB、EGLN3、等。该研究不仅加深了人们对参与猪低氧适应的分子机制的理解,还加深了对人类低氧疾病的理解。
5 展望多组学技术的特点是将各个组学多维层次的信息进行有机整合,构建基因的调控网络,全面探索和深层次理解各生物分子之间的调控及因果关系,从而正确解析生命体的生物功能和生理机制。多组学整合分析的策略就是针对生物体的特定生物学功能,对来自不同组学层次的批量数据在同一整合分析软件中进行归一化处理、比较分析和相关性分析,建立不同层次分子间数据相关性;同时结合GO功能分析、代谢通路富集、分子互作等生物功能分析,系统全面地解析生物分子功能和调控机制。而整合来自同一样本的多组学数据是很难实现的,因此就需要对来自不同样本组的数据进行标准化和统计学处理。目前满足多组学整合分析数据的公共平台越来越多,例如,组学发现索引数据库和国际上正努力开展的基因型-组织表达(GTEx)项目、Farm-GTEx项目、FarmGTEx-PigGTEx项目等为研究组织的特异性基因表达和调控提供了众多公开可用的资源[71-72]。而多组学技术目前在畜禽遗传育种、生长发育和疾病中应用较少,尤其是如何整合庞大的多组学数据,利用生物信息学(机器学习、共定位分析、网络)构建数据分析模型,从而准确、快速地筛选有用的信息,系统地解析畜禽复杂生命系统的机理和表型。
| [1] |
EISENSTEIN M. Closing in on a complete human genome[J]. Nature, 2021, 590(7847): 679-681. DOI:10.1038/d41586-021-00462-9 |
| [2] |
WU Z H, GUI S T, QUAN Z W, et al. A precise chloroplast genome of Nelumbo nucifera (Nelumbonaceae) e valuated with Sanger, Illumina MiSeq, and PacBio RS Ⅱ sequencing platforms: insight into the plastid evolution of basal eudicots[J]. BMC Plant Biol, 2014, 14(1): 289. DOI:10.1186/s12870-014-0289-0 |
| [3] |
HASIN Y, SELDIN M, LUSIS A. Multi-omics approaches to disease[J]. Genome Biol, 2017, 18(1): 83. DOI:10.1186/s13059-017-1215-1 |
| [4] |
ZHANG W W, LI F, NIE L. Integrating multiple 'omics' analysis for microbial biology: application and methodologies[J]. Microbiology, 2010, 156(2): 287-301. DOI:10.1099/mic.0.034793-0 |
| [5] |
冯勉, 张莉. 多组学联合分析在畜禽研究中的应用[J]. 中国畜牧杂志, 2022, 58(3): 1-6. FENG M, ZHANG L. Application of multi-omics joint analysis in the research of livestock and poultry[J]. Chinese Journal of Animal Science, 2022, 58(3): 1-6. (in Chinese) |
| [6] |
MARDIS E R. DNA sequencing technologies: 2006-2016[J]. Nat Protoc, 2017, 12(2): 213-218. DOI:10.1038/nprot.2016.182 |
| [7] |
CHEN P, SUN Z P, WANG J W, et al. Portable nanopore-sequencing technology: trends in development and applications[J]. Front Microbiol, 2023, 14: 1043967. DOI:10.3389/fmicb.2023.1043967 |
| [8] |
HU T S, CHITNIS N, MONOS D, et al. Next-generation sequencing technologies: an overview[J]. Human Immunol, 2021, 82(11): 801-811. DOI:10.1016/j.humimm.2021.02.012 |
| [9] |
SANGER F, NICKLEN S, COULSON A R. DNA sequencing with chain-terminating inhibitors[J]. Proc Natl Acad Sci USA, 1977, 74(12): 5463-5467. DOI:10.1073/pnas.74.12.5463 |
| [10] |
HAAS R, ZELEZNIAK A, IACOVACCI J, et al. Designing and interpreting 'multi-omic' experiments that may change our understanding of biology[J]. Curr Opin Syst Biol, 2017, 6: 37-45. DOI:10.1016/j.coisb.2017.08.009 |
| [11] |
WANG C S, HAN B. Twenty years of rice genomics research: From sequencing and functional genomics to quantitative genomics[J]. Mol Plant, 2022, 15(4): 593-619. DOI:10.1016/j.molp.2022.03.009 |
| [12] |
International Chicken Genome Sequencing Consortium. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution[J]. Nature, 2004, 432(7018): 695-716. DOI:10.1038/nature03154 |
| [13] |
GROENEN M A M, ARCHIBALD A L, UENISHI H, et al. Analyses of pig genomes provide insight into porcine demography and evolution[J]. Nature, 2012, 491(7424): 393-398. DOI:10.1038/nature11622 |
| [14] |
WANG K C, CHANG H Y. Epigenomics: technologies and applications[J]. Circ Res, 2018, 122(9): 1191-1199. DOI:10.1161/CIRCRESAHA.118.310998 |
| [15] |
LI H Y, XI Q Y, XIONG Y Y, et al. Identification and comparison of microRNAs from skeletal muscle and adipose tissues from two porcine breeds[J]. Anim Genet, 2012, 43(6): 704-713. DOI:10.1111/j.1365-2052.2012.02332.x |
| [16] |
LIU X F, DING X B, LI X, et al. An atlas and analysis of bovine skeletal muscle long noncoding RNAs[J]. Anim Genet, 2017, 48(3): 278-286. DOI:10.1111/age.12539 |
| [17] |
LOWE R, SHIRLEY N, BLEACKLEY M, et al. Transcriptomics technologies[J]. PLoS Comput Biol, 2017, 13(5): e1005457. DOI:10.1371/journal.pcbi.1005457 |
| [18] |
HARPER A L, HE Z, LANGER S, et al. Validation of an associative transcriptomics platform in the polyploid crop species Brassica juncea by dissection of the genetic architecture of agronomic and quality traits[J]. Plant J, 2020, 103(5): 1885-1893. DOI:10.1111/tpj.14876 |
| [19] |
LIAO Y H, LIU Z Y, ZHANG Y, et al. High-throughput and high-sensitivity full-length single-cell RNA-seq analysis on third-generation sequencing platform[J]. Cell Discov, 2023, 9(1): 5. DOI:10.1038/s41421-022-00500-4 |
| [20] |
SANGWAN R S, TRIPATHI S, SINGH J, et al. De novo sequencing and assembly of Centella asiatica leaf transcriptome for mapping of structural, functional and regulatory genes with special reference to secondary metabolism[J]. Gene, 2013, 525(1): 58-76. DOI:10.1016/j.gene.2013.04.057 |
| [21] |
JIN L, TANG Q Z, HU S L, et al. A pig BodyMap transcriptome reveals diverse tissue physiologies and evolutionary dynamics of transcription[J]. Nat Commun, 2021, 12(1): 3715. DOI:10.1038/s41467-021-23560-8 |
| [22] |
ZHAO Y X, HOU Y, XU Y Y, et al. A compendium and comparative epigenomics analysis of cis-regulatory elements in the pig genome[J]. Nat Commun, 2021, 12(1): 2217. DOI:10.1038/s41467-021-22448-x |
| [23] |
HARPER J W, BENNETT E J. Proteome complexity and the forces that drive proteome imbalance[J]. Nature, 2016, 537(7620): 328-338. DOI:10.1038/nature19947 |
| [24] |
MOOTHA V. The mitochondrial proteome and human disease[J]. Pathology, 2015, 47(S1): S28. |
| [25] |
GONZÁLEZ-GOMARIZ J, GURUCEAGA E, LÓPEZ-SÁNCHEZ M, et al. Proteogenomics in the context of the Human Proteome Project (HPP)[J]. Expert Rev Proteomics, 2019, 16(3): 267-275. DOI:10.1080/14789450.2019.1571916 |
| [26] |
PARK J, PIEHOWSKI P D, WILKINS C, et al. Informed-Proteomics: open-source software package for top-down proteomics[J]. Nat Methods, 2017, 14(9): 909-914. DOI:10.1038/nmeth.4388 |
| [27] |
WANG Z X, SHANG P, LI Q G, et al. iTRAQ-based proteomic analysis reveals key proteins affecting muscle growth and lipid deposition in pigs[J]. Sci Rep, 2017, 7(1): 46717. DOI:10.1038/srep46717 |
| [28] |
ZAMBONI N, SAGHATELIAN A, PATTI G J. Defining the metabolome: size, flux, and regulation[J]. Mol Cell, 2015, 58(4): 699-706. DOI:10.1016/j.molcel.2015.04.021 |
| [29] |
DAMIANI C, GAGLIO D, SACCO E, et al. Systems metabolomics: from metabolomic snapshots to design principles[J]. Curr Opin Biotechnol, 2020, 63: 190-199. DOI:10.1016/j.copbio.2020.02.013 |
| [30] |
WELZENBACH J, NEUHOFF C, HEIDT H, et al. Integrative analysis of metabolomic, proteomic and genomic data to reveal functional pathways and candidate genes for drip loss in pigs[J]. Int J Mol Sci, 2016, 17(9): 1426. DOI:10.3390/ijms17091426 |
| [31] |
BOVO S, MAZZONI G, GALIMBERTI G, et al. Metabolomics evidences plasma and serum biomarkers differentiating two heavy pig breeds[J]. Animal, 2016, 10(10): 1741-1748. DOI:10.1017/S1751731116000483 |
| [32] |
XIE F, XU L, WANG Y, et al. Metagenomic sequencing reveals that high-grain feeding alters the composition and metabolism of Cecal microbiota and induces Cecal mucosal injury in sheep[J]. mSystems, 2021, 6(5): e0091521. DOI:10.1128/mSystems.00915-21 |
| [33] |
BI Y L, TU Y, ZHANG N F, et al. Multiomics analysis reveals the presence of a microbiome in the gut of fetal lambs[J]. Gut, 2021, 70(5): 853-864. DOI:10.1136/gutjnl-2020-320951 |
| [34] |
BOIX C A, JAMES B T, PARK Y P, et al. Regulatory genomic circuitry of human disease loci by integrative epigenomics[J]. Nature, 2021, 590(7845): 300-307. DOI:10.1038/s41586-020-03145-z |
| [35] |
PREISSL S, GAULTON K J, REN B. Characterizing cis-regulatory elements using single-cell epigenomics[J]. Nat Rev Genet, 2023, 24(1): 21-43. DOI:10.1038/s41576-022-00509-1 |
| [36] |
ALDRIDGE S, TEICHMANN S A. Single cell transcriptomics comes of age[J]. Nat Commun, 2020, 11(1): 4307. DOI:10.1038/s41467-020-18158-5 |
| [37] |
RUSK N. Spatial transcriptomics[J]. Nat Methods, 2016, 13(9): 710. |
| [38] |
LIU X J, LOCASALE J W. Metabolomics: a primer[J]. Trends Biochem Sci, 2017, 42(4): 274-284. DOI:10.1016/j.tibs.2017.01.004 |
| [39] |
KNIGHT R, VRBANAC A, TAYLOR B C, et al. Best practices for analysing microbiomes[J]. Nat Rev Microbiol, 2018, 16(7): 410-422. DOI:10.1038/s41579-018-0029-9 |
| [40] |
MOHAMMADI-SHEMIRANI P, SOOD T, PARÉ G. From 'omics to multi-omics technologies: the discovery of novel causal mediators[J]. Curr Atheroscler Rep, 2023, 25(2): 55-65. DOI:10.1007/s11883-022-01078-8 |
| [41] |
ZHANG H W, LV C, ZHANG L J, et al. Application of omics- and multi-omics-based techniques for natural product target discovery[J]. Biomed Pharmacother, 2021, 141: 111833. DOI:10.1016/j.biopha.2021.111833 |
| [42] |
商鹏. 基于胚胎肌肉组织转录组和蛋白质组数据鉴定猪生长性状相关基因[D]. 北京: 中国农业大学, 2017. SHANG P. Identification of candidate genes on growth traits intergrating transcriptome and proteome of embryonic muscle in pigs[D]. Beijing: China Agricultural University, 2017. (in Chinese) |
| [43] |
LI X, YANG J, SHEN M, et al. Whole-genome resequencing of wild and domestic sheep identifies genes associated with morphological and agronomic traits[J]. Nat Commun, 2020, 11(1): 2815. DOI:10.1038/s41467-020-16485-1 |
| [44] |
ZHANG D, DENG Y X, KUKANJA P, et al. Spatial epigenome-transcriptome co-profiling of mammalian tissues[J]. Nature, 2023, 616(7955): 113-122. DOI:10.1038/s41586-023-05795-1 |
| [45] |
蒋可人, 马峥, 郑航, 等. 转录组与蛋白质组整合分析在生物学研究中的应用[J]. 生物技术通报, 2018, 34(12): 50-55. JIANG K R, MA Z, ZHENG H, et al. Review on the application of integrated transcriptome and proteome analysis in biology[J]. Biotechnol Bull, 2018, 34(12): 50-55. (in Chinese) |
| [46] |
PONSUKSILI S, TRAKOOLJUL N, HADLICH F, et al. Genetic regulation of liver metabolites and transcripts linking to biochemical-clinical parameters[J]. Front Genet, 2019, 10: 348. DOI:10.3389/fgene.2019.00348 |
| [47] |
LIU R X, HONG J, XU X Q, et al. Gut microbiome and serum metabolome alterations in obesity and after weight-loss intervention[J]. Nat Med, 2017, 23(7): 859-868. DOI:10.1038/nm.4358 |
| [48] |
LI Q, WANG Y H, HU X, et al. Transcriptional states and chromatin accessibility during bovine myoblasts proliferation and myogenic differentiation[J]. Cell Prolif, 2022, 55(5): e13219. DOI:10.1111/cpr.13219 |
| [49] |
YU T Y, TIAN X K, LI D, et al. Transcriptome, proteome and metabolome analysis provide insights on fat deposition and meat quality in pig[J]. Food Res Int, 2023, 166: 112550. DOI:10.1016/j.foodres.2023.112550 |
| [50] |
TIAN X K, LI D, ZHAO X, et al. Dietary grape pomace extract supplementation improved meat quality, antioxidant capacity, and immune performance in finishing pigs[J]. Front Microbiol, 2023, 14: 1116022. DOI:10.3389/fmicb.2023.1116022 |
| [51] |
TEKOLA-AYELE F, ZENG X H, CHATTERJEE S, et al. Placental multi-omics integration identifies candidate functional genes for birthweight[J]. Nat Commun, 2022, 13(1): 2384. DOI:10.1038/s41467-022-30007-1 |
| [52] |
JIN C, LEE B, SHEN L, et al. Integrating multi-omics summary data using a Mendelian randomization framework[J]. Brief Bioinform, 2022, 23(6): bbac376. DOI:10.1093/bib/bbac376 |
| [53] |
BODEIN A, SCOTT-BOYER M P, PERIN O, et al. Interpretation of network-based integration from multi-omics longitudinal data[J]. Nucleic Acids Res, 2022, 50(5): e27. DOI:10.1093/nar/gkab1200 |
| [54] |
REEL P S, REEL S, PEARSON E, et al. Using machine learning approaches for multi-omics data analysis: a review[J]. Biotechnol Adv, 2021, 49: 107739. DOI:10.1016/j.biotechadv.2021.107739 |
| [55] |
SHANG P, WANG Z X, CHAMBA Y, et al. A comparison of prenatal muscle transcriptome and proteome profiles between pigs with divergent growth phenotypes[J]. J Cell Biochem, 2019, 120(4): 5277-5286. DOI:10.1002/jcb.27802 |
| [56] |
HUANG X, ZHANG H Y, CAO H Y, et al. Transcriptomics and metabolomics analysis of the ovaries of high and low egg production chickens[J]. Animals, 2022, 12(16): 2010. DOI:10.3390/ani12162010 |
| [57] |
喇永富. 利用转录组和蛋白质组测序筛选绵羊多羔基因的研究[D]. 兰州: 甘肃农业大学, 2020. LA Y F. Study on screening polytocous genes in sheep based on transcriptome and proteomics sequencing[D]. Lanzhou: Gansu Agricultural University, 2020. (in Chinese) |
| [58] |
SUN Z P, LIU Y F, HE X Y, et al. Integrative proteomics and transcriptomics profiles of the oviduct reveal the prolificacy-related candidate biomarkers of goats (Capra hircus) in estrous periods[J]. Int J Mol Sci, 2022, 23(23): 14888. DOI:10.3390/ijms232314888 |
| [59] |
张壮彪. 基于下丘脑多组学分析筛选小尾寒羊多羔候选基因[D]. 北京: 中国农业科学院, 2020. ZHANG Z B. Screening polytocous candidate genes in small tail han sheep based on hypothalamic multi-omics analysis[D]. Beijing: Chinese Academy of Agricultural Sciences, 2020. (in Chinese) |
| [60] |
CHANG J Y, FAN D, LIU J X, et al. Transcriptomic and chromatin landscape analysis reveals that involvement of pituitary level transcription factors modulate incubation behaviors of magang geese[J]. Genes (Basel), 2023, 14(4): 815. DOI:10.3390/genes14040815 |
| [61] |
LI J J, XIANG Y, ZHANG L, et al. Enhancer-promoter interaction maps provide insights into skeletal muscle-related traits in pig genome[J]. BMC Biol, 2022, 20(1): 136. DOI:10.1186/s12915-022-01322-2 |
| [62] |
WANG L Y, ZHANG Y W, ZHANG B, et al. Candidate gene screening for lipid deposition using combined transcriptomic and proteomic data from Nanyang black pigs[J]. BMC Genomics, 2021, 22(1): 441. DOI:10.1186/s12864-021-07764-2 |
| [63] |
ZHAN H W, XIONG Y C, WANG Z C, et al. Integrative analysis of transcriptomic and metabolomic profiles reveal the complex molecular regulatory network of meat quality in Enshi black pigs[J]. Meat Sci, 2022, 183: 108642. DOI:10.1016/j.meatsci.2021.108642 |
| [64] |
LI J J, ZHANG D H, YIN L Q, et al. Integration analysis of metabolome and transcriptome profiles revealed the age-dependent dynamic change in chicken meat[J]. Food Res Int, 2022, 156: 111171. DOI:10.1016/j.foodres.2022.111171 |
| [65] |
WANG W L, HU H F, ZIJLSTRA R T, et al. Metagenomic reconstructions of gut microbial metabolism in weanling pigs[J]. Microbiome, 2019, 7(1): 48. DOI:10.1186/s40168-019-0662-1 |
| [66] |
REVERTER A, BALLESTER M, ALEXANDRE P A, et al. A gene co-association network regulating gut microbial communities in a Duroc pig population[J]. Microbiome, 2021, 9(1): 52. DOI:10.1186/s40168-020-00994-8 |
| [67] |
SAELAO P, WANG Y, CHANTHAVIXAY G, et al. Integrated proteomic and transcriptomic analysis of differential expression of chicken lung tissue in response to NDV infection during heat stress[J]. Genes (Basel), 2018, 9(12): 579. DOI:10.3390/genes9120579 |
| [68] |
YU H L, WANG Q, TANG J Q, et al. Comprehensive analysis of gut microbiome and host transcriptome in chickens after Eimeria tenella infection[J]. Front Cell Infect Microbiol, 2023, 13: 1191939. DOI:10.3389/fcimb.2023.1191939 |
| [69] |
许秋实. 基于多组学水平的酮病奶牛脂肪组织代谢适应分析[D]. 长春: 吉林大学, 2019. XU Q S. Integrated analysis of metabolic adaptation on adipose tissue in ketotic dairy cows based on multi-omics data[D]. Changchun: Jilin University, 2019. (in Chinese) |
| [70] |
ZHANG B, CHAMBA Y, SHANG P, et al. Comparative transcriptomic and proteomic analyses provide insights into the key genes involved in high-altitude adaptation in the Tibetan pig[J]. Sci Rep, 2017, 7(1): 3654. DOI:10.1038/s41598-017-03976-3 |
| [71] |
LIU S L, GAO Y H, CANELA-XANDRI O, et al. A multi-tissue atlas of regulatory variants in cattle[J]. Nat Genet, 2022, 54(9): 1438-1447. DOI:10.1038/s41588-022-01153-5 |
| [72] |
THE GTEX CONSORTIUM, ARDLIE K G, DELUCA D S, et al. The Genotype-Tissue Expression (GTEx) pilot analysis: multitissue gene regulation in humans[J]. Science, 2015, 348(6235): 648-660. DOI:10.1126/science.1262110 |
(编辑 孟培)



