2. 福建农林大学动物科学学院(蜂学学院),福州 350002
2. College of Animal Sciences (College of Bee Science), Fujian Agriculture and Forestry University, Fuzhou 350002, China
绵羊肺炎支原体(Mycoplasma ovipneumoniae, Movi)是引起山羊和绵羊发生羊支原体性肺炎(Mycoplasmal pneumonia of sheep and goats, MPSG)的主要病原之一,现被认为是一种分布在全球的家养绵羊和山羊的呼吸道病原体[1-3]。Movi宿主范围异常广泛,除山羊和绵羊外,在麝牛、驯鹿、大角羊等野生动物中均检测到该病原[4-6]。2001年以来,一些进行定期监测的国家报告了该病例,包括瑞典、美国、法国和英国[7-10]。其中在美国453个家养羊场中,88.0%的羊被检测出Movi[7];在法国,2007—2019年Movi占小反刍动物分离的所有支原体的16.4%[8],2005—2019年,在英格兰和威尔士,Movi占小反刍动物分离的所有支原体中的50.0%以上[10]。2012—2014年,对中国新疆6个地区羊Movi感染情况调查显示,血清样品的平均阳性率为17.8%,鼻拭子和肺组织样品的平均阳性率分别为10.2%和28.9%[11]。然而,在更多的国家建立全球监测系统进行大规模的流行病学调查之前,Movi感染的真正流行率还不得而知。
已有的研究报道显示,分离自不同地域、不同宿主来源的Movi的基因存在高度多态性[12-13]。热休克蛋白(heat shock protein,Hsp70)也称DnaK,在古细菌和植物到人类的所有生物体中均有发现,是机体内高度保守的生物分子,是生物细胞中含量最高的一种热休克蛋白,也是Movi重要的膜结构蛋白,具有免疫原性和免疫佐剂效应,其C-端抗原性优势明显,可诱导机体产生体液和细胞免疫反应[14-16],是疫苗开发的潜在候选蛋白。Hsp70基因在支原体种内保守,种间差异大[14-15],是分子生物学检测的重要靶点。由于体内Hsp70的变化与健康和患病状况相关,因此,它也可作为各种疾病的生理和病理状况以及药物靶标的生物标志物[17-19]。为了解Movi的流行情况及遗传演化规律,本研究建立基于Hsp70基因的Movi TaqMan荧光定量PCR检测方法,并对福建省Movi分离纯化菌株的Hsp70基因进行分析,以期为Movi临床诊断提供技术支撑,也为更进一步了解Movi遗传演化规律提供参考。
1 材料与方法 1.1 菌株与临床样品山羊支原体山羊肺炎亚种(Mycoplasma capricolum subsp.capripneumoniae,Mccp)F38株、山羊支原体山羊亚种(Mycoplasma capricolum capricolum,Mcc)、绵羊肺炎支原体(Movi)MO Y98标准株由中国农业科学院兰州兽医研究所储岳峰研究员惠赠;丝状支原体山羊亚种(Mycoplasma mycoides subsp. capri, Mmc)、莱氏无胆甾原体(Acholeplasmalaidlawii, AL)、无乳支原体(Mycoplasma agalactiae, Maga)、伪结核棒状杆菌(Corynebacterium pseudotuberculosis, CP)、羊口疮病毒(orf virus,ORFV)、牛支原体(Mycoplasma bovis, Mb)由福建省农业科学院畜牧兽医研究所草食动物病研究中心分离纯化并保存。
88份有呼吸道症状的山羊鼻拭子样品及解剖可见肺肝病变或胸膜肺炎症状的43份疑似MPSG的肺组织病料均来自福建省部分山羊养殖场。
1.2 主要试剂Probe qPCR Mix购自宝日医生物技术(北京)有限公司(TaKaRa);DL2000购自艾科瑞生物技术有限公司;DNA提取试剂盒购自天根生化科技有限公司;病毒DNA/RNA提取试剂盒、2×TransStartⓇFastPfuFly PCR SuperMix (-dye)以及pEASYⓇ-Blunt3 Cloning Kit试剂盒均购自北京全式金生物技术有限公司;Biospin Gel and PCR Purification Kit购自杭州博日科技股份有限公司。
1.3 TaqMan qPCR检测方法的建立1.3.1 引物设计 参考GenBank上登录的Movi strain 274菌株(登录号:CP079 199.1)的Hsp70基因保守序列,利用引物设计软件Oligo 7(v7.37)设计TaqMan qPCR的引物,Hsp70-qF1/R1和探针Hsp70-FAM以及Movi Hsp70全基因分段扩增引物Hsp70-F1/R1和Hsp70-F2/R2序列(表 1),引物均由生工生物工程(上海)股份有限公司合成。
|
|
表 1 本研究中所用的引物和探针 Table 1 Primers and probe used in this study |
1.3.2 反应条件优化 将Hsp70基因中位于921~1 400 bp的序列片段(由生工生物工程股份有限公司合成)插入PUC57载体的N和A酶切位点之间,经筛选后获得含有Hsp70基因的质粒(命名为Movi-Hsp70)为试验阳性标准品,利用微量核酸测定仪测定其浓度后,换算成拷贝数, 即5.72×107 copies ·μL-1),用EASY Dilution将标准品进行10倍连续倍比稀释。按照Probe qPCR Mix试剂盒说明书配制20 μL的TaqMan qPCR反应体系,并对反应体系和扩增条件进行优化,以最高荧光值、最小的样本阈值循环数(Cq值)确定建立的TaqMan qPCR方法的最佳反应条件。
1.3.3 标准曲线的建立 以Movi-Hsp70不同标准品浓度(5.72×102~5.72×105 copies ·μL-1)为模板,利用优化后的TaqMan qPCR方法的最佳反应条件,进行TaqMan qPCR扩增反应,获得扩增动力学曲线。以标准品起始拷贝数的常用对数(lgC)为横坐标,以循环数阈值(Cq值)为纵坐标,绘制出建立的TaqMan qPCR方法的标准曲线。
1.3.4 灵敏性试验 用优化后的TaqMan qPCR方法的最佳反应条件,以不同质粒浓度(5.72×102~5.72×10-1 copies ·μL-1)为模板,每个滴度进行3个平行试验,同时设阴性对照,确定建立的TaqMan qPCR方法的最低检测限。同时,利用表 1中的引物(Hsp70-qF1/Hsp70-qR1),经常规PCR扩增,比较二者的敏感性。
1.3.5 特异性试验 利用细菌基因组提取试剂盒提取牛羊常见病原菌(Maga、AL、Mmc、Mccp、Mcc、Mb、CP)基因组DNA,利用病毒DNA/RNA提取试剂盒对羊口疮病毒(ORFV)提取基因组DNA。以提取的核酸DNA为模板,用优化后的TaqMan qPCR方法的最佳反应条件,以质粒浓度(5.72×103 copies · μL-1)为阳性对照,对这些牛羊常见病原进行TaqMan qPCR扩增反应,评价建立的TaqMan qPCR方法的特异性。
1.3.6 重复性试验 用优化后的TaqMan qPCR方法的最佳反应条件,以不同质粒浓度(5.72×105、5.72×103、5.72×101 copies ·μL-1)为模板进行检测。每种标准品含量设置3个重复,计算组内变异系数(CV)。分别将上述标准品分装后置于-20 ℃保存,每隔7 d取出,用优化后的TaqMan qPCR方法进行检测,共检测3次,计算组间变异系数。
1.4 临床样品检测用本研究建立的Movi TaqMan qPCR方法及参考林裕胜等[20]建立的基于P113基因的TaqMan qPCR方法对福建省部分山羊场临床送检的88份羊鼻拭子样品及43份疑似MPSG的肺组织病料进行平行检测,抽取10份检测结果不符合的样品用Hsp70-F/R引物进行PCR扩增并测序分析;另外,将检测结果为Movi阳性的肺组织样品进行分离鉴定,将分离纯化成功的菌株进行Hsp70基因的扩增、克隆和测序。
1.5 Hsp70基因扩增与序列分析1.5.1 Hsp70基因分段扩增及克隆测序 利用基因组DNA提取试剂盒提取Movi分离株DNA。使用设计的Hsp70基因分段扩增引物(表 1)进行分段扩增。PCR扩增50 μL体系:2×TransStart FastPfu Fly PCR SuperMix 25 μL,模板5 μL,上、下游引物各1 μL(引物浓度均为10 μmol ·L-1),ddH2O 18 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃ 20 s,53 ℃ 20 s,72 ℃ 40 s,35个循环,最后72 ℃延伸10 min。PCR产物经1.0%的琼脂糖凝胶电泳鉴定。用PCR产物纯化试剂盒回收PCR扩增产物,将其与pEASYⓇ-Blunt3 CloningVector载体连接,转化感受态Trans1-T1;在SOC培养基中37 ℃振荡培养1 h,取适量菌液涂布含有X-Gal,IPTG和Amp的LB琼脂平板,37 ℃过夜培养。挑选白色菌落扩大培养,经过PCR验证为阳性的菌液送至福州铂尚生物技术有限公司测序。利用DNAStar7.0软件中的SeqMan软件对2个片段的序列进行拼接得到完整的Hsp70基因序列。
1.5.2 Hsp70序列及遗传进化分析 用DNAStar7.0软件中的MegAlign软件对从临床病料中分离到的Movi菌株及18个Movi参考菌株进行基因和氨基酸序列进行分析。利用MEGA 7.0软件构建Hsp70基因系统进化树,分析Hsp70基因的遗传进化关系。用NetNGlyc-1.0在线软件预测N-糖基化位点。
2 结果 2.1 引物和探针分析从GenBank数据库中共检索到24个Hsp70基因序列(21个山羊源Movi菌株和3个绵羊源Movi菌株)。对于正向引物Hsp70-qF1(5'-GTTCTTCTTGTTGGTGGATC-3')(位于943~962 bp),24个Hsp70基因序列中有23个(95.8%)与设计的正向引物相匹配,只有1个(菌株J1,GenBank登录号KC693 973.1)有不匹配的序列。对于反向引物Hsp70-qR1(5'-GCCACAACTTCATCAGGATTTATT-3')(位于1 026~1 049 bp),24个Hsp70基因序列中有20个(83.30%)与设计的反向引物相匹配,只有4个(菌株FJ-36/L16/L17/Y98,GenBank登录号OP121622/KC693 980.1/KC693 981.1/CP118 522.1)且仅在5'端第3位不匹配。对于探针Hsp70-FAM(5'-ACACGTATGCCAGCTGTTCAGACA-3')(位置964~987 bp),则与24个Hsp70基因序列均100%匹配。引物Hsp70-qF1和Hsp70-qR1以及TaqMan探针Hsp70-FAM的变化见表 2。
|
|
表 2 绵羊肺炎支原体引物和探针序列变异分析 Table 2 Sequence variation analysis of primers and probes of Movi |
优化后的TaqMan qPCR方法最佳反应体系(20 μL)如下:Probe qPCR Mix 10 μL,上、下游引物(浓度为10 μmol ·L-1)各0.4 μL、探针(浓度为10 μmol ·L-1)0.4 μL、模板2 μL、补充ddH2O至终体积20 μL。优化出的TaqMan qPCR方法最佳反应条件为:95 ℃预变性2 min;95 ℃ 10 s,60 ℃ 30 s,40个循环。
2.3 TaqMan qPCR标准曲线的建立以Movi-Hsp70标准品作为模板,利用优化后的反应条件进行TaqMan qPCR扩增,获得扩增曲线(图 1A)。以每个浓度标准品模板中拷贝数的常用对数(lgC)为横坐标,以出现的循环数阈值(Cq值)结果为纵坐标,获得基于检测Movi TaqMan qPCR方法的标准曲线(图 1B)。结果显示,建立的TaqMan qPCR方法在5.72×102~5.72×105 copies ·μL-1反应范围内有很好的线性关系,相关系数为1.00,扩增效率为96.0%,斜率为-3.411,截距为37.29。结果表明建立的TaqMan qPCR方法线性关系良好、扩增效率高。
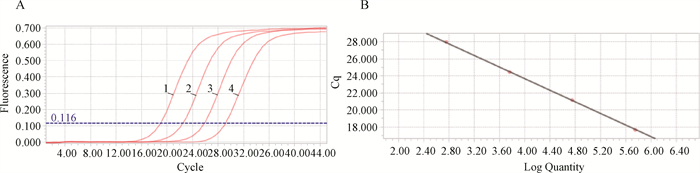
|
1~4. 不同稀释度的质粒(5.72×102~5.72×105 copies ·μL-1) 1-4. Plasmids at different dilutions (5.72×102~5.72×105 copies ·μL-1) 图 1 TaqMan qPCR扩增曲线(A)及标准曲线(B) Fig. 1 Amplification curve (A) and standard curve (B) of the TaqMan qPCR |
采用所建方法对牛羊常见病原核酸样品进行检测。从扩增曲线可见,建立的TaqMan qPCR方法仅对Movi检测出现阳性扩增,对其他牛羊常见病原(Maga、AL、Mmc、Mccp、Mcc、Mb、CP和ORFV)均未见荧光信号(图 2)。
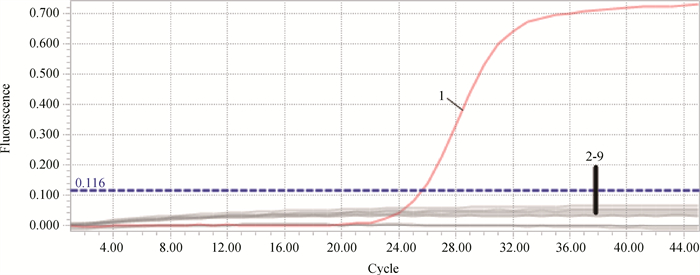
|
1. 绵羊肺炎支原体;2~9. 分别为无乳支原体、莱氏无胆甾原体、丝状支原体山羊亚种、山羊支原体山羊肺炎亚种、山羊支原体山羊亚种、牛支原体、伪结核棒状杆菌、羊口疮病毒 1. Movi; 2-9. Maga, AL, Mmc, MCCP, Mcc, Mb, CP, ORFV, respectively 图 2 TaqMan qPCR特异性试验结果 Fig. 2 Specificity analysis of TaqMan qPCR |
用建立的TaqMan qPCR方法对不同浓度质粒进行检测,结果显示,5.72×102~5.72 copies ·μL-1的质粒标准品均有特异性扩增曲线,因此确定该方法对重组质粒标准品最低检测限为5.72 copies ·μL-1(图 3),同时,用常规PCR对以上相同浓度质粒进行检测,结果显示常规PCR的最低检测限为5.72×102 copies ·μL-1,即TaqMan qPCR的敏感性是常规PCR的100倍。
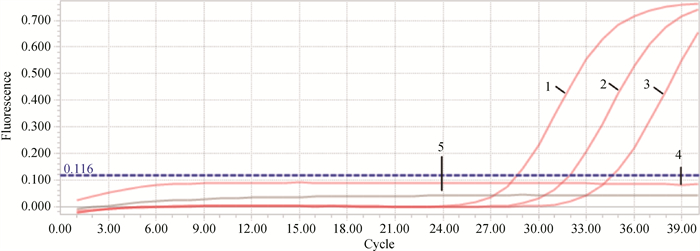
|
1~4. Movi-Hsp70质粒浓度5.72×102至5.72×10-1 copies.μL-1;5. 阴性对照 1-4. Movi-Hsp70 plasmid concentrations from 5.72×102 to 5.72×10-1 copies ·μL-1; 5. Negative control 图 3 TaqMan qPCR灵敏性试验结果 Fig. 3 Sensitivity analysis of TaqMan qPCR |
将不同稀释度的标准品分别进行组内和组间的重复性试验,结果显示(表 3),组内变异系数为0.09%~0.84%,组间变异系数0.35%~0.45%,Cq值变异系数均在1.00%以内,表明本研究建立的TaqMan qPCR方法重复性好。
|
|
表 3 TaqMan qPCR的重复性试验结果 Table 3 Repeatability analysis of the TaqMan qPCR |
临床样品的检测结果见表 4,基于Hsp70基因的TaqMan qPCR方法检测的临床样品阳性率高于基于P113基因的TaqMan qPCR方法;基于P113基因的TaqMan qPCR方法检测的阴性样品用基于Hsp70基因的TaqMan qPCR方法检测,结果也是阴性,符合率为100%。随机抽取基于Hsp70基因的TaqMan qPCR方法检测结果为阳性而基于P113基因的TaqMan qPCR方法检测结果为阴性的10份样品进行PCR扩增(用Hsp70基因分段扩增引物)后测序分析,测序结果显示均为目的基因序列。将检测结果为Movi阳性的40份肺组织样品进行分离鉴定,结果分离到6株Movi,将分离纯化成功的菌株进行Hsp70基因的扩增、克隆和测序。
|
|
表 4 临床样品检测结果 Table 4 Test results of clinical samples |
2.8.1 Hsp70基因的扩增 以提取的6个Movi菌株的DNA为模板,分别采用Hsp70基因引物分段扩增基因,PCR产物经琼脂糖凝胶鉴定,结果显示,出现与预期片段大小符合的条带,随后分别克隆后进行测序。利用DNAStar7.0软件中的SeqMan软件对Hsp70基因2个片段的序列进行拼接得到完整的Hsp70基因序列,结果显示6个Movi菌株Hsp70基因大小均为1 818 bp。6个Movi分离菌株分别命名为FJ-28、FJ-29、FJ-32、FJ-36、FJ-38、FJ-41,将序列上传至NCBI,获得序列登录号分别为OP121619~OP121624。
2.8.2 Hsp70序列分析 利用DNAStar7.0软件中的MegAlign软件对Movi分离株FJ-28、FJ-29、FJ-32、FJ-36、FJ-38、FJ-41与各Movi参考菌株(均参考自NCBI)的Hsp70基因和氨基酸序列进行分析。基因序列结果显示,本试验获取的6个Movi不同分离菌株之间核苷酸相似性在98.1%~100.0%,与山羊源菌株的亲缘关系较近,核苷酸相似性在97.7%~99.4%,与绵羊源菌株的亲缘关系较远,核苷酸相似性在96.0%~96.7%;与绵羊源菌株相比,山羊源的Hsp70基因在1 777~1 779 bp处均插入3个核苷酸(TCA);FJ-28、FJ-29、FJ-32、FJ-36、FJ-38、FJ-41分离株与Movi Y98标准株相比分别有64、59、57、58、65个核苷酸变异位点,这些发生变异的位点无规则地分布在整个基因中。基于Hsp70基因构建的进化树,结果显示,不同Movi菌株可以分成两个大分支,分别为ClusterⅠ(宿主均为山羊)和ClusterⅡ(宿主均为绵羊),ClusterⅠ下有ClusterⅠA和ClusterⅠB两个分支。FJ-28、FJ-29、FJ-32、FJ-36、FJ-38、FJ-41分离株的进化树分支均属于ClusterⅠA(图 4),研究表明本试验分离的菌株与同样来源于中国的山羊源菌株亲缘关系更近,而与绵羊源菌株的亲缘关系较远。

|
★. 表示本研究中分离的Movi菌株 ★. Indicates Movi strains isolated in the current study 图 4 基于Hsp70基因的遗传进化树分析结果 Fig. 4 Analysis of phylogenetic tree based on Hsp70 gene |
2.8.3 Hsp70氨基酸序列分析 氨基酸序列分析结果显示,本试验获取的6个Movi不同分离株之间氨基酸相似性在99.5%~100.0%,与山羊源的氨基酸相似性在99.2%~100.0%,与绵羊源的氨基酸相似性在98.0%~98.5%;与绵羊源菌株相比,所有山羊源分离株在25位(A→S)、89位(Q→D)、148位(E→A)、363位(E→D)、554位(L→V)均发生变异,且在593位点均插入Gln(谷氨酰胺)。Y98标准株、strain 150株与strain NXNK2203(均为绵羊源)在191、250、340、393、410、436位存在N末端糖基化位点,其它山羊源菌株(除了J1株外)均在586位多了1个N末端糖基化位点(图 5)。
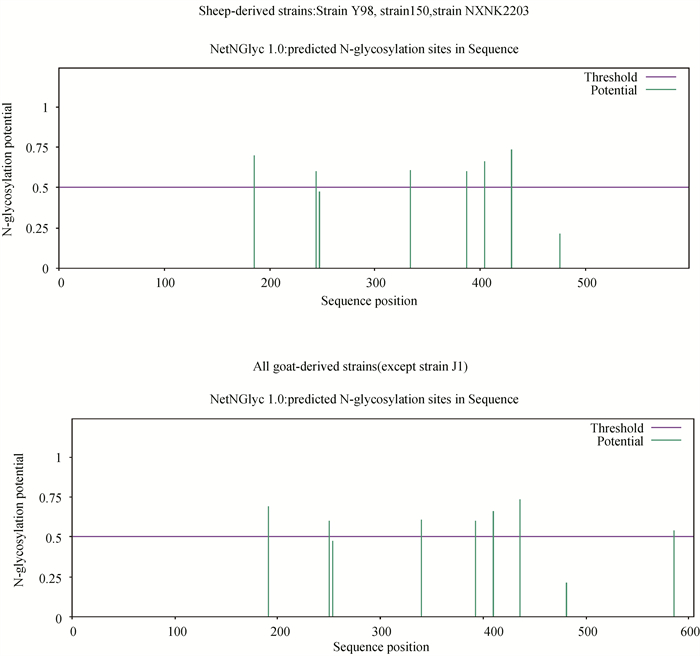
|
图 5 N末端糖基化位点分析 Fig. 5 Analysis of N-terminal glycosylation sites |
目前检测Movi最常用的方法为分子生物学检测方法,包括常规PCR、qPCR、LAMP等方法。但是,LAMP方法假阳性较多;常规PCR敏感性不如qPCR,对于核酸含量低的样品(如鼻拭子)可能存在漏检。目前已有基于16S rRNA基因、EF-Tu基因建立的SYBR Green实时荧光定量PCR方法及基于P113基因建立的TaqMan qPCR检测方法用于Movi流行病学调查[20-22]。Deshuillers等[23]报道称,Movi分离株的基因组序列操纵子上存在2个重复的16S rRNA基因序列,这2个重复序列可能分别对应不同的支原体属或为单一菌属的重复序列,这提示16S rRNA基因作为Movi系统鉴定靶标的特异性并不够强[24-26]。Zhang等[27]研究也表明,Hsp70基因比16S rRNA基因更适合用于评估Movi与其它支原体的遗传关系,可作为潜在的分子标记。另有研究报道,基于Hsp70基因建立的常规PCR方法比基于16S rRNA基因建立的常规PCR方法的灵敏度和特异性都有所提高[28-29]。
本研究基于Hsp70基因与基于P113基因的TaqMan qPCR检测方法对样品进行平行检测比较两者符合率,结果发现基于Hsp70基因的TaqMan qPCR检测方法对临床样品(尤其是鼻拭子样品)有更高的阳性率,可能的原因有两个方面:一是Hsp70基因是高度保守的生物分子,本研究设计的引物及探针几乎涵盖了不同Movi菌株Hsp70基因序列;二是Hsp70是在生物细胞中高效表达的一种热休克蛋白。本研究中临床鼻拭子样品检测结果的阳性率为52.27%,疑似MPSG的肺组织样品检测结果的阳性率为93.02%,如此高的阳性率主要是因为根据本团队前期对福建省MPSG的流行病学调查结果显示,福建省内该病的主要流行病原为Movi,其次为Mmc[30]。
Maksimović 等[31]通过SDS-PAGE、Western blotting、随机扩增多态性DNA标记(RAPD)和基于Hsp70基因序列这4种方法研究不同来源的Movi菌株,结果显示山羊和绵羊菌株之间具有高度的表型和基因型的异质性。研究还发现,从绵羊和山羊身上分离的大多数欧洲菌株与亚洲菌株也不同,表明Movi因其宿主和地理来源的不同呈现特征性差异。Jaÿ等[8]基于全基因组单核苷酸多态性(SNP)系统发育表明,无论其地理来源如何,菌株都以其宿主物种为基础进行聚类。本研究基于Hsp70基因构建的进化树结果显示,Movi分离株进化树可以分成两个大分支:Cluster Ⅰ(宿主均为山羊)和Cluster Ⅱ(宿主均为绵羊),说明同一宿主来源的Movi进化关系较近。
蛋白质的N-糖基化修饰是生物体调控蛋白质在组织和细胞中的定位、功能、活性、寿命和多样性的一种普遍的翻译后修饰,其有重要调节蛋白质功能的作用。郭鹏等[32]研究发现,突变N-糖基化位点使热休克蛋白gp96的ATPase活性、抗原交叉呈递能力和免疫佐剂功能减弱,提示糖基化水平对热休克蛋白gp96免疫功能产生直接影响。乙型脑炎病毒(Japanese enc ephalitis virus,JEV)的PrME蛋白和NS1蛋白的N-糖基化在免疫过程中起着重要的作用,一个N-糖基化消除的prME基因可以诱导较高的ELISA抗体、中和抗体和免疫保护,但两个N-糖基化都缺乏的基因则导致免疫力下降,在野生型和突变型NS1组中也观察到类似的情况[33]。禽流感病毒(Avian Influenza Virus,AIV)HA蛋白上受体结合区域的糖基化修饰对受体结合特性有较大影响。H7N1病毒HA蛋白在150-loop的第158位新增一个糖基化修饰,形成空间干扰从而阻碍受体的结合,最终降低病毒对受体的亲和力[34]。H5N6病毒HA蛋白第158位出现糖基化缺失后,不仅导致对人型受体的结合特异性增强,而且增强了其在豚鼠中的致病性[35]。以上研究表明,N-糖基化位点的增加、缺失可能对蛋白的生物学活性及免疫特性产生影响。本研究中发现Movi山羊源分离株(除了J1)比绵羊源分离株均在586位多了1个N末端糖基化位点,山羊源586位的糖基化位点对于Movi的感染、致病性及免疫应答有无影响及其作用机制还需更进一步深入研究。
4 结论本研究建立了特异性强、敏感性高、重复性优的基于Hsp70基因的Movi的qPCR检测方法。序列分析发现,不同来源Movi的Hsp70基因核苷酸同源性较高,所有山羊源的菌株均比绵羊源菌株多了1个N-糖基化位点;遗传进化分析证实,Movi福建株与山羊源分离株遗传关系较近(均处于山羊源ClusterⅠA亚分支)。本研究不仅为Movi临床诊断提供了检测技术支撑,更为后续进一步研究Movi遗传演化规律提供参考。
| [1] |
ZHAO J Y, DU Y Z, SONG Y P, et al. Investigation of the prevalence of Mycoplasma ovipneumoniae in southern Xinjiang, China[J]. J Vet Res, 2021, 65(2): 155-160. DOI:10.2478/jvetres-2021-0021 |
| [2] |
GAETA N C, DE SÁ GUIMARÃES A M, TIMENETSKY J, et al. The first Mycoplasma ovipneumoniae recovered from a sheep with respiratory disease in Brazil-draft genome and genomic analysis[J]. Vet Res Commun, 2022, 46(4): 1311-1318. DOI:10.1007/s11259-022-09972-x |
| [3] |
SPAAN R S, EPPS C W, CROWHURST R, et al. Impact of Mycoplasma ovipneumoniae on juvenile bighorn sheep (Ovis canadensis) survival in the northern basin and range ecosystem[J]. Peer J, 2021, 9: e10710. DOI:10.7717/peerj.10710 |
| [4] |
BESSER T E, CASSIRER E F, POTTER K A, et al. Association of Mycoplasma ovipneumoniae Infection with population-limiting respiratory disease in free-ranging rocky mountain bighorn sheep (Ovis canadensis canadensis)[J]. J Clin Microbiol, 2008, 46(2): 423-430. DOI:10.1128/JCM.01931-07 |
| [5] |
HANDELAND K, TENGS T, KOKOTOVIC B, et al. Mycoplasma ovipneumoniae-a primary cause of severe pneumonia epizootics in the Norwegian muskox (Ovibos moschatus) population[J]. PLoS One, 2014, 9(9): e106116. DOI:10.1371/journal.pone.0106116 |
| [6] |
HIGHLAND M A, HERNDON D R, BENDER S C, et al. Mycoplasma ovipneumoniae in wildlife species beyond subfamily Caprinae[J]. Emerg Infect Dis, 2018, 24(12): 2384-2386. DOI:10.3201/eid2412.180632 |
| [7] |
MANLOVE K, BRANAN M, BAKER K, et al. Risk factors and productivity losses associated with Mycoplasma ovipneumoniae infection in United States domestic sheep operations[J]. Prev Vet Med, 2019, 168: 30-38. DOI:10.1016/j.prevetmed.2019.04.006 |
| [8] |
JAŸ M, AMBROSET C, TRICOT A, et al. Population structure and antimicrobial susceptibility of Mycoplasma ovipneumoniae isolates in France[J]. Vet Microbiol, 2020, 248: 108828. DOI:10.1016/j.vetmic.2020.108828 |
| [9] |
LINDSTRÖM L, TAUNI F A, VARGMAR K. Bronchopneumonia in Swedish lambs: a study of pathological changes and bacteriological agents[J]. Acta Vet Scand, 2018, 60(1): 54. DOI:10.1186/s13028-018-0409-1 |
| [10] |
DEENEY A S, COLLINS R, RIDLEY A M. Identification of Mycoplasma species and related organisms from ruminants in England and wales during 2005-2019[J]. BMC Vet Res, 2021, 17(1): 325. DOI:10.1186/s12917-021-03037-y |
| [11] |
CHEN C, QIAO J, MENG Q L, et al. Serological and molecular survey of sheep infected with Mycoplasma ovipneumoniae in Xinjiang, China[J]. Trop Anim Health Prod, 2015, 47(8): 1641-1647. DOI:10.1007/s11250-015-0908-2 |
| [12] |
许健, 储岳峰, 赵萍, 等. 中国部分地区绵羊肺炎支原体的基因多态性分析[J]. 畜牧兽医学报, 2011, 42(9): 1295-1301. XU J, CHU Y F, ZHAO P, et al. Investigation of gene polymorphism on Mycoplasma ovipneumoniae isolates from some areas of China[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(9): 1295-1301. (in Chinese) |
| [13] |
许健, 储岳峰, 高鹏程, 等. 我国17株绵羊肺炎支原体分子分型及其菌体蛋白免疫印迹[J]. 微生物学报, 2011, 51(10): 1421-1426. XU J, CHU Y F, GAO P C, et al. Molecular typing and immunoblotting of 17 Mycoplasma ovipneumoniae isolates from China[J]. Acta Microbiologica Sinica, 2011, 51(10): 1421-1426. (in Chinese) |
| [14] |
MAKSIMOVIĆ Z, RIFATBEGOVIĆ M, LORIA G R, et al. Mycoplasma ovipneumoniae: a most variable pathogen[J]. Pathogens, 2022, 11(12): 1477. DOI:10.3390/pathogens11121477 |
| [15] |
LI M, MA C J, LIU X M, et al. Molecular cloning of HSP70 in Mycoplasma ovipneumoniae and comparison with that of other mycoplasmas[J]. Genet Mol Res, 2011, 10(2): 834-848. DOI:10.4238/vol10-2gmr1193 |
| [16] |
JIANG F, HE J Y, NAVARRO-ALVAREZ N, et al. Elongation factor Tu and heat shock protein 70 are membrane-associated proteins from Mycoplasma ovipneumoniae capable of inducing strong immune response in mice[J]. PLoS One, 2017, 11(8): e0161170. |
| [17] |
TIWARI S, THAKUR R, SHANKAR J. Role of heat-shock proteins in cellular function and in the biology of fungi[J]. Biotechnol Res Int, 2015, 2015: 132635. |
| [18] |
MULTHOFF G, POCKLEY A G, STREFFER C, et al. Dual role of heat shock proteins (HSPs) in anti-tumor immunity[J]. Curr Mol Med, 2012, 12(9): 1174-1182. DOI:10.2174/156652412803306666 |
| [19] |
MULTHOFF G, POCKLEY A G, SCHMID T E, et al. The role of heat shock protein 70 (Hsp70) in radiation-induced immunomodulation[J]. Cancer Lett, 2015, 368(2): 179-184. DOI:10.1016/j.canlet.2015.02.013 |
| [20] |
林裕胜, 江锦秀, 张靖鹏, 等. 绵羊肺炎支原体TaqMan荧光定量PCR检测方法的建立[J]. 中国预防兽医学报, 2018, 40(4): 316-320. LIN Y S, JIANG J X, ZHANG J P, et al. Establishment of a TaqMan real-time fluorescent quantitative PCR assay for detection of Mycoplasma ovipneumoniae[J]. Chinese Journal of Preventive Veterinary Medicine, 2018, 40(4): 316-320. (in Chinese) |
| [21] |
黄坚, 尹正军, 岳华, 等. 绵羊肺炎支原体荧光定量PCR检测方法的建立与应用[J]. 中国兽医科学, 2016, 46(10): 1270-1276. HUANG J, YIN Z J, YUE H, et al. Development and application of quantitative real-time PCR for the detection of Mycoplasma ovipneumoniae[J]. Chinese Veterinary Science, 2016, 46(10): 1270-1276. (in Chinese) |
| [22] |
程振涛, 张双翔, 王慧, 等. 绵羊肺炎支原体实时荧光定量PCR检测方法的建立[J]. 西北农业学报, 2013, 22(2): 7-12. CHENG Z T, ZHANG S X, WANG H, et al. Establishment of real-time fluorescent quantitative PCR assay for detection of Mycoplasma ovipneumoniae[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2013, 22(2): 7-12. (in Chinese) |
| [23] |
DESHUILLERS P L, SANTOS A P, DO NASCIMENTO N C, et al. Complete genome sequence of Mycoplasma ovis strain Michigan, a hemoplasma of sheep with two distinct 16S rRNA genes[J]. Genome Announc, 2014, 2(1): e01235-13. |
| [24] |
NOLL L W, HIGHLAND M A, HAMILL V A, et al. Development of a real-time PCR assay for detection and differentiation of Mycoplasma ovipneumoniae and a novel respiratory-associated Mycoplasma species in domestic sheep and goats[J]. Transbound Emerg Dis, 2022, 69(5): e1460-e1468. |
| [25] |
KAMLA V, HENRICH B, HADDING U. Phylogeny based on elongation factor Tu reflects the phenotypic features of mycoplasmas better than that based on 16S rRNA[J]. Gene, 1996, 171(1): 83-87. DOI:10.1016/0378-1119(95)00884-5 |
| [26] |
SÖDERLUND R, BÖLSKE G, HOLST B S, et al. Development and evaluation of a real-time polymerase chain reaction method for the detection of Mycoplasma felis[J]. J Vet Diagn Invest, 2011, 23(5): 890-893. DOI:10.1177/1040638711407479 |
| [27] |
ZHANG B, HAN X, YUE H, et al. Molecular characterization of the heat shock protein 70 gene in Mycoplasma ovipneumoniae[J]. Vet J, 2013, 198: 299-301. DOI:10.1016/j.tvjl.2013.07.001 |
| [28] |
MCAULIFFE L, HATCHELL F M, AYLING R D, et al. Detection of Mycoplasma ovipneumoniae in Pasteurella-vaccinated sheep flocks with respiratory disease in England[J]. Vet Rec, 2003, 153(22): 687-688. DOI:10.1136/vr.153.22.687 |
| [29] |
YANG F L, HAN X, TANG C, et al. A Hsp70 gene-based PCR for detection of Mycoplasma ovipneumoniae[J]. J Anim Vet Adv, 2013, 12(19): 1495-1497. |
| [30] |
江锦秀, 林裕胜, 游伟, 等. 福建省羊支原体性肺炎分子流行病学研究[J]. 福建农业学报, 2017, 32(1): 12-16. JIANG J X, LIN Y S, YOU W, et al. A molecular epidemiological study on Mycoplasma pneumonia of sheep and goats in Fujian[J]. Fujian Journal of Agricultural Sciences, 2017, 32(1): 12-16. (in Chinese) |
| [31] |
MAKSIMOVIĆ Z, DE LA FE C, AMORES J, et al. Comparison of phenotypic and genotypic profiles among caprine and ovine Mycoplasma ovipneumoniae strains[J]. Vet Rec, 2017, 180(7): 180. DOI:10.1136/vr.103699 |
| [32] |
郭鹏, 李长菲, 鞠莹, 等. N-糖基化修饰对热休克蛋白gp96免疫学功能的影响[J]. 生物工程学报, 2021, 37(11): 4036-4046. GUO P, LI C F, JU Y, et al. N-glycosylation modification of heat shock protein gp96 affects its immunological function[J]. Chinese Journal of Biotechnology, 2021, 37(11): 4036-4046. (in Chinese) |
| [33] |
张子重, 汪雪, 宰俊杰, 等. N-糖基化对乙型脑炎病毒prME和NS1基因免疫效果影响的研究[J]. 病毒学报, 2012, 28(3): 213-218. ZHANG Z Z, WANG X, ZAI J J, et al. Roles of N-glycosylation in immunity of prME and NS1 gene of JEV[J]. Chinese Journal of Virology, 2012, 28(3): 213-218. (in Chinese) |
| [34] |
BANKS J, SPEIDEL E S, MOORE E, et al. Changes in the haemagglutinin and the neuraminidase genes prior to the emergence of highly pathogenic H7N1 avian influenza viruses in Italy[J]. Arch Virol, 2001, 146(5): 963-973. DOI:10.1007/s007050170128 |
| [35] |
GU M, LI Q H, GAO R Y, et al. The T160A hemagglutinin substitution affects not only receptor binding property but also transmissibility of H5N1 clade 2.3.4 avian influenza virus in guinea pigs[J]. Vet Res, 2017, 48(1): 7. DOI:10.1186/s13567-017-0410-0 |
(编辑 范子娟)



