2. 中国科学院亚热带农业生态研究所,长沙 410125
2. Institute of Subtropical Agriculture, Chinese Academy of Sciences, Changsha 410125, China
牛、羊、猪可以为市场带来大量的肉、奶以及皮毛等重要产品,是社会发展中重要的经济动物。近年来,家畜重要性状如肉质、生长、繁殖、抗病能力等备受研究者关注。基因组遗传变异,是引起个体性状差异的重要遗传基础。基因组变异按大小可分为单核苷酸多态性(single nucleotide polymorphism,SNP。即单碱基变化)、插入缺失(insertion and deletion,Indel。即大小在2~50 bp之间的小结构变异)和结构变异(structural variation,SV。即大于50 bp的大结构变异)。CNV指大小从50 bp至5 Mb的DNA片段发生拷贝数目变化的结构变异,包括缺失、插入、重复、重排、倒位等[1]。其中最为常见的形式是单个DNA片段的重复增加,比如SD序列(segmental duplication,SD)[2]。相比于SNP等其他变异,CNV具有涉及碱基更多、覆盖范围更广以及检测更容易的特点,虽然CNV发生的频率较低,但是累积发生的片段长度明显超过了其他序列变异[3]。因此,CNV对家畜重要经济性状可能造成很大影响,其在动物分子育种应用中具有广阔前景。随着分子生物学和基因组学研究技术的发展,CNV的检测方法也不断革新。本文系统综述了在牛、羊、猪和马等家畜中基于全基因组CNV的主要检测方法发掘的功能CNV及相关分析进展,以期为家畜CNV研究提供参考。
1 CNV检测技术的应用现状分析对于基因组中未知序列的CNV,比较基因组杂交技术(comparative genome hybridization,CGH)、SNP芯片技术和基于新一代基因测序技术(next generation sequencing,NGS)的全基因组重测序(whole genome sequencing,WGS)被认为是目前全基因组范围内寻找CNV的主要有效手段[4-5]。其中,WGS技术包括基于二代测序(next generation sequencing,NGS)和三代测序(third generation sequencing,TGS)技术。
1.1 CGH技术CGH技术是将在一张芯片上的试验样本与对照样本分别标记上不同的荧光素,然后再进行杂交,根据特定分析软件中各位点的荧光信号强度和比值等,确定是否存在CNV及其在基因组中的位置等信息[6]。随着技术的发展,CGH芯片技术(comparative genome hybridization,CGH)被开发并且用于CNV的检测。CGH芯片技术具有灵敏度高、分辨率准确以及探针全基因组覆盖的优点,其探针密度与试验结果的准确性显著相关。
1.2 SNP芯片技术SNP芯片技术用于拷贝数鉴定和基因型分型,具有快速、方便的特点,检测时只需对待测样本进行酶切和荧光标记,然后与SNP芯片杂交,根据所得图像中荧光信号的强弱,比较待测样本与其他个体的相对强度,以此来确定每个位点的相对拷贝数[7]。但是,SNP芯片也存在一定的局限性。例如,SNP芯片的探针在基因组上经常出现分布不均匀的情况[8];并且在重复序列区域(如SD序列较多的拷贝数多态性区域)难以进行探针设计[7];此外,SNP芯片检测平台对较小的CNVs检出率较低。
1.3 WGS技术基于二代测序技术的WGS技术检测CNV时是将待测样本的DNA序列随机打断并加上接头,经过PCR扩增后,将这些reads与参考基因组的序列进行对比分析,然后通过隐马尔科夫模型(hidden markov model,HMM)以映射结果来判断CNV[9]。与其他的检测方法相比,NGS测序技术具有高通量、高分辨率、高重复性和速度快等显著优点,还能获得更多且更为精细的变异结构[10]。但是二代测序读长太短,基因组组装高度片段化,导致CNV检测结果的准确性受到限制。在进行NGS测序时需要提高对样品的测序深度要求,并且在结果分析前需对测序结果进行适当校正[11]。
基于第三代测序技术的WGS技术主要包括了单分子荧光测序(single molecule real time sequencing,SMRT)和纳米孔测序[12]。PacBio SMRT技术以SMRT芯片为核心,可以实现边合成边测序。纳米孔测序的基本原理是每个接头序列(adaptor)通过纳米孔时都会产生一个阻断电流,根据产生的阻断电流的变化识别各种碱基[13]。第三代测序技术可以减少PCR过程中碱基替换以及交叉污染等其他干扰因素,有效地减少测序结果的假阳性[14]。另外,长读长的特点还有助于提高SV检测效率[15]。近年来,新兴的第三代测序技术发展迅速,凭借长读长优势,三代测序在基因组从头测序和重测序方向具有广阔的发展前景,尤其是对二代测序难以发现的SV和较复杂的CNV,可利用三代测序进行鉴定。由此可见,第三代测序技术在CNV研究方面具有广泛的应用前景。
为综合分析CNV检测技术的应用现状,以“copy number variation;livestock”作为关键词,根据web of science 2005年至2022年发表文章数量进行统计分析(图 1)。CGH技术自2011年以后整体就呈现下降趋势,近年来已少有相关研究应用。而SNP芯片技术与WGS技术成为近年来用于检测家畜全基因组CNV的主要手段。但是,2014年以后,SNP技术的应用频率在逐年下降,推测应与第二代测序技术逐渐推广应用有关。第二代测序技术具有高通量、高准确度的优点,弥补了SNP技术难以检测较小CNV的不足。22年开始第二代测序技术使用频率有所下降,第三代测序技术逐渐兴起,其快速、读长更长、检测更准确的特点使得其被广泛应用于复杂CNV以及SV的检测。
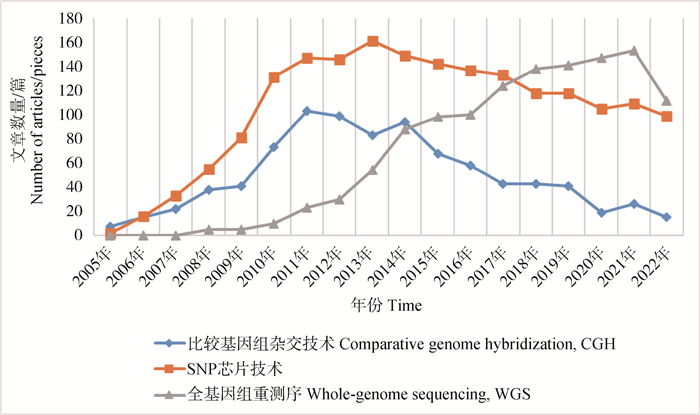
|
图 1 家畜基因组拷贝数变异检测技术使用热度分析 Fig. 1 Analysis of the detection technology for genome copy number variation in livestock |
基因组CNV可通过改变基因剂量、调控基因表达和暴露隐性等位基因等几种方式影响生物表型。家畜的生长发育受到多基因、多通路以及环境等多方面的综合调控,CNV可通过改变剂量敏感基因拷贝数影响该基因的表达量及相关表型。为此,研究者在家畜中开展了对于CNV的大量鉴定及其对表型性状影响的研究。下面将分别在牛、羊、猪和马4种家畜上综述基于CGH芯片、SNP芯片和NGS测序技术检测CNV并分析其功能的研究进展。
2.1 牛基因组CNV检测与分析对牛基因组CNV的部分研究汇总如表 1所示。
|
|
表 1 基于CGH、SNP和NGS技术对牛基因组CNV的检测及分析 Table 1 Detection and analysis of bovine genome CNV based on CGH, SNP and NGS techniques |
基于aCGH芯片技术:2010年,Liu等[16]首次使用了aCGH技术对5个品种的牛进行全基因组水平分析,结果表明存在着200多个特异性的拷贝数变异区域(copy number variation regions,CNVRs),其中约有67%的CNVRs完全或部分跨越了牛基因组,61%的CNVRs直接与片段重叠,并且与牛的免疫、繁殖、哺乳等性状显著相关。Zhang等[17]利用aCGH技术对3个品种的牛进行基因组CNV的检测,据此构建了黄牛基因组CNV草图。陈宏课题组[17-18]利用CGH芯片技术对荷斯坦奶牛、黄牛、水牛和牦牛进行了基因组CNV的检测,发现相关功能基因,如PLA2G2D(phospholipase a2 group IID)、CYP4A11(cytochrome p450 family 4 subfamily a member 11)、CTR9 (CTR9 homolog, paf1/RNA polymerase Ⅱ complex component) 等在牛的表型性状中发挥着重要的作用。
基于SNP芯片:在牛上,Illumina BovineSNP50芯片和BovineHD SNP芯片的开发对大群体进行CNV检测具有重要的意义。利用Illumina BovineSNP 50 K BeadChip,Bae等[19]对17个品种的248头牛进行全基因组CNV检测,由此绘制出牛CNVs图谱。Kumar等[20]在塔尔帕克牛中发现了447个CNVR,约占牛基因组的2.17%,许多与QTL(quantitative trait locus,QTL)重叠的CNVRs与奶牛乳腺炎和乳成分、蛋白质含量等产奶性状相关。利用BovineHD SNP芯片,Zhou等[21]在荷斯坦奶牛中将CNV与生产性状进行全基因组关联分析(genome-wide association studies,GWAS)分析,结果显示CNVs与采食量、乳品质、母牛繁殖力等生产性状显著相关;另外检测到与饲料转化效率和采食量相关的CNV与嗅觉受体基因OR2A2(olfactory receptor family 2 subfamily a member 2)重叠。Ahmad等[22]对96头Vrindavani牛进行CNV检测,结果发现存在71个CNVRs,其中有5个CNVRs与10个重要的生产与繁殖性状显著相关,包括峰值产奶量、泌乳天数、产犊间隔期等,并且与影响产奶量和乳成分以及繁殖和免疫功能性状的重要QTLs和WASHC4(WASH complex subunit 4)、HS6ST3(heparan sulfate 6-o-sulfotransferase 3)、MBNL2(muscleblind like splicing regulator 2)等基因重叠。
基于NGS技术:Gao等[23]在荷斯坦牛中检测到14 821个CNV,487个CNVR,CNVR区域内的的功能基因与牛的脂肪性状显著相关。Xu等[24]和Liu等[25-26]在南阳牛和秦川牛鉴定出2 907个CNV,利用qPCR技术发现LEPR(leptin receptor)、SHH(sonic hedgehog signaling molecule)、MAPK10(mitogen-activated protein kinase 10)基因的CNV在牛的肌肉脂肪沉积中发挥着重要的作用。Liu等[27]在水牛全基因组中检测到1 344个CNVR,与CNVRs重叠的1 245个基因与水牛的免疫反应、信号转导以及氧气转运等功能显著相关。Singh等[28]研究了多个瘤牛和普通牛品种的CNVRs模式,发现瘤牛品种在常染色体上共发现2 590个CNVs和335个CNVRs, CNVRs横跨嗅觉受体和免疫系统基因区域。
2.2 羊基因组CNV检测与分析由于山羊和绵羊的全基因组测序完成较晚,因此关于羊基因组CNV的研究较牛更晚。山羊和绵羊CNV研究进展较为有限,且人们在日常生活中常将绵羊和山羊都统称为羊,所以下面将绵羊与山羊基因组CNV研究进行整合综述如表 2所示。
|
|
表 2 基于CGH、SNP和NGS技术对羊基因组CNV的检测及分析 Table 2 Detection and analysis of sheep genome CNV based on CGH, SNP and NGS techniques |
基于aCGH技术:2010年,Fontanesi等[29]就利用了aCGH方法对4个欧洲山羊品种进行CNV检测,检测到161个CNVRs;除此之外,在绵羊基因组中发现了186个CNVRs,由此首次构建了绵山羊比较基因组图谱。Jenkins等[30]利用Roche-NimbleGen 2.1 M CGH对36头绵羊进行全基因组测定,确定了3488个CNVRs,总长度为67.6 Mb,相当于绵羊染色体的2.7%。但是由于绵羊的基因组序列不同于牛的基因组序列,所以跨物种杂交构建的图谱具有较大的误差,从而导致检测CNV结果不准确。
基于SNP芯片:随着绵羊和山羊基因组测序的完成,基于羊参考基因组的SNP芯片得以生产和应用。低密度芯片(5K和12K)、50K中密度SNP芯片(OvineSNP50 BeadChip array)和600K高密度SNP芯片(Ovine HD SNP BeadChip)在羊上发挥着重要的作用。Salehian-Dehkordi等[31]用绵羊Infinium HD(600K)SNP基因芯片对67个群体的绵羊进行CNV检测,在1 217个CNVRs中检测到7 547个独特的CNV,覆盖245 Mb,占整个绵羊基因组的10%。利用绵羊50K中密度SNP芯片,Yang等[32]利用50K中密度SNP芯片对全世界范围内的绵羊进行CNV检测,结果表明不同地理位置的绵羊品种之间的CNVRs存在种群差异。Kang等[33]对五个奶山羊品种进行CNV检测,鉴定出42个CNV,并且CNV5和CNV25与奶山羊的产奶性状显著相关。Moradi等[34]利用llumina Ovine SNP 50K BeadChip对45只阿富汗绵羊进行全基因组CNV检测,共鉴定出376个CNVRs,并首次绘制了阿富汗绵羊品种基因组CNV图谱;生物信息学分析表明这些CNVRs与许多与免疫系统发育、生长、繁殖和环境适应等各种途径相关的基因或QTL重叠;与伊朗绵羊中的CNVRs相比,有20个CNVRs为二者共有,且大多与影响生产、繁殖和免疫系统的基因重叠。在山羊ADAPTmap国际合作项目中,Liu等[35]研究了来源于世界不同地理位置的共50个山羊品种1 023头山羊的CNVs,检测出978个CNVRs,约占山羊基因组的8.96%,同时还检测出EDNRA(endothelin receptor type A)、ADAMTS20(ADAM metallopeptidase with thrombospondin type 1 motif 20)、ASIP(agouti signaling protein)、KDM5B(lysine demethylase 5B)、ADAM8(ADAM metallopeptidase domain 8)、DGAT1(diacylglycerol o-acyltransferase 1)等影响毛色、肌肉生长、脂肪生成和泌乳性能的基因也存在CNV。然后,Liu等[36]利用qPCR技术在中国的7个山羊品种中进行DGAT1 CNV验证,发现DGAT1 CNV与山羊的产奶性状显著相关。由于在山羊上基于CNV探寻与表型关联的功能基因还很匮乏,Liu等[37]选择Cameroon、West African Dwarf、Small East African和Landim四个非洲肉用山羊品种,进行了CNV与生长性状的GWAS,发现CNV4和CNV27与胸宽和髋骨宽两个体尺性状显著相关。
基于NGS技术:Nandolo等[38]基于NGS数据在182只非洲山羊中鉴定出6 231个CNVRs,占山羊基因组的59.2 Mb(2.4%),首次绘制出非洲山羊的精细CNV图谱。Zhang等[39]对崂山奶山羊产仔数不同的高低组分别进行CNV检测,发现PRP1和PRP6等与乳腺生长发育和泌乳性能相关的功能基因存在CNV。Yuan等[40]在3个细毛绵羊品种中确定了总长度56.06 Mb的7 228个CNVR,与CNVR相关的基因参与绵羊的生长发育和养分代谢的过程。通过比较家养山羊和野山羊的基因组,Dong等[41]发现了造成家山羊皮毛颜色变浅的主要原因是刺蛋白基因(ASIP)有关的拷贝数重复增加, 也有研究证明ASIP基因重复拷贝导致绵羊白色表型[42]。Chebii等[43]在努比亚山羊中发现191个CNV,这些CNV与编码蛋白质的基因重叠,参与山羊的免疫反应、外源性代谢以及能量代谢。Hu等[44]利用24个重测序数据集,对青藏高原3个生态群体的藏羊CNVs进行特征分析,共鉴定出2 777个CNVRs,其中1 098个共享CNVRs在与ABC转运蛋白、嗅觉转导和氧气转运有关的通路中显著富集,188个CNVRs与生长和胴体QTL, 免疫球蛋白QTL, 产奶量QTL等97个数量性状位点重叠。
2.3 猪基因组CNV检测与分析在以往的研究中,由于成本和技术的限制,猪CNVs图谱的分辨率还很低。现随着测序技术的不断发展和成本的不断降低,研究者获得更高分辨率的CNVs图谱。表 3汇总了目前对猪基因组CNVs的部分研究报道。
|
|
表 3 基于CGH、SNP和NGS技术对猪基因组CNV的检测及分析 Table 3 Detection and analysis of pig genome CNV based on CGH, SNP and NGS techniques |
基于aCGH技术:2008年,Fadista等[45]首次利用定制的385K aCGH芯片在杜洛克猪上进行CNV检测,一共鉴定出37个CNVRs。Wang等[46]利用定制的1 M aCGH芯片对9个猪种共12头进行CNV检测,鉴定出758个CNVRs,覆盖了猪基因组序列的47.43 Mb。马海明团队的刘晓琴等[47]以产肉量性状差异显著的大白猪和湖南地方品种沙子岭猪为研究对象,采用比较基因组杂交技术筛选猪CNVs,鉴定出与CNVs紧密连锁的Wnt蛋白。
基于SNP芯片:邱恒清等[48]利用件1.4 M高密度SNP芯片对319头巴马香猪进行CNV检测,采用PennCNV和R-Gada两种软件分析,结果显示PennCNV检测到6 327个CNVs,R-Gada软件检测到3 489个CNVs,结果表明,随着SNP芯片密度的增加,CNV的检测结果越加准确。黄路生课题组[49]利用SNP60 BeadChip和PennCNV对1693头猪检测发现565个CNVRs,相当于猪基因组的5.84%,还筛选出ANP32B(acidic nuclear phosphoprotein 32 family member B)、GDF3(growth differentiation factor 3)、GYS1(glycogen synthase 1)等基因上存在CNV,可作为胴体长度、背脂厚度、肩胛骨长度等性状的候选基因。Wang等[50]使用80K SNP BeadChip检测857头大白猪的全基因组CNV,一共检测到312个CNVs,覆盖了57.76 Mb的猪基因组。Xu等[51]基于Porcine 80K SNP BeadChip数据安徽地方猪(AHIP)和西方商品猪(WECP)进行了全基因组CNVs分析比较,基因功能富集分析发现在AHIP群体中与免疫(FOXJ1(forkhead box J1)、FOXK2(forkhead box K2)、MBL2(mannose binding lectin 2)、TNFRSF4(TNF receptor superfamily member 4)、SIRT1(sirtuin 1)、NCF1(neutrophil cytosolic factor 1)和肉质(DGAT1(diacylglycerol o-acyltransferase 1)、NT5E(5′-Nucleotidase ecto))相关的基因与CNVRs重叠;而这些基因在WECP群体中为缺失型。
基于NGS技术:Jiang等[52]利用NGS对10个猪种进行全基因组分析,构建了当时分辨率最高的猪CNV和SD图谱。此外,Zheng等[53]对梅山猪和杜洛克猪进行NGS测序,鉴定出梅山猪中的6 387个CNVRs与编码芳香烃受体(aryl hydrocarbon receptor,AHR)基因的繁殖相关基因存在重叠。Ran等[54]发现MTHFSD(methenyltetrahydrofolate synthetase domain containing)基因CNVs影响湘猪的产子数性状。Qiu等[55]对美国和加拿大的杜洛克猪进行全基因组分析,发现有10个CNVRs与美国杜洛克猪的平均日增重等性状有关,而9个CNVRs与加拿大杜洛克猪的平均日增重和背臀部厚度有关。另外,Ding等[56]对杜洛克猪进行全基因组重测序分析,发现23 856个CNVRs,相关性分析结果表明9个CNVRs与杜洛克猪腰肌面积、腰肌深度和瘦肉率相关,鉴定了DOK7(docking protein 7)、ARAP1(ArfGAP with RhoGAP domain, ankyrin repeat and PH domain 1)、ELMO2(engulfment and cell motility 2)和SLC13A3(solute carrier family 13 member 3)相关的候选功能基因。Wei等[57]在大白猪与民猪2代群体中共鉴定出1 185个CNV区域(CNVRs),将CNVs与IMF进行GWAS分析发现,19个CNVRs与IMF显著相关,CNVR可能通过调节PELP1基因的选择性剪接来影响IMF的含量,并最终影响PELP1蛋白的结构。Fan等[58]对安庆六白猪与亚洲野猪进行CNV检测,共鉴定出10 429个CNVRs,在96个CNVRs区域中鉴定出65个与生长、繁殖、代谢等性状相关的功能基因,如CD36(CD36 molecule)、CIT(citron rho-interacting serine/threonine kinase)、RLN(relaxin)、CYP3A29(cytochrome P450 family 3 subfamily a member 29)、ELOVL6(ELOVL fatty acid elongase 6)等,QTL重叠区域与肉质性状、生长和免疫相关,这与CNV分析一致。Zhang等[59]对皖南黑猪与亚洲野猪CNVs进行比较,对所选的288个CNVs中基因进行功能富集分析,鉴定出NDN(necdin, MAGE family member)、TMOD4(tropomodulin 4)、GJA1(gap junction protein alpha 1)、MAP3K5(mitogen-activated protein kinase kinase kinase 5)等基因,与肌肉生长、繁殖、剩余采食量、耳朵大小等性状相关。Long等[60]在杜洛克猪、长白猪和约克夏猪中发现罕见的CNV可能与猪脐疝疾病有着重要的联系。
2.4 马基因组CNV检测与分析由于国内现代马业起步晚,近年来人们才逐渐重视起国内马业的现代化进程,国内马CNVs相关研究还亟待探索。
基于CGH技术:王伟[61]在6匹不同品种的马(蒙古马, 阿巴嘎马, 河曲马, 哈萨克马, 德保矮马和纯血马)的常染色体中共鉴别了700个CNVs, 大小从6.1 kb到0.57 Mb。基于SNP芯片技术,2022年Wang等[62]在晋江马等10个中国本土马种共301个个体中鉴别了577个CNVs。
基于WGS技术:2021年Durward-Akhurst等[63]对534匹马进行了变异检测,并针对10个目标品种确定了单品种特有的变体。Al Abri等[64]针对来自6个不同品种的共6匹马(美国迷你马、佩尔什马、阿拉伯马、曼加拉加马查多尔马、蒙古马和田纳西走马)鉴定并功能注释了17 514 723个SNPs,1 923 693个Indels,以及平均每匹马1 540个CNVs和3 321个SVs。Gu等[65]首次在染色体水平上从头组装出10个全球代表马种的三维基因组,并对马基因组上的大片段SVs进行了全面解析,通过拓扑相关结构域(topologically associating domains,TAD)研究了SVs对染色质空间结构的潜在影响,还发现品种特异性SVs显著富集了品种特异性TADs。Tang等[66]对来自全世界的16个马品种共97匹马进行了全基因组重测序,各品种鉴定到5 023~44 681个CNVRs,CNVRs长度范围为1.9~8 kb,并鉴定到品种间特异性的CNVRs覆盖的基因(CTSL(cathepsin L)、RAB11FIP3(RAB11 family interacting protein 3) 和CTIF(cap binding complex dependent translation initiation factor))。
3 第三代测序技术检测CNV的应用现状分析以“Pacbio;SMRT; ONT;cattle; sheep; goat;pig”等作为关键词,根据web of science 2011年至2022年发表文章数量进行统计分析,如图 2所示,第三代测序技术主要应用在基因组结构变异鉴定、基因组组装和甲基化检测这3个方面上;受限于成本以及准确率,第三代测序技术在家畜全基因组测序方面尚未广泛应用,目前在牛和猪上的应用较多,在羊上相对较少。以下对目前第三代测序用于牛、羊、猪等家畜全基因组组装和结构变异的检测现状进行综述。

|
图 2 第三代测序技术在家畜中研究应用领域及热度分析 Fig. 2 Research applications and popularity analysis of third generation Sequencing technology in livestock |
在牛上,Gao等[67]对野牦牛和家牦牛进行了染色体水平全基因组组装,并通过牦牛和黄牛的长度数据筛选了SV,在携带高Fst SV的启动子区域基因富集筛选出对缺氧反应起重要作用的调控基因(ARNT(aryl hydrocarbon receptor nuclear translocator)、GATA1(GATA binding protein 1)、EPOR(erythropoietin receptor)、MAFG(MAF BZIP transcription factor G)、KLF5(KLF transcription factor 5))。罗西尔[68]利用第三代测序技术对水牛进行de novo基因组组装,并结合二代Illumina测序数据、Hi-C数据等进行数据的纠错与聚类,最后得到染色体水平的水牛参考基因组,沼泽型水牛和河流型水牛的N50分别为117 Mb和116 Mb。Liu课题组的Gao等[69]使用10x Genomics、PacBio continuous long read(CLR)和Circular consensus sequencing (CCS)以及ONT PromethION技术对牛进行了测序。结果表明,在SV检测方面,长读取覆盖了大多基因组,检测出更多SV,且鉴定出的SV显示出高度均匀性,明显优于短读测序。2020年,Lamb等[70]首次在肉牛中进行第三代测序,重点检测两种已知的与无角症有关的SVs,在澳大利亚婆罗门牛中证明了一个有关的SV。Low等[71]对安格斯牛和婆罗门牛单个杂交个体进行第三代测序,发现存在于婆罗门牛中的特异性SV在磷脂易位(GO: 0045332)、脂质易位(GO: 0034204)、脂质转运(GO: 0006869)和脂质定位(GO: 0010876)中具有3至5.7倍的富集,这表明脂质分布受SV的影响最大。Leonard等[72]对三个不同杂合度的牛进行测序分析,检测到9万个SVs,并且位于13号染色体上ASIP(agouti signaling protein)基因的SV与牛的色素沉淀显著相关。Zhou等[73]利用牛全基因组图谱数据进行分析,发现83 Mb的牛参考基因组中未发现的序列,在各个亚种和品种中发现数百个缺失变异,其中APPL2 SV与牛的品种地理分布有关。
3.2 利用三代测序技术在猪基因组结构变异鉴定在猪上,2012年,Du等[74]对杜洛克猪的长读长数据与短读数据利用不同的程序进行性能比较评估,构建出杜洛克猪基因组草图。同年,通过三代测序技术,五指山猪全基因组组装也完成。之后,研究者们进一步对陆川猪、梅山猪、宁乡猪、五指山猪、藏猪、民猪、巴马香猪等中国地方猪种也通过三代测序等技术陆陆续续完成了基因组组装(表 4)。这些研究为全面解析全基因组遗传信息及种质资源的开发利用提供了参考依据。
|
|
表 4 中国地方猪种三代测序技术研究进展 Table 4 Research progress of 3rd-generation sequencing technology for Chinese local pig breeds |
Ma等[80]利用三代测序技术、Hi-C和Bionano等新技术,首次绘制了高精度宁乡猪基因组图谱,揭示了宁乡猪基因组内丰富的遗传变异,并整合多组学数据鉴定了多个与外国瘦肉型猪杜洛克猪差异较大的基因组SV,并且这些SVs与376个基因关联,利用qPCR验证发现MYL4基因的SV可能是影响宁乡猪皮下脂肪沉积的关键候选基因,同时KEGG富集分析显示差异SVs相关基因与免疫系统、神经系统和脂质代谢等相关。Jiang等[81]整合11个长读长测序数据,构建了猪全基因组图谱,发现了183 352个非冗余SVs(63%为新发现SV),占参考基因组的12.12%。在梅山猪中,研究者检测出TEX11 SV与IL18RAP SV,推测其可能通过调控靶基因成为梅山猪高繁殖性能和抗病的重要候选变异。在藏猪中,研究者发现了7 568个特有SVs,这些SV显著富集在与脂肪和血液代谢相关的性状,如肌酐水平、接触球蛋白浓度、肌酸激酶水平和高密度脂蛋白胆固醇等。其中,SOD1(superoxide dismutase 1)、SEMA5A(semaphorin 5A)、REV1(REV1 DNA directed polymerase)、SGCD(sarcoglycan delta)、ADAMTS12(ADAM metallopeptidase with thrombospondin type 1 motif 12)、ATP6V0A1(ATPase H+ transporting v0 subunit a1)、EPHA2(EPH receptor A2)、HIPK2(homeodomain interacting protein kinase 2)等基因区域的SV在藏猪和低海拔猪之间表现出显著的群体差异,提示这些SV可能是影响藏猪对高海拔的适应性的关键功能变异。
3.3 利用三代测序技术在羊基因组结构变异鉴定在羊上,Bickhart等[82]对多品种山羊进行SMRT测序,结合SNP基因型数据以及Hi-C数据等,提出了一个近完整的家山羊(C.hircus)参考基因组,解决了超过1 kb的重复结构。2021年,姜雨团队的Li等[83-85]成功组装出湖羊参考基因组;接着构建了绵羊泛基因组,鉴定出HOXB13 SV导致绵羊的长尾性状。Li等[86]对萨能奶山羊进行第三代测序,一共确定了总长度11.7 Mb的16 714个SVs,并且,其中6 190个SVs与编码区和非编码区的功能基因有关。Kent等[87]对挪威Spael羊进行三代测序,发现可能在绵羊黄色脂肪产生过程发挥着重要作用的功能SV。
4 家畜全基因组CNV研究存在的问题与展望CNV在牛、羊和猪上已取得较好的研究进展,随着越来越多的CNV被研究者发现,极大的丰富了DNA变异的多样性,并发掘了大量可影响表型的候选功能CNV。但是,也存在着相对的问题,例如:①大多集中在CNV的鉴定及与表型的关联分析方面,对CNV如何影响到表型的功能机制的研究还有所欠缺。②CNV的检测与分析方法还有待完善,由于技术和分析方法等方面的限制,导致之前许多鉴定的CNV结果不准确且难以重复和验证。基于三代测序技术,并改进和利用相关分析方法,将有望解决CNV检测准确性等问题,但目前三代测序技术的成本仍较高,使其难以广泛应用。③CNV在动物育种的应用还十分有限,当前全基因组选择育种主要是依靠SNP芯片技术来实现。
CNV相比于传统的分子遗传标记具有更鲜明的特点,在未来家畜育种中具有作为重要分子标记的潜力。随着测序技术的发展、分析方法的改善、以及高密度SNP芯片的开发,可将CNV与SNP进行联合利用,以此来提高育种过程中的可靠性和选种效率。并且,我们相信,伴随着检测CNV技术的发展,也将推动基于CNV的全基因组选择在家畜育种上的应用,加速家畜的育种进程。
| [1] |
MACDONALD J R, ZIMAN R, YUEN R K C, et al. The Database of Genomic Variants: a curated collection of structural variation in the human genome[J]. Nucleic Acids Res, 2014, 42(D1): D986-D992. DOI:10.1093/nar/gkt958 |
| [2] |
PIROOZNIA M, GOES F S, ZANDI P P. Whole-genome CNV analysis: advances in computational approaches[J]. Front Genet, 2015, 6: 138. |
| [3] |
HOU Y L, LIU G E, BICKHART D M, et al. Genomic regions showing copy number variations associate with resistance or susceptibility to gastrointestinal nematodes in Angus cattle[J]. Funct Integr Genomics, 2012, 12(1): 81-92. DOI:10.1007/s10142-011-0252-1 |
| [4] |
SHEN W, SZANKASI P, DURTSCHI J, et al. Genome-wide copy number variation detection using NGS: data analysis and interpretation[J]. Methods Mol Biol, 2019, 1908: 113-124. |
| [5] |
裴生伟, 王丽, 曹学涛, 等. 牛全基因组拷贝数变异研究进展[J]. 畜牧兽医学报, 2018, 49(5): 871-878. PEI S W, WANG L, CAO X T, et al. Research progress on genomic copy number variations in cattle[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(5): 871-878. DOI:10.11843/j.issn.0366-6964.2018.05.001 (in Chinese) |
| [6] |
PINKEL D, SEGRAVES R, SUDAR D, et al. High resolution analysis of DNA copy number variation using comparative genomic hybridization to microarrays[J]. Nat Genet, 1998, 20(2): 207-211. DOI:10.1038/2524 |
| [7] |
REDON R, ISHIKAWA S, FITCH K R, et al. Global variation in copy number in the human genome[J]. Nature, 2006, 444(7118): 444-454. DOI:10.1038/nature05329 |
| [8] |
CARTER N P. Methods and strategies for analyzing copy number variation using DNA microarrays[J]. Nat Genet, 2007, 39(S7): S16-S21. DOI:10.1038/ng2028 |
| [9] |
ZHU M F, NEED A C, HAN Y J, et al. Using ERDS to infer copy-number variants in high-coverage genomes[J]. Am J Hum Genet, 2012, 91(3): 408-421. DOI:10.1016/j.ajhg.2012.07.004 |
| [10] |
YANG H, ZHU D M. Combinatorial detection algorithm for copy number variations using high-throughput sequencing reads[J]. Int J Patt Recogn Artif Intell, 2019, 33(14): 1950022. DOI:10.1142/S0218001419500228 |
| [11] |
CHEN Y, ZHAO L, WANG Y, et al. SeqCNV: a novel method for identification of copy number variations in targeted next-generation sequencing data[J]. BMC Bioinformatics, 2017, 18(1): 147. DOI:10.1186/s12859-017-1566-3 |
| [12] |
TREFFER R, DECKERT V. Recent advances in single-molecule sequencing[J]. Curr Opin Biotechnol, 2010, 21(1): 4-11. DOI:10.1016/j.copbio.2010.02.009 |
| [13] |
JAIN M, OLSEN H E, PATEN B, et al. The oxford nanopore MinION: delivery of nanopore sequencing to the genomics community[J]. Genome Biol, 2016, 17(1): 239. DOI:10.1186/s13059-016-1103-0 |
| [14] |
NORRIS A L, WORKMAN R E, FAN Y F, et al. Nanopore sequencing detects structural variants in cancer[J]. Cancer Biol Ther, 2016, 17(3): 246-253. DOI:10.1080/15384047.2016.1139236 |
| [15] |
LIAO Y C, LIN S H, LIN H H. Completing bacterial genome assemblies: strategy and performance comparisons[J]. Sci Rep, 2015, 5(1): 8747. DOI:10.1038/srep08747 |
| [16] |
LIU G E, HOU Y L, ZHU B, et al. Analysis of copy number variations among diverse cattle breeds[J]. Genome Res, 2010, 20(5): 693-703. DOI:10.1101/gr.105403.110 |
| [17] |
ZHANG L Z, JIA S G, YANG M J, et al. Detection of copy number variations and their effects in Chinese bulls[J]. BMC genomics, 2014, 15(1): 480. DOI:10.1186/1471-2164-15-480 |
| [18] |
LIU M, FANG L Z, LIU S L, et al. Array CGH-based detection of CNV regions and their potential association with reproduction and other economic traits in Holsteins[J]. BMC Genomics, 2019, 20(1): 181. DOI:10.1186/s12864-019-5552-1 |
| [19] |
BAE J S, CHEONG H S, KIM L H, et al. Identification of copy number variations and common deletion polymorphisms in cattle[J]. BMC Genomics, 2010, 11(1): 232. DOI:10.1186/1471-2164-11-232 |
| [20] |
KUMAR H, PANIGRAHI M, SARAVANAN K A, et al. Genome-wide detection of copy number variations in Tharparkar cattle[J]. Anim Biotechnol, 2023, 34(2): 448-455. DOI:10.1080/10495398.2021.1942027 |
| [21] |
ZHOU Y, CONNOR E E, WIGGANS G R, et al. Genome-wide copy number variant analysis reveals variants associated with 10 diverse production traits in Holstein cattle[J]. BMC Genomics, 2018, 19(1): 314. DOI:10.1186/s12864-018-4699-5 |
| [22] |
AHMAD S F, SINGH A, PANDA S, et al. Genome-wide elucidation of CNV regions and their association with production and reproduction traits in composite Vrindavani cattle[J]. Gene, 2022, 830: 146510. DOI:10.1016/j.gene.2022.146510 |
| [23] |
GAO Y H, JIANG J P, YANG S H, et al. CNV discovery for milk composition traits in dairy cattle using whole genome resequencing[J]. BMC Genomics, 2017, 18(1): 265. DOI:10.1186/s12864-017-3636-3 |
| [24] |
XU Y, JIANG Y, SHI T, et al. Whole-genome sequencing reveals mutational landscape underlying phenotypic differences between two widespread Chinese cattle breeds[J]. PLoS One, 2017, 12(8): e0183921. DOI:10.1371/journal.pone.0183921 |
| [25] |
LIU M, LI B, HUANG Y Z, et al. Copy number variation of bovine MAPK10 modulates the transcriptional activity and affects growth traits[J]. Livest Sci, 2016, 194: 44-50. DOI:10.1016/j.livsci.2016.09.014 |
| [26] |
LIU M, LI B, SHI T, et al. Copy number variation of bovine SHH gene is associated with body conformation traits in Chinese beef cattle[J]. J Appl Genet, 2019, 60(2): 199-207. DOI:10.1007/s13353-019-00496-w |
| [27] |
LIU S L, KANG X L, CATACCHIO C R, et al. Computational detection and experimental validation of segmental duplications and associated copy number variations in water buffalo (Bubalus bubalis)[J]. Funct Integr Genomics, 2019, 19(3): 409-419. DOI:10.1007/s10142-019-00657-4 |
| [28] |
SINGH V K, SINGH S, NANDHINI P B, et al. Comparative genomic diversity analysis of copy number variations (CNV) in indicine and taurine cattle thriving in Europe and Indian subcontinent[J]. Anim Biotechnol, 2023, 1-12. |
| [29] |
FONTANESI L, MARTELLI P L, BERETTI F, et al. An initial comparative map of copy number variations in the goat (Capra hircus) genome[J]. BMC Genomics, 2010, 11(1): 639. DOI:10.1186/1471-2164-11-639 |
| [30] |
JENKINS G M, GODDARD M E, BLACK M A, et al. Copy number variants in the sheep genome detected using multiple approaches[J]. BMC Genomics, 2016, 17(1): 441. DOI:10.1186/s12864-016-2754-7 |
| [31] |
SALEHIAN-DEHKORDI H, XU Y X, XU S S, et al. Genome-wide detection of copy number variations and their association with distinct phenotypes in the world′s sheep[J]. Front Genet, 2021, 12: 670582. DOI:10.3389/fgene.2021.670582 |
| [32] |
YANG L, XU L Y, ZHOU Y, et al. Diversity of copy number variation in a worldwide population of sheep[J]. Genomics, 2018, 110(3): 143-148. DOI:10.1016/j.ygeno.2017.09.005 |
| [33] |
KANG X L, LI M X, LIU M, et al. Copy number variation analysis reveals variants associated with milk production traits in dairy goats[J]. Genomics, 2020, 112(6): 4934-4937. DOI:10.1016/j.ygeno.2020.09.007 |
| [34] |
MORADI M H, MAHMODI R, FARAHANI A H K, et al. Genome-wide evaluation of copy gain and loss variations in three Afghan sheep breeds[J]. Sci Rep, 2022, 12(1): 14286. DOI:10.1038/s41598-022-18571-4 |
| [35] |
LIU M, ZHOU Y, ROSEN B D, et al. Diversity of copy number variation in the worldwide goat population[J]. Heredity (Edinb), 2019, 122(5): 636-646. DOI:10.1038/s41437-018-0150-6 |
| [36] |
LIU M, CHENG J, CHEN Y H, et al. Distribution of DGAT1 copy number variation in Chinese goats and its associations with milk production traits[J]. Anim Biotechnol, 2023, 34(4): 980-985. DOI:10.1080/10495398.2021.2007118 |
| [37] |
LIU M, WOODWARD-GREENE J, KANG X L, et al. Genome-wide CNV analysis revealed variants associated with growth traits in African indigenous goats[J]. Genomics, 2020, 112(2): 1477-1480. DOI:10.1016/j.ygeno.2019.08.018 |
| [38] |
NANDOLO W, MÉSZÁROS G, WURZINGER M, et al. Detection of copy number variants in African goats using whole genome sequence data[J]. BMC Genomics, 2021, 22(1): 398. DOI:10.1186/s12864-021-07703-1 |
| [39] |
ZHANG R Q, WANG J J, ZHANG T, et al. Copy-number variation in goat genome sequence: A comparative analysis of the different litter size trait groups[J]. Gene, 2019, 696: 40-46. DOI:10.1016/j.gene.2019.02.027 |
| [40] |
YUAN C, LU Z K, GUO T T, et al. A global analysis of CNVs in Chinese indigenous fine-wool sheep populations using whole-genome resequencing[J]. BMC Genomics, 2021, 22(1): 78. DOI:10.1186/s12864-021-07387-7 |
| [41] |
DONG Y, ZHANG X L, XIE M, et al. Reference genome of wild goat (capra aegagrus) and sequencing of goat breeds provide insight into genic basis of goat domestication[J]. BMC Genomics, 2015, 16(1): 431. DOI:10.1186/s12864-015-1606-1 |
| [42] |
NORRIS B J, WHAN V A. A gene duplication affecting expression of the ovine ASIP gene is responsible for white and black sheep[J]. Genome Res, 2008, 18(8): 1282-1293. DOI:10.1101/gr.072090.107 |
| [43] |
CHEBII V J, MPOLYA E A, OYOLA S O, et al. Genome scan for variable genes involved in environmental adaptations of nubian ibex[J]. J Mol Evol, 2021, 89(7): 448-457. DOI:10.1007/s00239-021-10015-3 |
| [44] |
HU L Y, ZHANG L Z, LI Q, et al. Genome-wide analysis of CNVs in three populations of Tibetan sheep using whole-genome resequencing[J]. Front Genet, 2022, 13: 971464. DOI:10.3389/fgene.2022.971464 |
| [45] |
FADISTA J, NYGAARD M, HOLM L E, et al. A snapshot of CNVs in the pig genome[J]. PLoS One, 2008, 3(12): e3916. DOI:10.1371/journal.pone.0003916 |
| [46] |
WANG J Y, JIANG J C, WANG H F, et al. Improved detection and characterization of copy number variations among diverse pig breeds by array CGH[J]. G3(Bethesda), 2015, 5(6): 1253-1261. |
| [47] |
刘晓琴, 蒋隽, 何俊, 等. Wnt信号转导调控猪肌内脂肪含量研究进展[J]. 国外畜牧学: 猪与禽, 2012, 32(1): 78-80. LIU X Q, JIANG J, HE J, et al. Research progress of wnt wignal transduction regulating ntramuscular fat content in pigs[J]. Animal Science Abroad: Pigs and Poultry, 2012, 32(1): 78-80. (in Chinese) |
| [48] |
邱恒清, 肖石军, 郭源梅. 利用猪1.4 M高密度SNP芯片检测巴马香猪全基因组拷贝数变异[J]. 畜牧兽医学报, 2020, 51(9): 2079-2088. QIU H Q, XIAO S J, GUO Y M. Detection of genome-wide copy number variation using porcine 1.4 M high-density SNP chips in Bama xiang pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(9): 2079-2088. DOI:10.11843/j.issn.0366-6964.2020.09.005 (in Chinese) |
| [49] |
CHEN C Y, QIAO R M, WEI R X, et al. A comprehensive survey of copy number variation in 18 diverse pig populations and identification of candidate copy number variable genes associated with complex traits[J]. BMC Genomics, 2012, 13(1): 733. DOI:10.1186/1471-2164-13-733 |
| [50] |
WANG Y, ZHANG T R, WANG C D. Detection and analysis of genome-wide copy number variation in the pig genome using an 80 K SNP Beadchip[J]. J Anim Breed Genet, 2020, 137(2): 166-176. DOI:10.1111/jbg.12435 |
| [51] |
XU C L, ZHANG W, JIANG Y, et al. Genome-wide detection and analysis of copy number variation in anhui indigenous and western commercial pig breeds using porcine 80K SNP BeadChip[J]. Genes (Basel), 2023, 14(3): 654. DOI:10.3390/genes14030654 |
| [52] |
JIANG J C, WANG J Y, WANG H F, et al. Global copy number analyses by next generation sequencing provide insight into pig genome variation[J]. BMC Genomics, 2014, 15(1): 593. DOI:10.1186/1471-2164-15-593 |
| [53] |
ZHENG X R, ZHAO P, JYANG K J, et al. CNV analysis of Meishan pig by next-generation sequencing and effects of AHR gene CNV on pig reproductive traits[J]. J Anim Sci Biotechnol, 2020, 11: 42. DOI:10.1186/s40104-020-00442-5 |
| [54] |
RAN X Q, PAN H, HUANG S H, et al. Copy number variations of MTHFSD gene across pig breeds and its association with litter size traits in Chinese indigenous Xiang pig[J]. J Anim Physiol Anim Nutr (Berl), 2018, 102(5): 1320-1327. DOI:10.1111/jpn.12922 |
| [55] |
QIU Y B, DING R R, ZHUANG Z W, et al. Genome-wide detection of CNV regions and their potential association with growth and fatness traits in Duroc pigs[J]. BMC Genomics, 2021, 22(1): 332. DOI:10.1186/s12864-021-07654-7 |
| [56] |
DING R R, ZHUANG Z W, QIU Y B, et al. A composite strategy of genome-wide association study and copy number variation analysis for carcass traits in a Duroc pig population[J]. BMC Genomics, 2022, 23(1): 590. DOI:10.1186/s12864-022-08804-1 |
| [57] |
WEI X, SHU Z, WANG L G, et al. Copy number variations contribute to intramuscular fat content differences by affecting the expression of PELP1 alternative splices in Pigs[J]. Animals(Basel), 2022, 12(11): 1382. |
| [58] |
FAN S H, KONG C C, CHEN Y G, et al. Copy number variation analysis revealed the evolutionary difference between Chinese indigenous pigs and asian wild boars[J]. Genes (Basel), 2023, 14(2): 472. DOI:10.3390/genes14020472 |
| [59] |
ZHANG W, ZHOU M, LIU L Q, et al. Population structure and selection signatures underlying domestication inferred from genome-wide copy number variations in Chinese indigenous pigs[J]. Genes (Basel), 2022, 13(11): 2026. DOI:10.3390/genes13112026 |
| [60] |
LONG Y, SU Y, AI H S, et al. A genome-wide association study of copy number variations with umbilical hernia in swine[J]. Anim Genet, 2016, 47(3): 298-305. DOI:10.1111/age.12402 |
| [61] |
王伟. 不同品种马基因组拷贝数变异研究[D]. 呼和浩特: 内蒙古农业大学, 2014. WANG W. Genome detection of copy number variations among diverse horse breeds by array CGH[D]. Hohhot: Inner Mongolia Agricultural University, 2014. (in Chinese) |
| [62] |
WANG M, LIU Y, BI X K, et al. Genome-wide detection of copy number variants in chinese indigenous horse breeds and verification of CNV-overlapped genes related to heat adaptation of the Jinjiang Horse[J]. Genes (Basel), 2022, 13(4): 603. DOI:10.3390/genes13040603 |
| [63] |
DURWARD-AKHURST S A, SCHAEFER R J, GRANTHAM B, et al. Genetic variation and the distribution of variant types in the horse[J]. Front Genet, 2021, 12: 758366. DOI:10.3389/fgene.2021.758366 |
| [64] |
AL ABRI M A, HOLL H M, KALLA S E, et al. Whole genome detection of sequence and structural polymorphism in six diverse horses[J]. PLoS One, 2020, 15(4): e0230899. DOI:10.1371/journal.pone.0230899 |
| [65] |
GU J J, LI S, ZHU B, et al. Genetic variation and domestication of horses revealed by 10 chromosome-level genomes and whole-genome resequencing[J]. Mol Ecol Resour, 2023, 23(7): 1656-1672. DOI:10.1111/1755-0998.13818 |
| [66] |
TANG X W, ZHU B, REN R M, et al. Genome-wide copy number variation detection in a large cohort of diverse horse breeds by whole-genome sequencing[J]. Front Vet Sci, 2023, 10: 1296213. DOI:10.3389/fvets.2023.1296213 |
| [67] |
GAO X, WANG S, WANG Y F, et al. Long read genome assemblies complemented by single cell RNA-sequencing reveal genetic and cellular mechanisms underlying the adaptive evolution of yak[J]. Nat Commun, 2022, 13(1): 4887. DOI:10.1038/s41467-022-32164-9 |
| [68] |
罗西尔. 沼泽型水牛和河流型水牛染色体水平全基因组组装及比较研究[D]. 南宁: 广西大学, 2020. LUO X E. Whole genome assembly and comparative genomics analysis in swamp buffalo and river buffal[D]. Nanning: Guangxi University, 2020. (in Chinese) |
| [69] |
GAO Y H, MA L, LIU G E. Initial analysis of structural variation detections in cattle using long-read sequencing methods[J]. Genes (Basel), 2022, 13(5): 828. DOI:10.3390/genes13050828 |
| [70] |
LAMB H J, ROSS E M, NGUYEN L T, et al. Characterization of the poll allele in Brahman cattle using long-read Oxford Nanopore sequencing[J]. J Anim Sci, 2020, 98(5): skaa127. DOI:10.1093/jas/skaa127 |
| [71] |
LOW W Y, TEARLE R, LIU R J, et al. Haplotype-resolved genomes provide insights into structural variation and gene content in Angus and Brahman cattle[J]. Nat Commun, 2020, 11(1): 2071. DOI:10.1038/s41467-020-15848-y |
| [72] |
LEONARD A S, CRYSNANTO D, FANG Z H, et al. Structural variant-based pangenome construction has low sensitivity to variability of haplotype-resolved bovine assemblies[J]. Nat Commun, 2022, 13(1): 3012. DOI:10.1038/s41467-022-30680-2 |
| [73] |
ZHOU Y, YANG L, HAN X T, et al. Assembly of a pangenome for global cattle reveals missing sequences and novel structural variations, providing new insights into their diversity and evolutionary history[J]. Genome Res, 2022, 32(8): 1585-1601. DOI:10.1101/gr.276550.122 |
| [74] |
DU H, DIAO C G, ZHAO P J, et al. Integrated hybrid de novo assembly technologies to obtain high-quality pig genome using short and long reads[J]. Brief Bioinform, 2021, 22(5): bbaa399. DOI:10.1093/bib/bbaa399 |
| [75] |
FANG X D, MOU Y L, HUANG Z Y, et al. The sequence and analysis of a Chinese pig genome[J]. Gigascience, 2012, 1(1): 16. DOI:10.1186/2047-217X-1-16 |
| [76] |
LI M Z, TIAN S L, JIN L, et al. Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars[J]. Nat Genet, 2013, 45(12): 1431-1438. DOI:10.1038/ng.2811 |
| [77] |
ZHANG L, HUANG Y M, SI J L, et al. Comprehensive inbred variation discovery in Bama pigs using de novo assemblies[J]. Gene, 2018, 679: 81-89. DOI:10.1016/j.gene.2018.08.051 |
| [78] |
YANG Y L, LIAN J M, XIE B K, et al. Chromosome-scale de novo assembly and phasing of a Chinese indigenous pig genome[Z]. BioRxiv, 2019, doi: 10.1101/770958.
|
| [79] |
ZHOU R, LI S T, YAO W Y, et al. The Meishan pig genome reveals structural variation-mediated gene expression and phenotypic divergence underlying Asian pig domestication[J]. Mol Ecol Resour, 2021, 21(6): 2077-2092. DOI:10.1111/1755-0998.13396 |
| [80] |
MA H M, JIANG J, HE J, et al. Long-read assembly of the Chinese indigenous Ningxiang pig genome and identification of genetic variations in fat metabolism among different breeds[J]. Mol Ecol Resour, 2022, 22(4): 1508-1520. DOI:10.1111/1755-0998.13550 |
| [81] |
JIANG Y F, WANG S, WANG C L, et al. Pangenome obtained by long-read sequencing of 11 genomes reveal hidden functional structural variants in pigs[J]. Iscience, 2023, 26(3): 106119. DOI:10.1016/j.isci.2023.106119 |
| [82] |
BICKHART D M, ROSEN B D, KOREN S, et al. Single-molecule sequencing and chromatin conformation capture enable de novo reference assembly of the domestic goat genome[J]. Nat Genet, 2017, 49(4): 643-650. DOI:10.1038/ng.3802 |
| [83] |
LI R, YANG P, LI M, et al. A Hu sheep genome with the first ovine Y chromosome reveal introgression history after sheep domestication[J]. Sci China Life Sci, 2021, 64(7): 1116-1130. DOI:10.1007/s11427-020-1807-0 |
| [84] |
LI R, GONG M, ZHANG X M, et al. A sheep pangenome reveals the spectrum of structural variations and their effects on tail phenotypes[J]. Genome Res, 2023, 33(3): 463-477. DOI:10.1101/gr.277372.122 |
| [85] |
LI R, GONG M, ZHANG X M, et al. The first sheep graph-based pan-genome reveals the spectrum of structural variations and their effects on tail phenotypes[J]. BioRxiv, 2021. DOI:10.1101/2021.12.22.472709 |
| [86] |
LI R, YANG P, DAI X L, et al. A near complete genome for goat genetic and genomic research[J]. Genet Sel Evol, 2021, 53(1): 74. DOI:10.1186/s12711-021-00668-5 |
| [87] |
KENT M, MOSER M, BOMAN I A, et al. Insertion of an endogenous Jaagsiekte sheep retrovirus element into the BCO2-gene abolishes its function and leads to yellow discoloration of adipose tissue in Norwegian Spælsau (Ovis aries)[J]. BMC Genomics, 2021, 22(1): 492. DOI:10.1186/s12864-021-07826-5 |
(编辑 郭云雁)



