2. 新疆维吾尔自治区昌吉回族自治州农产品质量安全中心, 昌吉 831100;
3. 新疆畜牧科学院兽医研究所, 乌鲁木齐 830013
2. Agricultural Products Quality and Safety Center, Changji 831100, China;
3. Institute of Veterinary Medicine, Xingjiang Academy of Animal Science, Urumqi 830013, China
牛传染性鼻气管炎病毒(infectious bovine rhinotracheitis virus,IBRV),即牛疱疹病毒Ⅰ型(bovine herpes virus type 1,BHV-1),是一种可造成牛发生严重呼吸障碍的病毒[1-2]。IBRV具有高度传染性,会引起牛严重的呼吸道感染[3]。牛感染后一般都会出现体温升高,精神、食欲不佳,呼吸不畅,鼻腔流出大量黏液,鼻黏膜伴有充血溃烂,鼻镜部分出现红肿,哺乳母牛还会出现产奶量减少等症状[4]。上呼吸道感染时常伴有结膜炎,病毒通过鼻泪管进入眼结膜使眼结膜出现炎症,当出现继发性细菌感染时眼角流出脓性液体,出现角膜炎[5]。IBRV感染有时还会造成患畜出现生殖道症状,包括母畜出现脓疱性外阴阴道炎和流产,公畜出现阴茎粘膜和包皮的炎症等。病毒大量存在于患畜的生殖器官中,一般在配种过程中传播。在病程初期表现为生殖器官肿胀充血有脓疮,频繁排尿并伴有疼痛感,轻度感染时数日就能痊愈,但感染严重时生殖器官会发生病变[6]。此外有时还会出现脑膜脑炎症状,主要出现在犊牛阶段,表现为脑膜充血出现出血点或血栓。体温升高到40 ℃以上,并伴有明显的神经症状,如有时精神萎靡有时亢奋,共济失调,不能正常行走等,一般此类型病程短死亡率高[7]。同时IBRV感染会使患畜出现免疫抑制的现象,从而出现继发感染,使患畜康复变得更加复杂[8-9]。隐性感染的患畜在受到外界刺激(如外伤、暴晒)时会再次激活体内病毒并向环境中排毒,从而造成BHV-1持续感染难以清除的特点[10-11]。
微生物群多样性和组成在控制传染病方面发挥着重要作用[12-13]。与肠道微生物群相比,鼻腔微生物群对呼吸道病原体感染的作用评价研究较少[14]。有研究表明,呼吸道合胞体病毒感染和婴儿鼻腔微生物菌群失调密切相关,感染该病毒的婴儿和未感染婴儿之间在微生物多样性和特定细菌类群丰度方面有差异,并显示这些差异在病前、病中和病后以一些不同的模式纵向表现[15]。据报道,由病毒、细菌病原体或环境应激因素单独或共同作用引起的猪呼吸道疾病综合征,相较于健康猪会显示出明显的菌属丰度变化[16]。由于呼吸道微生物群是高度可变的[17],在疾病发生时出现的改变是疾病发生的原因还是结果尚不能明确。因此,本研究通过对犊牛鼻腔菌群组成与差异特点进行研究,探索犊牛鼻腔菌群结构和功能变化与IBRV感染的相关性。以期为深入探究犊牛呼吸道病毒病的发病机制提供研究基础和理论依据。
1 材料与方法 1.1 犊牛呼吸道疾病流行病学调查于2021年,在中国新疆南疆地区4个规模化安格斯肉牛繁育场,以1月龄犊牛作为调查对象,进行为期一年的犊牛呼吸道疾病流行病学调查。所调查的犊牛呼吸道疾病相关临床症状包括发热(肛温39.5 ℃以上)、咳嗽(干咳、湿咳,活动时加剧)和呼吸困难(头颈伸直、气喘、腹式呼吸明显,伴随出现吞咽动作)。
1.2 样品采集样品分别在2020年12月至2021年11月采集于新疆南疆地区的4个安格斯肉牛繁育场,来源为出生1月龄犊牛,根据临床症状,随机抽取出现发热、咳嗽和呼吸困难等呼吸道症状犊牛和无呼吸道症状犊牛,通过佩戴无菌手套将无菌棉拭子深入犊牛鼻腔中,充分蘸取分泌物,将其密封于冻存管中置于液氮中保存,备用。
1.2.1 IBRV的PCR检测 根据GenBank收录的IBRV的UL27基因序列(登录号:MK654 723.1),利用DNAstar、DNAman及IEDB、Bepipred等在线软件分析其氨基酸序列。选择抗原性较强部位的氨基酸残基作为目的区域(第273-393位氨基酸)。利用Primer 5.0设计引物,由上海生物工程有限公司合成。引物序列为F: 5′CTCTACCGCACGGGCACCT3′,R: 5′GCGGCTCTCGTCTCGCA3′;目的基因(360 bp)扩增使用15 μL总反应体系,反应条件为95 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 10 min。
1.2.2 样本分组 依据PCR检测结果,在出现呼吸道临床症状犊牛样本中随机选取10个单纯IBRV阳性的犊牛样本定为P组,在无呼吸道临床症状犊牛样本中随机选取10个IBRV阴性且无其他呼吸道病毒感染的健康犊牛样本定为N组。
1.3 样品细菌16S rRNA测序使用Omega病毒基因组DNA快速提取试剂盒提取基因组DNA[18],基因组抽提成功后使用分光光度计测定DNA浓度和纯度,琼脂凝胶电泳试验检测提取DNA的质量是否标准。之后进行目标片段PCR扩增。对扩增产物使用1.2%琼脂糖凝胶电泳试验进行检测,之后回收目的片段,使用Axygen公司的凝胶回收试剂盒。根据电泳初步定量结果,对回收的PCR扩增产物进行荧光定量。选择V3和V4可变区使用Illumina平台对群落DNA片段进行双端(Paired-end)测序。
测序完成后,使用DADA2方法,进行去引物,质量过滤,去噪(denoise),拼接和去嵌合体步骤[19]。以上步骤针对每个文库分别进行分析。使用R语言脚本,对全部样本中所包含的高质量序列的长度分布进行统计。使用DADA2质控后产生的每个去重的序列称为ASVs(amplicon sequence variants)。将所得的ASV序列与Greengenes数据库进行对比[20],对ASV表中的每一个ASV进行分等级(门、纲、目、科、属)的物种注释。区分样本后用软件平台Usearch进行ASV聚类分析和物种分类学分析,基于ASV用分析软件Mothur进行α多样性和β多样性分析,基于分类学信息,在各个分类水平上进行群落结构的统计分析。使用R语言vegan包的adnois2函数进行adonis分析。
1.4 数据统计与分析数据采用“x±sx”表示。采用统计分析软件GraphPad Prism 9.0.0比较各组间差异菌门和菌属(T检验),P < 0.05表示差异显著,P < 0.01表示差异极显著。使用PICRUSt2方法对16 S rRNA基因序列在MetaCyc和KEGG数据库中进行代谢通路预测分析[21]。
2 结果 2.1 犊牛呼吸道疾病的流行病学调查与病原IBRV检测新疆南疆地区部分安格斯肉牛繁育场犊牛呼吸道疾病调查情况如下:2020年12—2021年11月,共计产犊11 215头,具有发热、咳嗽、呼吸困难等临床症状犊牛数共计922头,占产犊总数的8.2%(922/11 215);其中死亡98头,病死率为10.6%(98/922)。
随机抽取197头具有发热、咳嗽、呼吸困难等临床症状犊牛和30头无临床症状犊牛,采集鼻拭子进行IBRV的PCR检测。结果显示:具有临床症状犊牛中IBRV检出率为23.35%(46/197),无症状犊牛检出率为13.33%(4/30)。
在此基础上,随机选取10个单纯IBRV阳性的犊牛样本(P组)和10个IBRV阴性且无其他呼吸道病毒感染的健康犊牛样本(N组),对分组样本进行PCR检测。
2.2 鼻腔菌群物种组成分析2.2.1 各水平微生物分类单元ASV组成数 N组和P组犊牛鼻腔菌群各分类水平物种分类单元组成如图 1所示。与N组犊牛比较显示,P组犊牛鼻腔菌群在门、纲、目、科水平上的物种分类单元数呈极显著增加(P < 0.01),属水平物种分类单元数也呈增加趋势(P = 0.056)。种水平上无显著差异。
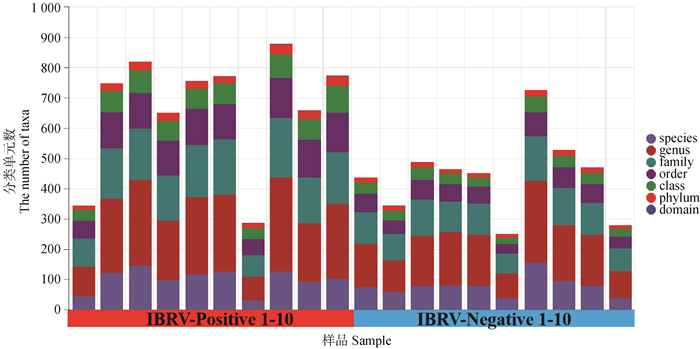
|
图 1 N组和P组犊牛鼻腔菌群物种分类学注释结果 Fig. 1 Taxonomic annotation of the nasal microbiota in group N and P |
2.2.2 鼻腔菌群物种组成差异分析 在N组和P组犊牛鼻腔菌群中相对丰度排名前20的菌门和前50的菌属如图 2A、B所示。LEfSe分析结果和物种组成差异如图 2C、D。进一步采用独立样本T检验显示,两组间共有13菌门存在显著差异(P<0.05),其中,与N组犊牛相比(图 2E),P组犊牛鼻腔菌群中的厚壁菌门(Firmicutes)相对丰度极显著降低(P<0.01),而NC10、浮霉菌门(Planctomycetes)、芽单胞菌门(Gemmatimonadetes)、蓝藻门(Cyanobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、绿菌门(Chlorobi)、WS3、硝化螺旋菌门(Nitrospirae)、疣微菌门(Verrucomicrobia)、TM7和软壁菌门(Tenericutes)相对丰度均显著升高(P<0.05或P < 0.01)。经独立样本T检验比较,发现:两组间属水平比较后共有15个差异显著菌属(P<0.05),与N组犊牛相比(图 2F和G),P组犊牛鼻腔菌群中阎氏菌属(Yaniella)、盐水球菌属(Salinicoccus)、动性球菌属(Planococcus)和短杆菌属(Brevibacterium)相对丰度显著降低(P<0.05),而红游动菌属(Rhodoplanes)和不动杆菌属(Acinetobacter)相对丰度极显著升高(P<0.01),此外德沃斯菌属(Devosia)、乳酸杆菌属(Lactobacillus)、嗜盐单胞菌属(Halomonas)、拟杆菌属(Bacteroidaceae_Bactericides)、普雷沃菌属(Prevotella)、Kaistobacter、芽胞杆菌属(Bacillaceae_Bacillus)、支原体(Mycoplasma)和根瘤菌属(Chelativorans)相对丰度均显著升高(P<0.05)。
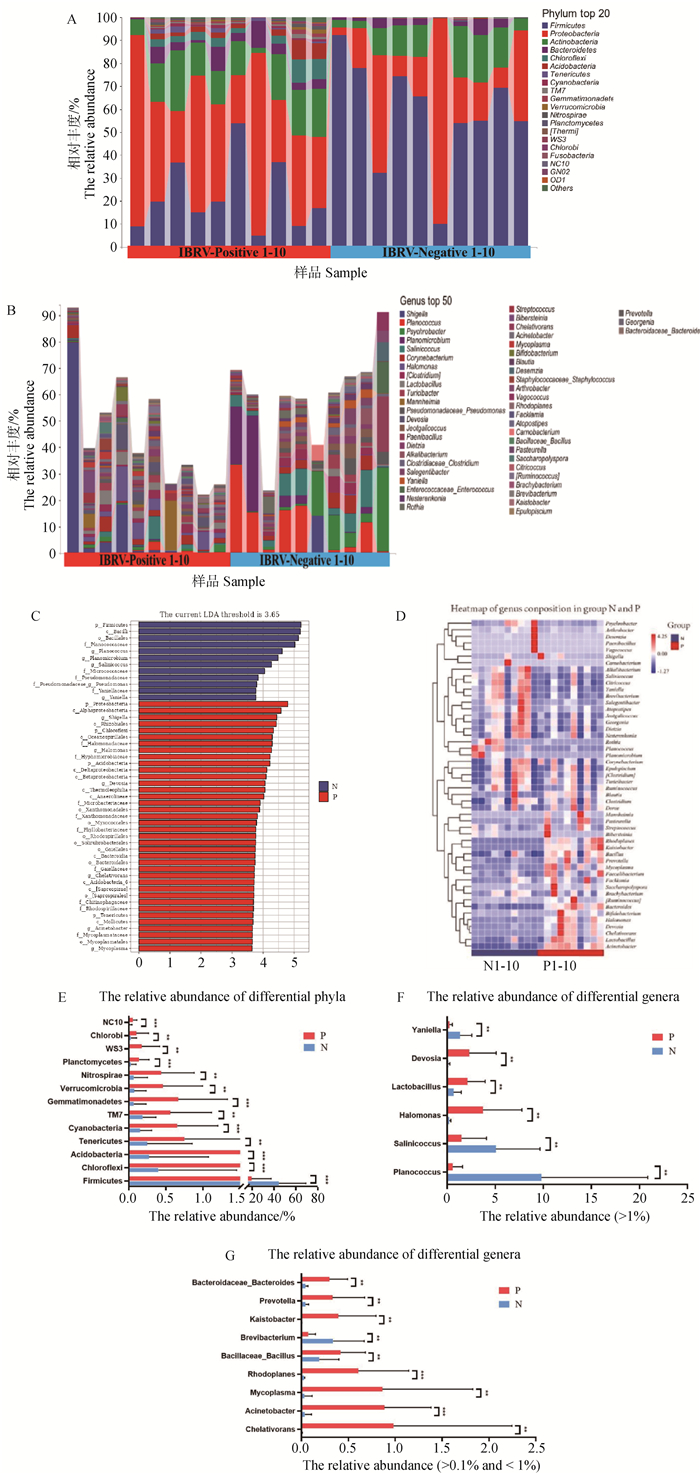
|
A、B为两组排名前20菌门和前50菌属;C为LEfSe图;D为属水平热图;E为两组差异菌门;F为两组相对丰度1%以上的差异菌属;G为两组差异菌属相对丰度平均值为:小于1%,但大于0.1%。**.P < 0.05, ***.P < 0.01 A and B are the top 20 phylum and top 50 genus in the two groups. C is the LEfSe plot. D is the genus level heat map. E is the difference phylum in the two groups. F is the difference genus with more than 1% relative abundance in the two groups. G represents the average abundance of two groups of different bacterial genera: less than 1%, but greater than 0.1%. **.P < 0.05, ***.P < 0.01 图 2 N组和P组犊牛菌门(前20)和菌属(前50)组成和差异 Fig. 2 The composition and differences of the relative abundance of phylum (top 20) and genus (top 50) in group N and P |
2.2.3 鼻腔菌群组成结构分析 对两组犊牛鼻腔菌群结构进行α多样性和β多样性分析(图 3A, B)。其中,两组犊牛在菌群丰富度(Chao1、Observed_species)和多样性(Shannon、Simpson)指数无显著性差异(P>0.05)。此外,P组犊牛基于进化的多样性(Faith_pd)指数与N组犊牛相比差异不显著(P>0.05)。然而,菌群均匀度(Pielou_e)和覆盖度(Goods_coverage)指数呈显著性差异(P<0.05)。两组犊牛鼻腔菌群β多样性PCoA分析显示,PCo1的贡献率为20%,PCo2的贡献率为12.7%。经降维处理后的NMDS分析结果如(图 3C)所示。进一步采用Unweighted_unifrac adonis分析显示P组和N组犊牛鼻腔菌群结构呈现极显著独立分布(P=0.001)(图 3B)。
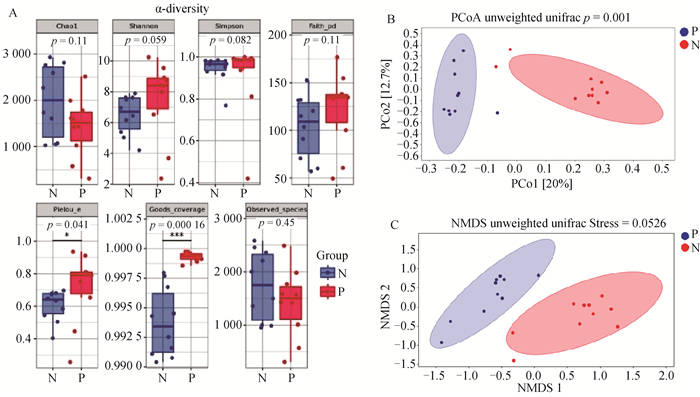
|
A.α-多样性指数;B.β-多样性指数;C.NMDS分析 A. α-diversity index; B. β-diversity index; C. NMDS analysis 图 3 N组和P组犊牛鼻腔菌群多样性指数 Fig. 3 The nasal microbiota diversities in groups N and P |
2.3.1 功能单元PCoA分析 N组和P组犊牛鼻腔菌群功能单元组成结构分布在MetaCyc和KEGG数据库中比较如图 4所示。在MetaCyc数据库中PCo1的贡献率为48.2%,PCo2的贡献率为21.72%。KEGG数据库中PCo1的贡献率为51.9%,PCo2的贡献率为19.7%。两组犊牛鼻腔菌群各自聚为一类。
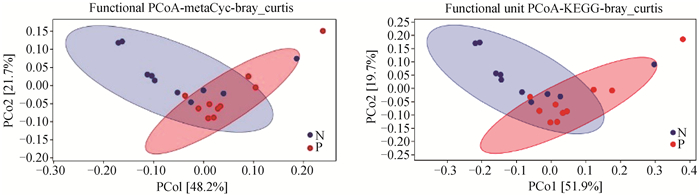
|
图 4 N组和P组犊牛鼻腔菌群结构PCoA分析 Fig. 4 PCoA analysis of nasal microbiota structure in groups N and P |
2.3.2 鼻腔菌群代谢通路统计 P组和N组犊牛鼻腔菌群在MetaCyc数据库中共富集到7类一级生物代谢功能,共计,主要涉及:生物合成(biosynthesis)、退化/利用/同化(degradation/utilization/assimilation)、解毒(detoxification)、前体代谢产物和能量产生(generation of precursor metabolite and energy)、聚糖途径(glycan pathways)、生物大分子修饰(macromolecule modification)和代谢簇(metabolic clusters)功能(图 5A)。在KEGG生物代谢功能中共富集到6类生物功能通路,主要涉及:细胞进程(cellular processes)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、代谢(metabolism)和生物体系统(organismal systems)(图 5B)。
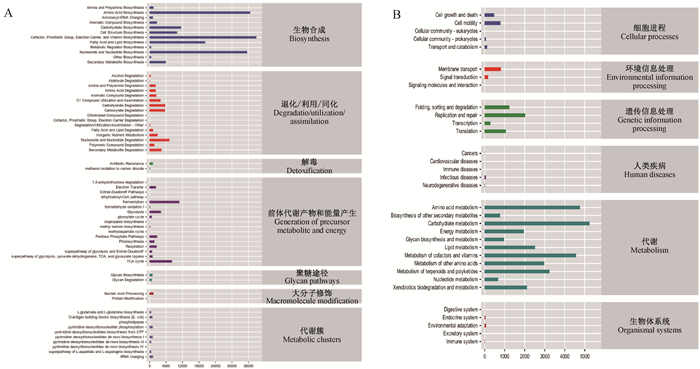
|
A. MetaCyc数据库;B. KEGG数据库 A. MetaCyc biometabolic function; B. KEGG biometabolic function 图 5 犊牛鼻腔菌群代谢通路统计 Fig. 5 Statistical analysis of metabolism pathway of calf nasal flora |
在MetaCyc一级功能层水平上,共检测出61条代谢通路。相对丰度较高的主要是生物合成(biosynthesis)功能,其中主要是氨基酸生物合成(amino acid biosynthesis)、辅因子、辅基、电子载体和维生素生物合成(cofactor,prosthetic group,electron carrier,and vitamin biosynthesis)、核苷与核苷酸生物合成(nucleoside and nucleotide biosynthesis)、脂肪酸与脂质生物合成(fatty acid and lipid biosynthesis)、碳水化合物生物合成(carbohydrate biosynthesis)、细胞结构生物合成(cell structure biosynthesis)。退化/利用/同化(degradation/utilization/assimilation)功能基因中,核苷和核苷酸降解(nucleoside and nucleotide degradation)相对丰度较高。前体代谢产物和能量的产生(generation of precursor metabolite and energy)功能基因中,发酵(fermentation)、三羧酸循环(TCA cycle)相对丰度较高。
在KEGG一级功能层水平上,共检测出33条代谢通路。相对丰度较高的生物功能通路为代谢(metabolism),其中主要是氨基酸代谢(amino acid metabolism)、碳水化合物代谢(carbohydrate metabolism)、能量代谢(energy metabolism)、脂质代谢(lipid metabolism)、辅助因子和维生素代谢(metabolism of cofactors and vitamins)、其他氨基酸的代谢(metabolism of other amino acids)、萜类和多酮类的代谢(metabolism of terpenoids and polyketides)和异种生物降解与代谢(xenobiotics biodegradation and metabolism)相对丰度较高。
2.3.3 鼻腔菌群代谢通路差异分析 如图 6所示,P组和N组犊牛鼻腔菌群在MetaCyc代谢通路丰度预测中存在9条差异通路,分别为甘露糖甘油酸生物合成I通路PWY-5656(mannosylglycerate biosynthesis I)、维生素B6降解通路PWY-5499(vitamin B6 degradation)、磷脂酶通路LIPASYN-PWY(phospholipases)、维生素E合成(生育酚)通路PWY-1422 [vitamin E biosynthesis(tocopherols)]、肌酐降解Ⅱ通路PWY-4722(creatinine degradation Ⅱ)、恩特纳-杜多罗夫途经Ⅲ(半磷酸化)通路PWY-2221 [(pathway Ⅲ (semi-phosphorylative)]、3-羟基丙酸循环超通路PWY-7024(superpathway of the 3-hydroxypropanoate cycle)、乙醛酸同化通路PWY-5744(glyoxylate assimilation)和3-羟基丙酸循环通路PWY-5743(3-hydroxypropanoate cycle),均为P组犊牛显著上调(P < 0.05)。
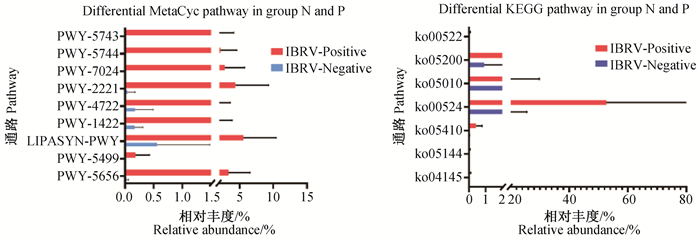
|
图 6 N组和P组犊牛鼻腔菌群差异代谢通路比较 Fig. 6 Comparison of different metabolic pathways of nasal microflora in N and P groups of calves |
P组和N组犊牛鼻腔菌群在KEGG代谢通路丰度预测中存在7条差异通路,分别为细胞过程(cellular processes)中的细胞生长与死亡(cell growth and death)和运输和分解(transport and catabolism)自噬信号ko04145(phagosome)功能、人类疾病(human diseaes)中的传染病(infectious diseases)疟原虫信号ko05144(malaria)功能、心血管疾病(cardiovascular diseases)肥厚性梗阻型心肌病ko05410[hypertrophic cardiomyopathy (HCM)]功能、神经退行性疾病(neurodegenerative diseases)阿尔茨海默病ko05010(Alzheimer′s disease)功能、癌症ko05200(pathways in cancer)功能、代谢(Metabolism)中的其他次级代谢产物的生物合成(biosynthesis of other secondary metabolites)、新霉素、卡那霉素和庆大霉素的生物合成ko00524(butirosin and neomycin biosynthesis)、功能和萜类化合物和聚酮化合物的代谢(metabolism of terpenoids and polyketones)、12、14和16元大环内酯的生物合成ko00522(biosynthesis of 12-, 14-and 16-membered macrolides)功能,均为P组犊牛显著上调(P < 0.05)。
3 讨论幼龄时期是牛生长过程的重要阶段,具有感染病原的潜在风险。有研究发现呼吸道病毒感染引起免疫变化会造成呼吸道局部环境改变,并影响局部菌群组成[22]。同时,若鼻腔菌群失调,会造成机体受到病毒的侵犯[23-24]。在慢性阻塞性肺病中,病毒可明显增加呼吸道细菌负荷量[25]。本研究结果显示,经IBRV感染的鼻腔菌群在物种丰度和多样性方面无显著变化。然而,鼻腔菌群的均匀度和覆盖度方面具有差异性。此外,β多样性分析表明IBRV感染能显著改变鼻腔菌群组成结构。因此,IBRV感染会造成犊牛鼻腔菌群结构更复杂造成菌群结构紊乱。LEfSe分析中发现志贺菌属(Shigella)在P组犊牛中被显著富集,这可能导致宿主发生肠道疾病[26],这与犊牛感染IBRV后出现腹泻等肠道疾病临床症状相符。Frank等[27]的研究证明,从鼻部中分离出潜在的病原微生物可能会成为局部和全身感染的病原体。Gracia-Rodriguez等[28]发现,患有慢性呼吸道疾病的宿主具有较高潜在病原体种类的鼻咽携带率。本研究中P组犊牛的厚壁菌门(Firmicutes)丰度低于N组犊牛,健康幼龄动物鼻腔中厚壁菌门(Firmicutes)为优势菌门,这与姜瑜琪等[29]的研究结果相似。然而,变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、蓝菌门(Cyanobacteria)、芽单胞菌门(Gemmatimonadetes)丰度高于N组犊牛。据报道,变形菌门(Proteobacteria)被认为是导致呼吸道疾病产生的主要诱因[30],进一步表明IBRV的感染与鼻腔菌群变化相关。
值得注意的是,P组犊牛在MetaCyc代谢通路丰度预测中的甘露糖甘油酸生物合成、维生素B6降解、磷脂酶、维生素E合成(生育酚)、肌酐降解、恩特纳-杜多罗夫途经Ⅲ(半磷酸化)、3-羟基丙酸循环和乙醛酸同化通路显著上调。其中,甘露糖甘油酸生物是细菌催化生成甘露糖甘油酸的重要物质,能增加微生物对高温的适应能力,从而加快微生物的代谢[31-32]。维生素B6的活性形式为吡哆醛5-磷酸(PLP),PLP在临床和基于人体的研究中与许多炎性标志物呈负相关[33-34],因此维生素B6降解提示机体出现炎症反应。维生素缺乏是由疾病或代谢紊乱引起的,但维生素E合成量的变化不具备一定的参考价值[35]。生育酚可以抑制乳腺癌细胞的生长并作为血清降胆固醇剂[36]。磷脂酶是参与合成溶血磷酸酯的主要物质,在维持机体正常生理功能和参与病理过程的发生发展方面均有重要作用[37]。肌酐降解是限制肌酐毒力的有力手段,降解不足会导致血液及肾脏乳脂醇的产生增加,典型例如尿毒症患者体内肌酐降解[38-39];但同时有研究证明,肌酐缺乏会造成线粒体能量代谢紊乱,会造成机体生长发育迟缓、脑损伤和偏瘫等[40-41]。恩特纳-杜多罗夫途经是大肠杆菌的主要糖酵解途经之一[42]。3-羟基丙酸是大肠杆菌、绿弯杆菌和肺炎克雷伯菌合成的关键物质,涵盖了菌株生产的各个方面[43]。L-苏氨酸是机体必需氨基酸之一,在L-苏氨酸生产过程中,乙醛酸能起到部分回补途经的功能,乙醛酸同化增加L-苏氨酸的积累有利于菌体的代谢[44]。在KEGG代谢通路丰度预测中,细胞生长与死亡、运输和分解、传染病、心血管疾病、神经退行性疾病、癌症和代谢功能均为P组犊牛显著上调。已有研究证明,吞噬作用是细胞吸收相对较大颗粒的过程,是组织重塑、炎症和抵御感染因子的核心机制。吞噬体和溶酶体的融合释放出有毒产物,可以杀死大多数细菌并将它们降解成碎片[45]。肥厚性梗阻型心肌病会导致肌丝Ca2+敏感性增加从而增加肌动球蛋白在Ca2+浓度变化时对ATP的利用,无法维持正常的ATP水平可能导致中枢异常[46]。阿尔茨海默症是由淀粉样蛋白(Abeta)造成细胞Ca2+失衡导致神经元凋亡、自噬缺陷、线粒体异常、神经传递缺陷、突触可塑性受损和神经变性[47]。P组犊牛鼻腔菌群发生改变,同时与代谢通路相互作用和影响导致代谢通路功能变化。基于以上,提示P组犊牛鼻腔菌群、代谢通路和功能出现变化,变化主要为门水平菌群差异,参加菌体合成、炎性反应标志物和影响机体生长发育相关代谢通路和细胞代谢、物质的运输、分解和合成,以及疾病的代谢通路功能。但IBRV感染与鼻腔菌群及代谢通路改变的因果关系尚不明确,具体机制还有待于进一步研究。
4 结论研究结果显示,鼻腔菌群失调与IBRV感染之间有着密切的联系。IBRV感染后犊牛的鼻腔菌群组成结构与功能都发生了显著改变。厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和绿弯菌门(Chloroflexi),红游动菌属(Rhodoplanes)和不动杆菌属(Acinetobacter)可能是IBRV感染犊牛鼻腔菌群中具有显著差异的标志性物种。此外,IBRV感染犊牛的菌体合成、炎性反应标志物和机体生长发育通路潜在功能显著上调。本研究解析了犊牛IBRV感染与其鼻腔菌群变化的相关性,即鼻腔菌群紊乱与IBRV感染密切相关,研究结果可为后期开展防治犊牛呼吸道疾病提供理论基础。
| [1] |
MUYLKENS B, THIRY J, KIRTEN P, et al. Bovine herpesvirus 1 infection and infectious bovine rhinotracheitis[J]. Vet Res, 2007, 38(2): 181-209. DOI:10.1051/vetres:2006059 |
| [2] |
PARDON B, DE BLEECKER K, DEWULF J, et al. Prevalence of respiratory pathogens in diseased, non-vaccinated, routinely medicated veal calves[J]. Vet Rec, 2011, 169(11): 278-278. DOI:10.1136/vr.d4406 |
| [3] |
JONES C, CHOWDHURY S. A review of the biology of bovine herpesvirus type 1 (BHV-1), its role as a cofactor in the bovine respiratory disease complex and development of improved vaccines[J]. Anim Health Res Rev, 2007, 8(2): 187-205. DOI:10.1017/S146625230700134X |
| [4] |
NANDI S, KUMAR M, MANOHAR M, et al. Bovine herpes virus infections in cattle[J]. Anim Health Res Rev, 2009, 10(1): 85-98. DOI:10.1017/S1466252309990028 |
| [5] |
SCHUDEL A A, CARRILLO B J, WYLER R, et al. Infections of calves with antigenic variants of bovine herpesvirus 1 (BHV-1) and neurological disease[J]. J Vet Med, Ser B, 1986, 33(1-10): 303-310. DOI:10.1111/j.1439-0450.1986.tb00036.x |
| [6] |
MURPHY F A, GIBBS E P J, HORZINEK M C, et al. Paramyxoviridae[M]//MURPHY F A, GIBBS E P J, HORZINEK M C, et al. Veterinary Virology. 3rd ed. Cambridge: Academic Press, 1999: 423.
|
| [7] |
GIBBS E P J, RWEYEMAMU M M. Bovine herpesviruses. Part Ⅰ. Bovine herpesvirus 1[J]. Vet Bull, 1977, 47: 317-343. |
| [8] |
LIU Y L, JONES C. Regulation of Notch-mediated transcription by a bovine herpesvirus 1 encoded protein (ORF2) that is expressed in latently infected sensory neurons[J]. J Neurovirol, 2016, 22(4): 518-528. DOI:10.1007/s13365-015-0394-3 |
| [9] |
BERTOLOTTI L, MURATORE E, NOGAROL C, et al. Development and validation of an indirect ELISA as a confirmatory test for surveillance of infectious bovine rhinotracheitis in vaccinated herds[J]. BMC Vet Res, 2015, 11: 300. DOI:10.1186/s12917-015-0612-5 |
| [10] |
BISWAS S, BANDYOPADHYAY S, DIMRI U, et al. Bovine herpesvirus-1 (BHV-1)-a re-emerging concern in livestock: a revisit to its biology, epidemiology, diagnosis, and prophylaxis[J]. Vet Quart, 2013, 33(2): 68-81. DOI:10.1080/01652176.2013.799301 |
| [11] |
KOOK I, JONES C. The serum and glucocorticoid-regulated protein kinases (SGK) stimulate bovine herpesvirus 1 and herpes simplex virus 1 productive infection[J]. Virus Res, 2016, 222: 106-112. DOI:10.1016/j.virusres.2016.06.007 |
| [12] |
KORTEN I, RAMSEY K, MIKA M, et al. Nasal microbiota and respiratory tract infections: the role of viral detection[J]. Am J Respir Crit Care Med, 2019, 199(7): 919-922. DOI:10.1164/rccm.201710-2020LE |
| [13] |
RULLO J, FAR P M, QUINN M, et al. Local oral and nasal microbiome diversity in age-related macular degeneration[J]. Sci Rep, 2020, 10(1): 3862. DOI:10.1038/s41598-020-60674-3 |
| [14] |
LEHTINEN M J, HIBBERD A A, MÄNNIKKÖ S, et al. Nasal microbiota clusters associate with inflammatory response, viral load, and symptom severity in experimental rhinovirus challenge[J]. Sci Rep, 2018, 8(1): 11411. DOI:10.1038/s41598-018-29793-w |
| [15] |
GRIER A, GILL A L, KESSLER H A, et al. Temporal dysbiosis of infant nasal microbiota relative to respiratory syncytial virus infection[J]. J Infect Dis, 2021, 223(9): 1650-1658. DOI:10.1093/infdis/jiaa577 |
| [16] |
CHRUN T, LENG J, LA RAGIONE R M, et al. Changes in the nasal microbiota of pigs following single or co-infection with porcine reproductive and respiratory syndrome and swine influenza a viruses[J]. Pathogens, 2021, 10(10): 1225. DOI:10.3390/pathogens10101225 |
| [17] |
DICKSON R P, ERB-DOWNWARD J R, FALKOWSKI N R, et al. The lung microbiota of healthy mice are highly variable, cluster by environment, and reflect variation in baseline lung innate immunity[J]. Am J Respir Crit Care Med, 2018, 198(4): 497-508. DOI:10.1164/rccm.201711-2180OC |
| [18] |
REHNER J, SCHMARTZ G P, GROEGER L, et al. Systematic cross-biospecimen evaluation of DNA extraction kits for long-and short-read multi-metagenomic sequencing studies[J]. Genomics, Proteomics Bioinf, 2022, 20(2): 405-417. DOI:10.1016/j.gpb.2022.05.006 |
| [19] |
CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al. DADA2:high-resolution sample inference from Illumina amplicon data[J]. Nat Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [20] |
DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Appl Environ Microbiol, 2006, 72(7): 5069-5072. DOI:10.1128/AEM.03006-05 |
| [21] |
DOUGLAS G M, MAFFEI V J, ZANEVELD J R, et al. PICRUSt2 for prediction of metagenome functions[J]. Nat bioTechnol, 2020, 38(6): 685-688. DOI:10.1038/s41587-020-0548-6 |
| [22] |
CHABAN B, ALBERT A, LINKS M G, et al. Characterization of the upper respiratory tract microbiomes of patients with pandemic H1N1 influenza[J]. PLoS One, 2013, 8(7): e69559. DOI:10.1371/journal.pone.0069559 |
| [23] |
FILLAUX J, BRÉMONT F, MURRIS M, et al. Assessment of Aspergillus sensitization or persistent carriage as a factor in lung function impairment in cystic fibrosis patients[J]. Scand J Infect Dis, 2012, 44(11): 842-847. DOI:10.3109/00365548.2012.695454 |
| [24] |
SAJJAN U S, JIA Y, NEWCOMB D C, et al. H. influenzae potentiates airway epithelial cell responses to rhinovirus by increasing ICAM-1 and TLR3 expression[J]. FASEB J, 2006, 20(12): 2121-2123. DOI:10.1096/fj.06-5806fje |
| [25] |
MOLYNEAUX P L, MALLIA P, COX M J, et al. Outgrowth of the bacterial airway microbiome after rhinovirus exacerbation of chronic obstructive pulmonary disease[J]. Am J Respir Crit Care Med, 2013, 188(10): 1224-1231. DOI:10.1164/rccm.201302-0341OC |
| [26] |
SIZEMORE D R, BRANSTROM A A, SADOFF J C. Attenuated Shigella as a DNA delivery vehicle for DNA-mediated immunization[J]. Science, 1995, 270(5234): 299-303. DOI:10.1126/science.270.5234.299 |
| [27] |
FRANK D N, FEAZEL L M, BESSESEN M T, et al. The human nasal microbiota and Staphylococcus aureus carriage[J]. PLoS One, 2010, 5(5): e10598. DOI:10.1371/journal.pone.0010598 |
| [28] |
GARCÍA-RODRÍGUEZ J Á, FRESNADILLO MARTÍNEZ M J. Dynamics of nasopharyngeal colonization by potential respiratory pathogens[J]. J Antimicrob Chemother, 2002, 50(S3): 59-74. |
| [29] |
姜瑜琦, 李昱辰, 张齐, 等. 16S rRNA测序分析枯草芽胞杆菌喷鼻对猪鼻腔菌群影响的研究[J]. 畜牧与兽医, 2023, 55(2): 43-49. JIANG Y Q, LI Y C, ZHANG Q, et al. Analysis of the effect of Bacillus subtilis on swine nasal flora based on the 16S rRNA sequencing technology[J]. Animal Husbandry & Veterinary Medicine, 2023, 55(2): 43-49. (in Chinese) |
| [30] |
RIZZATTI G, LOPETUSO L R, GIBⅡNO G, et al. Proteobacteria: a common factor in human diseases[J]. BioMed Res Int, 2017, 2017: 9351507. |
| [31] |
MARTINS L O, EMPADINHAS N, MARUGG J D, et al. Biosynthesis of mannosylglycerate in the thermophilic bacterium Rhodothermus marinus: biochemical and genetic characterization of a mannosylglycerate synthase[J]. J Biol Chem, 1999, 274(50): 35407-35414. DOI:10.1074/jbc.274.50.35407 |
| [32] |
BORGES N, JORGE C D, GONÇALVES L G, et al. Mannosylglycerate: structural analysis of biosynthesis and evolutionary history[J]. Extremophiles, 2014, 18(5): 835-852. DOI:10.1007/s00792-014-0661-x |
| [33] |
UELAND P M, MCCANN A, MIDTTUN Ø, et al. Inflammation, vitamin B6 and related pathways[J]. Mol Aspects Med, 2017, 53: 10-27. DOI:10.1016/j.mam.2016.08.001 |
| [34] |
MAYENGBAM S, CHLEILAT F, REIMER R A. Dietary vitamin B6 deficiency impairs gut microbiota and host and microbial metabolites in rats[J]. Biomedicines, 2020, 8(11): 469. DOI:10.3390/biomedicines8110469 |
| [35] |
BECHTHOLD A, ALBRECHT V, LESCHIK-BONNET E, et al. DGE Statement: evaluation of vitamin supplies in Germany. Part 2: critical vitamins and vitamin supplies in special situations[J]. Ernährungs Umschau, 2012, 59(7): 396-401. |
| [36] |
WAGNER K H, WOTRUBA F, ELMADFA I. Antioxidative potential of tocotrienols and tocopherols in coconut fat at different oxidation temperatures[J]. Eur J Lipid Sci Technol, 2001, 103(11): 746-751. DOI:10.1002/1438-9312(200111)103:11<746::AID-EJLT746>3.0.CO;2-P |
| [37] |
TOKUMURA A, MAJIMA E, KARIYA Y, et al. Identification of human plasma lysophospholipase D, a lysophosphatidic acid-producing enzyme, as autotaxin, a multifunctional phosphodiesterase[J]. J Biol Chem, 2002, 277(42): 39436-39442. DOI:10.1074/jbc.M205623200 |
| [38] |
OZASA H, WATANABE T, NAKAMURA K, et al. Changes in serum levels of creatol and methylguanidine in renal injury induced by lipid peroxide produced by vitamin E deficiency and GSH depletion in rats[J]. Nephron, 1997, 75(2): 224-229. DOI:10.1159/000189536 |
| [39] |
OHASHI F, AWAJI T, SHIMADA T, et al. Plasma methylguanidine and creatinine concentrations in cats with experimentally induced acute renal failure[J]. J Vet Med Sci, 1995, 57(5): 965-966. DOI:10.1292/jvms.57.965 |
| [40] |
RODRIGUEZ M C, MACDONALD J R, MAHONEY D J, et al. Beneficial effects of creatine, CoQ10, and lipoic acid in mitochondrial disorders[J]. Muscle Nerve, 2007, 35(2): 235-242. DOI:10.1002/mus.20688 |
| [41] |
WYSS M, KADDURAH-DAOUK R. Creatine and creatinine metabolism[J]. Physiol Rev, 2000, 80(3): 1107-1213. DOI:10.1152/physrev.2000.80.3.1107 |
| [42] |
CONWAY T. The Entner-Doudoroff pathway: history, physiology and molecular biology[J]. FEMS Microbiol Rev, 1992, 9(1): 1-27. |
| [43] |
ZHAO P, TIAN P F. Biosynthesis pathways and strategies for improving 3-hydroxypropionic acid production in bacteria[J]. World J Microbiol Biotechnol, 2021, 37(7): 117. DOI:10.1007/s11274-021-03091-6 |
| [44] |
ENSIGN S A. Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation[J]. Mol Microbiol, 2006, 61(2): 274-276. DOI:10.1111/j.1365-2958.2006.05247.x |
| [45] |
BRUMELL J H, GRINSTEIN S. Salmonella redirects phagosomal maturation[J]. Curr Opin Microbiol, 2004, 7(1): 78-84. DOI:10.1016/j.mib.2003.12.005 |
| [46] |
LANDSTROM A P, PARVATIYAR M S, PINTO J R, et al. Molecular and functional characterization of novel hypertrophic cardiomyopathy susceptibility mutations in TNNC1-encoded troponin C[J]. J Mol Cell Cardiol, 2008, 45(2): 281-288. DOI:10.1016/j.yjmcc.2008.05.003 |
| [47] |
BEDSE G, DI DOMENICO F, SERVIDDIO G, et al. Aberrant insulin signaling in Alzheimer's disease: current knowledge[J]. Front Neurosci, 2015, 9: 204. |
(编辑 白永平)



