2. 四川农业大学 畜禽遗传资源发掘与创新利用四川省重点实验室, 成都 611130;
3. 四川农业大学动物科技学院 猪禽种业全国重点实验室, 成都 611130;
4. 新希望六和股份有限公司, 北京 100102;
5. 国家生猪技术创新中心, 重庆 402460
2. Farm Animal Genetic Resources Exploration and Innovation Key Laboratory of Sichuan Province, Sichuan Agricultural University, Chengdu 611130, China;
3. State Key Laboratory of Swine and Poultry Breeding Industry, College of Animal Science and Technology, Sichuan Agricultural University, Chengdu 611130, China;
4. New Hope Liuhe Co. Ltd., Beijing 100102, China;
5. National Center of Technology Innovation for Pigs, Chongqing 402460, China
DNA甲基化由DNA甲基转移酶(Dnmts)家族催化,这些甲基转移酶将甲基从S-腺苷甲硫氨酸(SAM)转移到胞嘧啶残基的第五个碳上,形成5 mC[1]。DNA甲基化是一种被广泛研究的表观遗传修饰方式,与组蛋白修饰等方式一起,在沉默逆转录病毒元件[2],调节组织特异性基因表达,基因组印迹和X染色体失活等过程中至关重要[3]。曾有研究表明,DNA甲基化会影响骨骼肌从而影响运动[4],肥胖和糖尿病也与表观遗传学紧密相关[5-8]。DNA甲基化在哺乳动物发育中起着至关重要的作用,哺乳动物基因组通常高度甲基化,在人类胚胎干细胞中,DNA甲基化会在约80%的CpG中发生,其余未甲基化的CpG残基富集在位于基因启动子的CpG岛(CGI)中[9]。近些年来,表观遗传学和甲基化测序技术的发展为畜禽重要经济性状研究提供基础[10-12]。
一氧化氮(NO)由NOS亚型产生,这些亚型包含NOS1、NOS2和NOS3[13]。有研究表明,NOS2可治疗肌肉萎缩综合征,iNOS将L-精氨酸转化为瓜氨酸,在此过程中释放NO[14-16]。NOS也在血管疾病、卵巢功能[17]和糖尿病[18-19]中具有重要调控作用[20]。NO是一种信使分子,在调节组织、细胞和器官的各种生物过程中发挥着至关重要的作用[21-23]。NO能与OxyMb反应生成MetMb和硝酸盐[24],肌红蛋白存在DeoMb、OxyMb和MetMb三种状态。肌肉中三种状态肌红蛋白相对含量决定了肉的色泽[25]。此外,有研究表明,NO可抑制线粒体呼吸链[26],而线粒体是骨骼肌MyHC纤维类型的调节因子[27]。因此,本研究发现NOS2基因可能会通过调节细胞内NO的浓度来影响肌红蛋白和肌纤维组成,从而影响肌肉发育通路基因的表达。
1 材料与方法 1.1 细胞和载体本试验中使用的细胞株为猪肾细胞系PK-15细胞,使用的是MEM培养基,添加1%NEAA、1%丙酮酸钠和10%胎牛血清(FBS),将细胞置于37 ℃,5% CO2培养箱培养;根据Addgene #71666设计质粒载体pdCas9-DNMT3A-EGFP(E4692),融合蛋白pCLenti-U6-gRNA1(NOS2)-gRNA2(NOS2)-CMV-mCherry(Y14117)由和元生物构建,其中2个gRNA位于NOS2启动子上游(表 1),通过T4 DNA连接酶合成,并克隆到表达载体。
|
|
表 1 所有gRNA目标位点及序列 Table 1 The target site and sequence of all gRNAs |
将PK-15细胞进行消化后,计数后将每组细胞配成1.0×105个·mL-1后,每孔铺500 μL细胞,于37 ℃,5% CO2培养箱中培养过夜;12~20 h后,观察细胞的融合率达到60%~70%左右进行转染;配制质粒和转染试剂稀释液,0.5 μg E4692+0.5 μg Y14117+2 μL lipo2000进行转染。各组转染试剂混匀,常温孵育5 min;将稀释好的质粒和转染试剂混合均匀,以常温孵育20 min;在转染前加入400 μL optiMEM至24孔板中,然后再添加质粒和转染试剂转染混合物,约4~6 h后换成完全培养基;转染48 h后,荧光显微镜拍照; 转染结束后,倍比稀释法挑选3组单克隆细胞。
1.3 甲基化检测使用BSP(bisulfite sequencing PCR)方法对NOS2启动子区域前500 bp范围DNA甲基化水平进行检测,目标区域序列见图 1。使用细胞DNA提取试剂盒并严格遵照说明书操作步骤对细胞样本进行DNA提取,提取后的DNA保存备用;用亚硫酸氢盐对提取的DNA进行预处理,处理过后所有尚未发生甲基化的胞嘧啶被转化为尿嘧啶,而甲基化的胞嘧啶则不变;设计引物进行PCR扩增(表 2);目的产物纯化后进行TA克隆;在使用酶切法鉴别阳性菌落后,挑取质粒进行测序;将测序得到的序列与原始序列进行比对,并计算发生甲基化位点的数量及甲基化程度。

|
图 1 DNA甲基化编辑的目标区域序列 Fig. 1 Sequences of regions targeted for DNA methylation editing |
|
|
表 2 甲基化检测的引物信息 Table 2 Primers information for methylation detection |
使用CCK-8方法对细胞的增殖能力进行定量分析,具体步骤如下:细胞经过消化作用、重悬和计数后,铺于96孔板上,细胞贴壁后,根据分组进行对应处理,在每个检测时间点(0、24、48和72 h),弃上清,加入CCK-8:MEM完全培养基=1∶9混合液100 μL于每孔中,经过2 h后,用酶标仪检测450 nm OD值。
1.5 细胞RNA提取从培养皿中倒掉培养液,然后离心,弃去细胞上清,每孔加入1 mL Trizol,然后每孔加200 μL氯仿,剧烈震荡15 s,然后于室温静置15 min;在4 ℃条件下,12 000 r·min-1离心15 min;从每管中吸取上清至新1.5 mL离心管中,在其中添加同样体积的异丙醇,混合均匀后在4 ℃沉淀10 min;4 ℃,12 000 r·min-1离心10 min后,弃掉上清液;4 ℃预冷的75%乙醇洗涤两遍后弃去废液,然后在室温干燥;先添加30~40 μL RNase-free水,等待充分溶解后,用分光光度仪测定RNA的浓度与吸光值,检测合格后于-80 ℃冰箱贮存备用。
1.6 总RNA反转录使用反转录试剂盒(TaKaRa)进行总RNA反转录,主要步骤:1)遵照说明书进行gDNA Eraser反应混合液及RNA转录反应混合液的配置;2)将RNA反转录混合液先于42 ℃水浴中放置15 min,再于85 ℃水浴中放置5 s后立即置于冰上,获得反转录产物(cDNA);3)反转录产物(cDNA)可立即用于qPCR检测,或存于-80 ℃备用。
1.7 实时荧光定量PCR引物使用Primer 5.0设计,交由和元生物合成,具体引物序列如表 3所示。处理组中基因的表达量表示为相对表达量(2-△△Ct),其中△△Ct=△Ct1(处理组)-△Ct2(对照组),即:当2-△△Ct < 1且P < 0.05时,说明处理组中基因表达量显著下调,而当2-△△Ct>1且P < 0.05时,说明基因表达量显著上调。
|
|
表 3 实时荧光定量PCR引物序列 Table 3 Primers sequence of qPCR |
细胞总蛋白抽提后测定蛋白浓度,然后进行SDS-PAGE凝胶电泳。在电泳完成前,预先准备一张PVDF膜和六张滤纸。将PVDF膜用甲醇活化5 min,再在缓冲液中平衡15 min,随后将转膜用的夹子打开,将海绵、滤纸和膜组合形成海绵-滤纸-胶-膜-滤纸-海绵的结构,然后放在转移槽中,在槽的一边放置冰块降温,在300 mA转移2 h。使用1×TBST制成5%脱脂牛奶封闭液,将膜移至封闭液中封闭1 h,接着遵照说明书配置一抗稀释液,从封闭液中取出膜,在一抗中孵育过夜,在室温下用TBST脱色摇床上洗10 min,反复3次,然后配置二抗稀释液,将膜在二抗稀释液中室温孵育1~2 h后,于室温下在TBST脱色摇床上洗10 min,重复3次,用发光液浸湿PVDF膜,然后放入成像仪中拍照成像。
1.9 细胞内NO的检测利用一氧化氮(NO)试剂盒检测细胞中NO的含量,并严格遵照产品使用说明书执行,在细胞沉淀中添加500 μL PBS,混合均匀,使细胞悬浮于等渗缓冲液中;在冰水浴条件下,通过超声波破碎仪对细胞悬液进行超声破碎,重复5次,取破碎好的匀浆液进行下一步测定;根据说明书配置混合试剂(试剂一和试剂二等体积混合),混合均匀后备用;标准贮存液用双蒸水1∶100稀释成100 μmol·L-1的标准液, 提前计算好量后配好待用;每孔加入100 μL的待测样本(3重复孔)、空白对照,标准液(空白用蒸馏水,标准液为试剂一,各2个重复孔);每孔加入400 μL的混合试剂,混匀后,至37 ℃水浴准确反应1 h;每个测试管分别加入200 μL的试剂三和100 μL的试剂四;充分漩涡混匀30 s, 室温静置40 min后,在3 000~4 000 r·min-1条件下离心10 min,取上清显色;根据说明书溶解试剂五和试剂六,按照试剂五:试剂六:试剂七=2.5∶1∶1的比例配制显色剂,每个测试管要加入600 μL显色剂,提前计算好量后配好待用;每管分别加入500 μL上清和600 μL显色剂,混合均匀后,在室温下静置10 min,并在550 nm的波长下,测定吸光度;根据公式计算NO含量:NO(μmol·L-1)= C标准×(A测定-A空白)/(A标准-A空白)。
1.10 统计分析使用R v4.1.1软件对本试验的结果进行统计分析,每个试验3次重复,结果表示为“平均数±标准差(Mean±SD)”,设置检验标准为P=0.05。
2 结果 2.1 细胞转染结果在本试验中,通过lipo2000将载体转染到PK-15细胞中。使用0.5 μg的pdCas9-DNMT3A-EGFP和0.5 μg的pCLenti-U6-gRNA1(NOS2)-gRNA2(NOS2)-CMV-mCherry以及2 μL的lipo2000进行转染。转染结果如图 2所示(图为100×放大结果)。

|
A.转染前和转染48 h白光组结果比较;B.转染前和转染48 h荧光染色结果比较 A. Comparison of white light before transfection and 48 h after transfection; B. Comparison of fluorescence staining results before transfection and 48 h after transfection 图 2 PK-15细胞转染结果(100×) Fig. 2 The results of PK-15 cell transfection (100×) |
转染48 h后,利用倍比稀释法挑选3组单克隆细胞(即处理组)进行后续试验,3组单克隆细胞结果如图 3所示(图为100×放大结果)。

|
图 3 转染后挑选3组单克隆细胞结果(100×) Fig. 3 The results of monoclonal cells in 3 groups after transfection (100×) |
挑选的3组单克隆细胞检测NOS2基因启动子前500 bp范围的DNA甲基化水平,结果发现,处理组的NOS2基因启动子DNA甲基化水平比对照组降低,但差异不显著(P>0.05),如图 4所示。gRNA1结合位点附近有2个CpG位点,在gRNA2结合位点附近有2个CpG位点。然后比较对照组和处理组中gRNA结合位点附近CpG位点的甲基化水平,结果发现,对照组的gRNA1结合位点的1个CpG位点平均甲基化为0.8,gRNA2结合位点的2个CpG位点平均甲基化为0.2,编辑过后的处理组中gRNA1结合位点的1个CpG位点平均甲基化为0.8,gRNA2结合位点的2个CpG位点平均甲基化为0.32。与对照组相比,gRNA2结合位点附近的CpG位点甲基化水平增加。
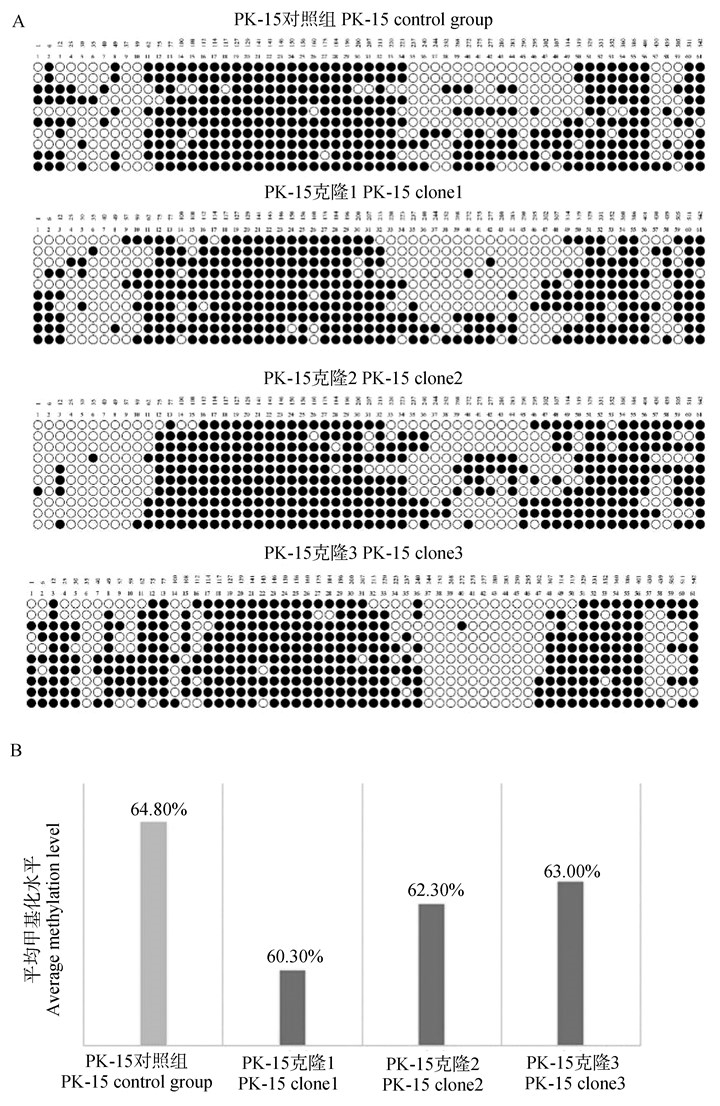
|
A.对照组和处理组NOS2启动子甲基化检测结果;B.对照组和处理组NOS2启动子平均甲基化水平 A. Results of the DNA methylation of NOS2 promoter in control group and treatment groups; B. Average methylation level of NOS2 promoter in control group and treatment groups 图 4 NOS2启动子甲基化在对照组和处理组中的比较 Fig. 4 Comparison of the DNA methylation of NOS2 promoter in control and treated groups |
在不同时间点对细胞增殖检测后,结果发现:在0 h时,对照组的细胞增殖与处理组之间无明显差异(P>0.05);在24 h时,与对照组相比,处理组的细胞增殖倍数显著降低(P < 0.05);在48 h时,与对照组相比,对照组的细胞增殖与处理组之间无明显差异(P>0.05);在72 h时,与对照组相比,对照组的细胞增殖与处理组之间无明显差异(P>0.05),结果如图 5所示。
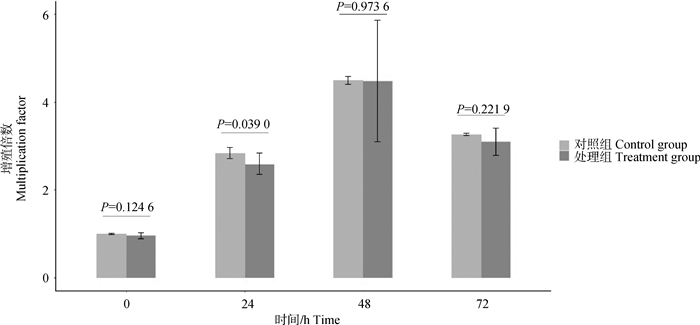
|
图 5 NOS2甲基化编辑后不同时间点对照组和处理组的细胞中细胞增殖倍数的柱状图 Fig. 5 Histograms of cell proliferation in control group and treatment groups at different times after NOS2 methylation editing |
在PK-15细胞中对NOS2进行甲基化编辑后,检测NOS2基因表达和iNOS蛋白表达结果发现,与对照组相比,处理组的NOS2基因相对表达量和iNOS蛋白质相对表达量均显著下降(P < 0.05),如图 6A和6B所示。
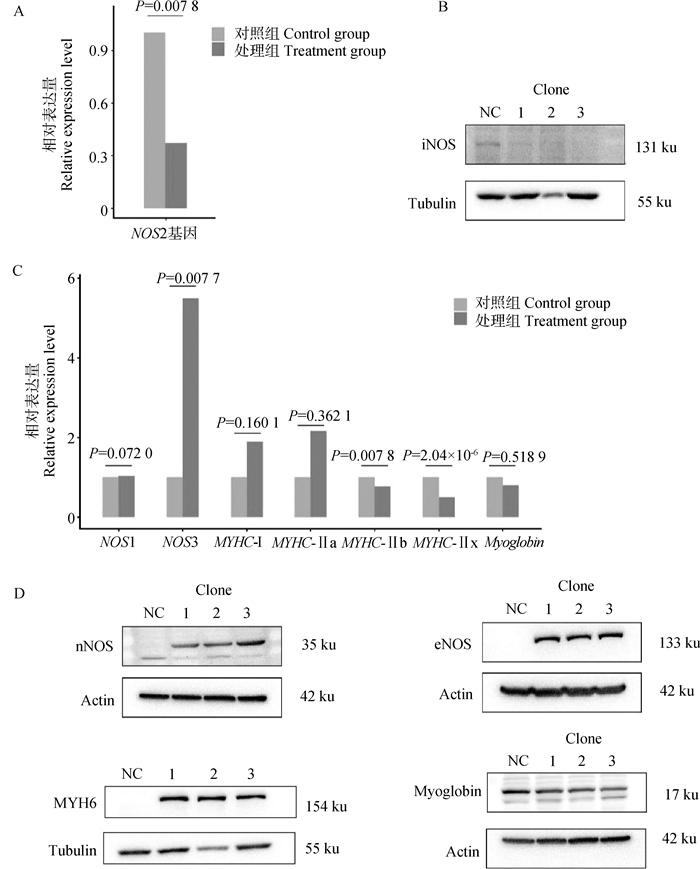
|
A.对照组和处理组细胞中NOS2基因表达水平;B. 对照组和处理组细胞中iNOS蛋白表达水平;C.对照组和处理组细胞中相关基因表达水平柱状图;D.对照组和处理组细胞中相关蛋白表达水平 A. The histogram of NOS2 gene expression levels in control group and treatment groups; B. iNOS protein expression levels in control group and treatment groups; C. The histogram of related gene expression levels in control group and treatment groups; D. Related protein expression levels in control group and treatment groups 图 6 NOS2甲基化编辑后对照组和处理组相关基因mRNA和蛋白水平的表达结果 Fig. 6 The expression results of mRNA and protein levels of relevant genes after NOS2 methylation editing in control group and treatment groups |
利用qPCR检测NOS2的同源基因以及几个与肌纤维相关基因的表达量发现:与对照组相比,NOS1基因的表达差异不显著,NOS3基因的表达量显著上调(P < 0.05);MYHC-Ⅰ和MYHC-Ⅱa表达量上调,但差异不显著(P>0.05),MYHC-Ⅱb和MYHC-Ⅱx基因的表达量显著下调(P < 0.05),Myoglobin表达量下降,但差异不显著(P>0.05),如图 6C所示。
利用WB检测NOS2的同源基因以及两个与肌纤维相关基因编码蛋白的相对表达量发现:与对照组相比,nNOS蛋白和eNOS蛋白相对表达量明显上升,MYH6的蛋白相对表达量也明显上升,而Myoglobin蛋白相对表达量明显下降。该结果表明,随着NOS2表达下降,肌纤维和肌红蛋白相关基因及相应蛋白的表达也发生变化,如图 6D所示。
2.4 细胞中NO浓度检测结果通过测定对照组和处理组的细胞NO含量来检测NOS2甲基化编辑对NO浓度的影响。结果显示, 对照组中NO浓度平均值为8.66 μmol·L-1, 处理组的NO浓度平均值为1.88 μmol·L-1, 与对照组相比,处理组的NO含量显著降低(P < 0.05),如图 7所示。
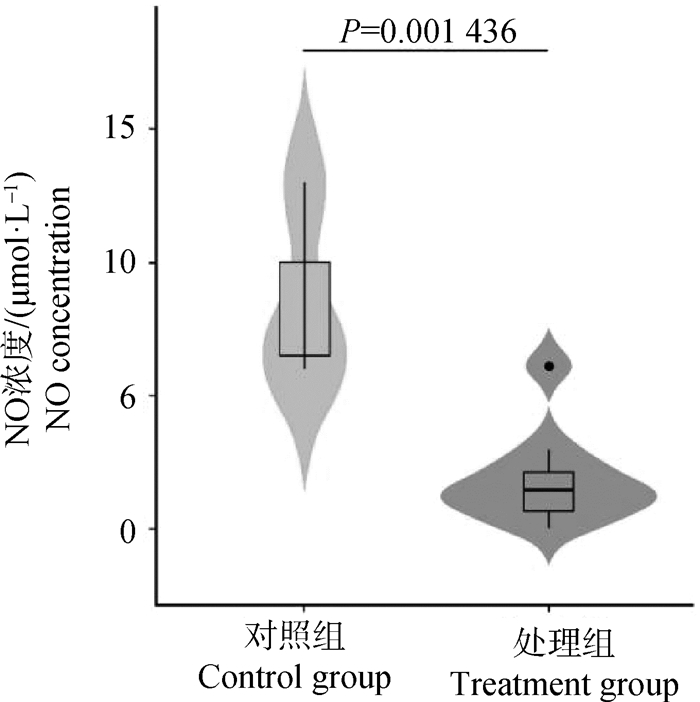
|
图 7 对照组和处理组细胞中NO含量的小提琴图 Fig. 7 The violin plot of NO concentration in control group and treatment groups |
NOS2基因属于一氧化氮合酶家族,一氧化氮合酶催化L-精氨酸产生NO和L-瓜氨酸[28],一氧化氮合酶家族有3种亚型,包括神经元一氧化氮合酶(nNOS)与诱导性一氧化氮合酶(iNOS)和内皮一氧化氮合酶(eNOS)[29],分别由NOS1、NOS2和NOS3编码。本研究通过对NOS2基因进行甲基化编辑,结果发现NOS2基因启动子前500 bp区域平均DNA甲基化水平降低,但差异不显著,而gRNA2结合位点附近的CpG位点甲基化水平增加。此外,NOS2基因表达量和其iNOS蛋白质表达量显著降低。通常位于启动子区域的DNA甲基化会抑制基因的表达,而在本研究中NOS2基因启动子前500 bp区域平均DNA甲基化水平降低但NOS2基因表达量下降,可能的原因是NOS2基因的表达活性可能受其启动子区域某个或某些关键CpG位点的甲基化水平影响,在一项NOS2甲基化与帕金森综合征的研究中,作者评估了NOS2基因3个CpG位点,其中一个CpG位点与帕金森综合征显著相关[30]。尽管本研究检测了NOS2基因启动子区域前500 bp的甲基化状态,但gRNA2结合位点附近的CpG位点甲基化水平增加。因此,NOS2基因表达量降低可能是由一个或几个关键CpG位点的甲基化引起的。
3.2 NOS2基因对肉品质的作用Hattori等[31]研究表明,在脂多糖和细胞因子刺激的血管平滑肌细胞中,PI3K-Akt通路可以通过NF-kappa B(核因子kappa B)调控iNOS的表达。Silva和Garvin[32]在小鼠上的研究发现,ATP通过刺激PI3激酶和Akt来增强一氧化氮酶的活性,进而增加NO的产生。NO是一种信使分子,在许多组织、细胞和器官中的核心生物过程中为关键的调节分子[33]。Reisberg等[34]验证了NO与绵羊血红蛋白结合的动力学和平衡,并证实NO结合得更快,亲和力比CO高1 500倍。有研究表明,肌红蛋白也可以通过氧化、配体结合以及形成生物活性S-亚硝基肌红蛋白的方式调节NO平衡[35]。因此,NOS2基因可能通过影响细胞内NO的浓度进而影响肌红蛋白的形成[36]。本研究发现,随着NOS2表达量的降低,细胞内NO含量显著降低,这个结果表明了NOS2在内源性NO合成的作用,这也与前人研究结果一致。
对于肉品质的形成,肌纤维和肌红蛋白都起着至关重要的作用。肌纤维是肌肉组织的基本结构单位,肌纤维数目和直径决定了肌肉产量。肌纤维依据代谢特征和收缩特性可以分为4种类型,分别为Ⅰ型(慢速氧化型)、Ⅱa型(快速氧化型)、Ⅱb型(快速酵解型)和Ⅱx型(中间型),而在分子生物学中,根据肌球蛋白重链(myosin heavy chain,MYHC)亚型的不同,猪骨骼肌中含有MYHCⅠ、MYHCⅡa、MYHCⅡb和MYHCⅡx四种异构体,对应不同的编码基因,特异性的存在于Ⅰ型、Ⅱa型、Ⅱb型和Ⅱx型肌纤维中[37]。不同肌纤维类型组成与猪肉品质密切相关,是影响肉质性状表现的关键因素[38]。猪肌肉色素主要由肌红蛋白、血红蛋白以及微量有色代谢物组成。在放血充分的情况下,对肉色起主要作用的是肌红蛋白。本研究结果显示,随着NOS2表达降低,MYHC-Ⅰ的表达水平增加,MYHC-Ⅱa表达水平下降,MYHC-Ⅱb和MYHC-Ⅱx的表达水平显著下降,而Myoglobin基因表达量下降。这表明NOS2表达可能通过影响肌纤维相关基因和肌红蛋白的表达从而影响猪肉色。
此外,本研究还注意到NOS2同家族的另外两个基因NOS1和NOS3表达量的变化,结果显示随着NOS2表达降低,PK-15细胞中nNOS和eNOS蛋白均显著上调,二者均属于钙离子依赖型NOS。相比于iNOS,nNOS和eNOS对NO的合成高度依赖于足够的底物和辅因子的参与,而且nNOS和eNOS的调控更加复杂,涉及许多翻译后调控,包括位点磷酸化[24]、蛋白质互作[39]以及亚细胞定位的调节[40]。因此,NOS表达的变化不总是和实际细胞NO合成水平一致。例如,在一项高盐和低盐饮食对NO合成影响的研究中,Welch和Wilcox[41]发现在肾远直小管末端的致密斑中NO合成与nNOS的表达呈负相关。因此,NOS2表达对NOS1和NOS3表达的影响以及三者的相互调控还需进一步研究。
4 结论通过在PK-15细胞上对NOS2基因进行甲基化编辑,发现NOS2基因的异常表达能够影响细胞中的NO的生成,同时影响MYHC-Ⅰ、MYHC-Ⅱa、MYHC-Ⅱb、MYHC-Ⅱx以及Myoglobin的表达,初步阐述了NOS2基因对猪肌肉发育相关基因表达的影响机制。
| [1] |
ZHANG L, ZHENG Y L, WANG R, et al. Exercise for osteoporosis: a literature review of pathology and mechanism[J]. Front Immunol, 2022, 13: 1005665. DOI:10.3389/fimmu.2022.1005665 |
| [2] |
SAVIANA M, LE P, MICALO L, et al. Crosstalk between miRNAs and DNA methylation in cancer[J]. Genes (Basel), 2023, 14(5): 1075. DOI:10.3390/genes14051075 |
| [3] |
MENG H, CAO Y, QIN J Z, et al. DNA methylation, its mediators and genome integrity[J]. Int J Biol Sci, 2015, 11(5): 604-617. DOI:10.7150/ijbs.11218 |
| [4] |
MCGEE S L, HARGREAVES M. Epigenetics and exercise[J]. Trends Endocrinol Metab, 2019, 30(9): 636-645. DOI:10.1016/j.tem.2019.06.002 |
| [5] |
LING C, RÖNN T. Epigenetics in human obesity and type 2 diabetes[J]. Cell Metab, 2019, 29(5): 1028-1044. DOI:10.1016/j.cmet.2019.03.009 |
| [6] |
SAYOLS-BAIXERAS S, SUBIRANA I, FERNÁNDEZ-SANLÉS A, et al. DNA methylation and obesity traits: an epigenome-wide association study.The REGICOR study[J]. Epigenetics, 2017, 12(10): 909-916. DOI:10.1080/15592294.2017.1363951 |
| [7] |
BAGHERIPOUR F, JEDDI S, KASHFI K, et al. Metabolic effects of L-citrulline in type 2 diabetes[J]. Acta Physiol (Oxf), 2023, 237(3): e13937. DOI:10.1111/apha.13937 |
| [8] |
TEKWE C D, LUAN Y Y, MEININGER C J, et al. Dietary supplementation with L-leucine reduces nitric oxide synthesis by endothelial cells of rats[J]. Exp Biol Med (Maywood), 2023, 248(18): 1537-1549. DOI:10.1177/15353702231199078 |
| [9] |
CHEN P Y, FENG S H, JOO J W J, et al. A comparative analysis of DNA methylation across human embryonic stem cell lines[J]. Genome Biol, 2011, 12(7): R62. DOI:10.1186/gb-2011-12-7-r62 |
| [10] |
NERY DA SILVA A, SILVA ARAUJO M, PÉRTILLE F, et al. How epigenetics can enhance pig welfare?[J]. Animals (Basel), 2022, 12(1): 32. |
| [11] |
ZHAO Y X, HOU Y, XU Y Y, et al. A compendium and comparative epigenomics analysis of cis-regulatory elements in the pig genome[J]. Nat Commun, 2021, 12(1): 2217. DOI:10.1038/s41467-021-22448-x |
| [12] |
THOMPSON R P, NILSSON E, SKINNER M K. Environmental epigenetics and epigenetic inheritance in domestic farm animals[J]. Anim Reprod Sci, 2020, 220: 106316. DOI:10.1016/j.anireprosci.2020.106316 |
| [13] |
MATTILA J T, THOMAS A C. Nitric oxide synthase: non-canonical expression patterns[J]. Front Immunol, 2014, 5: 478. |
| [14] |
HALL D T, MA J F, DI MARCO S, et al. Inducible nitric oxide synthase (iNOS) in muscle wasting syndrome, sarcopenia, and cachexia[J]. Aging (Albany NY), 2011, 3(8): 702-715. |
| [15] |
MACCALLINI C, AMOROSO R. Preface to nitric oxide modulators in health and disease I[J]. Molecules, 2022, 27(20): 6820. DOI:10.3390/molecules27206820 |
| [16] |
ALMANNAI M, EL-HATTAB A W. Nitric oxide deficiency in mitochondrial disorders: the utility of arginine and citrulline[J]. Front Mol Neurosci, 2021, 14: 682780. DOI:10.3389/fnmol.2021.682780 |
| [17] |
NATH P, MAITRA S. Physiological relevance of nitric oxide in ovarian functions: an overview[J]. Gen Comp Endocrinol, 2019, 279: 35-44. DOI:10.1016/j.ygcen.2018.09.008 |
| [18] |
HE W X, ZHOU H F, HE X Y. Aloperine protects beta-cells against streptozocin-induced injury to attenuate diabetes by targeting NOS1[J]. Eur J Pharmacol, 2022, 916: 174721. DOI:10.1016/j.ejphar.2021.174721 |
| [19] |
KRÓL M, KEPINSKA M. Human nitric oxide synthase-its functions, polymorphisms, and inhibitors in the context of inflammation, diabetes and cardiovascular diseases[J]. Int J Mol Sci, 2021, 22(1): 56. |
| [20] |
PAUTZ A, LI H G, KLEINERT H. Regulation of NOS expression in vascular diseases[J]. Front Biosci (Landmark Ed), 2021, 26(5): 85-101. DOI:10.52586/4926 |
| [21] |
PAPPAS G, WILKINSON M L, GOW A J. Nitric oxide regulation of cellular metabolism: adaptive tuning of cellular energy[J]. Nitric Oxide, 2023, 131: 8-17. DOI:10.1016/j.niox.2022.11.006 |
| [22] |
ROY R, WILCOX J, WEBB A J, et al. Dysfunctional and dysregulated nitric oxide synthases in cardiovascular disease: mechanisms and therapeutic potential[J]. Int J Mol Sci, 2023, 24(20): 15200. DOI:10.3390/ijms242015200 |
| [23] |
ALMAZROUE H, JIN Y, NELIN L D, et al. Human pulmonary microvascular endothelial cell DDAH1-mediated nitric oxide production promotes pulmonary smooth muscle cell apoptosis in co-culture[J]. Am J Physiol Lung Cell Mol Physiol, 2023, 325(3): L360-L367. DOI:10.1152/ajplung.00433.2021 |
| [24] |
BOO Y C, JO H. Flow-dependent regulation of endothelial nitric oxide synthase: role of protein kinases[J]. Am J Physiol Cell Physiol, 2003, 285(3): C499-C508. DOI:10.1152/ajpcell.00122.2003 |
| [25] |
MANCINI R A, HUNT M C. Current research in meat color[J]. Meat Sci, 2005, 71(1): 100-121. DOI:10.1016/j.meatsci.2005.03.003 |
| [26] |
BROWN G C, BORUTAITE V. Nitric oxide and mitochondrial respiration in the heart[J]. Cardiovasc Res, 2007, 75(2): 283-290. DOI:10.1016/j.cardiores.2007.03.022 |
| [27] |
VENHOFF N, LEBRECHT D, PFEIFER D, et al. Muscle-fiber transdifferentiation in an experimental model of respiratory chain myopathy[J]. Arthritis Res Ther, 2012, 14(5): R233. DOI:10.1186/ar4076 |
| [28] |
SESSA W C. The nitric oxide synthase family of proteins[J]. J Vasc Res, 1994, 31(3): 131-143. DOI:10.1159/000159039 |
| [29] |
MOSKALEVA P V, SHNAYDER N A, PETROVA M M, et al. The role of single nucleotide variants of NOS1, NOS2, and NOS3 genes in the development of the phenotype of migraine and arterial hypertension[J]. Brain Sci, 2021, 11(6): 753. DOI:10.3390/brainsci11060753 |
| [30] |
SEARLES NIELSEN S, CHECKOWAY H, CRISWELL S R, et al. Inducible nitric oxide synthase gene methylation and parkinsonism in manganese-exposed welders[J]. Parkinsonism Relat Disord, 2015, 21(4): 355-360. DOI:10.1016/j.parkreldis.2015.01.007 |
| [31] |
HATTORI Y, HATTORI S, KASAI K. Lipopolysaccharide activates Akt in vascular smooth muscle cells resulting in induction of inducible nitric oxide synthase through nuclear factor-kappa B activation[J]. Eur J Pharmacol, 2003, 481(2-3): 153-158. DOI:10.1016/j.ejphar.2003.09.034 |
| [32] |
SILVA G B, GARVIN J L. Akt1 mediates purinergic-dependent NOS3 activation in thick ascending limbs[J]. Am J Physiol Renal Physiol, 2009, 297(3): F646-F652. DOI:10.1152/ajprenal.00270.2009 |
| [33] |
MACMICKING J, XIE Q W, NATHAN C. Nitric oxide and macrophage function[J]. Annu Rev Immunol, 1997, 15: 323-350. DOI:10.1146/annurev.immunol.15.1.323 |
| [34] |
REISBERG P, OLSON J S, PALMER G. Kinetic resolution of ligand binding to the α and β chains within human hemoglobin[J]. J Biol Chem, 1976, 251(14): 4379-4383. DOI:10.1016/S0021-9258(17)33307-0 |
| [35] |
RAYNER B S, HUA S S, SABARETNAM T, et al. Nitric oxide stimulates myoglobin gene and protein expression in vascular smooth muscle[J]. Biochem J, 2009, 423(2): 169-177. DOI:10.1042/BJ20090716 |
| [36] |
VANEK T, KOHLI A. Biochemistry, myoglobin[M]//StatPearls[Internet]. Treasure Island (FL): StatPearls Publishing, 2023.
|
| [37] |
CHANG K C, FERNANDES K. Developmental expression and 5' cloning of the porcine 2x and 2b myosin heavy chain genes[J]. DNA Cell Biol, 1997, 16(12): 1429-1437. DOI:10.1089/dna.1997.16.1429 |
| [38] |
RYU Y C, KIM B C. The relationship between muscle fiber characteristics, postmortem metabolic rate, and meat quality of pig longissimus dorsi muscle[J]. Meat Sci, 2005, 71(2): 351-357. DOI:10.1016/j.meatsci.2005.04.015 |
| [39] |
KONE B C, KUNCEWICZ T, ZHANG W Z, et al. Protein interactions with nitric oxide synthases: controlling the right time, the right place, and the right amount of nitric oxide[J]. Am J Physiol Renal Physiol, 2003, 285(2): F178-F190. DOI:10.1152/ajprenal.00048.2003 |
| [40] |
SHULTZ P J, TOLINS J P. Adaptation to increased dietary salt intake in the rat.Role of endogenous nitric oxide[J]. J Clin Invest, 1993, 91(2): 642-650. DOI:10.1172/JCI116244 |
| [41] |
WELCH W J, WILCOX C S. Macula densa arginine delivery and uptake in the rat regulates glomerular capillary pressure.Effects of salt intake[J]. J Clin Invest, 1997, 100(9): 2235-2242. DOI:10.1172/JCI119761 |
(编辑 郭云雁)



