2. 宁夏大学生命科学学院, 银川 750021;
3. 甘肃省甘南州合作市佐盖曼玛镇畜牧兽医站, 甘南 747000
2. School of Life Sciences, Ningxia University, Yinchuan 750021, China;
3. Animal Husbandry and Veterinary Station, Zogimenma Town, Hezuo City, Gannan Prefecture, Gansu Province, Gannan 747000, China
牛冠状病毒(bovine coronavirus,BCoV)于1973年[1]首次在美国的牛场中发现,随后在五大洲流行,呈世界范围内广泛分布。BCoV是新生犊牛腹泻、成年牛冬季痢疾和牛呼吸道疾病的重要病原体之一。病毒的感染会导致牛出血性腹泻、产奶量减少甚至死亡,给养牛业造成巨大的经济损失。BCoV的刺突(spike,S)蛋白是诱导中和抗体的主要结构蛋白,产生的中和抗体能够阻断病毒附着和入侵,在抗病毒感染中有着重要作用。而对于目前流行的SARS-CoV-2,其通过S蛋白与细胞血管紧张素转化酶2(angiotensin converting enzyme 2, ACE2)受体结合感染宿主,S蛋白突变导致不同的新变异接连出现,显著影响着病毒的感染能力和致病性,如D614G[2]、N501Y[3]、S477N[4]等突变株,威胁着人类健康和公共安全。由于大多数冠状病毒的S蛋白承担病毒与宿主细胞膜受体结合及膜融合功能,是宿主中和抗体的重要作用位点以及疫苗设计的关键靶标,因此,详细深入地了解S蛋白的基础生物学信息,对研究BCoV的感染机制、病毒与宿主之间的相互作用及研制新型基因工程疫苗具有重要借鉴意义。
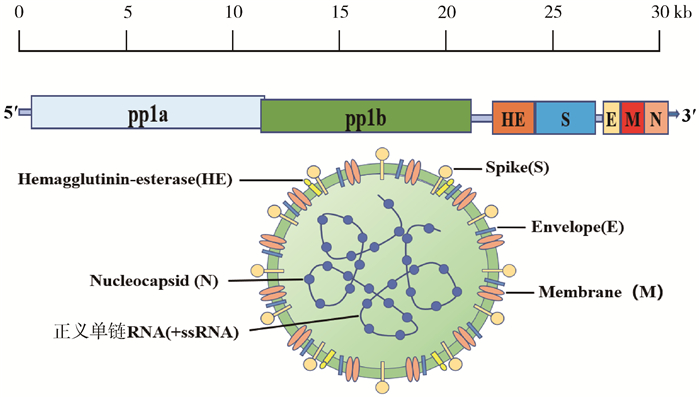
1 牛冠状病毒概述BCoV是一种肺肠病毒,属于尼多病毒目、冠状病毒科、β冠状病毒属2a亚群成员,与马冠状病毒、HCoV-OC43、犬呼吸道冠状病毒属于同一类病毒,研究表明它们在抗原性和基因遗传性上密切相关,这可能由于同一亲本病毒在遗传过程中发生种间传播以及多种重组事件造成的宿主范围变异[5]。BCoV基因组全长约31 kb,包括13个开放阅读框(ORFs),ORFs相互重叠或被基因间序列分隔,其两侧为5′和3′非翻译区域。ORF1a和ORF1b编码多蛋白,这些多蛋白被进一步切割为成熟RNA聚合酶和其他非结构蛋白。基因组编码了5种主要结构蛋白:血凝素酯酶蛋白(HE)、刺突蛋白(S)、小膜蛋白(M)、膜蛋白(E)和核衣壳蛋白(N),是病毒的基本组成粒子,其余的ORFs编码未知或特征较少的非结构蛋白(图 1)。和其他包膜病毒一样,BCoV对洗涤剂和脂质溶剂(如乙醚、氯仿)敏感,并能够被常规消毒剂福尔马林和热灭活。一些抗生素如伊维菌素[6]、氯喹及其衍生物[7]也能抑制病毒的复制。
|
图 1 BCoV基因组及结构示意图(笔者团队制作) Fig. 1 BCoV genome and structure diagram(made by the writers group) |
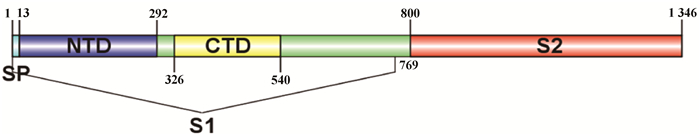
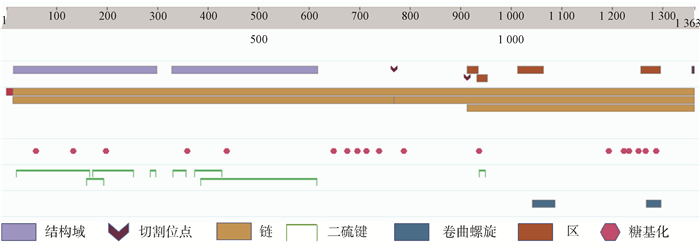
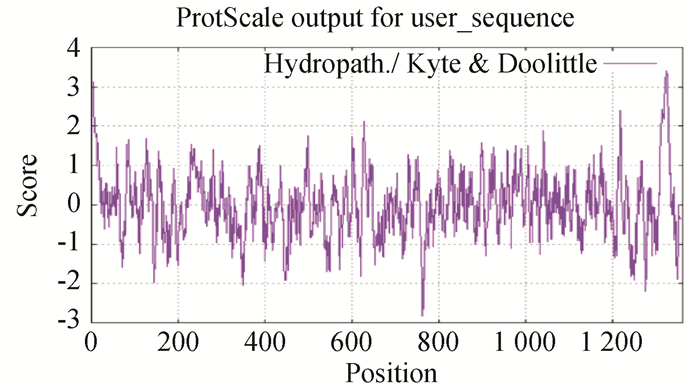
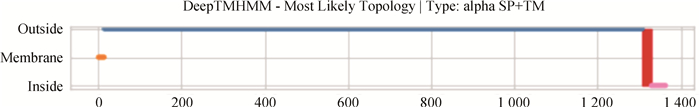
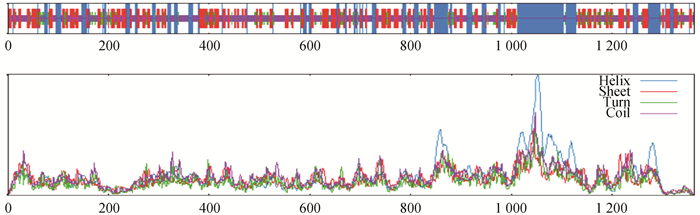
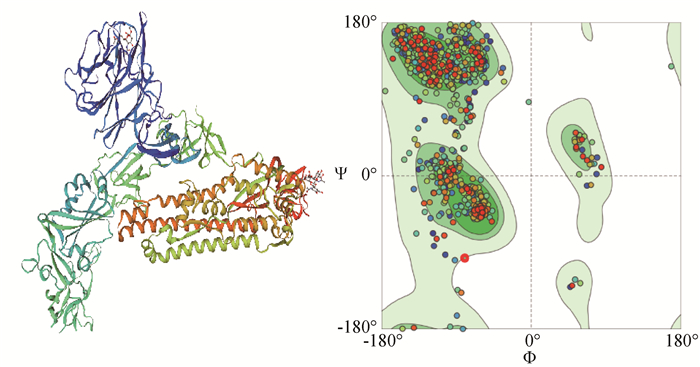
BCoV的S蛋白是一种由1 363个氨基酸编码的高度糖基化I型膜蛋白,包括一个含13个氨基酸的N末端疏水信号序列和18个N-糖基化修饰位点以及靠近C末端的两个卷曲螺旋结构[8],同时有稳定的空间拓扑结构的8个二硫键(图 2和图 3)。利用ProtScale对S蛋白的预测表明,整条多肽链中疏水性氨基酸残基数量明显居多(图 4),这些疏水性氨基酸构成卷曲螺旋的疏水核心,对形成三聚体结构起决定性作用[9]。DeepTMHMM预测S蛋白有1个位于1 308~1 328aa的跨膜区(图 5),这可能对BCoV刺突三聚体锚定、稳定和调控有着显著影响[10]。SOPMA预测分析S蛋白的二级结构主要构成原件为无规卷曲和延伸链,使抗原结构更易呈现在蛋白表面,有利于与受体结合(图 6)。利用SWISS-MODEL软件对S蛋白进行同源建模,序列一致性为91.69%,全局模型质量估计为0.75。Ramachandran图评价所建模型96.22%的氨基酸位于允许区域,得分为0.95,表明所建模型合理可靠(图 7)。S蛋白在位于氨基酸768和769之间被蛋白酶切割成两个100 ku左右的片段,形成2个结构和功能不同的S1、S2亚基。S1亚基在蛋白的N端,作为信号序列,通常显示出低水平的序列同源性,其含有N端结构域(N-terminal domain, NTD)和C端结构域(C-terminal domain, CTD),两者都可以作为病毒受体结合结构域[11]。S1亚基与宿主细胞受体结合,能刺激中和抗体产生并负责血凝素活性,因此,该区域的突变可能影响蛋白抗原性和病毒致病性[12]。S2亚基位于蛋白的C端,序列高度保守,负责膜融合活性和将糖蛋白锚定到病毒包膜的跨膜结构域。膜融合活性是由S2亚基内的内部疏水序列赋予[13]。因此,BCoV毒株中S2亚基内的亲水性氨基酸被取代会改变病毒的融合活性和感染能力。
|
图 2 S蛋白示意图(笔者团队制作) Fig. 2 Schematic diagram of the S protein(made by the writers group) |
|
图 3 S蛋白的翻译后修饰和加工示意图(UniProt) Fig. 3 Schematic diagram of post-translational modification and processing of S protein(UniProt) |
|
图 4 ProtScale预测S蛋白疏水性(笔者团队制作) Fig. 4 ProtScale predicts hydrophobicity of S protein(made by the writers group) |
|
图 5 DeepTMHMM预测S蛋白的跨膜结构(笔者团队制作) Fig. 5 DeepTMHMM predicts the transmembrane structure of S protein(made by the writers group) |
|
图 6 SOPMA预测S蛋白的二级结构(笔者团队制作) Fig. 6 SOPMA predicted the secondary structure of S protein(made by the writers group) |
|
图 7 SWISS-MODEL预测S蛋白三级结构及结构模型评价(笔者团队制作) Fig. 7 Prediction of tertiary structure of S protein by SWISS-MODEL and evaluation of structural model(made by the writers group) |
S蛋白对于病毒感染和发病机制很重要,S1亚基结合靶细胞上的受体,而S2亚基促进病毒和宿主膜的融合,从而允许病毒基因组进入宿主中复制。这种对S蛋白独特的水解切割增强了病毒感染能力,例如SARS-CoV中S蛋白存在呋喃裂解位点,被细胞内的弗林蛋白酶裂解为S1和S2亚基,增强细胞间融合和病毒感染能力[14]。MERS-CoV中S蛋白的有效切割使得该病毒能够从蝙蝠感染到人类,扩大了物种传播范围及增强了其对宿主的适应性[15]。最近研究发现该位点的缺失减弱了SARS-CoV-2的致病性,诱导了中和抗体的产生从而赋予仓鼠保护性免疫力[16]。由此推断,裂解位点中氨基酸的变化可能对细胞向性以及致病性产生显著影响,基于此特性该位点有希望被选为一种潜在的治疗靶点。
2.2 S蛋白的多态性区域冠状病毒作为目前已知基因组最大的RNA病毒组,以其低复制保真和高遗传变异的特点成为快速进化的病毒组[17]。其中S蛋白是冠状病毒研究最多的一种结构蛋白,宿主范围和组织向性的变化主要归因于S蛋白的突变[12]。有关系统发育的研究表明,S1比S2亚基对遗传变异性更敏感,且该研究观察到BCoV的S1基因的452至593区域之间的氨基酸比其他区域更多变,被鉴定为多态性区域,故常被选为分子分析的靶标[18]。之前研究显示,小鼠肝炎病毒和猪传染性肠炎病毒的细胞受体结合区位于S蛋白的多态性区域[19-20],而BCoV的S蛋白与唾液酸结合感染宿主细胞,但尚未确定细胞受体结合区[21]。多态性区域在多数冠状病毒中表现出高变异性,这可能与细胞受体结合区有关[17]。同时呼吸道和肠道BCoV分离株的序列差异表明,趋向性决定因素也可能出现在多态性区域[22]。
S1亚基这种可塑性可以在快速变化的环境中提供进化优势,允许其适应不同的组织和宿主,逃避免疫反应,对该区域的分子分析有助于研究BCoV的分子流行病学。此外,基于对不同分离株的氨基酸差异的研究清楚地揭示出:在多态性区域中存在某些致病性和临床症状的遗传标记,但多态性区域是否在牛的BCoV发病机制中起重要作用还需要更直接的证据。不过,有研究已经发现S1亚基的确能够诱导宿主体内中和抗体产生[23],这已强烈表明它在病毒-宿主作用中确实发挥重要作用。合理利用反向遗传学系统将定向修饰技术引入冠状病毒基因组生产感染性cDNA克隆,对于进一步研究S蛋白的基础生物学意义重大。
3 S蛋白的生物学功能与应用研究 3.1 S蛋白的致病特性研究S蛋白能够决定病毒的传染能力及其在宿主中的传播范围。在所有患病的临床样本中检测到的BCoV的S序列都表现出高度的相似性,不同的核苷酸序列并不是随机分布,而是聚集在多态性区域[24]。S1亚基多态性区域中氨基酸位点突变能使突变株毒力发生改变,甚至导致由强毒株到无毒株的变化[25],这为S1亚基多态性区域参与病毒致病性提供了有力的证据,同时也表明单个关键氨基酸的变化能够影响病毒的感染性,这在SARS-CoV-2中也有所体现[26]。但S基因中多态性区域是否存在其他决定病毒毒力强弱的单独特异性位点仍需要深入研究。
一些报告表明,所有BCoV在基因组和抗原水平上是相似的[18],而另一些报告则表明,不同毒株在遗传和抗原方面存在差异[27],有研究在野生反刍动物和人类肠道及呼吸道分离株的S基因中检测到明显的遗传差异,但体内和体外试验证明,这些分离株之间具有高水平的交叉保护[28]。因此,尚不确定BCoV与不同疾病表现相关的遗传标记,疾病的发生可能是病原体、宿主和环境因素之间的相互作用的结果。同时,多项针对BCoV不同临床症状的遗传决定因素的研究提出多态性区域中的多种氨基酸(113、148、501、531氨基酸位点)可能与不同疾病的发生有关[27, 29-30],数据统计显示少数氨基酸的变化与不同病症的发生具有一定的关联性[31],然而具有不同疾病的遗传标记仍然很难确认,因为临床症状的差异也可能取决于宿主因素,这需要使用反向遗传学系统或假病毒系统技术进行进一步探索验证。为了详细阐述BCoV致病特性的遗传决定因素,需要分析疾病发生中包括S基因在内的其他基因的致病性以及基因与基因之间的相关性,同时收集足够数量的不同类型BCoV基因组样本,以检测与这些疾病类型密切相关的遗传标记。
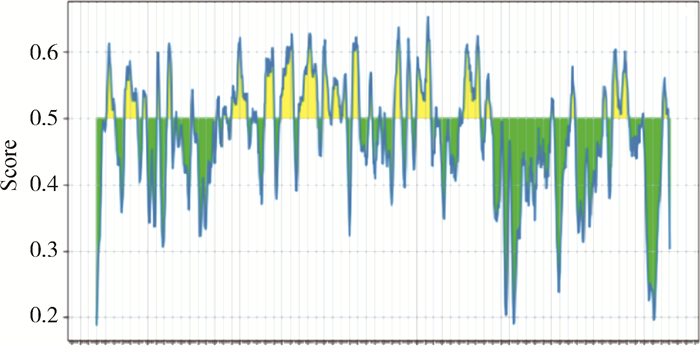
3.2 S蛋白的免疫原性S蛋白是冠状病毒附着并感染宿主细胞的关键性靶标,对病毒侵染很重要,其中一个重要因素是S蛋白的抗原性[32]。为了更好地评价S蛋白的抗原性,本课题组利用IEBD服务器分析预测其潜在的抗原表位(图 8和表 1)。S蛋白是BCoV的主要中和抗原,其两个亚基都有负责诱导宿主体内中和抗体的抗原结构域,但S1亚基能诱导更高中和活性的单克隆抗体(monoclonal antibodys,mAbs)的产生,甚至比HE诱导的抗体更稳定[33]。对S1亚基抗原表位的深入研究可能有助于开发针对BCoV有效安全的基因工程疫苗,如人们已基于其S1亚基成功研制出具有显著预防作用的SARS-CoV-2商业化疫苗,并进行大量生产和广泛应用[34]。
|
图 8 IEBD预测S蛋白的B细胞表位信息(笔者团队制作) Fig. 8 IEBD predicts B cell epitope information of S protein(made by the writers group) |
有研究人员在BCoV中和抗S蛋白可识别竞争结合试验中定义了A(氨基酸残基位点324至403)和B(氨基酸残基位点517至720)两组不同位点的抗原表位,发现虽然A组和B组mAb在体外都可以中和BCoV,但只有A组mAb表现出了体内病毒中和保护反应[35]。因此,S蛋白的抗原位点A在宿主物种中具有重要功能,如病毒抗原性及宿主特异性。有研究将B组mAb的中和表位定位到氨基酸残基位点517至621(结构域II),将结构域II中氨基酸位置528的丙氨酸突变为缬氨酸,定点突变导致B组失去中和活性,单个氨基酸的变化赋予了其对病毒中和的抵抗力,证实了S蛋白中和表位的单点突变是BCoV从免疫选择性压力中逃逸的原因[13]。有研究表明,免疫逃逸可能是暴露于宿主免疫系统的自然进化模式,而S1亚基是最有可能因免疫选择压力而发生变化的结构[36]。
多项研究使用针对BCoV的mAbs对S蛋白进行表征,表明S1亚基上含有最多抗原识别位点,也说明主要的中和表位位于BCoV的S1亚基上,而S2亚基被较少部分具有中和活性的mAbs识别[37]。这和SARS-CoV-2免疫血清中存在的90%中和抗体都是针对SARS-CoV-2刺突蛋白S1亚基的受体结合结构域(receptor binding domain,RBD)的结果基本保持一致[38]。RBD由诱导高水平的S蛋白特异性中和抗体产生和Th1偏置反应的免疫显性表位组成,已被认为是理想的疫苗靶标[39]。同时,目前大多数SARS[40]和SARS-CoV-2[41]疫苗也是基于RBD这一结构域设计的,且单独的RBD比S1亚基或完整的S基因所构建的疫苗策略具有更高的体液免疫和细胞免疫水平和保护效力,这可能是由于S蛋白其他结构域的部分抗原表位具有抗体依赖性[42]。尽管据报道称冠状病毒的S2亚基引发的中和免疫反应较弱,但其依旧可以诱导特异性细胞免疫反应和高水平的总IgG,并能够针对SARS-CoV毒株给予小鼠免疫保护[43]。由于S2亚基在不同变异毒株中高度保守,这为广谱BCoV疫苗开发提供了重要参考信息。目前,对于BCoV的RBD区域尚无清楚的描述,但是冠状病毒具有明显的共同特征,因此BCoV可能与SARS-CoV-2的RBD区域在结构和功能上存在一定的相似性,SARS-CoV-2的研究成果也许能够为BCoV的RBD区域研究提供一些参考。同时,目前BCoV疫苗大多为灭活疫苗或者减毒活疫苗,尚无商业化的牛冠状病毒疫苗上市,SARS-CoV-2疫苗研究成果对BCoV疫苗开发和抗病毒药物的设计具有重要借鉴意义。
|
|
表 1 IEBD预测S蛋白的T细胞表位信息(笔者团队制作) Table 1 cell epitope information of S protein predicted by IEBD(made by the writers group) |
尽管S蛋白的S2亚基在BCoV全基因组中相当保守,但完整的S基因仍然被允许进行分子比较。人们通过对S蛋白部分特定基因或者全长基因进行系统发育分析,从而揭示BCoV的分子流行病学和遗传进化信息,以及剖析S基因中遗传信息与毒力强弱的相关性(表 2)。
|
|
表 2 完整S蛋白或部分S蛋白基因用于系统发育分析 Table 2 Complete S protein or partial S protein genes were used for phylogenetic analysis |
由于研究者在研究中仅使用部分或整个S序列进行分析,因此被认为推断进化路径或共同祖先的依据并不充分。HE序列甚至于全基因组序列也应该被考虑,使得溯源分析趋于完善。分析不同BCoV毒株之间S基因中氨基酸差异,了解其存在的抗原表位与遗传关系,能够为确定最适合冠状病毒疫苗的成分提供有用的信息。同时有必要从分子水平进行病毒监测和深度分析,以发现抗原特性改变的新BCoV谱系,确保为有效疫苗的研发提供准确信息以应对预出现的新变异株。
4 S蛋白变异分析 4.1 重组事件近三年SARS-CoV-2的出现和传播再次引起了人们对冠状病毒的关注,高突变率和重组率是冠状病毒基因型和表型变异的重要原因之一。重组已被报道为冠状病毒基因组扩增和基因型变异的驱动力,并有助于新病毒的诞生和新病例的出现[49]。之前有研究表明,BCoV的HE基因存在明显的重组事件,后来人们也发现了S基因内的潜在重组事件[50]。新重组毒株的出现可能有利于病毒进化与提高其对环境适应性,如与鸡传染性支气管炎病毒的重组导致跨物种传播和致病性变化[51],这是由于重组导致新冠状病毒出现的第一个证据。随后在雪貂冠状病毒[52]、犬呼吸道冠状病毒[53]的S基因区域均检测到重组事件。此外,最近研究发现,牛的双重感染可能导致SARS-CoV-2和BCoV重组,产生具有这两种病毒特征的嵌合病毒[54]。
由此推断BCoV之间的跨物种传播,除了本身的基因突变外,冠状病毒之间S基因和HE基因的重组事件也是造成宿主范围转变的主要原因之一,应强调限制接触冠状病毒库的重要性,因为病毒库为新病毒的产生提供了巨大的遗传物质来源[55]。另外,通过深度测序技术对不同BCoV种群进行遗传进化分析,以监测BCoV新的基因型和其他β冠状病毒重组体的出现,并评估它们在未来流行病中的致病潜力,以便更好地揭示可能发生的重组事件与预测未来BCoV的分子流行病学。
4.2 免疫压力选择Franzo等[36]发现,免疫逃逸可能是暴露于宿主免疫系统的自然进化模式,而S1亚基是最有可能因免疫选择压力而发生变化的结构。研究人员利用收集到的大量的可用S蛋白数据集进行了选择性压力分析,证明了免疫压力选择是主要影响S蛋白变异的因素,特别是在S1区域检测到不同位点的突变,高数量氨基酸的变化证明了突变是偶然发生的多样化选择[24]。这表明进化不是普遍存在的,主要通过选择性爆发来发挥作用,例如由环境、宿主等因素引起[56]。遗传多样性的压力作用于暴露在S表面的氨基酸,同时宿主免疫压力也会反作用于S蛋白,致使S蛋白不断发生循环进化,使BCoV不断适应新宿主与增强自身在宿主体内存留的持久性。
4.3 蛋白质的相互作用蛋白质之间的相互作用能够促进S蛋白的变异,而S蛋白通过与唾液酸结合可以形成稳定的复合物来阻止蛋白质多样化,使结合位点高度保守,进而保持S蛋白的稳定,这一猜测在HCoV-OC43和猪血凝性脑脊髓炎病毒中得到了证明[33]。蛋白质功能是长期进化过程的结果,涉及不同领域的精细相互作用,因此,在一个位点发生突变后,往往需要其他位点的补偿性突变才能获得适度的功能性增益,从而在进化中确认一个病毒与宿主相互兼容的模式[57]。S蛋白氨基酸多个位点的共同进化表明S蛋白在与宿主相互作用中保持整体蛋白质稳定性的相关性。由此推测,不同位点共同进化的发生可能是保持蛋白质稳定性和功能的补偿性突变的结果。
4.4 宿主向性除上述原因外,宿主向性是有利于S蛋白变异的因素之一。对冠状病毒宿主范围的评估确定了牛是BCoV引入散养、圈养和野生物种的主要宿主来源,也证实了BCoV作为主要病毒库与其它新型冠状病毒的出现具有明确的相关性[58]。令人惊讶的是,人类感染HCoV-OC43的病例和动物冠状病毒的起源也可以追溯到牛(图 9),目前的结果进一步显示该病毒具有巨大的人畜共患感染潜力,从而有必要对S蛋白结构和功能进行深入研究[59]。同一种BCoV在不同宿主中有不同程度的变异,不同的BCoV在同一宿主中的致病性也不尽相同,这可能是S蛋白与宿主内的其他病毒或者细菌以及环境因素相互作用的结果。
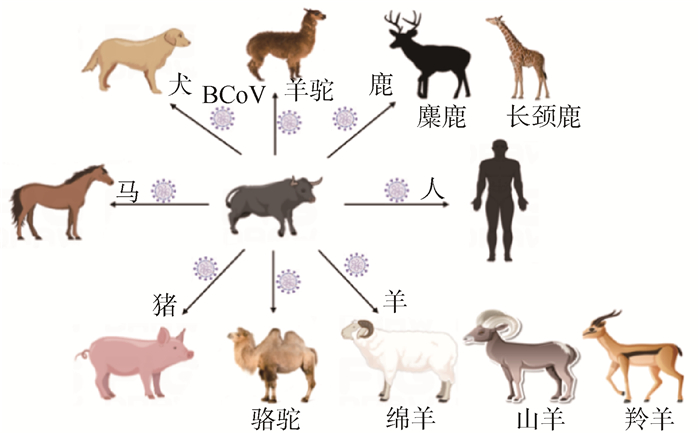
|
图 9 BCoV的潜在跨物种传播 Fig. 9 Potential cross-species transmission of BCoV |
总体而言,CoV作为一种快速进化的新兴病原体,能够短时间内进行跨物种传播和新宿主的定植,其中最主要的原因之一是S蛋白变异所导致。免疫压力选择的作用已被证明是导致S蛋白遗传变异的主要原因,有利于S蛋白的进化,进而扩大宿主范围和重塑病毒的传染性。
5 S蛋白的选择模式S1亚基中的两个结构域具有明确的正选择模式。正选择模式通常用于物种与环境之间的适应性进化机制研究,例如在猪圆环病毒[60]和猪细小病毒[61]中记录了编码病毒蛋白的正选择基因片段,并阐述了其与环境之间的进化机制。Zhu等[62]利用生物信息学软件检测到处于正选择状态的氨基酸位点为113、499、501、509、1 238,N端结构域中含有的正选择位点通过受体结合亚基在宿主免疫系统起作用,这可能与其含有的中和表位有关[8]。研究表明,S蛋白中强正向选择序列与免疫反应和受体结合有关,因此连续监测阳性选择位点的变化可能为识别未来的主要流行株提供潜在的有用数据,在将来有效预防BCoV的疫苗研发中提供关键信息。
S2亚基具有的是一种负选择模式,病毒生命周期中具有基本功能的基因通常具有负选择模式,该模式是限制和调节病毒表型的主导力量[63]。与其他模式相比,这种模式更灵活且能够减弱有害突变的积累[64],例如,在感染了BCoV的S2重组体的细胞中观察到大量合胞体形成,这加速了病毒的复制和造成免疫逃避,从而提高病毒的感染能力[65]。由此推测,S2亚基的主要功能是与细胞区室而不是宿主的免疫系统元件相作用,但是N端除外,因为S2序列N末端的突变经常导致抗原性的改变[66]。S蛋白的受体结合亚基具有强阳性选择位点,而融合亚基处于负选择模式,基于这种选择模式暗示了BCoV的适应性进化,这解释了BCoV在牛和其他动物甚至到人之间循环传播而不会消失的作用机制,同时也表明其感染的持久性以及跨物种传播的巨大潜力。
6 基于S蛋白的BCoV侵染的潜在双受体系统冠状病毒的病毒进入依赖于病毒粒子表面的S-三聚体与宿主细胞受体之间的特异性相互作用,宿主受体的参与使S-三聚体不稳定而暴露出S1和S2亚基之间的切割位点,然后被宿主中的组织蛋白酶或细胞外蛋白酶切割,启动S2介导的膜融合和病毒进入[67]。最近有人对SADS-CoV结构进行了详细分析,提出了一种基于S蛋白的双受体结合基序系统的独特见解[67],这可能也适用于BCoV。对于大多数冠状病毒而言,S1-NTD能识别细胞表面碳水化合物,S1-CTD可以特异性地与细胞蛋白受体结合。之前的报告中提出BCoV使用5-N-乙酰基-9-O-乙酰神经氨酸(Neu5, 9Ac2)作为S1-NTD识别的受体(图 10)。然而,最近发现Neu5, 9Ac2只是BCoV的附着受体,识别受体还未确定[68]。此外,Szczepanski等[69]使用多克隆抗体阻断了病毒与人类白细胞抗原I (human leukocyte antigen, HLA-I)分子之间的相互作用,防止了BCoV的感染,这表明HLA-1是BCoV的进入受体,根据目前研究推测,BCoV也可能使用双受体结合基序系统,其中Neu5, 9Ac2作为S1-NTD识别的聚糖附着受体,HLA-I作为S1-CTD识别的蛋白质附着受体。
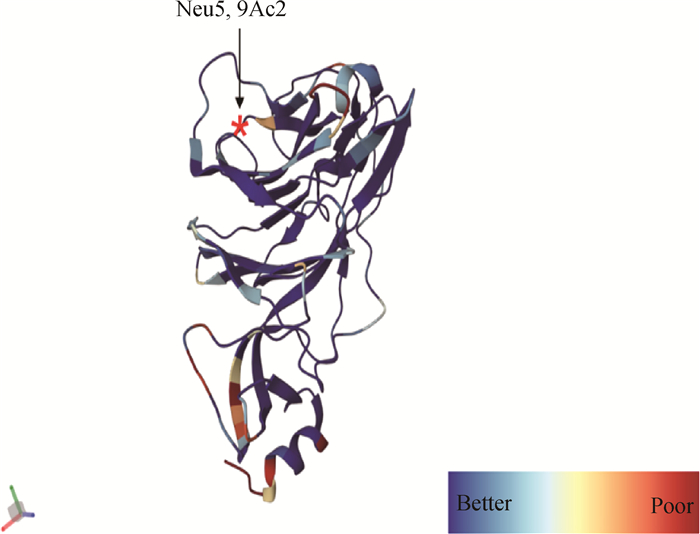
|
结构使用密度拟合方案对蛋白结构进行着色,*表示糖受体Neu5,9Ac2的结合位点 The protein structure was colored using a density fitting scheme, and the * indicated the binding site of the sugar receptor Neu5, 9Ac2 图 10 BCoV S1-NTD晶体结构(PDB ID:4H14) Fig. 10 Crystal structure of BCoV S1-NTD (PDB ID: 4H14) |
研究表明,宿主受体结合的S1亚基是宿主向性及传播范围的主要决定因素。Millet等[70]发现,冠状病毒的S1在一定程度上可以被认为是已经进化出NTD和CTD结合模块,作为扩大宿主范围以及不同宿主物种之间的细胞向性的策略。推测NTD允许新出现的冠状病毒适应新的宿主环境并尽量保持低水平的结合,以促进对宿主细胞的感染,这可能在跨越物种屏障中发挥关键作用。相反,CTD通过调整并获得适应性突变,以优化与新宿主蛋白受体的结合。
假定的双受体识别系统可能在BCoV的跨物种传播和感染过程中发挥关键作用。具体机制的阐述仍需深入了解S蛋白的整体结构以便进一步解读和验证。迄今为止,细胞识别受体尚不确定,病毒进入的机理尚不清楚,RBD的功能仍需阐明。这限制了人们对BCoV致病特性和潜在跨物种传播研究以及S蛋白详细机理分析。
7 结语与展望S糖蛋白是BCoV的主要结构蛋白之一,在病毒的初始感染、致病毒力、宿主向性、跨越种间屏障和诱导宿主免疫应答等多个过程中发挥着重要作用。在当今强传染性和高致病率的SARS-CoV-2全球流行的趋势下,对于S蛋白的基础和应用研究已经成为国内外冠状病毒研究的一个热点和重要内容。人们利用S蛋白的各种截断形式以及完整的三聚体结构开发疫苗,有些经过三期试验后已进入商业化生产和临床应用,并得到了良好的预防效果。但就目前而言,人们对BCoV的关注度较低,对该病毒S蛋白相关研究阐述较少,且尚未生产出安全的商业化疫苗用于预防BCoV大规模化感染。BCoV的S蛋白作为病毒的多功能性蛋白,应当充分发挥其在诊断、预防和治疗方面的应用价值。对BCoV的S蛋白深入研究将有利于BCoV感染疾病的治疗与防控以及明确该病毒对人类跨物种屏障传播风险,以降低该病的发病率、致死率和人畜共患病概率,以促进全球畜牧业经济稳定发展和公共卫生安全保障。
| [1] |
MEBUS C A, STAIR E L, RHODES M B, et al. Pathology of neonatal calf diarrhea induced by a coronavirus-like agent[J]. Vet Pathol, 1973, 10(1): 45-64. DOI:10.1177/030098587301000105 |
| [2] |
KORBER B, FISCHER W M, GNANAKARAN S, et al. Tracking changes in SARS-CoV-2 Spike: evidence that D614G increases infectivity of the COVID-19 virus[J]. Cell, 2020, 182(4): 812-827. DOI:10.1016/j.cell.2020.06.043 |
| [3] |
LIU H L, ZHANG Q Q, WEI P C, et al. The basis of a more contagious 501Y.V1 variant of SARS-CoV-2[J]. Cell Res, 2021, 31(6): 720-722. DOI:10.1038/s41422-021-00496-8 |
| [4] |
CHEN A T, ALTSCHULER K, ZHAN S H, et al. COVID-19 CG enables SARS-CoV-2 mutation and lineage tracking by locations and dates of interest[J]. Elife, 2021, 10: e63409. DOI:10.7554/eLife.63409 |
| [5] |
JEVŠNIK VIRANT M, AČG ERNE D, PETROVEC M, et al. Genetic characterisation and comparison of three human coronaviruses (HKU1, OC43, 229E) from patients and bovine coronavirus (BCoV) from cattle with respiratory disease in Slovenia[J]. Viruses, 2021, 13(4): 676. DOI:10.3390/v13040676 |
| [6] |
YESILBAG K, TOKER E B, ATES O. Ivermectin also inhibits the replication of bovine respiratory viruses (BRSV, BPIV-3, BoHV-1, BCoV and BVDV) in vitro[J]. Virus Res, 2021, 297: 198384. DOI:10.1016/j.virusres.2021.198384 |
| [7] |
GALAN L E B, SANTOS N M D, ASATO M S, et al. Phase 2 randomized study on chloroquine, hydroxychloroquine or ivermectin in hospitalized patients with severe manifestations of SARS-CoV-2 infection[J]. Pathog Glob Health, 2021, 115(4): 235-242. DOI:10.1080/20477724.2021.1890887 |
| [8] |
BIDOKHTI M R M, TRÅVÉN M, KRISHNA N K, et al. Evolutionary dynamics of bovine coronaviruses: natural selection pattern of the spike gene implies adaptive evolution of the strains[J]. J Gen Virol, 2013, 94(Pt 9): 2036-2049. |
| [9] |
SUZUKI K, HIROAKI H, KOHDA D, et al. An isoleucine zipper peptide forms a native-like triple stranded coiled coil in solution[J]. Protein Eng, 1998, 11(11): 1051-1055. DOI:10.1093/protein/11.11.1051 |
| [10] |
SHAIK M M, PENG H Q, LU J M, et al. Structural basis of coreceptor recognition by HIV-1 envelope spike[J]. Nature, 2019, 565(7739): 318-323. DOI:10.1038/s41586-018-0804-9 |
| [11] |
FEHR A R, PERLMAN S. Coronaviruses: an overview of their replication and pathogenesis[M]//MAIER H, BICKERTON E, BRITTON P. Coronaviruses. New York: Humana Press, 2015: 1-23.
|
| [12] |
GALLAGHER T M, BUCHMEIER M J. Coronavirus spike proteins in viral entry and pathogenesis[J]. Virology, 2001, 279(2): 371-374. DOI:10.1006/viro.2000.0757 |
| [13] |
LIU L H, HAGGLUND S, HAKHVERDYAN M, et al. Molecular epidemiology of bovine coronavirus on the basis of comparative analyses of the S gene[J]. J Clin Microbiol, 2006, 44(3): 957-960. DOI:10.1128/JCM.44.3.957-960.2006 |
| [14] |
FOLLIS K E, YORK J, NUNBERG J H. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell-cell fusion but does not affect virion entry[J]. Virology, 2006, 350(2): 358-369. DOI:10.1016/j.virol.2006.02.003 |
| [15] |
MENACHERY V D, DINNON Ⅲ K H, YOUNT B L JR, et al. Trypsin treatment unlocks barrier for zoonotic bat coronavirus infection[J]. J Virol, 2020, 94(5): e01774-19. |
| [16] |
SASAKI M, TOBA S, ITAKURA Y, et al. SARS-CoV-2 bearing a mutation at the S1/S2 cleavage site exhibits attenuated virulence and confers protective immunity[J]. mBio, 2021, 12(4): e0141521. DOI:10.1128/mBio.01415-21 |
| [17] |
YUAN H W, WEN H L. Research progress on coronavirus S proteins and their receptors[J]. Arch Virol, 2021, 166(7): 1811-1817. DOI:10.1007/s00705-021-05008-y |
| [18] |
VILČEK S, JACKOVÁ A, KOLESÁROVÁ M, et al. Genetic variability of the S1 subunit of enteric and respiratory bovine coronavirus isolates[J]. Acta Virol, 2017, 61(2): 212-216. DOI:10.4149/av_2017_02_12 |
| [19] |
KUBO H, YAMADA Y K, TAGUCHI F. Localization of neutralizing epitopes and the receptor-binding site within the amino-terminal 330 amino acids of the murine coronavirus spike protein[J]. J Virol, 1994, 68(9): 5403-5410. DOI:10.1128/jvi.68.9.5403-5410.1994 |
| [20] |
KREMPL C, SCHULTZE B, LAUDE H, et al. Point mutations in the S protein connect the sialic acid binding activity with the enteropathogenicity of transmissible gastroenteritis coronavirus[J]. J Virol, 1997, 71(4): 3285-3287. DOI:10.1128/jvi.71.4.3285-3287.1997 |
| [21] |
LANG Y F, LI W T, LI Z S, et al. Coronavirus hemagglutinin-esterase and spike proteins coevolve for functional balance and optimal virion avidity[J]. Proc Natl Acad Sci U S A, 2020, 117(41): 25759-25770. DOI:10.1073/pnas.2006299117 |
| [22] |
YOO D, DEREGT D. A single amino acid change within antigenic domain Ⅱ of the spike protein of bovine coronavirus confers resistance to virus neutralization[J]. Clin Diagn Lab Immunol, 2001, 8(2): 297-302. DOI:10.1128/CDLI.8.2.297-302.2001 |
| [23] |
SAIF L J. Bovine respiratory coronavirus[J]. Vet Clin North Am Food Anim Pract, 2010, 26(2): 349-364. DOI:10.1016/j.cvfa.2010.04.005 |
| [24] |
HODNIK J J, JEŽEK J, STARIČ J. Coronaviruses in cattle[J]. Trop Anim Health Prod, 2020, 52(6): 2809-2816. DOI:10.1007/s11250-020-02354-y |
| [25] |
JEONG J H, KIM G Y, YOON S S, et al. Molecular analysis of S gene of spike glycoprotein of winter dysentery bovine coronavirus circulated in Korea during 2002-2003[J]. Virus Res, 2005, 108(1-2): 207-212. DOI:10.1016/j.virusres.2004.07.003 |
| [26] |
ZHANG L Z, JACKSON C B, MOU H H, et al. SARS-CoV-2 spike-protein D614G mutation increases virion spike density and infectivity[J]. Nat Commun, 2020, 11(1): 6013. DOI:10.1038/s41467-020-19808-4 |
| [27] |
HASOKSUZ M, SREEVATSAN S, CHO K O, et al. Molecular analysis of the S1 subunit of the spike glycoprotein of respiratory and enteric bovine coronavirus isolates[J]. Virus Res, 2002, 84(1-2): 101-109. DOI:10.1016/S0168-1702(02)00004-7 |
| [28] |
HAN M G, CHEON D S, ZHANG X M, et al. Cross-protection against a human enteric coronavirus and a virulent bovine enteric coronavirus in gnotobiotic calves[J]. J Virol, 2006, 80(24): 12350-12356. DOI:10.1128/JVI.00402-06 |
| [29] |
BOK M, MIÑO S, RODRIGUEZ D, et al. Molecular and antigenic characterization of bovine Coronavirus circulating in Argentinean cattle during 1994-2010[J]. Vet Microbiol, 2015, 181(3-4): 221-229. DOI:10.1016/j.vetmic.2015.10.017 |
| [30] |
The Bovine Respiratory Disease Complex Coordinated Agricultural Project Research Team. Rapid Communication: Subclinical bovine respiratory disease-loci and pathogens associated with lung lesions in feedlot cattle[J]. J Anim Sci, 2017, 95(6): 2726-2731. |
| [31] |
KISER J N, NEIBERGS H L. Identifying loci associated with bovine corona virus infection and bovine respiratory disease in dairy and feedlot cattle[J]. Front Vet Sci, 2021, 8: 679074. DOI:10.3389/fvets.2021.679074 |
| [32] |
SUZUKI T, OTAKE Y, UCHIMOTO S, et al. Genomic characterization and phylogenetic classification of bovine coronaviruses through whole genome sequence analysis[J]. Viruses, 2020, 12(2): 183. DOI:10.3390/v12020183 |
| [33] |
LI F. Evidence for a common evolutionary origin of coronavirus spike protein receptor-binding subunits[J]. J Virol, 2012, 86(5): 2856-2858. DOI:10.1128/JVI.06882-11 |
| [34] |
YANG S L, LI Y, DAI L P, et al. Safety and immunogenicity of a recombinant tandem-repeat dimeric RBD-based protein subunit vaccine (ZF2001) against COVID-19 in adults: two randomised, double-blind, placebo-controlled, phase 1 and 2 trials[J]. Lancet Infect Dis, 2021, 21(8): 1107-1119. DOI:10.1016/S1473-3099(21)00127-4 |
| [35] |
DEREGT D, GIFFORD G A, IJAZ M K, et al. Monoclonal antibodies to bovine coronavirus glycoproteins E2 and E3:demonstration of in vivo virus-neutralizing activity[J]. J Gen Virol, 1989, 70(Pt 4): 993-998. |
| [36] |
FRANZO G, DRIGO M, LEGNARDI M, et al. Bovine coronavirus: variability, evolution, and dispersal patterns of a No longer neglected betacoronavirus[J]. Viruses, 2020, 12(11): 1285. DOI:10.3390/v12111285 |
| [37] |
VAUTHEROT J F, MADELAINE M F, BOIREAU P, et al. Bovine coronavirus peplomer glycoproteins: detailed antigenic analyses of S1, S2 and HE[J]. J Gen Virol, 1992, 73(Pt 7): 1725-1737. |
| [38] |
PICCOLI L, PARK Y J, TORTORICI M A, et al. Mapping neutralizing and immunodominant sites on the SARS-CoV-2 spike receptor-binding domain by structure-guided high-resolution serology[J]. Cell, 2020, 183(4): 1024-1042. DOI:10.1016/j.cell.2020.09.037 |
| [39] |
WANG Z Z, POPOWSKI K D, ZHU D S, et al. Exosomes decorated with a recombinant SARS-CoV-2 receptor-binding domain as an inhalable COVID-19 vaccine[J]. Nat Biomed Eng, 2022, 6(7): 791-805. DOI:10.1038/s41551-022-00902-5 |
| [40] |
JIANG S B, BOTTAZZI M E, DU L Y, et al. Roadmap to developing a recombinant coronavirus S protein receptor-binding domain vaccine for severe acute respiratory syndrome[J]. Expert Rev Vacc, 2012, 11(12): 1405-1413. DOI:10.1586/erv.12.126 |
| [41] |
SUN Y S, ZHOU J J, ZHU H P, et al. Development of a recombinant RBD subunit vaccine for SARS-CoV-2[J]. Viruses, 2021, 13(10): 1936. DOI:10.3390/v13101936 |
| [42] |
ROGERS T F, ZHAO F Z, HUANG D L, et al. Isolation of potent SARS-CoV-2 neutralizing antibodies and protection from disease in a small animal model[J]. Science, 2020, 369(6506): 956-963. DOI:10.1126/science.abc7520 |
| [43] |
GUO Y J, SUN S H, WANG K Y, et al. Elicitation of immunity in mice after immunization with the S2 subunit of the severe acute respiratory syndrome coronavirus[J]. Dna Cell Biol, 2005, 24(8): 510-515. DOI:10.1089/dna.2005.24.510 |
| [44] |
ZHU Q H, SU M J, LI Z J, et al. Epidemiological survey and genetic diversity of bovine coronavirus in Northeast China[J]. Virus Res, 2022, 308: 198632. DOI:10.1016/j.virusres.2021.198632 |
| [45] |
DAVID D, STORM N, ILAN W, et al. Characterization of winter dysentery bovine coronavirus isolated from cattle in Israel[J]. Viruses, 2021, 13(6): 1070. DOI:10.3390/v13061070 |
| [46] |
SEVINC TEMIZKAN S, ALKAN F. Bovine coronavirus infections in Turkey: molecular analysis of the full-length spike gene sequences of viruses from digestive and respiratory infections[J]. Arch Virol, 2021, 166(9): 2461-2468. DOI:10.1007/s00705-021-05147-2 |
| [47] |
MARTÍNEZ N, BRANDÃO P E, DE SOUZA S P, et al. Molecular and phylogenetic analysis of bovine coronavirus based on the spike glycoprotein gene[J]. Infect Genet Evol, 2012, 12(8): 1870-1878. DOI:10.1016/j.meegid.2012.05.007 |
| [48] |
KANNO T, HATAMA S, ISHIHARA R, et al. Molecular analysis of the S glycoprotein gene of bovine coronaviruses isolated in Japan from 1999 to 2006[J]. J Gen Virol, 2007, 88(Pt 4): 1218-1224. |
| [49] |
WANG Y Q, LIU D, SHI W F, et al. Origin and possible genetic recombination of the middle east respiratory syndrome coronavirus from the first imported case in China: phylogenetics and coalescence analysis[J]. mBio, 2015, 6(5): e01280-15. |
| [50] |
SALEM E, DHANASEKARAN V, CASSARD H, et al. Global transmission, spatial segregation, and recombination determine the long-term evolution and epidemiology of bovine coronaviruses[J]. Viruses, 2020, 12(5): 534. DOI:10.3390/v12050534 |
| [51] |
DOMANSKA-BLICHARZ K, SAJEWICZ-KRUKOWSKA J. Recombinant turkey coronavirus: are some S gene structures of gammacoronaviruses especially prone to exchange?[J]. Poult Sci, 2021, 100(4): 101018. DOI:10.1016/j.psj.2021.101018 |
| [52] |
MINAMI S, KURODA Y, TERADA Y, et al. Detection of novel ferret coronaviruses and evidence of recombination among ferret coronaviruses[J]. Virus Genes, 2016, 52(6): 858-862. DOI:10.1007/s11262-016-1365-3 |
| [53] |
LU S, WANG Y Q, CHEN Y Z, et al. Discovery of a novel canine respiratory coronavirus support genetic recombination among betacoronavirus1[J]. Virus Res, 2017, 237: 7-13. DOI:10.1016/j.virusres.2017.05.006 |
| [54] |
ULRICH L, WERNIKE K, HOFFMANN D, et al. Experimental infection of cattle with SARS-CoV-2[J]. Emerg Infect Dis, 2020, 26(12): 2979-2981. DOI:10.3201/eid2612.203799 |
| [55] |
ISLAM A, FERDOUS J, ISLAM S, et al. Evolutionary dynamics and epidemiology of endemic and emerging coronaviruses in humans, domestic animals, and wildlife[J]. Viruses, 2021, 13(10): 1908. DOI:10.3390/v13101908 |
| [56] |
FRANZO G, LEGNARDI M, TUCCIARONE C M, et al. Evolution of infectious bronchitis virus in the field after homologous vaccination introduction[J]. Vet Res, 2019, 50(1): 92. DOI:10.1186/s13567-019-0713-4 |
| [57] |
CAMPS M, HERMAN A, LOH E, et al. Genetic constraints on protein evolution[J]. Crit Rev Biochem Mol Biol, 2007, 42(5): 313-326. DOI:10.1080/10409230701597642 |
| [58] |
GENG H L, MENG X Z, YAN W L, et al. Prevalence of bovine coronavirus in cattle in China: A systematic review and meta-analysis[J]. Microb Pathog, 2023, 176: 106009. DOI:10.1016/j.micpath.2023.106009 |
| [59] |
AMER H M. Bovine-like coronaviruses in domestic and wild ruminants[J]. Anim Health Res Rev, 2018, 19(2): 113-124. DOI:10.1017/S1466252318000117 |
| [60] |
OLVERA-CHÁVEZ A, MEDINA-FIGUEROA A M. Clinical problem solving as a measurement instrument in a diploma course for respiratory technicians[J]. Cir Cir, 2007, 75(4): 271-274. |
| [61] |
SHANGJIN C, CORTEY M, SEGALÉS J. Phylogeny and evolution of the NS1 and VP1/VP2 gene sequences from porcine parvovirus[J]. Virus Res, 2009, 140(1-2): 209-215. DOI:10.1016/j.virusres.2008.11.003 |
| [62] |
ZHU Q H, LI B, SUN D B. Advances in bovine coronavirus epidemiology[J]. Viruses, 2022, 14(5): 1109. DOI:10.3390/v14051109 |
| [63] |
YANG Z H. The power of phylogenetic comparison in revealing protein function[J]. Proc Natl Acad Sci U S A, 2005, 102(9): 3179-3180. DOI:10.1073/pnas.0500371102 |
| [64] |
YOO D, PARKER M D, BABIUK L A. The S2 subunit of the spike glycoprotein of bovine coronavirus mediates membrane fusion in insect cells[J]. Virology, 1991, 180(1): 395-399. DOI:10.1016/0042-6822(91)90045-D |
| [65] |
RAJAH M M, HUBERT M, BISHOP E, et al. SARS-CoV-2 Alpha, Beta, and Delta variants display enhanced Spike-mediated syncytia formation[J]. EMBO J, 2021, 40(24): e108944. DOI:10.15252/embj.2021108944 |
| [66] |
KANNO T, ISHIHARA R, HATAMA S, et al. Antigenic variation among recent Japanese isolates of bovine coronaviruses belonging to phylogenetically distinct genetic groups[J]. Arch Virol, 2013, 158(5): 1047-1053. DOI:10.1007/s00705-012-1587-1 |
| [67] |
GUAN H X, WANG Y W, PERČULIJA V, et al. Cryo-electron microscopy structure of the swine acute diarrhea syndrome coronavirus spike glycoprotein provides insights into evolution of unique coronavirus spike proteins[J]. J Virol, 2020, 94(22): e01301-20. |
| [68] |
QIAN Z H, OU X Y, GÓES L G B, et al. Identification of the receptor-binding domain of the spike glycoprotein of human betacoronavirus HKU1[J]. J Virol, 2015, 89(17): 8816-8827. DOI:10.1128/JVI.03737-14 |
| [69] |
SZCZEPANSKI A, OWCZAREK K, BZOWSKA M, et al. Canine Respiratory coronavirus, bovine coronavirus, and human coronavirus OC43:receptors and attachment factors[J]. Viruses, 2019, 11(4): 328. DOI:10.3390/v11040328 |
| [70] |
MILLET J K, JAIMES J A, WHITTAKER G R. Molecular diversity of coronavirus host cell entry receptors[J]. FEMS Microbiol Rev, 2021, 45(3): fuaa057. DOI:10.1093/femsre/fuaa057 |
(编辑 范子娟)



