2. 中国农业科学院北京畜牧兽医研究所, 北京 100193;
3. 佛山市高明区新广农牧有限公司, 佛山 528000
2. Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193, China;
3. Xinguang Farming & Animal Husbandry Co. in Gaoming District of Foshan City, Foshan 528000, China
随着肉鸡养殖规模化和集约化的发展,商业化肉鸡的选育目标逐渐变为生长周期短、增重快、生产性能显著提高,这给腿部骨骼尚未发育完全的肉鸡带来健康隐患的同时还导致机体代谢紊乱。这些问题使得我国肉鸡腿病发生率显著上升,整体可高达2.07%[1],严重影响了肉鸡养殖的经济效益和动物福利[2-3]。在肉鸡腿病中,跛足病约占60%,已被认为是造成禽肉行业经济损失的主要原因之一[4]。肉鸡骨骼腿病发生原因错综复杂,如生长速度、机体吸收代谢、饲养管理方式、光照时间等均会导致腿病发生的风险。而大多数影响跛足和腿部畸形的非传染性原因与骨代谢紊乱有关, 伴随着骨骼变小、骨密度和骨强度降低[5]。有研究发现,雄性肉鸡比雌性生长速度更快、腿病的发生率高出两倍、行走能力也更差[6],也有研究表明肉鸡腿病可遗传[7-9],其遗传力在0.10~0.40之间[10-12]。因此,通过遗传选择手段能有效降低肉鸡腿病发生率,为肉鸡育种工作打下基础。
随着基因芯片测序技术的日渐成熟,全基因组关联分析(genome-wide association study, GWAS)技术作为一种识别遗传区域和性状间关联的无假设方法,可用于鉴定与复杂疾病或性状相关的单核苷酸多态性(single nucleotide polymorphism,SNP),挖掘与表型性状变异相关的基因,已被广泛使用于人类和畜禽的研究中[13-15]。在人类骨质疏松症的研究中,已经通过GWAS发现了多个与骨骼生物学和代谢相关的基因,其中一些基因位点的突变或多态性变异与骨质疏松症的发生和进展密切相关[16-17];Guo等[18]通过对哈伯德肉鸡样本进行双重基因分型测序(double-digest genotyping by sequencing, ddGBS),鉴定出5个显著单核苷酸多态性(SNPs)位点和70个候选基因作为肉鸡肢体内外翻畸形(valgus-varus deformity, VVD)的潜在标记;Li等[19]将GWAS和选择特征分析(固定指数值和核苷酸多样性比率)相结合,发现与骨性状相关的显著SNPs主要集中在鸡1、4和27号染色体上,确定了21个可能调控鸡骨生长发育的候选基因。肉鸡腿病发生一直以来是家禽生产中的重大难题,因其发生原因极其复杂,目前尚不清楚确切的发生原因和遗传机制。因此,本研究采用我国自主研发的“京芯一号”55K SNP基因芯片,对我国自主培育的“广明2号”白羽肉鸡多个群体共2 331只白羽肉鸡进行基因组测序,通过GWAS技术鉴定与肉鸡腿病发生相关的遗传位点和候选基因,阐明肉鸡腿病发生的机理和遗传基础,为肉鸡腿部疾病的发病机制提供更为科学合理的解释。
1 材料与方法 1.1 试验动物及样品采集本次试验采用我国自主培育的“广明2号”白羽肉鸡A、B、C、D这4个资源群体,各有1 321、711、264、35只不同世代和批次共2 331只公鸡个体,所有肉鸡均在相同环境条件下以双层单笼饲养条件管理,采用自由采食、饮水和一般免疫程序(中国肉鸡饲养标准,NY 33-2004)。在肉鸡自然生长状态下,于42日龄时通过翅间静脉穿刺采集2 mL血液于EDTA抗凝管中,-20 ℃保存用于后续DNA测序。
1.2 表型测定有研究表明,肉鸡胫骨弯曲更容易出现畸形和病变,增加跛足甚至腿病发生的风险[20]。于是本研究通过X光测定肉鸡42日龄时腿部健康状况及骨骼具体形态,将胫骨形变角度≤10°个体作为健康组,定义为0;形变角度>10°个体作为患病组,定义为1,具体测定方法和仪器均参照Zheng等[21]的研究。
1.3 DNA提取与质量检测本研究采用磁珠法提取肉鸡血液基因组DNA。步骤如下:1)使用dsDNA Fragmentase对DNA样品进行酶切,以实现片段化并修复酶切末端。2)通过2%琼脂糖凝胶电泳检测,确认在300~500 bp范围内存在明显的亮带,从而选取效果良好的样品进行后续反应。3)使用连接酶将测序接头与片段化DNA连接,并利用磁珠对连接产物进行纯化。4)运用PCR对连接产物进行扩增,并使用磁珠对PCR产物进行片段筛选。筛选后的片段产物经过Qubit荧光定量仪检测浓度,2%琼脂糖凝胶电泳确认片段大小,并通过Qsep400生物分析仪进一步核准文库片段大小,仅对质控合格的样品进行后续文库制备。
1.4 基因分型和质量控制采用中国农业科学院北京畜牧兽医研究所研发的“京芯一号”55K SNP芯片对2 331只肉鸡质控合格的血液样品进行基因分型[22],由北京康普森生物技术有限公司进行测定。通过plink(Plink v1.90)软件对所有个体分型后得到的基因组数据进行共有位点合并,再严格按照最小等位基因频率≥5%、基因分型率≥90%的位点以及个体分型率≥90%的标准进行质控,剔除不符合要求的个体和位点。在进行全基因组分析之前,位于性染色体(Z染色体和W染色体)中的SNPs被去除。
1.5 群体结构分析和固定效应检验在建立模型之前,使用主成分分析(principal component analysis, PCA)进行群体分层检验,对可解释方差最大的前2个主成分绘制散点图。
将腿部健康表型(即健康或患病)作为因变量,世代、批次作为自变量,使用广义线性回归模型进行固定效应检验,使用二项分布和logit链接函数拟合逻辑回归模型,并使用卡方检验评估世代和批次是否对腿病发生有显著影响。本研究发现,肉鸡的批次和世代与腿病发生有显著的相关性(P<0.05),因此将其作为协变量加入模型中。
1.6 全基因组关联分析参照此前对肉鸡腿部健康表型的分类方式将样本进行分类,采用plink软件中的逻辑回归模型进行GWAS分析。用“—dummy-coding”命令将固定效应转换成0、1格式的二分类变量,将其和前两个主成分作为协变量加入逻辑回归模型中。使用逻辑回归对腿部健康表型进行GWAS分析建立的模型如下:
| $ \begin{gathered} {Logit}(P)=\ln (P /(1-P))= \\ \beta_0+\beta_1 \chi_1, \beta_2 \chi_2 \cdots \cdots, \beta_m \chi_m \end{gathered} $ |
其中,P表示性状发生的概率,取值范围为0到1;χ1, χ2, ……, χm表示输入的m个特征,是模型的自变量;β0,β1,β2,……, βm表示模型参数,需要通过优化算法学习得到合适的数值。
采用plink软件中的参数“-indep-Pairwise 25 5 0.2”推测独立检验的有效SNP数量,以确保SNP单独具有统计显著性且互相独立,从而根据独立的SNP数量调整全基因组显著阈值和全基因组水平潜在显著阈值[23]。计算得到有效独立检验SNPs为7 468个,即GWAS分析的显著性阈值为0.05/7 468=6.70×10-6,潜在显著阈值为1/7 468=1.34×10-4。使用R(V4.2.3)软件包“CMplot”(https://github.com/YinLiLin/CMplot)对GWAS结果进行曼哈顿图和Q-Q图绘制。
1.7 基因注释及其表型解释率为了筛选可能与腿病发生相关的候选基因,选择了显著SNPs基因区段。利用Ensembl基因组数据库(https://asia.ensembl.org/Sus_scrofa/Info/Index)获取SNP的信息,对照Gullas(GRCg6a)参考基因组,并对GWAS所得SNPs进行注释和基因本体(gene ontology,GO)和京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)功能富集分析,以确定蛋白编码基因的重要生物学功能。为衡量所注释到的基因对表型变异的贡献程度,通过计算其表型解释率(phenotypic variation explained,PVE):
| $ P V E=\frac{2 \times { beta }^2 \times { MAF } \times(1- { MAF })}{2 \times { beta }^2 \times { MAF } \times(1- { MAF })+s e^2 \times 2 \times N \times { MAF } \times(1- { MAF })} $ |
beta为基因的效应大小或效应大小的估计值;MAF为最小等位基因频率(minor allele frequency);se为基因效应的标准误差(standard error);N为样本数量。
识别出对特定表型特征具有较高遗传影响的基因,以初步筛选影响肉鸡腿病发生的重要候选基因。
2 结果 2.1 腿部健康表型评价X光测定可以精准评价肉鸡腿部是处于健康状况(图 1a)或病变状态(图 1b),根据腿部骨骼形变程度和特征区分得到2 257只健康和74只腿病个体[24],总体腿病发生率为3.17%。各群体腿病发生具体情况统计如表 1所示,A、B、C、D群体发病率分别为4.09%、0.70%、4.55%和8.57%,D群体由于个体数较少结果不予考虑,A群体和C群体腿病发生率均较高,而B群体腿病发生率较低。

|
a.健康个体(0)腿部骨骼形态X光图像;b.腿病个体(1)腿部骨骼形态X光图像 a. X-ray images of leg bone morphology in healthy individuals (0); b. X-ray images of leg bone morphology in leg disease individuals (1) 图 1 X光测定评价肉鸡腿部健康情况 Fig. 1 X-ray assessment to evaluate the health condition of broiler legs |
|
|
表 1 试验群体肉鸡腿病发生情况 Table 1 Leg disease of broiler in experimental group |
利用“京芯一号”55K SNP芯片对2 331只“广明2号”白羽肉鸡进行基因分型,基于最小等位基因频率≥5%、基因分型率≥90%的位点和个体分型率≥90%的标准对基因型数据进行质量控制,保留了2 330只肉鸡个体和30 414个有效SNPs位点用于后续分析, 各染色体有效位点密度图见图 2。其中1号染色体上位点数量最多,有6 601个;16号染色体上位点数量最少,有134个。

|
图 2 基因分型质控后染色体密度图 Fig. 2 Chromosome density map after genotyping quality control |
使用plink软件对A、B、C、D群体进行主成分分析,结果(图 3)显示这4个群体有明显的结构分层,说明这4个白羽肉鸡群体在人工选育过程中群体结构产生差异,且PC1和PC2分别可解释方差的比例为28.4%和6.20%。因此,在后续全基因组关联分析中将前2个主成分作为协变量加入模型中,以校正群体结构,降低群体分层对模型稳定性的影响。
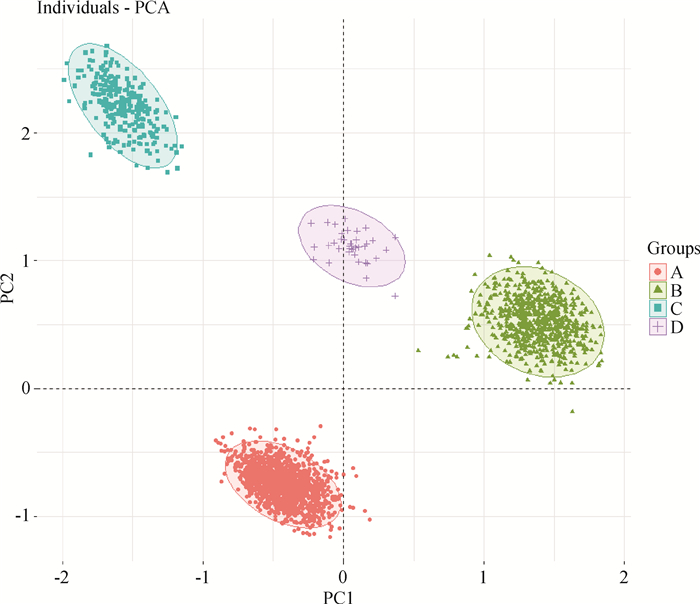
|
图中的每个点对应一个家庭中的个体,红色、绿色、蓝色和紫色的圆点分别代表来自A、B、C、D群体的个体,代表 4个群体 Each dot in the figure corresponds to an individual in a family, and the red, green, blue, and purple dots represent individuals from group A, B, C, and D, respectively, representing the 4 groups 图 3 A、B、C、D群体结构主成分分析 Fig. 3 Principal component analysis of the structure of A, B, C and D groups |
本研究基于肉鸡腿部健康表型,将群体分为对照和患病群体进行GWAS分析,以获得与腿病相关的位点和候选基因,结果用曼哈顿图(图 4a)和QQ图表示(图 4b)。QQ图膨胀因子为1.018,表示分析结果的假阳性较低。曼哈顿图显示没有与腿病发生相关的显著SNP位点,在潜在显著水平上共检测到5个SNPs位点(P < 9.90×10-5),分别位于6号染色体(rs7317511、rs7963346、rs11152911、rs11277299)和18号染色体(rs3239706)。在6号染色体上,最显著的SNP是rs11277299(P=1.23×10-5)。

|
a.曼哈顿图:水平黑色虚线表示全基因组潜在阈值线(P=1.34×10-4),黑色实线表示全基因组显著阈值线(P=6.70×10-6);b.Q-Q图 a. Manhattan plot: Horizontal black dashed line indicates the genome-wide potential threshold line (P=1.34×10-4), and the black solid line indicates the genome-wide significant threshold line (P=6.70×10-6); b; Q-Q plot 图 4 全基因组水平上腿部健康表型性状的曼哈顿图和Q-Q图 Fig. 4 Manhattan and Q-Q plots of leg health phenotypic traits at the genome-wide level |
所鉴定到的显著SNP位点解释的表型变异解释(phenotypic variation explanation, PVE)范围为0.65%~0.81%,这5个位点PVE值较其他位点高,说明对表型的贡献较大。rs11277299位点的表型变异率最高,为0.81%,认为这几个SNPs位点可能是重要的遗传位点,在肉鸡腿部健康发生发展调控中发挥重要作用。对上述5个显著SNPs位点进行注释,共得到TBCD、SIRT1和PBLD这3个候选基因(表 2),通过计算各SNP位点在健康组(0)和患病组(1)间的比例,发现SIRT1基因中携带GG基因型的个体、PBLD基因中携带AA和GA基因型的个体、TBCD基因中携带CC基因型的个体患病比例较大,均有可能增加肉鸡患腿病的风险(图 5)。
|
|
表 2 白羽肉鸡腿部健康性状显著相关的SNPs及其候选基因 Table 2 SNPs and their candidate genes significantly associated with leg health traits in white feather broilers |

|
a, b, c分别为显著SNPs rs7317511、rs11277299和rs3239706的基因型频率在健康组(0)和腿病组(1)间的比例,即不同等位基因对腿部健康性状的影响 a, b, c are the ratios of genotype frequencies of significant SNPs rs7317511, rs11277299, and rs3239706, respectively, between the healthy group (0) and the leg disease group (1), i.e., the effect of different alleles on leg health traits 图 5 注释到基因的显著SNPs在健康和腿病组间的基因型比例 Fig. 5 Genotype proportions of significant SNPs annotated to genes between healthy and leg disease groups |
对腿病表型注释到的TBCD、SIRT1和PBLD这3个候选基因通过KOBAS网站(http://kobas.cbi.pku.edu.cn)进行GO和KEGG功能富集分析,结果显示共有82个GO项达到了具有统计学意义的水平(P<0.05),这些GO条目主要涉及细胞分化、细胞生物合成、细胞因子结合和信号通路负调控等过程,转化生长因子受体信号通路的负向调控GO条目(GO: 0030512)是GO富集分析结果中最显著的GO条目,其中细胞质过程(GO: 0005737)在这3个基因中均显著富集到(P<0.05)(图 6)。KEGG分析结果表明,仅有SIRT1基因富集到FoxO信号通路(gga04068)和细胞衰老信号通路(gga04218)并达到显著水平(P<0.05),可能在腿部疾病和骨骼发育中起重要作用。

|
图 6 候选基因GO富集分析 Fig. 6 GO enrichment analysis of candidate genes |
随着对现代商业化肉鸡增重和生产性能的不断选育,骨骼生长发育不足以支撑肉鸡快速生长的体重导致多种类型腿病发生,腿病发生已引起养殖产业极大关注。本研究采用我国自主培育的“广明2号”白羽肉鸡,通过X光方法对已在人工选育过程中群体结构产生差异的A、B、C、D四个群体测定腿部骨骼健康状况,对实践应用有重要的指导意义。腿病个体在品系培育过程中经历了严格淘汰,可能导致已上市的配套系中相应性状遗传力降低,而本研究中特定的通过X光方法定义表型也可能对遗传力估计造成影响。需要继续探究腿病个体淘汰对整个品系性状的影响且分析造成遗传力较低的原因,进一步完善通过X光方法评价肉鸡腿病发生的状况,为改进肉鸡品系培育和预防腿病发生提供借鉴。
本研究基于多个品系、批次和世代群体腿病发病率的统计,发现腿病发生率总体高达3.17%,A、C群体发病率分别高达4.09%、4.55%,这极度加大了家禽生产中的死淘率,造成了严重的经济损失,但群体内世代和批次间的腿病发生率无显著差异。而B群体腿病发生率较低,可进一步探究B群体腿部健康状况遗传机制,从遗传选育角度入手改良腿部健康性状。肉鸡腿病发生受遗传、营养、饲养管理、环境等多方面的影响,其中定位其主效遗传因和位点是作为突破口进行遗传改良以改善肉鸡腿部疾病的关键[25]。
全基因组关联分析(GWAS)作为揭示基因与疾病之间关联性的有力研究方法,能为疾病的机制研究和治疗提供新的方向和策略,已被广泛应用于挖掘复杂疾病的易感基因[26-27]。在畜禽生产中,GWAS方法已被广泛应用于经济性状的研究中,如体重、产蛋性状、免疫功能和骨骼等[28]。通过全基因组关联研究和基因组选择等手段能迅速加快腿部健康的遗传改良进程,然而目前关于肉鸡腿部健康表型相关遗传变异位点的研究仍然十分缺乏,没有大样本用于鉴定与腿病相关的基因位点和候选基因。因此,本研究基于白羽肉鸡腿部疾病发生的病例和对照样本,将2 257例健康和74例腿病发生白羽肉鸡通过“京芯一号”55K SNP芯片基因分型测序方法进行全基因组关联分析,以期鉴定出与白羽肉鸡腿部健康相关的候选基因和变异位点,为肉鸡腿部健康遗传改良提供参考。
本研究通过全基因组关联分析定位到5个潜在SNPs位点与腿病发生显著相关,其中4个位点位于6号染色体,1个位点位于18号染色体。PVE值可以作为评估基因位点对表型变异解释能力的指标,本研究发现在肉鸡腿部健康表型变异中,位点rs7317511、rs7963346、rs11152911、rs11277299和rs3239706的基因突变或多态性分别解释了0.65%、0.66%、0.72%、0.81%和0.72%表型的变异,这些位点较其他位点PVE值大,一定程度上能作为评估肉鸡腿部健康表型的变异位点的指标。其中,rs7317511注释得到SIRT1基因,rs3239706注释得到TBCD基因,rs11277299注释得到PBLD基因,这3个已知候选基因相对来说对腿部健康表型具有较为重要的潜在意义。
骨骼作为鸡的一个重要性状,它的质量与肉鸡的产肉量和骨质疏松症有关。有研究采用高密度基因分型平台检测骨骼性状的候选基因,发现位于不同染色体上的3个基因位点包含候选基因HTR2A、LPAR6、CAB39L、TRPC4、WNT9A、SPOP、NGFR、GIP和HOXB3与骨骼质量和长度有关[29];Zhang等[30]对鸡龙骨表型差异进行选择消除分析,发现了10个具有强选择信号且与骨骼相关的重要候选基因并富集到了MAPK和破骨细胞分化两条重要的骨信号通路。重要的是,基于本研究注释到的基因SIRT1、TBCD和PBLD具体功能及其如何参与机体骨骼代谢过程也已有多项研究。鸡SIRT1基因cDNA全长约2 271 bp,位于6号染色体,可编码756个氨基酸[31],它参与调节肿瘤、氧化应激、凋亡、衰老和炎症等一系列细胞代谢过程[32-33]。有研究发现,SIRT1敲除小鼠体内骨量减少,成骨细胞与破骨细胞比例失衡,这表明该基因是通过直接作用于骨细胞来调控骨代谢过程[34],它的表达水平与骨矿物质密度和骨脆性有关,其可能成为系统性骨代谢和骨相关疾病的潜在生物标志物[35],还能介导FoxO(gga04068)和细胞衰老(gga04218)等重要信号通路。FoxO是一类转录因子,参与调控多种细胞过程,而FoxO信号通路通过参与调控骨母细胞的增殖分化和吸收等过程影响骨细胞的功能和骨骼组织的形成[36-37]。细胞衰老信号通路主要包括端粒缩短、DNA损伤响应、染色质重塑、细胞周期调控和细胞死亡等多个分子机制。有研究发现了细胞衰老信号通路与骨骼生长发育过程的关联,即端粒缩短和端粒酶的活性与骨质疏松和骨折风险增加相关[38],但目前对于细胞衰老信号通路在骨骼生长发育中的具体功能还需要进一步研究证实。关于SIRT1基因对骨骼代谢和生长发育的调控机制和互作网络在未来的研究中值得进一步关注,特别是GG基因型的肉鸡。PBLD通过抑制NF-κB和上皮到间充质转化信号通路而发挥肿瘤抑制作用,控制炎症的关键调节,对骨关节炎等疾病都有着重要影响[39];TBCD主要参与微管相关的生物学过程,如细胞分裂、细胞轴向极化和细胞运输等[40],尚未发现该基因与骨骼之间的联系,但微管在骨骼生长和骨骼细胞的某些功能中也发挥着重要作用[41]。关于这3个基因如何具体影响骨骼生长发育的生理过程及其分子遗传机理,以及携带不同基因型的肉鸡腿部健康是否会有差异,还亟待进一步的理论和试验研究进行探索。
本研究还发现了特定的潜在SNP位点rs7963346,其位置被定位到了lncRNA上,这个发现提供了进一步研究该SNP位点对腿病发生影响的线索。lncRNA是一类长链非编码RNA,它的功能相对较为复杂和多样,可以调控基因表达、细胞生物学过程等。可通过进一步试验深入研究该位点所在的lncRNA的功能,还可通过构建SNP-lncRNA-disease网络来探究该位点与腿病之间的关系,从而揭示其在腿部疾病发生发展机制中的作用,为肉鸡腿病的预防、诊断和治疗提供重要的理论依据。大多数疾病发生发展都是极其复杂的,受多种环境条件和基因控制。本研究在全基因组水平上发现多个显著与肉鸡腿病发生相关的位点,但这些位点未定位到相似的区域和基因。骨骼生长发育及病变过程受遗传和环境因素共同影响调控,基因组的复杂性和多样性决定了不同环境背景下基因的功能可能会呈现不同的效应。推测肉鸡腿病作为复杂性状,受到了多种效应的影响,进一步的功能注释、环境相关研究以及更大规模的验证研究将有助于深入理解这些位点在肉鸡腿部健康中的潜在作用和复杂关系。SIRT1、PBLD和TBCD均是可能与腿病发生相关的候选基因,有望深入研究这些基因的潜在生物学意义,并为肉鸡腿部健康研究提供新的见解。
4 结论本研究基于多个品系及世代的2 331只白羽肉鸡个体,通过大量样本数据基于55K SNP基因芯片利用GWAS方法研究白羽肉鸡腿部健康的遗传机制,挖掘与腿病发生相关的重要遗传信息。研究发现6号染色体(rs7317511、rs7963346、rs11152911、rs11277299)和18号染色体(rs3239706)上的5个SNPs位点可能是影响肉鸡腿病发生的变异位点,SIRT1、PBLD和TBCD可能是影响白羽肉鸡腿病发生的重要候选基因。本试验为研究肉鸡腿病的发生机制和降低腿病发生率提供了重要遗传参考。
| [1] |
李鹏飞, 姜敏, 史超颖, 等. 规模化养殖场肉鸡腿病情况调查[J]. 畜牧与兽医, 2016, 48(6): 116-118. LI P F, JIANG M, SHI C Y, et al. An investigation of broiler leg disorders in large-scale farms[J]. Animal Husbandry & Veterinary Medicine, 2016, 48(6): 116-118. (in Chinese) |
| [2] |
李岩. 肉鸡发生腿病的原因及防治[J]. 畜牧兽医科技信息, 2019(2): 127. LI Y. Causes and prevention of leg disease in broilers[J]. Animal Husbandry and Veterinary Science and Technology Information, 2019(2): 127. DOI:10.3969/J.ISSN.1671-6027.2019.02.115 (in Chinese) |
| [3] |
顾敏清. 商品代肉鸡的腿部健康[J]. 山东家禽, 2004(9): 29-32. GU M Q. Leg health of commercial broilers[J]. Shandong Poultry, 2004(9): 29-32. DOI:10.3969/j.issn.1673-1085.2004.09.014 (in Chinese) |
| [4] |
JULIAN R J. Production and growth related disorders and other metabolic diseases of poultry-A review[J]. Vet J, 2005, 169(3): 350-369. DOI:10.1016/j.tvjl.2004.04.015 |
| [5] |
HUANG S C, ZHANG L H, ZHANG J L, et al. Role and regulation of growth plate vascularization during coupling with osteogenesis in tibial dyschondroplasia of chickens[J]. Sci Rep, 2018, 8(1): 3680. DOI:10.1038/s41598-018-22109-y |
| [6] |
LOUTON H. Exploring the association between slight gait defects and sex, body morphology and leg pathology in broilers[J]. Vet Rec, 2021, 189(7): 284-285. DOI:10.1002/vetr.1039 |
| [7] |
KAPELL D N R G, HILL W G, NEETESON A M, et al. Genetic parameters of foot-pad dermatitis and body weight in purebred broiler lines in 2 contrasting environments[J]. Poult Sci, 2012, 91(3): 565-574. DOI:10.3382/ps.2011-01934 |
| [8] |
KAPELL D N R G, HILL W G, NEETESON A M, et al. Twenty-five years of selection for improved leg health in purebred broiler lines and underlying genetic parameters[J]. Poult Sci, 2012, 91(12): 3032-3043. DOI:10.3382/ps.2012-02578 |
| [9] |
KUHLERS D L, MCDANIEL G R. Estimates of heritabilities and genetic correlations between tibial dyschondroplasia expression and body weight at two ages in broilers[J]. Poult Sci, 1996, 75(8): 959-961. DOI:10.3382/ps.0750959 |
| [10] |
GONZÁLEZ-CERÓN F, REKAYA R, ANTHONY N B, et al. Genetic analysis of leg problems and growth in a random mating broiler population[J]. Poult Sci, 2015, 94(2): 162-168. DOI:10.3382/ps/peu052 |
| [11] |
MOGHADAM H K, MCMILLAN I, CHAMBERS J R, et al. Estimation of genetic parameters for ascites syndrome in broiler chickens[J]. Poult Sci, 2001, 80(7): 844-848. DOI:10.1093/ps/80.7.844 |
| [12] |
郑炬梅, 刘大伟, 唐鑫鑫, 等. 利用X光检测白羽肉鸡腿部健康表型分类及遗传力估计[J]. 中国家禽, 2023, 1-5. ZHENG J M, LIU D W, TANG X X, et al. Classification of leg health traits and estimation of heritability in white feather broiler by X-ray detection[J]. China Poultry, 2023, 1-5. (in Chinese) |
| [13] |
EINARSDOTTIR E, HAFRÉN L, LEINONEN E, et al. Genome-wide association analysis reveals variants on chromosome 19 that contribute to childhood risk of chronic otitis media with effusion[J]. Sci Rep, 2016, 6(1): 33240. DOI:10.1038/srep33240 |
| [14] |
王晓薇, 樊毅, 杨阳, 等. 肉牛生长性状和肉质性状全基因组关联分析的研究进展[J]. 现代畜牧兽医, 2022(1): 78-83. WANG X W, FAN Y, YANG Y, et al. Progress in genome-wide association studies for growth traits and meat quality traits in beef cattle[J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2022(1): 78-83. (in Chinese) |
| [15] |
吴骏, 蔡晓钿, 林清, 等. 大白猪眼肌面积、估计瘦肉率和背膘厚的加权一步法全基因组关联分析[J]. 畜牧兽医学报, 2023, 54(4): 1403-1414. WU J, CAI X D, LIN Q, et al. Weighted single-step GWAS of eye muscle area, predicted lean meat percentage and average backfat thickness in a Yorkshire pig population[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(4): 1403-1414. DOI:10.11843/j.issn.0366-6964.2023.04.006 (in Chinese) |
| [16] |
KIEL D P, DEMISSIE S, DUPUIS J, et al. Genome-wide association with bone mass and geometry in the Framingham Heart Study[J]. BMC Med Genet, 2007, 8(Suppl 1): S14. DOI:10.1186/1471-2350-8-S1-S14 |
| [17] |
CHEN Y C, GUO Y F, HE H, et al. Integrative analysis of genomics and transcriptome data to identify potential functional genes of BMDs in females[J]. J Bone Miner Res, 2016, 31(5): 1041-1049. DOI:10.1002/jbmr.2781 |
| [18] |
GUO Y P, HUANG H T, ZHANG Z Z, et al. Genome-wide association study identifies SNPs for growth performance and serum indicators in Valgus-varus deformity broilers (Gallus gallus) using ddGBS sequencing[J]. BMC Genomics, 2022, 23(1): 26. DOI:10.1186/s12864-021-08236-3 |
| [19] |
LI Y D, LIU X, LI Z W, et al. A combination of genome-wide association study and selection signature analysis dissects the genetic architecture underlying bone traits in chickens[J]. Animal, 2021, 15(8): 100322. DOI:10.1016/j.animal.2021.100322 |
| [20] |
PULCINI D, MEO ZILIO D, CENCI F, et al. Differences in tibia shape in organically reared chicken lines measured by means of geometric morphometrics[J]. Animals (Basel), 2021, 11(1): 101. |
| [21] |
ZHENG J, LIU D, ZHANG G, et al. Improve the accuracy of chicken leg disease assessment by combination of gait score and X-ray scan[J]. J Appl Poultry Res, 2023, 32(2): 100339. DOI:10.1016/j.japr.2023.100339 |
| [22] |
刘冉冉, 赵桂苹, 文杰. 鸡基因组育种和保种用SNP芯片研发及应用[J]. 中国家禽, 2018, 40(15): 1-6. LIU R R, ZHAO G P, WEN J. Development of genome-wide SNP genotyping arrays for chicken breeding and conservation[J]. China Poultry, 2018, 40(15): 1-6. (in Chinese) |
| [23] |
LIU R R, XING S Y, WANG J, et al. A new chicken 55K SNP genotyping array[J]. BMC Genomics, 2019, 20(1): 410. DOI:10.1186/s12864-019-5736-8 |
| [24] |
SHIM M Y, KARNUAH A B, ANTHONY N B, et al. The effects of broiler chicken growth rate on valgus, varus, and tibial dyschondroplasia[J]. Poult Sci, 2012, 91(1): 62-65. DOI:10.3382/ps.2011-01599 |
| [25] |
GUO J, SUN C J, QU L, et al. Genetic architecture of bone quality variation in layer chickens revealed by a genome-wide association study[J]. Sci Rep, 2017, 7(1): 45317. DOI:10.1038/srep45317 |
| [26] |
MIR Z A, CHANDRA T, SAHARAN A, et al. Recent advances on genome-wide association studies (GWAS) and genomic selection (GS); prospects for Fusarium head blight research in Durum wheat[J]. Mol Biol Rep, 2023, 50(4): 3885-3901. DOI:10.1007/s11033-023-08309-4 |
| [27] |
GUNDERSON K L, STEEMERS F J, LEE G, et al. A genome-wide scalable SNP genotyping assay using microarray technology[J]. Nat Genet, 2005, 37(5): 549-554. DOI:10.1038/ng1547 |
| [28] |
杨梦园, 宁中华. 鸡重要性状GWAS分析的研究进展[J]. 中国家禽, 2023, 45(4): 105-110. YANG M Y, NING Z H. Research progress on GWAS analysis of important traits in chicken[J]. China Poultry, 2023, 45(4): 105-110. (in Chinese) |
| [29] |
GUO J, QU L, DOU T C, et al. Genome-wide association study provides insights into the genetic architecture of bone size and mass in chickens[J]. Genome, 2020, 63(3): 133-143. DOI:10.1139/gen-2019-0022 |
| [30] |
ZHANG Z H, YANG W F, ZHU T, et al. Genetic parameter estimation and whole sequencing analysis of the genetic architecture of chicken keel bending[J]. Front Genet, 2022, 13: 833132. DOI:10.3389/fgene.2022.833132 |
| [31] |
刘三凤, 任军, 郭源梅, 等. 鸡SCD和Sirt1基因的SNP搜寻及其遗传多样性分析[J]. 畜牧兽医学报, 2008, 39(3): 278-285. LIU S F, REN J, GUO Y M, et al. Identification of single nucleotide polymorphisms of SCD and Sirt1 and evaluation of their genetic diversities in chickens[J]. Acta Veterinaria et Zootechnica Sinica, 2008, 39(3): 278-285. (in Chinese) |
| [32] |
KARBASFOROOSHAN H, ROOHBAKHSH A, KARIMI G. SIRT1 and microRNAs: the role in breast, lung and prostate cancers[J]. Exp Cell Res, 2018, 367(1): 1-6. |
| [33] |
ZHANG W J, HUANG Q B, ZENG Z H, et al. Sirt1 inhibits oxidative stress in vascular endothelial cells[J]. Oxid Med Cell Longev, 2017, 2017: 7543973. |
| [34] |
ZAINABADI K, LIU C J, CALDWELL A L M, et al. SIRT1 is a positive regulator of in vivo bone mass and a therapeutic target for osteoporosis[J]. PLoS One, 2017, 12(9): e0185236. |
| [35] |
CHEN Y, ZHOU F, LIU H H, et al. SIRT1, a promising regulator of bone homeostasis[J]. Life Sci, 2021, 269: 119041. |
| [36] |
JIANG Y X, LUO W Q, WANG B, et al. Resveratrol promotes osteogenesis via activating SIRT1/FoxO1 pathway in osteoporosis mice[J]. Life Sci, 2020, 246: 117422. |
| [37] |
HWANG J W, YAO H W, CAITO S, et al. Redox regulation of SIRT1 in inflammation and cellular senescence[J]. Free Radic Biol Med, 2013, 61: 95-110. |
| [38] |
HU H, GUO X D, MU T T, et al. Long non-coding RNA telomerase RNA elements improve glucocorticoid-induced osteoporosis by EZH2 to regulate DKK1[J]. Int J Rheum Dis, 2023, 26(4): 638-647. |
| [39] |
LI A M, YAN Q, ZHAO X M, et al. Decreased expression of PBLD correlates with poor prognosis and functions as a tumor suppressor in human hepatocellular carcinoma[J]. Oncotarget, 2016, 7(1): 524-537. |
| [40] |
EDVARDSON S, TIAN G L, CULLEN H, et al. Infantile neurodegenerative disorder associated with mutations in TBCD, an essential gene in the tubulin heterodimer assembly pathway[J]. Hum Mol Genet, 2016, 25(21): 4635-4648. |
| [41] |
KIM P, PARK J, LEE D J, et al. Mast4 determines the cell fate of MSCs for bone and cartilage development[J]. Nat Commun, 2022, 13(1): 3960. |
(编辑 郭云雁)



