2. 安徽省动物疫病预防与控制中心, 合肥 230091;
3. 安徽省农业科学院畜牧兽医研究所, 合肥 230031
2. Anhui Animal Disease Prevention and Control Center, Hefei 230091, China;
3. Institute of Animal Husbandry and Veterinary Medicine, Anhui Academy of Agricultural Sciences, Hefei 230031, China
猪流行性腹泻(porcine epidemic diarrhea, PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起的接触性传染病[1]。PED于1971年首次在英国报道,随后在整个欧洲流行[2]。2010年前,PED在我国区域性流行,但在2010年10月以后,PED在我国南方开始大规模流行,并很快在全国各地流行,严重损害了我国的养猪业健康发展[3-4]。
PEDV属于冠状病毒属成员,为单链正义RNA,基因组全长约为28 kb,包含5′、3′端非编码区、ORF1a、ORF1b、ORF2、ORF3、ORF4、ORF5、ORF6等非结构蛋白及S、M、E和N等结构蛋白[5]。其中,N基因在不同的PEDV毒株之间高度保守,可以作为PEDV感染诊断的靶标[4]。
目前,通常采用病毒分离、定量实时PCR、环介导等温扩增和ELISA等方法来检测猪是否感染PEDV[6-7],但这些方法操作相对复杂,工作量较大。因此,需要一种新的技术方案来解决上述问题。研究发现,CRISPR/Cas13a在特异性crRNA的引导下切割目标RNA后,其“附属切割”活性被激活,可高效切割体系中非特异单链RNA[8-9]。基于这一原理,通过设计两端标记荧光基团的RNA探针,可使CRISPR/Cas13对RNA模板的检测和信号放大,实现对目标分子的特异性检测。重组酶辅助扩增(recombinase aided amplification, RAA) 技术是一种恒温快速扩增核酸的技术。该技术在37~42 ℃等温条件下可短时间内对模板进行指数级扩增,但常规RAA需要琼脂糖凝胶电泳,结果判读复杂[10]。基于CRISPR/cas13a技术的检测方法已被应用于检测猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus, PRRSV)、禽流感病毒(avian influenza virus, AIV)、埃博拉病毒(Ebola virus, EBOV)及登革热病毒(dengu evirus, DENV)等病毒[11-13]。因此,本研究将CRISPR/Cas13a与RAA技术相结合,针对PEDV N基因设计特异性引物和探针,建立操作简单,特异性强,灵敏度高的PEDV检测方法,以期为PED的防控提供技术支持。
1 材料与方法 1.1 毒株与临床样本猪圆环病毒1型(porcine circovirus type 1, PCV1)、猪圆环病毒2型(porcine circovirus type 2, PCV2)、猪圆环病毒3型(porcine circovirus type 3, PCV3)、猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus, PRRSV)、猪瘟病毒(classical swine fever virus, CSFV)及伪狂犬病病毒(pseudorabies virus,PRV)等阳性病料均由作者所在实验室保存。40份腹泻哺乳仔猪肠道和肛门拭子样品由第三方检测机构收集并提供。
1.2 主要试剂病毒基因组DNA/RNA提取试剂盒购自天根生化科技(北京)有限公司;LwCas13a和RAA检测试剂盒购自安徽微分基因科技有限公司;HiScribe T7 Quick High Yield RNA合成试剂盒、T7 RNA聚合酶及RNase酶抑制剂购自NEB公司;NucawayTM离心柱购自Invitrogen公司;RNAXP磁珠购自南京诺唯赞生物科技股份有限公司。
1.3 引物设计与合成N基因长度为1 326 bp,其在不同的PEDV毒株之间高度保守,根据GenBank公布的PEDV毒株N基因序列(登录号:KU646831、JN173276、MK584552、KM089829、MZ364314、JX647847、ON571549、MN759311、ON649885、MH726391),参照RAA引物设计原则,设计N基因特异性RAA引物见表 1(单下划线为T7启动子序列)。此外,根据Cas13a蛋白crRNA识别靶序列的特性,在PEDV的N基因序列上设计3对特异性的crRNA探针(PEDV-crRNA1-3),引物序列如表 1所示(双下划线为茎环结构序列)。引物由生工生物工程(上海)股份有限公司合成。
|
|
表 1 PEDV crRNA引物序列 Table 1 PEDV crRNA primer sequences |
根据GenBank公布的PEDV AH2012/12株(登录号:KU646831),由生工生物工程(上海)股份有限公司将N基因合成,并连接至载体pUC57,重组质粒分别命名为pUC-N,测序正确的重组质粒即为质粒标准品。将“1.3”中合成的两条PEDV-crRNA1-3的上、下游引物退火形成双链(终浓度为10 μmol ·L-1),随后利用HiScribe T7 Quick High Yield RNA合成试剂盒进行体外转录,转录产物利用RNAXP磁珠进行纯化,-80 ℃保存备用。
1.5 RAA扩增参照RAA核酸检测试剂盒说明书配制反应体系(50 μL):25 μL预混液,3 μL MgOAc,RAA上、下游引物(10 μmol ·L-1)各2 μL,1 μL待检样本cDNA或DNA,用RNase free H2O补至50 μL,将反应体系震荡离心,37 ℃孵育30 min,制备RAA产物。
1.6 CRISPR/Cas13a检测利用LwCas13a核酸酶(45 nmol ·L-1),crRNA探针(22.5 nmol ·L-1),FT-RNA报告分子(125 nmol ·L-1),RNase inhibitor(0.25 μL),dNTPs(1 mmol ·L-1)和T7聚合酶(0.4 μL),检测缓冲液补充至9 μL,RAA扩增产物1 μL,在QuantStudioTM 6 Flex实时荧光定量PCR 37 ℃反应40 min,每间隔5 min收集荧光信号。
1.7 crRNA的筛选以pUC-N为模板,采用“1.5”和“1.6”中的检测方法,分别对3组crRNA进行筛选,通过扩增曲线的荧光值及起峰时间,选择最佳检测PEDV的crRNA探针。
1.8 灵敏度试验将pUC-N标准质粒稀释为1.0×108 ~ 1.0×100 copies ·μL-1等9个浓度。用已建立的RAA-Cas13a检测方法,以稀释后的各浓度阳性质粒为模板,通过扩增曲线确定该方法的灵敏度,同时设置RNase free H2O作为阴性对照,每组进行3次重复。
1.9 特异性试验选取PCV1、PCV2、PCV3、PRRSV、CSFV和PRV等病毒作为特异性检测对象,参照病毒基因组DNA/RNA提取试剂盒说明书对上述病毒进行核酸提取。用已建立的RAA-Cas13a方法进行检测,同时设置阴性对照,分析该检测方法的特异性。
1.10 临床样本检测对40份临床腹泻哺乳仔猪肠道和肛门拭子样本进行检测,采用本研究建立的RAA-Cas13a检测方法和RT-qPCR检测方法[14]进行PEDV检测,并使用SPSS软件进行Kappa一致性检验,以评估该方法的临床实用性。
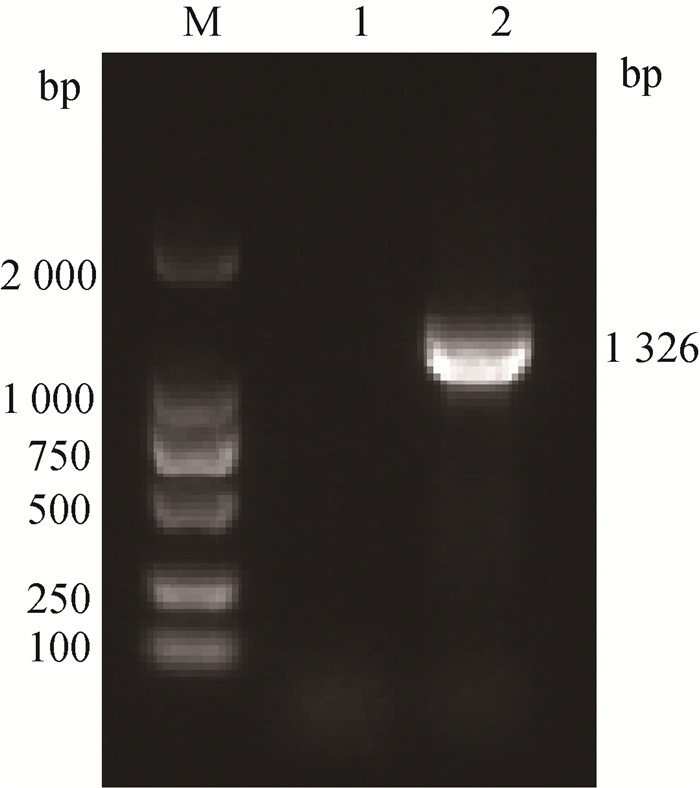
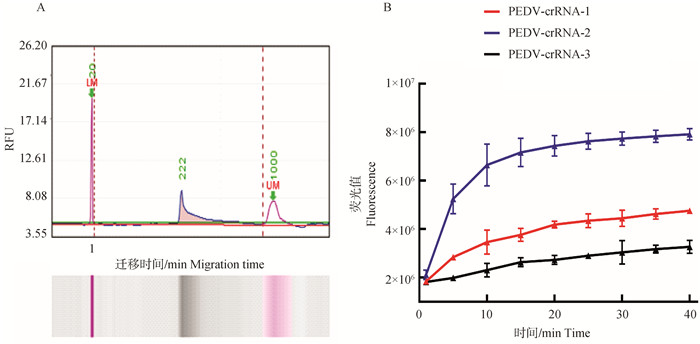
2 结果 2.1 crRNA的筛选将合成的N基因进行PCR验证,结果显示, 在目标大小处有特异性条带,经测序证实为PEDV N基因(图 1)。此外,由图 2A可知,RAA引物扩增产物检测峰图在222 bp处有单峰,表明引物特异性良好。通过建立的RAA-Cas13a方法比较和筛选已制备的3组PEDV-crRNA,结果如图 2B所示crRNA-2与其他2组相比,荧光值最高且起峰时间最短。因此,选择PEDV-crRNA-2用于后续试验。
|
图 1 pUC-N质粒PCR扩增结果 Fig. 1 The results of PCR amplification of pUC-N plasmid |
|
A. RAA引物扩增产物检测峰图;B. crRNA筛选结果 A. RAA primer amplification product detection peak; B. The screening result of crRNA 图 2 RAA引物验证及crRNA筛选 Fig. 2 The results of validation of RAA primers and crRNA screening |
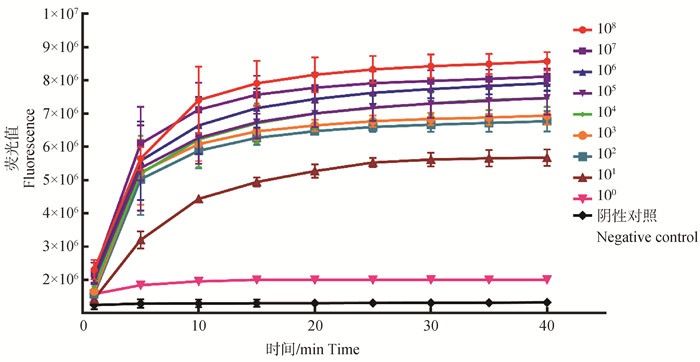
RAA-Cas13a检测方法的灵敏度评价以9个梯度稀释(1.0×108 ~ 1.0×100 copies ·μL-1) 的标准质粒及RNase free H2O作为模板进行RAA-Cas13a方法的灵敏度评价。结果如图 3所示,在反应5 min时即可检测到1.0×101 copies ·μL-1的信号,表明本研究建立的RAA-Cas13a检测方法的检测限为101 copies ·μL-1。
|
图 3 RAA-Cas13a灵敏度试验结果 Fig. 3 Sensitivity detection results of RAA-Cas13a |
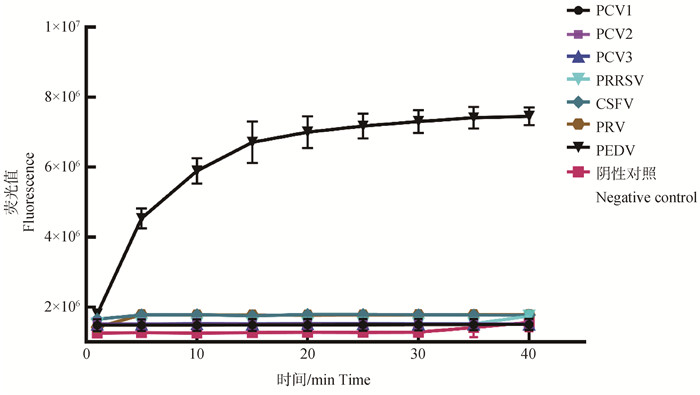
RAA-Cas13a的分析特异性试验检测结果如图 4所示,仅PEDV出现扩增曲线,PCV1、PCV2、PCV3、PRRSV、CSFV、PRV及阴性对照均无检测信号,表明本研究建立的方法能特异性扩增检测出PEDV,而不与其他病毒核酸发生交叉反应,说明本检测方法特异性强。
|
图 4 RAA-Cas13a特异性试验结果 Fig. 4 Specificity detection results of RAA-Cas13a |
RAA-Cas13a分别应用本试验建立的RAA-Cas13a方法和RT-qPCR方法,对临床上采集的40份样品进行检测。结果显示,RAA-Cas13a的阳性率为72.5%(29/40),RT-qPCR方法的阳性率为67.5%(27/40),阳性符合率为100%,阴性符合率为84.6%(表 2),总一致性为95%,Kappa=0.881(Kappa≥0.75,表示可重复性极好)。
|
|
表 2 RAA-Cas13a和RT-qPCR方法对临床样本中的PEDV检测结果 Table 2 Detection results of clinical samples in RAA-Cas13a and RT-qPCR assays |
PED是一种高度传染性的传染病,可引起哺乳仔猪的腹泻、呕吐和脱水等临床症状,有高致死率,给我国养猪业带来严重的经济损失[3, 15-16]。目前,PEDV的疫苗保护效果较差,甚至注射过疫苗都能感染。因此,建立高效、灵敏且特异性强的PEDV检测方法,对PEDV的流行病学调查和早期诊断具有重要意义。PEDV的实验室检测方法很多,但大多数检测方法需要专业操作或特殊设备,需要在专业实验室进行。CRISPR/Cas13a系统可以特异性切割目标RNA,且具有非特异性附带切割活性,基于该原理可在反应体系中加入非特异RNA报告探针,实现对靶基因的特异检测,这为CRISPR/Cas13a系统在核酸检测领域的应用提供了新思路[8, 17]。此外,RAA技术引物设计简单,扩增效率高,因此,本研究将RAA技术与CRISPR/Cas13a系统结合,实现了两种技术优势互补,提高检测系统性,增强特异性。
本研究建立的RAA-Cas13a检测方法的检测限达到101copies ·μL-1。Wang等[18]建立的实时荧光定量PCR方法仅能检测出300 copies ·μL-1的标准质粒;Liu等[6]开发的基于ORF3基因建立的TaqMan探针的荧光定量PCR的检测限为37 copies ·μL-1;翟刚等[19]基于M基因建立的TaqMan检测方法最低检测下限为1.09×101 copies ·μL-1;冉伟等[20]建立SYBR Green Ⅰ荧光定量PCR方法,最低可检测102 copies ·μL-1的质粒。本研究建立的RAA-Cas13a检测法比上述方法的灵敏度更高。同时,该方法与其他6种猪源病原核酸没有交叉反应,表明建立的方法对PEDV具有强特异性。为了评估该技术在临床应用中的可靠性,计算了RAA-Cas13a检测与RT-qPCR的符合率,结果显示,40份疑似样品中,用RAA-Cas13a方法检测出29份阳性,RT-qPCR法检测出27份阳性,这27份阳性样品在RAA-Cas13a中的检测结果均为阳性,说明建立的RAA-Cas13a检测方法比RT-qPCR灵敏度更高。此外,RAA反应和CRSIRR/Cas13a检测均在37 ℃下进行,全部反应都可在一台便携的恒温荧光检测仪中完成,对硬件要求不高。
综上所述,本研究建立的PEDV RAA-Cas13a检测方法具有特异性强、灵敏度高、对实验室设备要求不高等优势,本研究结果可为PEDV检测提供一种新方法,为今后PED的诊断及流行病学调查提供技术支持。
4 结论本研究将RAA技术与CRISPR/Cas13a系统结合,建立了PEDV RAA-Cas13a检测方法,该方法特异性强、灵敏度高、对实验室设备要求不高、操作简单,可为PED的诊断及流行病学调查提供技术支持。
| [1] |
HAVE P, MOVING V, SVANSSON V, et al. Coronavirus infection in mink (Mustela vision). Serological evidence of infection with a coronavirus related to transmissible gastroenteritis virus and porcine epidemic diarrhea virus[J]. Vet Microbiol, 1992, 31(1): 1-10. DOI:10.1016/0378-1135(92)90135-G |
| [2] |
WOOD E N. An apparently new syndrome of porcine epidemic diarrhoea[J]. Vet Rec, 1977, 100(12): 243-244. DOI:10.1136/vr.100.12.243 |
| [3] |
SUN R Q, CAI R J, CHEN Y Q, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerg Infect Dis, 2012, 18(1): 161-163. DOI:10.3201/eid1801.111259 |
| [4] |
SONG D, PARK B. Porcine epidemic diarrhoea virus: a comprehensive review of molecular epidemiology, diagnosis, and vaccines[J]. Virus Genes, 2012, 44(2): 167-175. DOI:10.1007/s11262-012-0713-1 |
| [5] |
KOCHERHANS R, BRIDGEN A, ACKERMANN M, et al. Completion of the porcine epidemic diarrhoea coronavirus (PEDV) genome sequence[J]. Virus Genes, 2001, 23(2): 137-144. DOI:10.1023/A:1011831902219 |
| [6] |
LIU J, LI L M, HAN J Q, et al. A TaqMan probe-based real-time PCR to differentiate porcine epidemic diarrhea virus virulent strains from attenuated vaccine strains[J]. Mol Cell Probes, 2019, 45: 37-42. DOI:10.1016/j.mcp.2019.04.003 |
| [7] |
YU X W, SHI L, LV X P, et al. Development of a real-time reverse transcription loop-mediated isothermal amplification method for the rapid detection of porcine epidemic diarrhea virus[J]. Virol J, 2015, 12: 76. DOI:10.1186/s12985-015-0297-1 |
| [8] |
KELLNER M J, KOOB J G, GOOTENBERG J S, et al. SHERLOCK: nucleic acid detection with CRISPR nucleases[J]. Nat Protoc, 2019, 14(10): 2986-3012. DOI:10.1038/s41596-019-0210-2 |
| [9] |
MYHRVOLD C, FREIJE C A, GOOTENBERG J S, et al. Field-deployable viral diagnostics using CRISPR-Cas13[J]. Science, 2018, 360(6387): 444-448. DOI:10.1126/science.aas8836 |
| [10] |
LI J, MACDONALD J, VON STETTEN F. Review: a comprehensive summary of a decade development of the recombinase polymerase amplification[J]. Analyst, 2019, 144(1): 31-67. DOI:10.1039/C8AN01621F |
| [11] |
CHANG Y F, DENG Y, LI T Y, et al. Visual detection of porcine reproductive and respiratory syndrome virus using CRISPR-Cas13a[J]. Transbound Emerg Dis, 2020, 67(2): 564-571. DOI:10.1111/tbed.13368 |
| [12] |
LIU Y F, XU H P, LIU C, et al. CRISPR-Cas13a nanomachine based simple technology for Avian Influenza a (H7N9) virus on-site detection[J]. J Biomed Nanotechnol, 2019, 15(4): 790-798. DOI:10.1166/jbn.2019.2742 |
| [13] |
QIN P W, PARK M, ALFSON K J, et al. Rapid and fully microfluidic ebola virus detection with CRISPR-Cas13a[J]. ACS Sens, 2019, 4(4): 1048-1054. DOI:10.1021/acssensors.9b00239 |
| [14] |
陈浩, 鞠永政, 王文文, 等. 猪流行性腹泻病毒SYBR Green I荧光定量RT-PCR检测方法的建立与应用[J]. 中国动物检疫, 2022, 39(9): 110-114. CHEN H, JU Y Z, WANG W W, et al. Establishment and application of SYBR Green I fluorescent quantitative RT-PCR for detecting porcine epidemic diarrhea virus[J]. China Animal Health Inspection, 2022, 39(9): 110-114. (in Chinese) |
| [15] |
WANG D, FANG L R, XIAO S B. Porcine epidemic diarrhea in China[J]. Virus Res, 2016, 226: 7-13. DOI:10.1016/j.virusres.2016.05.026 |
| [16] |
LIANG W, ZHOU D N, GENG C, et al. Isolation and evolutionary analyses of porcine epidemic diarrhea virus in Asia[J]. PeerJ, 2020, 8: e10114. DOI:10.7717/peerj.10114 |
| [17] |
ABUDAYYEH O O, GOOTENBERG J S, ESSLETZBICHLER P, et al. RNA targeting with CRISPR-Cas13[J]. Nature, 2017, 550(7675): 280-284. DOI:10.1038/nature24049 |
| [18] |
WANG Z L, LI X R, SHANG Y J, et al. Rapid differentiation of PEDV wild-type strains and classical attenuated vaccine strains by fluorescent probe-based reverse transcription recombinase polymerase amplification assay[J]. BMC Vet Res, 2020, 16(1): 208. DOI:10.1186/s12917-020-02424-1 |
| [19] |
翟刚, 顾文源, 刘涛, 等. 猪流行性腹泻病毒TaqMan检测方法的建立及基于S基因的遗传变异分析[J]. 畜牧兽医学报, 2023, 54(2): 847-854. ZHAI G, GU W Y, LIU T, et al. Establishment of TaqMan detection method of porcine epidemic diarrhea virus and analysis of genetic variation based on S gene[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(2): 847-854. (in Chinese) |
| [20] |
冉伟, 田宇, 李梓健, 等. 基于ORF3基因检测猪流行性腹泻病毒荧光定量RT-PCR方法的建立与应用[J]. 中国预防兽医学报, 2021, 43(4): 394-398. RAN W, TIAN Y, LI Z J, et al. Development and preliminary application of ORF3-based fluorescent quantitative RT-PCR method for detection of porcine epidemic diarrhea virus[J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(4): 394-398. (in Chinese) |
(编辑 白永平)



