随着我国经济实力和人民生活水平的逐步提高,牛肉的需求量也在不断增加,宁夏作为肉牛养殖大省,肉牛饲养量增长迅猛。“十四五”规划提出“到2025年,全区肉牛饲养量将达到263万头”。肉牛养殖模式向着规模化、集约化和标准化不断发展。目前,宁夏存在两种极具特色的肉牛养殖模式:散养模式和规模化养殖模式。散养模式作为最原始、最基础的自繁自养自育养殖模式,存在于半农半牧区;规模化养殖模式具有高投资、高产出、养殖周期短等特点,存在于农场及城市周边。尽管肉牛养殖是宁夏地区的优势产业,但腹泻问题一直是困扰养殖户的重要难题之一,其病因极其复杂,除饲养管理引起的非感染性因素外,以病毒为主的感染性因素引起的腹泻病防控难度较大,造成了巨大的经济损失。
目前,已有研究人员对包括牛病毒性腹泻病毒(bovine viral diarrhea virus, BVDV)、牛冠状病毒(bovine coronavirus, BCoV)、牛轮状病毒(bovine rotavirus, BRV)、牛诺瓦病毒(bovine norovirus, BNoV)及牛星状病毒(bovine astrovirus, BAstV)在内的牛腹泻相关病毒的结构、致病机制及分子免疫方面做了大量研究[1-6],但新腹泻病原不断出现,且各病毒存在混合感染,导致肉牛腹泻病防控难度不断增加。因此,本研究对宁夏地区肉牛腹泻相关病毒进行RT-PCR检测,分析不同养殖模式、不同地区的病毒感染及混合感染情况,并进行主要病原的遗传进化分析,以期为宁夏肉牛腹泻病的综合防控提供理论依据。
1 材料与方法 1.1 样本采集本实验室于2021年1月—2022年7月在宁夏银川市、固原市和吴忠市随机采集肉牛拭子样本293份,其中散养肉牛样本139份、规模化养殖肉牛样本154份。
1.2 试剂与仪器病毒基因组RNA提取试剂盒(DP315)、DNA Marker(MD102)购自天根生化科技有限公司,反转录试剂盒(6210A)购自宝生物工程(大连)有限公司,2×PCR Mix(BL553D)、琼脂糖(BS081)以及核酸染料(BS354B)购自Biosharp生物公司。TC-512 PCR扩增仪购自英国Bibbby科技有限公司,凝胶成像仪、电泳仪购自美国伯乐科技有限公司,离心机购自赛默飞世尔科技公司。
1.3 引物设计及合成根据GenBank公布的各腹泻病毒序列,针对BRV VP7基因、BVDV 5′UTR基因、BCoV N基因、BNoV RdRp基因以及BAstV ORF1ab基因运用DNA star、Primer Premier 5.0软件设计特异性引物(表 1)。引物由生工生物工程股份有限公司合成。
|
|
表 1 牛腹泻相关病毒的引物信息 Table 1 Primer information for bovine diarrhea-associated viruses |
将采集的样本从液氮取出,置于4 ℃融化。涡旋振荡10 min使棉签上样本完全溶解,取悬液4 ℃,12 000 r ·min-1离心10 min,弃沉淀后进行核酸提取。
1.5 RNA提取及RT-PCR检测按照天根病毒基因组提取试剂盒提取拭子上清中RNA,根据TaKaRa反录说明书将RNA进一步反转录为cDNA。以cDNA为模板进行PCR扩增,体系(25 μL)如下:2×Taq plus Master Mix 12.5 μL,上、下游引物各1 μL,DNA模板1 μL,dd H2O 9.5 μL。PCR扩增程序:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,反应35个循环;72 ℃延伸10 min,4 ℃ 10 min。取5 μL PCR产物进行1%琼脂糖凝胶电泳检测,并在凝胶成像系统上观察结果、拍照。
1.6 扩增产物序列测定与分析按照DNA胶回收试剂盒说明书进行PCR产物回收纯化,将胶回收产物送至库美生物科技有限公司测序。通过NCBI-BLAST将测序结果与GenBank上发表的国内外病毒序列进行比对分析。利用MEGA 7.0软件的邻位相接法(Neighbor-joining法)进行遗传进化分析;采用DNA star软件Meg Align进行序列比对和同源性比较分析。
2 结果 2.1 不同腹泻病毒检测结果对宁夏地区293份肉牛样本进行RT-PCR检测,扩增产物电泳后在紫外线光下可观察到大小为300 bp(BVDV)、444 bp(BCoV)、652 bp(BRV)、286 bp(BNoV)和392 bp(BAstV)的条带(图 1),与预期片段大小相符,表明本试验设计的引物特异性强。RT-PCR检测发现,宁夏全区肉牛BVDV、BCoV、BRV、BNoV、BAstV阳性率分别为41.64%、41.98%、59.39%、50.51%、25.26%。
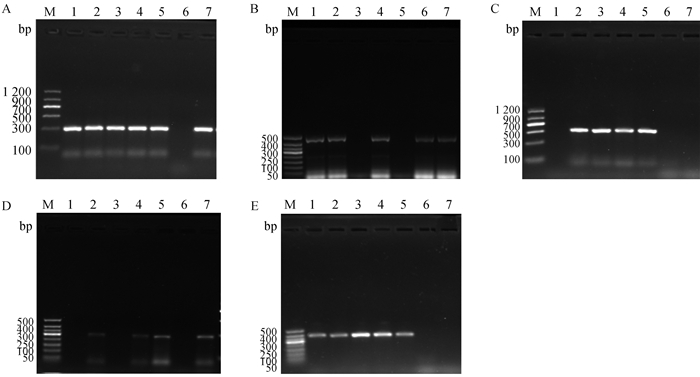
|
M. DNA相对分子质量标准;1~7. 肉牛拭子样本 M. DNA marker; 1-7. Swap samples of beef cattle 图 1 肉牛部分样本BVDV(A)、BCoV(B)、BRV(C)、BNoV(D)和BAstV(E)的PCR扩增结果 Fig. 1 PCR amplification results of BVDV(A), BCoV(B), BRV(C), BNoV(D), and BAstV(E) in beef cattle partial samples |
5种腹泻病原混合感染情况统计结果如图 2所示,0种、1种、2种、3种、4种及5种病毒感染及混合感染占比分别为9.9%、20.48%、27.30%、22.87%、18.09%、1.37%。单种病毒感染数最多的是BRV,有20份;两种病毒感染数最多的是BRV+ BNoV,感染数为20;3种病毒混合感染数最多的是BVDV+BRV+BNoV,感染数为19;4种病毒混合感染数最多的是BVDV+BCoV+BRV+BoAstV,感染数高达17。结果表明,宁夏地区病毒腹泻主要以BRV、BNoV、BVDV感染及其混合感染为主。
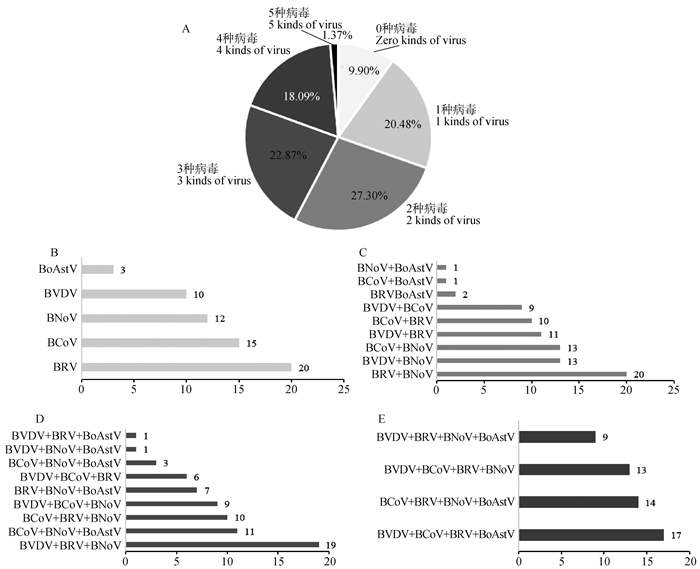
|
A. 腹泻病毒混合感染占比;B~E. 0种、1种、2种、3种、4种病毒混合感染 A. Proportion of diarrhea virus mixed infection; B-E. Zero, one, two, three, four kinds of virus mixed infection 图 2 不同腹泻病毒感染情况 Fig. 2 Different diarrhoea virus infections |
对139份散养肉牛样本、154份规模化养殖肉牛样本RT-PCR检测发现,散养模式下腹泻相关病毒阳性率普遍高于规模化养殖,如散养模式下BVDV、BRV、BNoV、BAstV阳性率远高于规模化养殖(图 3A);散养模式下病毒混合感染情况也较规模化养殖严重,散养模式下两种以上腹泻病毒混合感染率高达56.43%,规模化养殖模式下阳性率仅为29.40%(图 3B)。因此,规模化养殖一定程度上降低了腹泻相关病毒的感染及混合感染风险。
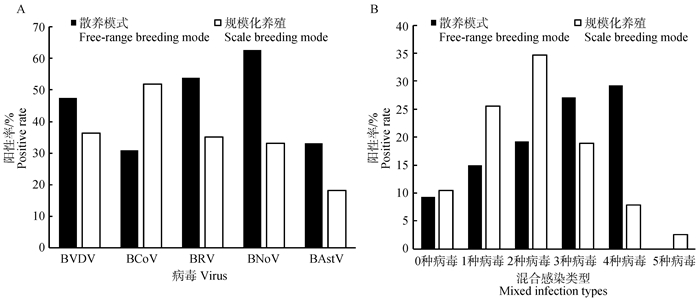
|
A.各腹泻病毒阳性率;B. 腹泻病毒混合感染情况 A. Positive rate of each diarrhea virus; B. Mixed infection of various diarrhea viruses 图 3 不同养殖模式下腹泻病毒检测结果 Fig. 3 Detection results of diarrhea virus under different breeding modes |
对宁夏82份银川市、71份固原市、140份吴忠市肉牛样本的RT-PCR检测发现,银川市肉牛腹泻相关病毒总阳性率为86.59%;固原市腹泻病毒总阳性率为92.96%;吴忠市腹泻病毒总阳性率为90.71%(图 4A)。银川市以1种、2种腹泻相关病毒感染为主,阳性率均为32.93%;固原市以2、3种病毒混合感染为主,阳性率分别为36.62%、25.35%;吴忠市以3、4种病毒感染混合为主,阳性率分别为27.14%、29.29%。各地区5种病毒混合感染普遍较低,均低于4%(图 4B)。由此说明,宁夏地区病毒性腹泻在不同地区、不同病毒差异明显,且混合感染较为严重。

|
A. 各腹泻病毒阳性率;B. 腹泻病毒混合感染情况 A. Positive rate of each diarrhea virus; B. Mixed infection of various diarrhea viruses 图 4 不同地区腹泻病毒检测结果 Fig. 4 Diarrheal virus test results in different regions |
对BVDV阳性样本的5′UTR基因片段进行回收纯化并测序,获得17条核苷酸序列,上传至GenBank(登录号为OQ513857~OQ513873)。应用DNA star软件对5′UTR基因序列与NCBI中上传的BVDV序列进行同源性分析,发现此研究中所有5′UTR基因序列均与GenBank中已公布的BVDV-1e的同源性最高,核苷酸序列一致性为75.30%~99.62%。通过Mega软件构建遗传进化树发现,测序的17个BVDV 5′UTR基因序列均与BVDV-1e聚集在同一分支上,与同亚型的IT99-3755、3186V6株亲缘关系较近(图 5A)。

|
A. BVDV遗传进化树;B. BNoV遗传进化树;C. BRV遗传进化树;D. BCoV遗传进化树 A. BVDV genetic evolution tree; B. BNoV genetic evolutionary tree; C. BRV genetic evolutionary tree; D. BCoV genetic evolutionary tree 图 5 肉牛腹泻相关病毒遗传进化分析 Fig. 5 Genetic evolution analysis of diarrhea related viruses in beef cattle |
对BNoV阳性样本的RdRp基因片段进行回收纯化并测序,获得的25条核苷酸序列,上传至GenBank(登录号:OQ430676~OQ3430682)。应用DNA star软件对RdRp基因序列与NCBI中上传的BNoV序列进行同源性分析,发现所有RdRp基因序列均与GenBank中已公布的BNoV GⅢ.2型同源性最高,核苷酸序列一致性为74.30%~99.18%。通过Mega软件构建遗传进化树发现,测序的25个BNoV RdRp基因序列均位于BNoV GⅢ.2分支上,与大多数中国株(Bo/SDA-3/18/CH等)和埃及株(GIII.2/Bo/BNoV/EGY)遗传进化关系较近(图 5B)。
对BRV阳性样本的VP7基因片段进行回收纯化并测序,获得的2条核苷酸序列,上传至GenBank(登录号:OQ513855、OQ513856)。应用DNA star软件对VP7基因序列与NCBI中上传的BRV序列进行同源性分析,发现2条VP7基因序列与GenBank中已公布的BRV G1亚型同源性最高,核苷酸序列一致性为86.43%~100.00%。通过Mega软件构建遗传进化树发现,VP7基因序列与BRV G1亚型遗传距离最近,与同亚型的T449株聚在同一分支上,一致性高达100%(图 5C)。
对BCoV阳性样本的N基因PCR片段进行回收纯化并测序,获得的14条核苷酸序列,上传至GenBank(登录号:OQ513841~OQ513854)。应用DNA star软件对N基因序列与NCBI中收录的BCoV序列进行同源性分析,发现此研究中的N基因序列与GenBank中已公布的BCoV核苷酸序列一致性为78.26%~100.00%。通过Mega软件构建遗传进化树发现,测序的BCoV N基因序列与法国株(登录号:KT318096、KT318085)同源性最近,聚在一个分支上(图 5D)。
3 讨论近年来,以腹泻为主要特征的疫病与日俱增,给宁夏地区肉牛养殖业造成了严重的经济损失。研究发现,宁夏地区肉牛的散户养殖质量及经济效益均低于规模化养殖;肉牛养殖规模越大,养殖技术效率水平越高,生产效益也随之增加[7]。病毒性病原引起的腹泻严重影响肉牛发育及成活率,目前国内除BVDV有灭活疫苗外,其他腹泻病毒(BCoV、BRV、BNoV、BAstV)尚无特效治疗方法[8]。此外,牛腹泻相关病毒均为RNA病毒,其基因组极易发生突变、重组和缺失等变异,使其毒力和致病力增强,同时也增加了疫苗研制难度[9]。牛腹泻性疾病难以根除另一大难题在于肉牛腹泻往往不是由单一病原引起的,而是多种病原微生物混合感染所致[10-12]。因此,肉牛腹泻疾病的防控在安全和效益方面都起着至关重要的作用。流行病学调查是疫病防控的基础,而防控的根本是检测。PCR/RT-PCR作为分子生物学检测的核心技术之一,相较于其他检测方法,具有特异性强、灵敏度高、操作简便等优点,是病原流行病学调查最常用的检测手段[13]。Zhang等[14]对中国西部385头奶牛鼻黏液进行PCR检测,结果显示,BVDV阳性率为42.34%,与BCoV混合感染率为0.78%。Zhu等[15]对东北黑龙江省奶牛场和肉牛场1 016头腹泻牛样本进行BCoV N基因的RT-PCR检测,结果显示BCoV阳性率为15.45%。Chen等[16]对中国BRV感染率进行系统评价和荟萃分析,发现牛BRV汇总患病率高达46%。孙吉等[17]应用多重RT-PCR方法对四川省220份牦牛腹泻样本进行BCoV、BVDV、BNoV检测,结果显示BNoV检出率为5.9%,BVDV和BCoV无检出;也证实牛腹泻病通常由两种及以上的病毒共同感染所致。
本研究对宁夏地区肉牛腹泻相关病毒(BVDV、BCoV、BRV、BNoV、BAstV)进行了RT-PCR检测,发现宁夏全区肉牛各腹泻病毒阳性率在25.26%~59.39%,病毒腹泻主要以BRV、BNoV、BVDV感染极其混合感染为主。宁夏地区普遍存在腹泻病毒的感染,可能是由于肉牛的频繁引种与交易;宁夏极端天气导致肉牛抵抗力下降,使得病毒在牛群中迅速传播[18]。混合感染的发生,一方面是由于肉牛感染某种病毒后产生免疫耐受,免疫力下降,造成易感染其他病毒[19];另一方面,牛群接触病原不断积累的同时也增加了病毒变异风险[20]。散养模式下肉牛腹泻相关病毒感染及混合感染情况较规模化养殖严重。究其原因,主要是由于规模化肉牛场具有专业的饲养模式、饲养人员、兽医人员,有效预防了腹泻病毒感染及进一步传播,从而规模牛场表现为较低的肉牛腹泻病毒阳性率及轻微的混合感染[21]。宁夏银川市、固原市、吴忠市肉牛腹泻病毒的感染及混合感染差异明显,这可能与各地免疫程序、养殖规模、饲养方式以及病毒本身变异及多样性有关。此外,研究发现有些肉牛并不表现出临床症状,但在其体内检测到了腹泻相关病毒,可能是由于肉牛感染病毒后表现为持续性感染或者隐性感染,成为潜在的长期性传染源[22]。
通过对各腹泻病毒RT-PCR检测阳性样本进行遗产进化分析发现,BVDV阳性样本属于BVDV-1e型。据国内外研究报道,亚洲主要流行BVDV-1m、1n、1o、1p、1q和1v亚型,而欧洲国家主要流行BVDV-1a、1b、1d、1e和1f[23]。一方面说明BVDV在适应环境的过程中表现出高度突变频率,另一方面说明牛群广泛的贸易与引种存在问题,需加大检查力度。BNoV阳性样本属于为GⅢ.2亚型,与已报道的大多数中国株和埃及株遗传进化关系较,提示宁夏地区存在国内和国际两种病毒传播途径[24]。BCoV阳性样本N基因与法国株同源性较近。由于N基因编码核衣壳蛋白,其高度保守[5],因此该地区存在BCoV的国际间传播途径,应当加强本地区流行毒株的监控。BRV阳性样本与G1亚型的猪轮状病毒C95株聚在同一小支上,表明该地区可能存在感染猪的BRV流行株,演化方向较广,需要进一步扩大对当地其他家畜的流行病学调查。
结合宁夏地区肉牛腹泻病的流行及感染情况,应采取综合措施进行防控和进化。在环境卫生方面,定时清理牛舍粪便和污水,开展牛舍消毒、进出场消毒。在饲养管理方面,提倡牛初乳喂养犊牛,从而获得母源抗体;病牛及时隔离治疗,合理使用抗生素类药物。在疫苗接种方面,制定合理的疫苗接种方案,将BVDV等疫苗纳入待产牛和犊牛的疫苗计划中。在病原监测方面,加强引种检疫工作;定期进行腹泻相关病毒的流行病学调查,及时作出相关疾病的预警。同时,应加快腹泻病毒流行毒株的分离,筛选出免疫原性好的毒株进行疫苗研制。
4 结论本研究对宁夏地区肉牛腹泻相关病毒进行RT-PCR检测与遗传进化分析,发现该地区肉牛普遍存在腹泻病毒的感染及混合感染;散养模式下肉牛腹泻病毒的感染及混合感染情况较规模化养殖严重;病毒性腹泻在不同地区、不同病毒差异明显;宁夏地区BVDV流行株为1e亚型,BRV流行株为G1亚型,BNoV流行株为GⅢ.2亚型,BCoV流行株与法国株遗传进化关系较近。本研究为宁夏肉牛腹泻疾病的防控提供了参考。
| [1] |
AL-KUBATI A A G, HUSSEN J, KANDEEL M, et al. Recent advances on the bovine viral diarrhea virus molecular pathogenesis, immune response, and vaccines development[J]. Front Vet Sci, 2021, 8: 665128. DOI:10.3389/fvets.2021.665128 |
| [2] |
ZHU Q H, LI B, SUN D B. Bovine astrovirus-a comprehensive review[J]. Viruses, 2022, 14(6): 1217. DOI:10.3390/v14061217 |
| [3] |
CUI Y C, CHEN X, YUE H, et al. First detection and genomic characterization of bovine norovirus from yak[J]. Pathogens, 2022, 11(2): 192. DOI:10.3390/pathogens11020192 |
| [4] |
GELETU U S, USMAEL M A, BARI F D. Rotavirus in calves and its zoonotic importance[J]. Vet Med Int, 2021, 2021: 6639701. |
| [5] |
VLASOVA A N, SAIF L J. Bovine coronavirus and the associated diseases[J]. Front Vet Sci, 2021, 8: 643220. DOI:10.3389/fvets.2021.643220 |
| [6] |
PARK G N, CHOE S, CHA R M, et al. Genetic diversity of bovine group a rotavirus strains circulating in korean calves during 2014 and 2018[J]. Animals (Basel), 2022, 12(24): 3555. |
| [7] |
ZHU L Y, OUDE LANSINK A. Dynamic sustainable productivity growth of Dutch dairy farming[J]. PLoS One, 2022, 17(2): e0264410. DOI:10.1371/journal.pone.0264410 |
| [8] |
MOSENA A C S, FALKENBERG S M, MA H, et al. Use of multivariate analysis to evaluate antigenic relationships between US BVDV vaccine strains and non-US genetically divergent isolates[J]. J Virol Methods, 2022, 299: 114328. DOI:10.1016/j.jviromet.2021.114328 |
| [9] |
VILLA T G, ABRIL A G, SÁNCHEZ S, et al. Animal and human RNA viruses: genetic variability and ability to overcome vaccines[J]. Arch Microbiol, 2021, 203(2): 443-464. DOI:10.1007/s00203-020-02040-5 |
| [10] |
GAUDINO M, NAGAMINE B, DUCATEZ M F, et al. Understanding the mechanisms of viral and bacterial coinfections in bovine respiratory disease: a comprehensive literature review of experimental evidence[J]. Vet Res, 2022, 53(1): 70. DOI:10.1186/s13567-022-01086-1 |
| [11] |
SOCHA W, LARSKA M, ROLA J, et al. Occurrence of bovine coronavirus and other major respiratory viruses in cattle in poland[J]. J Vet Res, 2022, 66(4): 479-486. DOI:10.2478/jvetres-2022-0059 |
| [12] |
PRATELLI A, LUCENTE M S, CORDISCO M, et al. Natural bovine coronavirus infection in a calf persistently infected with bovine viral diarrhea virus: viral shedding, immunological features and S gene variations[J]. Animals (Basel), 2021, 11(12): 3350. |
| [13] |
ZHU H L, ZHANG H Q, XU Y, et al. PCR past, present and future[J]. Biotechniques, 2020, 69(4): 317-325. DOI:10.2144/btn-2020-0057 |
| [14] |
ZHANG K, ZHANG J Y, QIU Z Y, et al. Prevalence characteristic of BVDV in some large scale dairy farms in Western China[J]. Front Vet Sci, 2022, 9: 961337. DOI:10.3389/fvets.2022.961337 |
| [15] |
ZHU Q H, SU M J, LI Z J, et al. Epidemiological survey and genetic diversity of bovine coronavirus in Northeast China[J]. Virus Res, 2022, 308: 198632. DOI:10.1016/j.virusres.2021.198632 |
| [16] |
CHEN S Y, ZHANG W, ZHAI J J, et al. Prevalence of bovine rotavirus among cattle in mainland China: a meta-analysis[J]. Microb Pathog, 2022, 170: 105727. DOI:10.1016/j.micpath.2022.105727 |
| [17] |
孙吉, 岳华, 汤承. 检测牛5种腹泻病毒的多重RT-PCR方法的建立及应用[J]. 畜牧兽医学报, 2022, 53(1): 209-218. SUN J, YUE H, TANG C. Establishment and application of multiplex RT-PCR for detection of five bovine diarrhea viruses[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(1): 209-218. (in Chinese) |
| [18] |
DIAO N C, GONG Q L, LI J M, et al. Prevalence of bovine viral diarrhea virus (BVDV) in yaks between 1987 and 2019 in mainland China: a systematic review and meta-analysis[J]. Microb Pathog, 2020, 144: 104185. DOI:10.1016/j.micpath.2020.104185 |
| [19] |
RIDPATH J F, FULTON R W, BAUERMANN F V, et al. Sequential exposure to bovine viral diarrhea virus and bovine coronavirus results in increased respiratory disease lesions: clinical, immunologic, pathologic, and immunohistochemical findings[J]. J Vet Diagn Invest, 2020, 32(4): 513-526. DOI:10.1177/1040638720918561 |
| [20] |
ROMEO F, LOUGE URIARTE E, DELGADO S G, et al. Effect of bovine viral diarrhea virus on subsequent infectivity of bovine gammaherpesvirus 4 in endometrial cells in primary culture: an in vitro model of viral co-infection[J]. J Virol Methods, 2021, 291: 114097. DOI:10.1016/j.jviromet.2021.114097 |
| [21] |
CHAI J M, LIU X T, USDROWSKI H, et al. Geography, niches, and transportation influence bovine respiratory microbiome and health[J]. Front Cell Infect Microbiol, 2022, 12: 961644. DOI:10.3389/fcimb.2022.961644 |
| [22] |
HAUSE B M, PILLATZKI A, CLEMENT T, et al. Persistent infection of American bison (Bison bison) with bovine viral diarrhea virus and bosavirus[J]. Vet Microbiol, 2021, 252: 108949. DOI:10.1016/j.vetmic.2020.108949 |
| [23] |
ZHU J, WANG C, ZHANG L N, et al. Isolation of BVDV-1a, 1m, and 1v strains from diarrheal calf in China and identification of its genome sequence and cattle virulence[J]. Front Vet Sci, 2022, 9: 1008107. DOI:10.3389/fvets.2022.1008107 |
| [24] |
KHAN M K, ALAM M M. Norovirus gastroenteritis outbreaks, genomic diversity and evolution: an overview[J]. Mymensingh Med J, 2021, 30(3): 863-873. |
(编辑 白永平)



