2. 新疆农垦科学院畜牧兽医研究所, 石河子 832000;
3. 石河子大学动物科技学院, 石河子 832000
2. Institute of Animal Husbandry and Veterinary, Xinjiang Academy of Agricultural and Reclamation Sciences, Shihezi 832000, China;
3. College of Animal Science and Technology, Shehezi University, Shihezi 832000, China
细粒棘球绦虫(Echinococcus granulosus, E.granulosus)属于扁形动物门,绦虫纲,圆叶目,带科,棘球属物种,是囊型包虫病的主要病原[1]。该病呈世界性分布,我国是囊型包虫病的高发国家之一。全国20多个省份均有该病发生,主要流行于青海、西藏、新疆、四川等畜牧业较发达的地区。据估计,我国包虫病患者人数约为100万,受威胁人口近6 600万。与现有的全球包虫病流行数据相比,中国包虫病受威胁人口数和患者人数仍居世界首位[1-2]。该病已严重威胁到广大农牧区群众的生命健康和财产安全,是导致我国西部农牧区群众因病致贫、因病返贫的主要病因之一。该病已被纳入“健康中国2030”规划中的五大寄生虫病,同时也是我国免费救治的重大传染病之一[3]。
细粒棘球绦虫的成虫寄生于犬科动物的小肠,成熟的虫卵和孕卵节片随粪便排出体外,污染草场和水源,被中间宿主牛、羊、骆驼等误食,虫卵在胃肠液的刺激下发育为六钩蚴,六钩蚴钻过肠壁,随血液循环到达宿主全身各处,形成包囊,里面含有大量原头蚴,其中主要以肝包囊为主。如宿主体内的包囊因机械损伤而破裂,每个原头蚴又可以发育为新的包囊。终末宿主犬科动物因采食被感染的含有包囊的动物脏器而感染,包囊中的原头蚴在终末宿主小肠内经大约45 d发育为成熟的虫体。其中,细粒棘球绦虫的原头蚴是虫体生长发育的主要阶段,在中间宿主体内可以经无性生殖发育为新的包囊,在终末宿主体内可以经过有性生殖发育为成虫,进而产生具有感染性的虫卵。同时原头蚴也是细粒棘球绦虫感染宿主的主要阶段,既可以在中间宿主造成二次感染形成新的包囊,也可以感染终末宿主,发育为成虫,进而形成新的循环[4-5]。由此可见,原头蚴无论是在虫体的生长发育,还是感染过程中都发挥着重要作用。但目前对其发育调控机制还知之甚少。近年的研究发现,circRNA是一种重要的非编码RNA调控分子。该分子呈封闭环状结构,可通过结合miRNA,作为翻译模板,调控基因转录,与RNA结合蛋白作用等,在表观遗传、转录及转录后水平上等多种水平调控基因表达[6-7]。目前,有关circRNA分子在细粒棘球绦虫生长发育过程中的生物学功能的研究鲜有报道。基于此,本研究拟采用RNA-seq技术对细粒棘球绦虫原头蚴,成虫以及包囊壁中circRNA表达谱特性进行系统解析,筛选各阶段高表达以及特异表达circRNA分子,对其生物学功能进行预测,进而为进一步研究其在虫体生长发育中的调控机制奠定基础。
1 材料与方法 1.1 试验材料、试剂及主要仪器在石河子牛羊屠宰场收集被细粒棘球绦虫感染的带有包囊的羊肝3个,4℃保温箱带回实验室,无菌条件下抽取含有原头蚴的包囊液,3 000 r·min-1离心5 min收集原头蚴,分离包囊壁,用加有1%双抗(青霉素+链霉素)的PBS洗涤3~5次,最后将收集的原头蚴、包囊壁保存至-80℃冰柜备用。细粒棘球绦虫成虫全虫样品为本实验室前期-80℃冰柜保存样品,采自人工感染犬。细粒棘球绦虫原头蚴和成虫的分子鉴定按照参考文献[8]的方法进行。
胎牛血清购自公司Gibco公司(美国);RPMI 1640细胞培养液购自Sigma公司(美国);小鼠单核巨噬细胞RAW 264.7购自ATCC细胞库(中国);SMARTer Ultra Low RNA Kit for Illumina Sequencing试剂盒购自Clontech公司(美国);AMPure XP beads试剂盒购自Beckman公司(美国);Invitrogen TRIzol Reagent购自Invitrogen公司(美国);CW2582 cDNA合成试剂盒购自康为世纪生物技术有限公司(中国);SYBR Premix Ex Taq Mix购自TaKaRa公司(日本);实时荧光定量PCR仪Lightcycle 2.0购自罗氏公司(德国)。
1.2 方法1.2.1 RNA的提取及质控 准备足量样品,按照TRIzol试剂盒提取样品总RNA,提取后使用微量核酸蛋白测定仪(scandrop100)检测OD260 nm/OD280 nm值以鉴定RNA样品浓度,排除RNA污染可能性。Bioanalyzer 2100(Agilent, Santa Clara, CA)用于评估总RNA质量,以RIN≥7且260/280≥1.8为阈值,RNase-free DNase I (Ambion Inc., Texas, USA)用于消除潜在的基因组DNA污染。分离的RNA样本保存在-80℃环境下。
1.2.2 cDNA文库构建和RNA测序 RNA质检合格后,分别从3个发育阶段样品中各取出约10 μg RNA,分别构建测序文库,主要包括酶促RNA片段化、cDNA合成、测序接头衔接及PCR扩增等过程。首先,根据EpicentreRibo-Zero Gold试剂盒(Illumina,San Diego,USA)的操作说明,消耗样品中的核糖体RNA、线性RNA并纯化后,在高温下使其片段化,随后转录成cDNA文库,最后分别利用Illumina Hiseq 4000进行测序。
1.2.3 数据处理及转录水平分析 采用Cutadapt软件对测序产生的原始数据进行过滤[9],除去低质量、含N的序列后,通过Bowtie2软件[10]和Tophat2软件[11]将clean reads比对到细粒棘球绦虫的基因组(GenBank登录号NW_020170381.1)。通过StringTie[12]软件识别新的转录本,并在此基础上采用Ballgown[13]软件估计转录物的表达水平。进行基因表达定量前,先通过FPKM算法消除偏好性。在本研究中,采用edgeR软件包确定差异表达基因[14],同时使用上四分位数算法校正数据,即设定|log2(两个样本的FPKM比率)|≥1且P≤0.05为阈值。采用TopHat-fusion[15]和CIRCExplorer2[16]软件对未映射序列进行识别,进而通过反向剪接读数来估计circRNA的表达水平[17],通过Pearson相关系数R2值估算确定circRNA及其相应亲本基因的共表达关系。本研究采用TMeV软件对差异表达的circRNA进行热图绘制,直观展示各样本的表达一般规律、差别及变化,从而通过聚类预测新型circRNA的功能及作用。circRNA-miRNA互作分析主要采用Targetscan与miRanda软件进行靶标预测后取交集,将得到的数据上传至Cytoscape绘制可视化互作网络图。
1.2.4 差异表达circRNA的GO及KEGG通路富集分析 经定量确定的差异表达circRNA仍需深入了解其生物学功能进而发掘重要的调控因子,在生物信息学分析中,常采用基因本体论(gene ontology,GO)进行分析[18]。在GO体系中,可以依次从细胞成分、生物学过程和分子功能3个方面定位基因功能,使用其注释富集功能将具有相似注释的转录物整合归类。在预测各类未知circRNA时,除了通过circRNA的宿主基因进行功能预测外,后期还可间接通过它们在生物信号通路中的参与情况进行功能分析。生物通路富集分析主要依赖于京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes, KEGG)数据库[19],其中途径富集分析可以精确定位差异表达基因的主要代谢转导和信号传导途径。本研究使用Scripts in house软件进行GO(http://geneontology.org)和KEGG(http://www.kegg.jp/kegg)富集分析,设置统计P≤0.05为结果显著阈值。
1.2.5 实时荧光定量PCR验证 为确保测序结果的准确性和有效性,需要通过qRT-PCR验证基因表达水平。经过组间对比和筛选,最终选择4个circRNAs分子进行验证。根据转录本数据库设计转录物引物(表 1),使用罗氏实时荧光定量PCR仪Lightcycle 2.0进行定量PCR。试验结果用采用2-ΔΔ Ct法计算各个基因的相对表达量[20],利用SPSS 22.0软件对所得表达量进行单因素方差分析(One-way ANOVA),结果分为差异显著(P≤0.05)和差异极显著(P≤0.01)。
|
|
表 1 实时定量PCR试验相关引物 Table 1 Primer used for qRT-PCR |
采用RNA-Seq和Small RNA-Seq深度测序,成虫每个样本文库内获得约90 000 000的原始数据,质量控制后平均得到约90 000 000的有效读段;包囊壁每个样本文库内获得约92 000 000的原始数据,质量控制后平均得到约90 000 000的有效读段;原头蚴每个样本文库内获得约86 000 000的原始数据,质量控制后平均得到约86 000 000的有效读段。Q-score>20的reads在99%以上,Q-score>30的reads在97%。综上,充分证明样本数据处理得当,质量可靠,符合下游数据分析要求。在深度分析样本注释结果,评估细粒棘球绦虫不同发育阶段中转录本丰度及表达差异情况前,计算评估了各生物学重复间皮尔森积矩相关系数平方(R2)均大于0.98,均满足统计学要求。
经筛选鉴定,最终鉴定得到635个circRNAs分子,其中有573个属于外显子来源的circRNAs分子,有14个属于内含子来源的circRNAs分子,有48个属于外显子-内含子circRNAs分子。对获得的circRNA分子的碱基组成分析发现,来源于成虫的circRNA分子,其GC为58.88%,来源于包囊壁的circRNA分子,其GC为60.09%,而来源于原头蚴的circRNA分子,其GC为59.74%。对circRNA分子的长度分析发现,成虫的crcRNA分子,其平均长度为2 100 nt左右,最长为17 013 nt,最小为191 nt;原头蚴的circRNA分子,其平均长度为1 560 nt左右,最长为20 176 nt,最小为179 nt;包囊壁的其平均长度为1 580 nt左右,最长为20 176 nt,最小为179 nt。
2.2 差异表达circRNA的鉴定在细粒棘球绦虫的3个不同发育阶段,共鉴定635个circRNAs分子,利用FPKM算法标准化基因转录水平差异,以细粒棘球绦虫原头蚴为参照,和成虫相比,有74个差异表达的circRNAs分子,其中上调表达的有68个,下调表达的有6个(图 1a);和包囊壁相比,有64个差异表达的circRNAs分子,其中上调表达的有55个,下调表达的有9个(图 1b);成虫和包囊壁相比,有45个差异表达的circ-RNAs分子,其中上调表达有1个,下调表达的有44个(图 1c);图 1d显示了circRNA分子在细粒棘球绦虫原头蚴、包囊壁和成虫差异表达的热图分析结果。其中在原头蚴阶段特异表达的circRNA有44个,包括novel_circ_0000037、novel_circ_0000254、novel_circ_0000307、novel_circ_0000379、novel_circ_0000501、novel_circ_0000537、novel_circ_0000704、novel_circ_0000855、novel_circ_0001062、novel_circ_0001105、novel_circ_0001221、novel_circ_0001250、novel_circ_0001375、novel_circ_0001424、novel_circ_0001490、novel_circ_0001554、novel_circ_0001585、novel_circ_0001781、novel_circ_0001932、novel_circ_0002021、novel_circ_0002060、novel_circ_0002101、novel_circ_0002298、novel_circ_0002332、novel_circ_0002371、novel_circ_0002391、novel_circ_0002416、novel_circ_0002509、novel_circ_0002635、novel_circ_0002713、novel_circ_0002843、novel_circ_0002909、novel_circ_0003207、novel_circ_0003410、novel_circ_0003417、novel_circ_0003436、novel_circ_0003558、novel_circ_0003815、novel_circ_0003867、novel_circ_0003978、novel_circ_0003986、novel_circ_0004074、novel_circ_0004131和novel_circ_0004185;包囊壁特异表达的circRNA分子有2个,为novel_circ_0002919和novel_circ_0003163;成虫阶段未发现特异表达的circRNA分子。

|
a.原头蚴(psc)和成虫(adult)中差异表达circRNA火山图;b.成虫和包囊壁(csw)中差异表达circRNA火山图;c.原头蚴和包囊壁中差异表达circRNA火山图;d.原头蚴、成虫和包囊壁中差异表达circRNA的热图 a.The volcanoplot of the differentlly expressed circRNAs in protoscoleces(psc) and adult worms(adult); b.The volcanoplot of the differentlly expressed circRNAs in adult worms and cyst wall csw; c.The volcanoplot of the differentlly expressed circRNAs in protoscoleces and cyst wall; d.The heatmap of the differentlly expressed circRNAs in protoscoleces, cyst wall and adult worms 图 1 CircRNA在细粒棘球绦虫原头蚴、包囊壁和成虫的差异表达分析 Fig. 1 Differential expression of circRNA in Echinococcus granulosus protoscoleces, cyst wall and adult worms |
以原头蚴为参照,分别和包囊壁以及成虫相比,其特异性差异表达的circRNA分子数量分别为58和68个,共同差异表达的circRNAs分子为6个,分别为novel_circ_0000017、novel_circ_0000654、novel_circ_0002024、novel_circ_0002781、novel_circ_0003640和novel_circ_0004249(图 2a)。以包囊壁为参照,分别和原头蚴以及成虫相比,其特异性差异表达的circRNA分子分别为60和41个,共同差异表达的circRNA分子为4个,分别为novel_circ_0002781、novel_circ_0003060、novel_circ_0003061和novel_circ_00032370(图 2b)。

|
a.成虫vs.原头蚴和包囊壁vs.原头蚴差异表达circRNA分子的Venn分析;b.原头蚴vs.包囊壁和成虫vs.包囊壁差异表达circRNA分子的Venn分析 a.The Venn analysis of the differentlly expressed circRNAs in adult vs. psc and csw vs. psc; b.The Venn analysis of the differentlly expressed circRNAs in psc vs. csw and adult vs. csw 图 2 细粒棘球绦虫原头蚴、包囊壁和成虫差异表达CircRNA分子的Venn图 Fig. 2 The Venn map of differential expression CircRNAs in Echinococcus granulosus protoscoleces, cyst wall and adult worms |
进一步的分析发现,在细粒棘球绦虫原头蚴阶段高表达top10的circRNA分子包括novel_circ_0003572、novel_circ_0003080、novel_circ_0002505、novel_circ_0003345、novel_circ_0002695、novel_circ_0000126、novel_circ_0004249、novel_circ_0003763、novel_circ_0001419和novel_circ_0004199。在成虫阶段高表达top10的circRNA分子包括novel_circ_0003572、novel_circ_0001343、novel_circ_0003080、novel_circ_0002505、novel_circ_0003345、novel_circ_0001341、novel_circ_0000582、novel_circ_0002695、novel_circ_0000126和novel_circ_0002031。在原头蚴和成虫两个阶段均高表达的circRNA分子包括novel_circ_0003572、novel_circ_0003080、novel_circ_0002505、novel_circ_0003345和novel_circ_0002695。在包囊壁阶段高表达top10的circRNA分子包括novel_circ_0000654、novel_circ_0002781、novel_circ_0003237、novel_circ_0003060、novel_circ_0003061、novel_circ_0004249、novel_circ_0002463、novel_circ_0002024、novel_circ_0003163和novel_circ_0002278。
2.3 差异表达circRNA的GO分析GO分析结果显示,原头蚴和成虫相比,差异表达的CircRNA的显著富集于75个条目,包括14个分子功能条目,22个细胞组分条目,39个生物学过程条目。主要富集于分子功能的核苷酸酶活性、腺苷酰转移酶活性、水解酶活性和FDA结合等;细胞组分的细胞内、细胞以及细胞部分等;生物学过程的多细胞生物的生殖、单细胞生物的生殖、氰酸酯代谢、有性生殖、精子发生、雄性配子产生、轴系发生以及神经生成、分化等(图 3)。原头蚴成包囊壁相比,差异表达的circRNA的显著富集于55个条目,包括34个生物学过程条目,12个细胞组分条目,9个分子功能条目。主要富集于生物学过程的脂质定位、氰酸酯代谢过程、调节脂质沉积以及细胞壁大分子分解代谢过程等;细胞组分的固有的细胞器膜、细胞器膜组成部分、固有的高尔基膜以及高尔基膜组成部分等。分子功能的酰辅酶A脱氢酶活性、溶菌酶活性、ADP结合以及结构分子活性等(图 4)。成虫和包囊壁相比,差异表达的差异表达的CircRNA的显著富集于65个条目,包括18个分子功能条目,14个细胞组分条目,33个生物学过程条目。主要富集于分子功能的FDA结合、二甲基丙酸锡脱硫甲基酶活性和6-磷酸果糖激酶活性等;细胞组分的病毒膜、细胞前缘及细胞色素b6f蛋白复合体等;生物学过程的磷酸烯醇式丙酮酸依赖性糖磷酸转移酶系统、碳水化合物运输、精子发生、雄性配子产生、轴系发生以及神经生成、分化等(图 5)。

|
图 3 细粒棘球绦虫原头蚴和成虫差异表达circRNA分子的GO功能分析 Fig. 3 The GO anlysis of differential expression circRNAs in Echinococcus granulosus protoscoleces and adult worms |

|
图 4 细粒棘球绦虫原头蚴和包囊壁差异表达circRNA分子的GO功能分析 Fig. 4 The GO anlysis of differential expression circRNAs in Echinococcus granulosus protoscoleces and cyst wall |
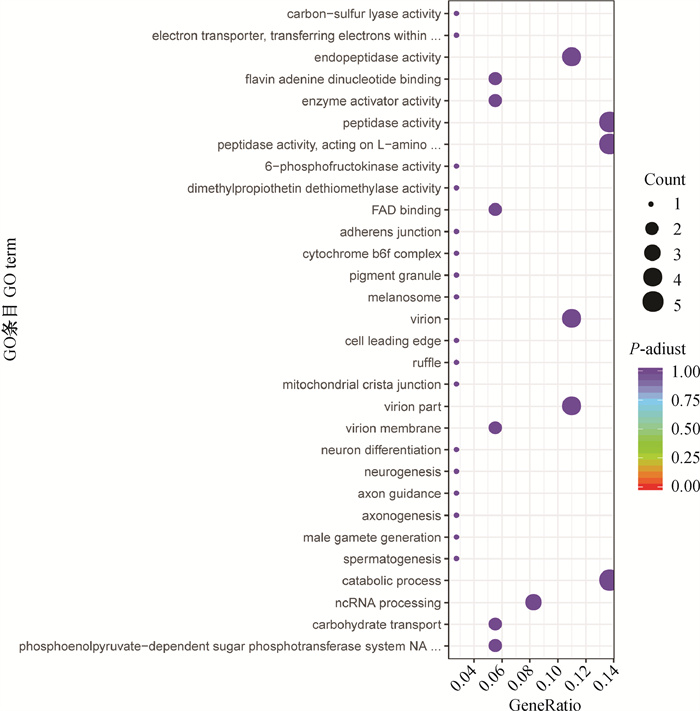
|
图 5 细粒棘球绦虫成虫和包囊壁差异表达circRNA分子的GO功能分析 Fig. 5 The GO anlysis of differential expression circRNAs in Echinococcus granulosus adult worms and cyst wall |
KEGG分析结果显示,原头蚴和包囊壁相比,差异表达的circRNA的显著富集于背腹轴形成、Notch、wnt以及FoxO信号通路等(图 6a)。原头蚴和成虫相比,差异表达的circRNA的显著富集于Notch、三羧酸循环、磷脂酰肌醇、核黄素代谢通路以及脂肪酸合成和降解等信号通路。成虫和包囊壁相比,差异表达的circRNA的显著富集于Notch、碳代谢、三羧酸循环以及糖酵解/糖异生通路。图 6b显示了差异表达的circRNA在Notch信号通路的富集情况,图 6c显示了circRNA在背腹轴形成信号通路的富集结果。

|
a.原头蚴和包囊壁中差异表达circRNA的KEGG信号通路显著富集结果;b.差异表达circRNA在NOTCH信号通路的富集结果;c.差异表达circRNA在DORSO-VENTRAL AXIS FORMATION(Grk/Egfr)信号通路的富集结果 a.The KEGG pathway enrichment result of the differential expression circRNAs in protoscoleces and cyst wall; b.The NOTCH signaling pathway enrichment result of the differential expression circRNAs; c.The DORSO-VENTRAL AXIS FORMATION(Grk/Egfr) signaling pathway enrichment result of the differential expression circRNAs 图 6 细粒棘球绦虫原头蚴、包囊壁和成虫差异表达circRNAs分子的KEGG信号通路分析 Fig. 6 The KEGG anlysis of differential expression circRNAs in Echinococcus granulosus protoscoleces, cyst wall and adult worms |
将筛选得到的差异表达circRNA及其靶向的miRNA和mRNA进行转录调控分析和共表达网络分析。原头蚴和成虫相比,有33个差异表达的circRNAs与24个miRNAs以及108个mRNAs构成302个circRNA-miRNA-mRNA调控网络。其中24个差异表达circRNAs和其互作的14个miRNAs、55个mRNAs形成164个调控网络符合circRNA上调表达,miRNA下调表达,mRNA下调表达的规律(图 7)。原头蚴和包囊壁相比,有2个差异表达的circRNAs与1个miRNA以及1个mRNA构成2个circRNA-miRNA-mRNA调控网络。成虫和包囊壁相比,有16个差异表达的circRNAs与10个miRNAs以及37个mRNAs构成73个circRNA-miRNA-mRNA调控网络。其中有13个差异表达的circRNAs与7个miRNAs以及16个mRNAs构成36个下调-上调-下调的circRNA-miRNA-mRNA调控网络(图 8)。

|
图 7 细粒棘球绦虫原头蚴和成虫差异表达circRNA分子上调-下调-上调型circRNA-miRNA-mRNA的调控通路分析 Fig. 7 The up-down-up circRNA-miRNA-mRNA regulated network anlysis of differential expression circRNAs in Echinococcus granulosus protoscoleces and adult worms |

|
图 8 细粒棘球绦虫成虫和包囊壁差异表达circRNA分子下调-上调-下调型circRNA-miRNA-mRNA的调控通路分析 Fig. 8 The down-up-down circRNA-miRNA-mRNA regulated network anlysis of differential expression circRNAs in Echinococcus granulosus adult worms and cyst wall |
为验证从转录组测序数据中获得差异基因结果的可靠性,随机选取4个差异表达的circRNAs进行qRT-PCR验证,其中包括novel_circ_0003572、novel_circ_0002024、novel_circ_0003223和novel_circ_0003080,结果显示qRT-PCR验证结果与RNA-seq测序结果一致,进一步说明转录组测序结果可信(图 9)。

|
图 9 差异表达circRNA的qRT-PCR验证分析 Fig. 9 Validation analysis of the differntially expressed circRNA by qRT-PCR |
环状RNA(CircRNAs)是一类不具有5′端帽子和3′端ploy(A)尾巴结构的共价闭合环状RNA分子[21-22]。早在1976年,Sanger等[23]就在植物类病毒中发现了CircRNA。随后的几十年间,科学家又相继在小鼠、猴、猪、人等物种中发现了circRNA[24-27]。由于传统的分子生物学方法对circRNA数量和丰度的检测能效都非常有限,因此一直以来circRNA被认为是RNA异常剪接的产物。近年来随着生物信息学的快速发展和高通量测序技术的不断革新,目前已经在真核细胞中发现了大量内源circRNAs,大部分circRNA结构稳定,丰度高且呈现出时空表达特异性,说明circRNA可能在调控基因表达过程中发挥着重要作用[21, 28]。根据序列构成的差异可以将circRNA分为3类:外显子circRNA (exonic circRNA, ecRNA),内含子circRNA(circular intronic RNA, ciRNA),外显子-内含子circRNA (exon-intron circRNA, Elci-RNA)[29-31]。这些circRNA具有自身特有的产生方式,同时它们的生物学功能也有一定差异。与其他非编码RNA一样,circRNA的序列和结构决定了它的生物学功能。circRNA可能通过以下几种方式发挥生物学功能:miRNA海绵,作为翻译模板,调控基因转录,与RNA结合蛋白作用[32-34]。
目前有关寄生虫circRNA的研究报道相对较少。Broadbent等[35]研究发现疟原虫有数百个circRNA分子,并对其中部分分子进行了初步验证。Zhou等[36]测序分析发现circRNA分子在捻转血矛线虫3期幼虫、成熟雌虫和雄虫中存在差异表达,且其功能主要与机体代谢、MAPK信号通路以及磷脂酰肌醇信号通路相关。同时也有部分学者对宿主应对寄生虫感染时宿主circRNA分子的差异表达进行了研究,挖掘出一些与宿主应对寄生虫感染相关的circRNA分子[37-39]。近年来,也有棘球绦虫circRNA相关的研究报道,但多数研究者把研究的焦点放在宿主方面,如Kalifu等[40]研究发现,和正常肝组织相比,患者包囊组织中有177个ccircRNAs分子的表达出现上调,166个出现下调。基因功能聚类分析发现这些差异表达的circRNA分子可能参与有机化合物循环、内源性刺激以及细胞生物过程等。之后,Liu等[41]研究发现,在多房棘球绦虫感染的小鼠肝细胞、肝星状细胞和肝枯否细胞中共鉴定到6 290个circRNAs分子,进一步的分析发现在多房棘球绦虫感染后2和3个月的时候,小鼠肝细胞、肝星状细胞和肝枯否细胞中分别有834个、834个以及760个差异表达的circRNA分子。目前,仅有1篇有关细粒棘球绦虫虫体本身circRNA相关的研究报道,此研究发现在细粒棘球绦虫原头蚴与包囊液外泌体中分别有1 277和512个circRNAs分子,其中有183个circRNAs分子是二者共有的[42]。由以上研究可以发现,目前对棘球绦虫circRNA的研究,基本还是停留在组学测序分析阶段,有关其具体生物学功能的研究还未见报道。同时,有关系统解析细粒棘球绦虫不同发育阶段,即原头蚴,成虫以及包囊壁相关circRNA的研究亦未见报道。基于此,为了揭示circRNA分子在细粒棘球绦虫不同发育阶段的差异表达以及发生的分子事件,本研究采用RNA-seq技术系统解析了circRNA分子在细粒棘球绦虫原头蚴,成虫以及包囊壁中的表达谱特性,筛选了各阶段高表达以及特异表达circRNA分子,并对其生物学功能进行了预测,为进一步揭示这些差异表达的circRNA分子在虫体生长发育中的调控机制提供了理论依据。
本研究的结果显示,circRNA在细粒棘球绦虫原头蚴、包囊壁和成虫的表达均存在差异。同时,本研究首次在细粒棘球绦虫原头蚴和包囊壁中分别鉴定到44和2个特异表达的circRNA分子,推测其在虫体原头蚴和包囊壁的发育过程中可能发挥重要作用。对差异表达circRNA分子的GO分析结果显示,当成虫和原头蚴、包囊壁相比,部分差异表达的circRNA分子的功能显著富集于有性生殖、精子发生、雄性配子产生、轴系发生以及神经生成、分化等过程。这一结果和细粒棘球绦虫发育规律恰好吻合,相对于虫体原头蚴和包囊壁,成虫是虫体有性生殖、精子发生和雄性配子产生的主要阶段,也是虫体前后轴发育,神经生成、分化的主要阶段[43],此结果提示这些差异表达的circRNA分子可能参与调控了细粒棘球绦虫以上的生物学过程。对差异表达circRNA分子的KEGG分析结果显示,原头蚴和包囊壁相比,差异表达的circRNA显著富集于背腹轴形成和Notch信号通路。
本研究对差异表达circRNA进行ceRNA调控网络分析发现,有164个ircRNA-miRNA-mRNA调控通路属于up-down-up的调控形式,有36个circRNA-miRNA-mRNA调控通路属于down-up-down的调控形式。同时,分析发现有8个编码基因既参与up-down-up调控通路,又参与down-up-down调控通路,包括thymidine kinase、ccaat enhancer binding protein beta、caspase 8、THUMP domain containing protein 1、ETS transcription factor Elf 2、SH2、tetraspanin和Heat shock 70 kDa protein 4。其中ccaat enhancer binding protein beta是一种神经发育相关调控蛋白,已有研究显示此蛋白在成年啮齿动物大脑的不同部位表达[44-45]。在突触可塑性和记忆形成中发挥重要作用,尤其是在海马体[46-47]、神经元分化[48-49]和海马神经原的发育中发挥重要调控作用[50],与其互作的miRNA为egr-miR-10227a-5p,circRNA为novel_circ_0000152。而SH2是一种精子发育相关调控基因,具体参与调控精子核的形状[51],与其互作的miRNA为novel_19,circRNA分别为novel_circ_0002024、novel_circ_0003572、novel_circ_0003223和novel_circ_0001419。
在up-down-up的调控通路中,还有1个参与调控雄性生殖细胞生长发育相关的基因,即Tesmin TSO1 CXC,此基因在雄性配子的发育以及精子的分化过程中起到调控作用[52],与其互作的miRNA为egr-miR-4990,circRNA分别为novel_circ_0002152、novel_circ_0000017、novel_circ_0002024、novel_circ_0000654、novel_circ_0000253和novel_circ_0003999。Tetraspanin是一类四跨膜蛋白,此家族成员在绦虫基因组中发生扩张[53],并且可能是宿主/病原体微界面的主要成分,同时也是绦虫释放到宿主体内外泌体的主要组成部分[54],此类蛋白可以结合宿主抗体的Fc区域[55],并且其本身具有很高的免疫原性[56],与其互作的miRNA为novel_19,circRNA为novel_circ_0002024、novel_circ_0003572、novel_circ_0003223和novel_circ_0001419。对结果的分析发现,novel_circ_0003572既在虫体原头蚴和成虫阶段高表达,又可能参与调控虫体雄性生殖系统的发育以及虫体的免疫调控等过程,提示其可能具有重要的生物学功能,可以作为后续深入研究的一个靶标。除此之外,还发现novel_circ_0002024、novel_circ_0003223和novel_circ_0001419同样也参与调控多个生物学过程,提示这些分子也可能在细粒棘球绦虫的生长发育过程中发挥重要功能,但其具体的生物学功能还有待进一步研究。
4 结论综上所述,本研究首次系统解析了circRNA在细粒棘球绦虫原头蚴、包囊壁以及成虫的差异表达谱特性。筛选了细粒棘球绦虫各发育阶段高表达以及特异表达的circRNA分子,并且初步预测了虫体精子生长发育以及虫体神经生成、分化相关的circRNA分子及其circRNA-miRNA-mRNA调控通路,为进一步阐明circRNA分子在细粒棘球绦虫生长发育中的作用提供了新的视角。
| [1] |
SIRACUSANO A, DELUNARDO F, TEGGI A, et al. Host-parasite relationship in cystic echinococcosis: an evolving story[J]. Clin Dev Immunol, 2012, 2012: 639362. |
| [2] |
SAVIOLI L, DAUMERIE D. Working to overcome the global impact of neglected tropical diseases-Summary[J]. Wkly Epidemiol Rec, 2011, 86(13): 113-120. |
| [3] |
汪天平, 操治国. 中国棘球蚴病防控进展及其存在的问题[J]. 中国寄生虫学与寄生虫病杂志, 2018, 36(3): 291-296. WANG T P, CAO Z G. Current status of echinococcosis control in China and the existing challenges[J]. Chinese Journal of Parasitology and Parasitic Diseases, 2018, 36(3): 291-296. (in Chinese) |
| [4] |
MCMANUS D P, ZHANG W B, LI J, et al. Echinococcosis[J]. Lancet, 2003, 362(9392): 1295-1304. DOI:10.1016/S0140-6736(03)14573-4 |
| [5] |
ZHENG H J, ZHANG W B, ZHANG L, et al. The genome of the hydatid tapeworm Echinococcus granulosus[J]. Nat Genet, 2013, 45(10): 1168-1175. DOI:10.1038/ng.2757 |
| [6] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [7] |
EBBESEN K K, HANSEN T B, KJEMS J. Insights into circular RNA biology[J]. RNA Biol, 2017, 14(8): 1035-1045. DOI:10.1080/15476286.2016.1271524 |
| [8] |
BOWLES J, BLAIR D, MCMANUS D P. Genetic variants within the genus Echinococcus identified by mitochondrial DNA sequencing[J]. Mol Biochem Parasitol, 1992, 54(2): 165-173. DOI:10.1016/0166-6851(92)90109-W |
| [9] |
KECHIN A, BOYARSKIKH U, KEL A, et al. cutPrimers: a new tool for accurate cutting of primers from reads of targeted next generation sequencing[J]. J Comput Biol, 2017, 24(11): 1138-1143. DOI:10.1089/cmb.2017.0096 |
| [10] |
LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [11] |
KIM D, PERTEA G, TRAPNELL C, et al. TopHat2:accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions[J]. Genome Biol, 2013, 14(4): R36. DOI:10.1186/gb-2013-14-4-r36 |
| [12] |
MIHAELA P, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290-295. DOI:10.1038/nbt.3122 |
| [13] |
FRAZEE A C, GEO P, JAFFE A E, et al. Ballgown bridges the gap between transcriptome assembly and expression analysis[J]. Nat Biotechnol, 2015, 33(3): 243-246. DOI:10.1038/nbt.3172 |
| [14] |
ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Genome Biol, 2010, 11(10): R106. DOI:10.1186/gb-2010-11-10-r106 |
| [15] |
KIM D, SALZBERG S L. TopHat-Fusion: an algorithm for discovery of novel fusion transcripts[J]. Genome Biol, 2011, 12(8): R72. DOI:10.1186/gb-2011-12-8-r72 |
| [16] |
ZHANG X O, WANG H B, ZHANG Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 |
| [17] |
ASHWAL-FLUSS R, MEYER M, PAMUDURTI N R, et al. circRNA biogenesis competes with Pre-mRNA splicing[J]. Mol Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 |
| [18] |
CAMON E, MAGRANE M, BARRELL D, et al. The gene ontology annotation (GOA) database: sharing knowledge in uniprot with gene ontology[J]. Nucleic Acids Res, 2004, 32(S1): D262-D266. |
| [19] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2008, 36(S1): D480-D484. |
| [20] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [21] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [22] |
EBBESEN K K, HANSEN T B, KJEMS J. Insights into circular RNA biology[J]. RNA Biol, 2017, 14(8): 1035-1045. DOI:10.1080/15476286.2016.1271524 |
| [23] |
SANGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci U S A, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 |
| [24] |
CAPEL B, SWAIN A, NICOLIS S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-1030. DOI:10.1016/0092-8674(93)90279-Y |
| [25] |
ABDELMOHSEN K, PANDA A C, DE S, et al. Circular RNAs in monkey muscle: age-dependent changes[J]. Aging (Albany NY), 2015, 7(11): 903-910. |
| [26] |
VENØ M T, HANSEN T B, VENØ S T, et al. Spatio-temporal regulation of circular RNA expression during porcine embryonic brain development[J]. Genome Biol, 2015, 16: 245. DOI:10.1186/s13059-015-0801-3 |
| [27] |
BURD C E, JECK W R, LIU Y, et al. Expression of linear and novel circular forms of an INK4/ARF-associated non-coding RNA correlates with atherosclerosis risk[J]. PLoS Genet, 2010, 6(12): e1001233. DOI:10.1371/journal.pgen.1001233 |
| [28] |
WANG P L, BAO Y, YEE M C, et al. Circular RNA is expressed across the eukaryotic tree of life[J]. PLoS One, 2014, 9(6): e90859. |
| [29] |
ZHANG X O, WANG H B, ZHANG Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 |
| [30] |
ZHANG Y, ZHANG X O, CHEN T, et al. Circular intronic long noncoding RNAs[J]. Mol Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [31] |
LI Z Y, HUANG C, BAO C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nat Struct Mol Biol, 2015, 22(3): 256-264. DOI:10.1038/nsmb.2959 |
| [32] |
HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [33] |
WANG Y, WANG Z F. Efficient backsplicing produces translatable circular mRNAs[J]. RNA, 2015, 21(2): 172-179. DOI:10.1261/rna.048272.114 |
| [34] |
JECK W R, SHARPLESS N E. Detecting and characterizing circular RNAs[J]. Nat Biotechnol, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890 |
| [35] |
BROADBENT K M, BROADBENT J C, RIBACKE U, et al. Strand-specific RNA sequencing in Plasmodium falciparum malaria identifies developmentally regulated long non-coding RNA and circular RNA[J]. BMC Genomics, 2015, 16(1): 454. DOI:10.1186/s12864-015-1603-4 |
| [36] |
ZHOU C X, ZHANG Y, WU S M, et al. Genome-wide Identification of circRNAs of infective larvae and adult worms of parasitic nematode, Haemonchus contortus[J]. Front Cell Infect Microbiol, 2021, 11: 764089. DOI:10.3389/fcimb.2021.764089 |
| [37] |
REN G J, FAN X C, LIU T L, et al. Genome-wide analysis of differentially expressed profiles of mRNAs, lncRNAs and circRNAs during Cryptosporidium baileyi infection[J]. BMC Genomics, 2018, 19(1): 356. DOI:10.1186/s12864-018-4754-2 |
| [38] |
ZHOU C X, AI K, HUANG C Q, et al. miRNA and circRNA expression patterns in mouse brain during toxoplasmosis development[J]. BMC Genomics, 2020, 21(1): 46. DOI:10.1186/s12864-020-6464-9 |
| [39] |
YU H L, MI C H, WANG Q, et al. Comprehensive analyses of circRNA expression profiles and function prediction in chicken cecums after Eimeria tenella infection[J]. Front Cell Infect Microbiol, 2021, 11: 628667. DOI:10.3389/fcimb.2021.628667 |
| [40] |
KALIFU B, MAITISEYITI A, GE X H, et al. Expression profile of circular RNAs in cystic echinococcosis pericystic tissue[J]. J Clin Lab Anal, 2021, 35(3): e23687. |
| [41] |
LIU T L, WANG L Q, LI H, et al. circRNA expression pattern and circRNA-miRNA-mRNA network in HCs, HSCs, and KCs of murine liver after Echinococcus multilocularis infection[J]. Front Vet Sci, 2022, 9: 825307. DOI:10.3389/fvets.2022.825307 |
| [42] |
ZHANG X F, GONG W C, CAO S K, et al. Comprehensive analysis of Non-coding RNA profiles of exosome-like vesicles from the protoscoleces and hydatid cyst fluid of Echinococcus granulosus[J]. Front Cell Infect Microbiol, 2020, 10: 316. DOI:10.3389/fcimb.2020.00316 |
| [43] |
PALUDO G P, THOMPSON C E, MIYAMOTO K N, et al. Cestode strobilation: prediction of developmental genes and pathways[J]. BMC Genomics, 2020, 21(1): 487. DOI:10.1186/s12864-020-06878-3 |
| [44] |
NADEAU S, HEIN P, FERNANDES K J L, et al. A transcriptional role for C/EBP β in the neuronal response to axonal injury[J]. Mol Cell Neurosci, 2005, 29(4): 525-535. DOI:10.1016/j.mcn.2005.04.004 |
| [45] |
STERNECK E, PAYLOR R, JACKSON-LEWIS V, et al. Selectively enhanced contextual fear conditioning in mice lacking the transcriptional regulator CCAAT/enhancer binding protein δ[J]. Proc Natl Acad Sci U S A, 1998, 95(18): 10908-10913. DOI:10.1073/pnas.95.18.10908 |
| [46] |
TAUBENFELD S M, WⅡG K A, MONTI B, et al. Fornix-dependent induction of hippocampal CCAAT enhancer-binding proteinβ and δ Co-localizes with phosphorylated cAMP response element-binding protein and accompanies long-term memory consolidation[J]. J Neurosci, 2001, 21(1): 84-91. DOI:10.1523/JNEUROSCI.21-01-00084.2001 |
| [47] |
ALBERINI C M, GHIRARDL M, METZ R, et al. C/EBP is an immediate-early gene required for the consolidation of long-term facilitation in Aplysia[J]. Cell, 1994, 76(6): 1099-1114. DOI:10.1016/0092-8674(94)90386-7 |
| [48] |
CORTÉS-CANTELI M, PIGNATELLI M, SANTOS A, et al. CCAAT/enhancer-binding protein β plays a regulatory role in differentiation and apoptosis of neuroblastoma cells[J]. J Biol Chem, 2002, 277(7): 5460-5467. DOI:10.1074/jbc.M108761200 |
| [49] |
MÉNARD C, HEIN P, PAQUIN A, et al. An essential role for a MEK-C/EBP pathway during growth factor-regulated cortical neurogenesis[J]. Neuron, 2002, 36(4): 597-610. DOI:10.1016/S0896-6273(02)01026-7 |
| [50] |
CORTES-CANTELI M, AGUILAR-MORANTE D, SANZ-SANCRISTOBAL M, et al. Role of C/EBPβ transcription factor in adult hippocampal neurogenesis[J]. PLoS One, 2011, 6(10): e24842. DOI:10.1371/journal.pone.0024842 |
| [51] |
L'HÔTE D, SERRES C, LAISSUE P, et al. Centimorgan-range one-step mapping of fertility traits using interspecific recombinant congenic mice[J]. Genetics, 2007, 176(3): 1907-1921. DOI:10.1534/genetics.107.072157 |
| [52] |
JIANG J Q, BENSON E, BAUSEK N, et al. Tombola, a tesmin/TSO1-family protein, regulates transcriptional activation in the Drosophila male germline and physically interacts with Always early[J]. Development, 2007, 134(8): 1549-1559. DOI:10.1242/dev.000521 |
| [53] |
International Helminth Genomes Consortium. Comparative genomics of the major parasitic worms[J]. Nat Genet, 2019, 51(1): 163-174. DOI:10.1038/s41588-018-0262-1 |
| [54] |
COAKLEY G, MAIZELS R M, BUCK A H. Exosomes and other extracellular vesicles: the new communicators in parasite infections[J]. Trends Parasitol, 2015, 31(10): 477-489. DOI:10.1016/j.pt.2015.06.009 |
| [55] |
WU C, CAI P F, CHANG Q C, et al. Mapping the binding between the tetraspanin molecule (Sjc23) of Schistosoma japonicum and human non-immune IgG[J]. PLoS One, 2011, 6(4): e19112. DOI:10.1371/journal.pone.0019112 |
| [56] |
KRAUTZ-PETERSON G, DEBATIS M, TREMBLAY J M, et al. Schistosoma mansoni infection of mice, rats and humans elicits a strong antibody response to a limited number of reduction-sensitive epitopes on five major tegumental membrane proteins[J]. PLoS Negl Trop Dis, 2017, 11(1): e0005306. DOI:10.1371/journal.pntd.0005306 |
(编辑 范子娟)



