2. 宁夏农林科学院动物科学研究所, 银川 750002
2. Institute of Animal Science, Ningxia Academy of Agriculture and Forestry Sciences, Yinchuan 750002, China
养羊业是我国畜牧业的重要组成部分,也是广大农村、牧区乡村振兴的主导产业之一[1]。众所周知,瘤胃是反刍动物特有的消化器官,反刍动物可通过瘤胃中细菌、原虫和真菌发酵对饲料进行分解从而获取营养物质。瘤胃中各种微生物相互作用,能够影响瘤胃中脂肪、蛋白质等沉积与纤维素的降解速率、改善反刍动物消化功能[2]。此外,瘤胃作为一个厌氧发酵罐,正常稳定的微生物区系是瘤胃健康发育的重要保证,而这些微生物在瘤胃结构形态发育、免疫功能调节、抵御外源致病因子侵袭等方面发挥着重要作用[3-4]。然而,由于反刍动物瘤胃的特殊生理环境,绝大多数瘤胃微生物很难在体外培养,这一特点极大限制了人们对瘤胃生物资源的探索研究。近年来,随着测序技术的迅速发展,宏基因组测序已广泛运用于特定环境中的微生物组成分析、代谢通路研究以及酶蛋白的功能分析等[5]。
通过调控反刍动物的日粮组成和饲养方式等,可以促进瘤胃内不同微生物群落的定植并产生持久影响[6-8],这也是反刍动物饲料转化效率不同的原因。姚喜喜等[9]发现,饲养方式显著影响了大通牦牛的瘤胃组织结构及瘤胃菌群多样性和组成。张洁等[10]发现不同饲养方式因饲喂日粮的不同导致滩羊瘤胃真菌菌群结构发生了变化。研究表明,运动能够促进动物肠道菌群改变,如改变菌种丰度和菌群多样性[11-12]。Kang等[13]研究发现,运动增加了小鼠肠道内有益菌厚壁菌门和拟杆菌门的丰度。因此,探索不同饲养方式对羊瘤胃微生物群落的种类和丰度的影响,对于制定科学合理的饲养模式是非常必要的。
滩羊作为中国珍贵的裘皮羊品种,适应当地的自然条件,抗逆性突出,耐粗饲,在草质差、产草量低的情况下仍能很好的生长,可见滩羊瘤胃内蕴藏着独特的木质纤维素降解微生物和酶系。然而,目前关于饲喂方式对家畜的影响多集中在生长性能、消化代谢和肉品质[14-17],对瘤胃微生物菌群的研究比较匮乏。因此,本试验以滩羊为研究对象,通过高通量测序技术研究不同运动量对滩羊瘤胃菌群多样性的影响,旨在为健康养殖滩羊提供参考依据。
1 材料与方法 1.1 时间和地点试验于2021年4—8月在宁夏回族自治区盐池滩羊养殖基地进行。
1.2 试验动物与试验设计选取同龄、体况良好(体重(20±0.5)kg)的2月龄断奶滩羊羔羊60只,随机分为3组(公母各半):即对照组(NC组)、5公里组(5 km组)和10公里组(10 km组),每组20只,每组5个重复,每个重复4只羊,各组分别饲养在同一栏。NC组采取全舍饲饲养,5 km组每日沿生态牧场运动围栏运动5公里,时间控制在2 h以内;10 km组每日沿生态牧场运动围栏运动10公里,时间控制在5 h以内。试验采用单因素试验设计,试验羊佩戴计步器,适度驱赶,实验人员通过手机端APP,查看运动轨迹和运动量,然后记录每日运动量,预试期10 d,正试期120 d。
1.3 试验日粮参照中华人民共和国肉羊饲养标准NY/T 816—2004配制试验日粮,试验日粮及营养水平见表 1。
|
|
表 1 基础日粮组成及营养水平(干物质基础) Table 1 Basal diet composition and nutritional level (dry matter basis) |
试验期间,每只羊定时定量饲喂。每天早上(6:30)和下午(18:30)喂料2次,期间自由饮水。试验开始前用2%的NaOH溶液对羊舍进行喷洒消毒,试验期内每天定时打扫圈舍,保持圈舍清洁、通风,并记录试验羊健康状况。
1.5 样品采集与处理试验结束时,各组随机选取体重相近、健康状况一致的7只试验羊,禁食12 h后屠宰采集瘤胃内容物样,并在2 h内运送至实验室,-80 ℃冰箱中储存用于瘤胃微生物区系多样性的测定。
1.6 测定指标及方法1.6.1 DNA提取 按照QIAamp DNA Micro kit(德国Qiagen)试剂盒说明书的要求,进行瘤胃内容物宏基因组DNA的分离和纯化。DNA样品先用超微量紫外分光光度(Thermo,美国)测定浓度和纯度,后用1%的琼脂糖凝胶电泳定性检测。符合上机建库要求的DNA样本送至上海伟寰生物科技有限公司进行测序。
1.6.2 Illumina PE150测序 纯化后的样品利用Illumina PE150高通量测序平台进行测序。
1.6.3 数据分析 首先对测序数据进行宿主基因的过滤和质量剪切等优化处理;其次使用软件SOAP denovo 2.04对测序数据进行拼接组装,组装时选用的kmer参数为65;使用软件prodigal 2.6.3对基因组序列进行基因预测;最后进行数据库比对分析。通过BLAST将预测得到的基因序列与GenBank的非冗余核苷酸序列数据库(Nonredundant database, NR)进行比对(ftp://ftp.ncbi.nih.gov/ pub/taxonomy),并获得物种注释信息;最后将预测基因与EggNOG数据库(http://eggnog5.embl.de)、GO数据库(http://www.geneontology.prg/)和KEGG数据库(http://www.genome.jp/kegg/pathway.html)进行比对,获得基因功能注释。最后使用CAZY数据库(http://www.cazy.org/)的对应工具hmmscan(http://hmmer.janelia.org/search/hmmscan)将预测基因与CAZY数据库进行比对,获得基因对应的碳水化合物活性酶注释信息,并使用碳水化合物活性酶对应的基因丰度总合计算该碳水化合物活性酶的丰度。
1.6.4 数据统计与分析 试验数据以Excel 2016软件进行统计预处理,采用SPSS 23.0软件对所得数据进行正态分布检验,符合正态分布后进行单因素方差分析(One-way ANOVA),若有显著差异则采用LSD法对组间差异进行多重比较。结果以平均值和标准误表示,P<0.05表示结果差异显著,P<0.01表示结果差异极显著。
2 结果 2.1 测序结果本次瘤胃内容物样本宏基因组测序共获得1 331 858 536对原始序列数(Raw Reads);双端Reads质控、拼接后共产生1 324 887 370条高质量序列数(Clean Reads),GC平均含量为52%,说明测序量和测序深度合理,测序结果可信。
2.2 Alpha多样性分析从表 2可知,5 km组和10 km组的瘤胃微生物Shannon、Simpson和Chao1指数均高于NC组,但三组之间无显著差异(P>0.05)。
|
|
表 2 Alpha多样性指数分析 Table 2 Alpha diversity index analysis |
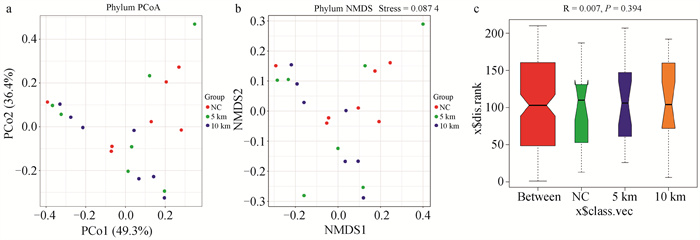
通过主坐标分析(PCoA)发现,NC组和5 km组瘤胃微生物区系分布较散乱,没有规律性,而10 km组聚类相对较规律,说明10 km组中瘤胃微生物相似性高(图 1、2)。NMDS分析结果Stress=0.087 4和0.098 3 < 0.2,表明NMDS分析具有一定的可靠性。相似性分析(ANOSIM)结果显示,5 km组、10 km组和NC组间R值的范围为[-1, 1],P值为0.394和0.159,说明组间差异大于组内差异,但差异不显著(P>0.05)。
|
a. PCoA分析;b. NMDS分析;c. ANOSIM分析 a. PCoA analysis; b. NMDS analysis; c. ANOSIM analysis 图 1 门水平Beta多样性分析 Fig. 1 Beta diversity analysis at phylum level |

|
a. PCoA分析;b. NMDS分析;c. ANOSIM分析 a. PCoA analysis; b. NMDS analysis; c. ANOSIM analysis 图 2 属水平Beta多样性分析 Fig. 2 Beta diversity analysis at genus level |
不同运动量条件下滩羊瘤胃细菌蛋白质编码基因来源主要的菌门有拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria),分别占据36.85%、26.32%和22.42%(表 3)。试验组乳酸杆菌门和蓝藻门的相对丰度显著高于NC组(P < 0.05)。此外,将不同处理中相对丰度排名前20的物种用柱状图和热图展示(图 3),发现在门水平上,10 km组中物种数最多,试验组和NC组共有物种数是5个,且三组各有1个特有物种。
|
|
表 3 不同运动量对滩羊瘤胃菌群门水平相对丰度的影响 Table 3 Effects of different exercise amounts on the relative abundance of rumen flora at phylum level of Tan sheep |

|
a.门水平组间物种柱状图;b.门水平韦恩图;c.门水平热图。lw1~21为瘤胃样品编号,其中lw2、4、6、8、13、14、16为NC组样品,lw5、7、10、11、15、17、21为5 km组样品,lw1、3、9、12、18、19、20为10 km组样品。下同 a. Column stacking chart of species among groups; b. Venn diagram at phylum level; c. Heatmap at phylum level.lw1-21 are the number of rumen samples, in which lw2, 4, 6, 8, 13, 14, 16 belong to NC group, lw5, 7, 10, 11, 15, 17, 21 belong to 5 km group, lw1, 3, 9, 12, 18, 19, 20 belong to 10 km group. The same as below 图 3 瘤胃菌群门水平组间物种统计图 Fig. 3 Chart of rumen flora species among groups at phylum level |
瘤胃微生物属水平上相对丰度大于0.5%的物种列于表 4。属水平上的普雷沃氏菌属(Prevotella)、硒单胞菌属(Selenomonas)、乳杆菌属(Lactobacillus)、小杆菌属(Dialister)和链霉菌属(Streptomyces)的相对丰度较高,分别占15.63%、8.31%、1.45%、1.84%和2.20%,但组间差异均不显著(P>0.05)。试验组的瘤胃球菌属(Ruminococcus)和乳杆菌属(Lactobacillus)相对丰度显著高于NC组(P < 0.05)。样品共有物种分析结果显示(图 4),在属水平上5 km组、10 km组和NC组有24个共有微生物菌,10 km组存在3种特有菌且10 km组菌的多样性依次高于5 km组和NC组,进一步说明运动能够增加滩羊瘤胃微生物的多样性。
|
|
表 4 不同运动量对滩羊瘤胃菌群属水平相对丰度的影响 Table 4 Effects of different exercise amounts on the relative abundance of rumen flora at genus level of Tan sheep |

|
A. 属水平组间物种柱状图;B.属水平韦恩图;C.属水平热图 A. Column stacking chart of species between groups; B. Venn diagram at genus level; C. Heatmap at genus level 图 4 瘤胃菌群属水平组间物种统计图 Fig. 4 Chart of rumen flora species among groups at genus level |
经hmmscan分析,将基因比对到CAZy数据库中,结果如表 5所示,5 km和10 km组的糖苷水解酶(GHs)、碳水化合物酯酶(CEs)、碳水化合物结合模块(CBM)和辅助活性酶系(AAs)丰度均增加。5 km组的糖基转移酶(GTs)相对丰度增加,多聚体裂解酶(PLs)的相对丰度下降;10 km组的多聚体裂解酶(PLs)相对丰度增加,糖基转移酶(GTs)相对丰度下降。
|
|
表 5 不同运动量对滩羊瘤胃微生物CAZy酶类相对丰度的影响 Table 5 Effects of different exercise amounts on the relative abundance of CAZy enzymes in the rumen of Tan sheep |
线性判别分析(LDA)结果显示,10公里组糖苷水解酶家族中GH43-17和GH34,糖水化合物脂酶类家族中CE-19的功能具有显著差异;5 km组糖苷水解酶家族中GH102的功能具有显著差异,差异功能的LDA值分布如图 5A和图 5B所示。

|
A.10公里组;B.5公里组 A.10 km group; B.5 km group 图 5 差异功能的LDA值分布图 Fig. 5 Distribution of LDA values for difference function |
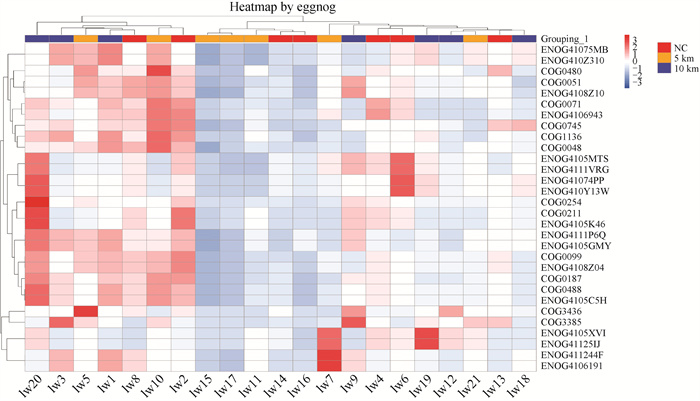
对得到的基因进行EggNOG功能分类预测。发现碳水化合物转运及代谢显著富集,并基于比对获得的COG相对丰度表,筛选出相对丰度Top 30的COG,如图 6所示。
|
图 6 EggNOG热图 Fig. 6 Heatmap by EggNOG |
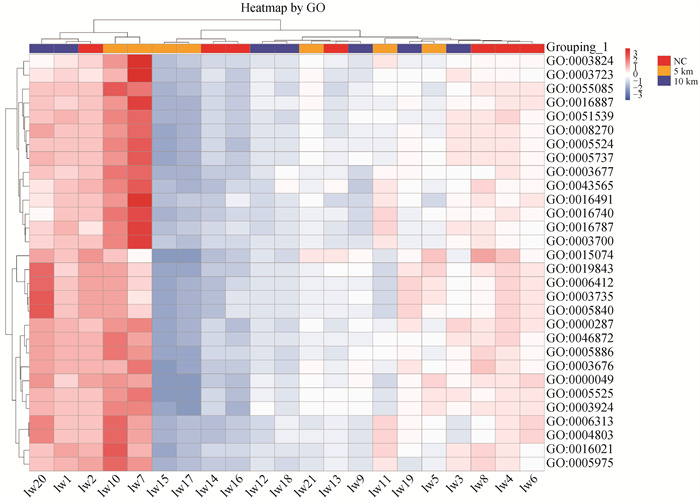
在GO分类中,统计基因在生物过程(biological process, BP)细胞成分(cellular component, CC)和分子功能(molecular function, MF)3个GO类别中。在BP类中,基因多集中在代谢、细胞加工过程;在CC类中,集中在细胞组分的基因居多;在MF类中,集中在结合以及催化功能的基因最多(图 7)。
|
图 7 前30个GO条目热图 Fig. 7 Heatmap by top 30 GO terms classification |
Level 2层级上相对丰度最高的通路富集分析结果表明,10 km组中主要富集到脂肪酸生物合成,丙酮酸盐代谢及细菌趋化性等通路;5 km组中主要富集到双组分系统、碳代谢、辅助因子的代谢、丙酮酸代谢、淀粉和蔗糖代谢、丙酸代谢、同源重组、脂肪酸生物合成、原核生物中的碳固定途径、肽聚糖生物合成、鞭毛组装和脂肪酸代谢通路。

|
a.10公里组;b. 5公里组 a. 10 km group; b. 5 km group 图 8 KEGG通路富集分析 Fig. 8 KEGG enrichment analysis bubble chart |
众所周知,运动对动物心肺健康、肌肉力量、葡萄糖代谢、免疫系统和心理健康都有积极的影响[18]。前期研究表明运动能够改善滩羊体内造血功能、促进免疫细胞的激活,有利于增强机体免疫功能[19]。王柏辉[20]研究发现,放牧组的苏尼特羊瘤胃和肠道微生物的差异明显大于舍饲组,特别是普氏菌属、理研菌属、拟杆菌属、丁酸弧菌属和奎因氏菌属的含量增高,而舍饲组琥珀酸弧菌属的含量最多。孙国平[21]发现,与自然放牧育肥相比,舍饲育肥能明显提高成熟母羊瘤胃液中的琥珀酸丝状杆菌和原虫数量,产甲烷菌数量呈明显上升趋势,而对总细菌、瘤胃球菌数量无显著影响。这些结果证实了运动与微生物种群之间的关联,揭示了微生物区系对运动成绩、康复和疾病模式的各种指数的影响是通过肌动因子和其他细胞因子发出信号,调节下丘脑-垂体-肾上腺轴,进而影响与运动相关的代谢途径,但未确定运动量与菌群改变的因果关系。本研究首次探讨了滩羊采食后运动5 km和10 km对其瘤胃菌群多样性的影响。
3.2 不同运动量对滩羊瘤胃菌群Alpha和Beta多样性的影响Alpha多样性指标用于反映特定样本或单个群落中物种的丰富度和均匀度[22]。Simpson和Chao1反映微生物群落的丰富度[23]。在本试验中,5 km组和10 km组中两者的数值较NC组均升高,说明适当的增加运动量会增加瘤胃微生物的丰富度,这与Guo等[13]的研究结果相似。Shannon指数反映微生物群落的均匀度,5 km组和10 km组值高于NC组,说明增加运动量会影响瘤胃微生物群落的均匀度。Beta多样性分析主要用于比较各样本中微生物群落整体结构的差异,通过PCoA分析发现,10 km组菌群坐标值更趋近,而5 km组和NC组显示较分散,可见每日运动10 km可提高滩羊个体间肠道微生物相似性,表明滩羊每日运动10 km有利于滩羊肠道微生物群落的稳定。
3.3 不同运动量对滩羊瘤胃微生物组成的影响研究表明,反刍动物瘤胃微生物组成复杂多样,其中厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)在成年反刍动物瘤胃菌群中的门水平上占比最高[24-25]。本试验共发现14个门的相对丰度在0.1%以上,其中拟杆菌门、厚壁菌门和变形菌门(Proteobacteria)这3个门的相对丰度最高,分别占据36.85%、26.32%和22.42%。拟杆菌门作为第一优势菌门在NC组中的相对丰度为42.43%,数值上高于试验组中的30.92%和37.2%,这与Jami和Mizrahi[26]在奶牛瘤胃中的研究结果相一致。此外,滩羊瘤胃中的微生物大部分为细菌界,其中瘤胃球菌产生的纤维素酶能降解饲草料中的木质纤维素,被认为是瘤胃中降解木质素的主要微生物,而瘤胃普雷沃菌科能够大量降解木聚糖及果胶[27-28]。本试验中5 km和10 km组中瘤胃球菌数显著高于NC组,在5 km组值最高,随着运动量的增加瘤胃球菌属的数量又逐渐降低,说明运动量为5 km时动物机体对木质素的降解效果最好。与NC组相比,5 km和10 km组中的瘤胃普雷沃菌属的含量有所升高,由此推测,增加运动量能够促进机体对木聚糖及果糖的降解。之前的研究表明运动可以加大对碳水化合物的利用,为机体提供营养物质[29]。
厚壁菌门是促进动物瘤胃微生物分解纤维素最主要的菌群[30-31],5 km组和10 km组中厚壁菌门的丰度高于NC组,说明运动可提高瘤胃消化纤维素的能力。本试验发现,变形菌门的相对丰度在门水平上仅次于拟杆菌门和厚壁菌门,但含量远低于这两种门,这与前人的研究一致[32]。Pitta等[33]发现,变形菌门在瘤胃蛋白质降解、淀粉降解和致病菌的形成中起着重要作用,本试验中的5 km组和10 km组变形菌门的相对丰度低于NC组,说明运动在一定程度上能抑制致病菌的繁殖。疣微菌门(Verrucomicrobia)是影响哺乳动物免疫功能的重要因素,其丰度的降低与机体免疫功能下降有关[34],10 km组中疣微菌门的相对丰度高于5 km组和NC组,且NC组的丰度最低,说明运动可增强机体免疫功能。
在属水平上发现11个丰度在1%以上的属,其中以普雷沃菌属(Prevotella)和硒单胞菌属(Selenomonas)的相对丰度最高,分别为15.63%和8.31%。双歧杆菌能在胃肠道建立具有保护力的真菌膜且代谢产物过氧化氢等具有一定的抗菌能力[35-36],能够有效抵抗病原菌的入侵和定植。本试验中5 km组和10 km组双歧杆菌丰度均高于NC组,说明运动可能会增加滩羊瘤胃中的益生菌数量,并抑制有害菌的增殖。Ze等[37]研究发现,瘤胃球菌属(Ruminococcus)在不同饮食结构中的相对丰度存在显著性差异,并发现该属参与瘤胃纤维素和淀粉的降解,本试验中5 km组和10 km组瘤胃球菌属相对丰度显著高于NC组,由此可推测运动能够通过促进瘤胃球菌属的繁殖,从而增强机体对纤维素和淀粉的降解能力。样品共有物种分析结果显示,10 km组富集到的物种数依次高于5 km组和NC组,该结果表明,运动能够提高滩羊瘤胃微生物的多样性。
3.4 不同运动量对滩羊瘤胃微生物功能的影响碳水化合物的合成与分解是一个复杂的过程[38]。瘤胃微生物菌群是CAZy的重要资源,不但能将纤维素转化为反刍动物可利用的生物能源,而且在机体碳水化合物代谢、蛋白质糖基化和生态环境中都有着重要作用[39-40]。CAZy酶可分为6个功能基团,其中葡萄糖苷酶(GH)、碳水化合物酯酶(CE)和多糖裂解酶(PL)能协同降解木质纤维素物质[41-42]。糖苷水解酶(GHs)家族中的GH10、GH26和GH28具有半纤维素酶活性,能降解少量的木糖[43],而碳水化合物酯酶(CEs)中的CE8家族的酶具有果胶甲基酯酶活性,可脱去果胶中的甲酯基团,生成果胶酸[44]。试验组的糖苷水解酶(GHs)、碳水化合物酯酶(CEs)、碳水化合物模块(CBM)和辅助活性酶系(AAs)丰度均增加,表明增加运动量,改变了机体内碳水化合物酶多样性。Lynd等[45]研究发现,碳水化合物结合模块(CBM)是纤维素酶的重要组成部分,它通过与多糖基体结合增加底物浓度和活力的方式促进酶与纤维素的结合,从而促进纤维素的降解。本试验结果显示,5 km和10 km中CBM和辅助模块酶类数值均高于NC组,表明运动能够提高滩羊降解纤维素的能力。这与本试验中瘤胃球菌属的研究结果类似。在KEGG结果中,5 km组和10 km组的差异表达基因显著富集在氨基酸代谢、丙酮酸代谢和脂肪酸生物合成等通路中,说明运动有助于增强CAZy酶的活力,并影响反刍动物瘤胃微生物对碳水化合物的运输代谢、能量生成和辅酶转运等过程。
4 结论在本试验条件下,滩羊运动量的增加能够提高瘤胃微生物的丰富度和多样性,影响瘤胃球菌属(Ruminococcus)和乳杆菌属(Lactobacillus)等菌属的相对丰富度,但对滩羊瘤胃微生物的均匀度没有显著影响。
| [1] |
马友记, 李发弟. 中国养羊业现状与发展趋势分析[J]. 中国畜牧杂志, 2011, 47(14): 16-20. MA Y J, LI F D. Analysis of current situation and development trend of sheep industry in China[J]. Chinese Journal of Animal Science, 2011, 47(14): 16-20. (in Chinese) |
| [2] |
EISLER M C, LEE M R F, TARLTON J F, et al. Agriculture: Steps to sustainable livestock[J]. Nature, 2014, 507(7490): 32-34. DOI:10.1038/507032a |
| [3] |
HOOPER L V, LITTMAN D R, MACPHERSON A J. Interactions between the microbiota and the immune system[J]. Science, 2012, 336(6086): 1268-1273. DOI:10.1126/science.1223490 |
| [4] |
TREMAROLI V, BÄCKHED F. Functional interactions between the gut microbiota and host metabolism[J]. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [5] |
王禄禄, 王立志, 周美丽. 宏基因组学技术在反刍动物瘤胃微生态系统上的应用研究进展[J]. 中国微生态学杂志, 2017, 29(2): 223-228. WANG L L, WANG L Z, ZHOU M L. Application of metagenomics technology on the microecological system in rumen of ruminant: research progress[J]. Chinese Journal of Microecology, 2017, 29(2): 223-228. DOI:10.13381/j.cnki.cjm.201702028 (in Chinese) |
| [6] |
REY M, ENJALBERT F, COMBES S, et al. Establishment of ruminal bacterial community in dairy calves from birth to weaning is sequential[J]. J Appl Microbiol, 2014, 116(2): 245-257. DOI:10.1111/jam.12405 |
| [7] |
SARO C, HOHENESTER U M, BERNARD M, et al. Effectiveness of interventions to modulate the rumen microbiota composition and function in pre-ruminant and ruminant lambs[J]. Front Microbiol, 2018, 9: 1273. DOI:10.3389/fmicb.2018.01273 |
| [8] |
ZHANG Y D, CHENG J, ZHENG N, et al. Different milk replacers alter growth performance and rumen bacterial diversity of dairy bull calves[J]. Livest Sci, 2020, 231: 103862. DOI:10.1016/j.livsci.2019.103862 |
| [9] |
姚喜喜, 王伟, 徐成体, 等. 饲养方式对大通牦牛瘤胃组织结构和瘤胃菌群多样性和组成的影响[J]. 动物营养学报, 2022, 34(6): 3777-3787. YAO X X, WANG W, XU C T, et al. Effects of feeding pattern on rumen tissue structure and rumen bacterial community diversity and composition of Datong yaks (Bos grunniens)[J]. Chinese Journal of Animal Nutrition, 2022, 34(6): 3777-3787. (in Chinese) |
| [10] |
张洁, 李娜, 郭婷婷, 等. ITS rDNA高通量测序研究不同饲养方式对滩羊瘤胃真菌菌群的影响[J]. 甘肃农业大学学报, 2019, 54(5): 25-34. ZHANG J, LI N, GUO T T, et al. Effects of different feeding methods on rumen fungus flora of Tan sheep by high-throughput sequencing of ITS rDNA[J]. Journal of Gansu Agricultural University, 2019, 54(5): 25-34. (in Chinese) |
| [11] |
DENOU E, MARCINKO K, SURETTE M G, et al. High-intensity exercise training increases the diversity and metabolic capacity of the mouse distal gut microbiota during diet-induced obesity[J]. Am J Physiol Endocrinol Metab, 2016, 310(11): E982-E993. DOI:10.1152/ajpendo.00537.2015 |
| [12] |
EVANS C C, LEPARD K J, KWAK J W, et al. Exercise prevents weight gain and alters the gut microbiota in a mouse model of high fat diet-induced obesity[J]. PLoS One, 2014, 9(3): e92193. DOI:10.1371/journal.pone.0092193 |
| [13] |
KANG S S, JERALDO P R, KURTI A, et al. Diet and exercise orthogonally alter the gut microbiome and reveal independent associations with anxiety and cognition[J]. Mol Neurodegener, 2014, 9: 36. DOI:10.1186/1750-1326-9-36 |
| [14] |
HUANG C, GE F, YAO X X, et al. Microbiome and metabolomics reveal the effects of different feeding systems on the growth and ruminal development of yaks[J]. Front Microbiol, 2021, 12: 682989. DOI:10.3389/fmicb.2021.682989 |
| [15] |
LIU H J, XU T W, XU S X, et al. Effect of dietary concentrate to forage ratio on growth performance, rumen fermentation and bacterial diversity of Tibetan sheep under barn feeding on the Qinghai-Tibetan plateau[J]. PeerJ, 2019, 7: e7462. DOI:10.7717/peerj.7462 |
| [16] |
KRAUSE T R, LOURENCO J M, WELCH C B, et al. The relationship between the rumen microbiome and carcass merit in Angus steers[J]. J Anim Sci, 2020, 98(9): skaa287. DOI:10.1093/jas/skaa287 |
| [17] |
MARTINEZ-FERNANDEZ G, JIAO J Z, PADMANABHA J, et al. Seasonal and nutrient supplement responses in rumen microbiota structure and metabolites of tropical rangeland cattle[J]. Microorganisms, 2020, 8(10): 1550. DOI:10.3390/microorganisms8101550 |
| [18] |
廖沁, 曾来生, 陈杨, 等. 基于16S rRNA测序分析游泳运动对小鼠肠道菌群的影响[J]. 江西医药, 2022, 57(5): 437-443. LIAO Q, ZENG L S, CHEN Y, et al. Bacterial flora composition of swimming exercise mice based on 16S rRNA sequencing[J]. Jiangxi Medical Journal, 2022, 57(5): 437-443. (in Chinese) |
| [19] |
陈鹏飞. 不同运动量对滩羊生产性能和瘤胃微生物菌群的影响[D]. 兰州: 甘肃农业大学, 2022. CHEN P F. Effects of different exercise levels on production performance and rumen microflora of Tan sheep[D]. Lanzhou: Gansu Agricultural University, 2022. |
| [20] |
王柏辉. 饲养方式对苏尼特羊胃肠道菌群、脂肪酸代谢和羊肉品质的影响及机理研究[D]. 呼和浩特: 内蒙古农业大学, 2019. WANG B H. Effects of feeding regimens on gastrointestinal microbiota, fatty acid metabolism and meat quality of Sunit sheep and its underlying mechanism[D]. Hohhot: Inner Mongolia Agricultural University, 2019. |
| [21] |
孙国平. 两种育肥方式下绒山羊瘤胃发酵及几种瘤胃微生物数量的比较研究[D]. 呼和浩特: 内蒙古农业大学, 2013. SUN G P. Comparative study of two fattening ways on rumen fermentation and the number of several rumen microorganisms of cashmere goats[D]. Hohhot: Inner Mongolia Agricultural University, 2013. |
| [22] |
LOZUPONE C A, KNIGHT R. Species divergence and the measurement of microbial diversity[J]. FEMS Microbiol Rev, 2008, 32(4): 557-578. DOI:10.1111/j.1574-6976.2008.00111.x |
| [23] |
DENG R, CHEN X, QIU L P, et al. Bacterial community structure and diversity in the aqueous environment of Shihou Lake and its relationship with environmental factors[J]. Indian J Microbiol, 2021, 61(4): 475-486. DOI:10.1007/s12088-021-00974-y |
| [24] |
QIU Q H, GAO C Y, AZIZ UR RAHMAN M, et al. Digestive ability, physiological characteristics, and rumen bacterial community of Holstein finishing steers in response to three nutrient density diets as fattening phases advanced[J]. Microorganisms, 2020, 8(3): 335. DOI:10.3390/microorganisms8030335 |
| [25] |
JAMI E, ISRAEL A, KOTSER A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. ISME J, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [26] |
JAMI E, MIZRAHI I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [27] |
FLINT H J, BAYER E A. Plant cell wall breakdown by anaerobic microorganisms from the Mammalian digestive tract[J]. Ann N Y Acad Sci, 2008, 1125(1): 280-288. DOI:10.1196/annals.1419.022 |
| [28] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria: potential for new insights from genomic analysis[J]. Nat Rev Microbiol, 2008, 6(2): 121-131. DOI:10.1038/nrmicro1817 |
| [29] |
TIPTON K D, WOLFE R R. Exercise, protein metabolism, and muscle growth[J]. Int J Sport Nutr Exerc Metab, 2001, 11(1): 109-132. DOI:10.1123/ijsnem.11.1.109 |
| [30] |
LA REAU A J, SUEN G. The ruminococci: key symbionts of the gut ecosystem[J]. J Microbiol, 2018, 56(3): 199-208. DOI:10.1007/s12275-018-8024-4 |
| [31] |
BABA Y, MATSUKI Y, MORI Y, et al. Pretreatment of lignocellulosic biomass by cattle rumen fluid for methane production: bacterial flora and enzyme activity analysis[J]. J Biosci Bioeng, 2017, 123(4): 489-496. DOI:10.1016/j.jbiosc.2016.11.008 |
| [32] |
FERNANDO S C, PURVIS Ⅱ H T, NAJAR F Z, et al. Rumen microbial population dynamics during adaptation to a high-grain diet[J]. Appl Environ Microbiol, 2010, 76(22): 7482-7490. DOI:10.1128/AEM.00388-10 |
| [33] |
PITTA D W, INDUGU N, KUMAR S, et al. Metagenomic assessment of the functional potential of the rumen microbiome in Holstein dairy cows[J]. Anaerobe, 2016, 38: 50-60. DOI:10.1016/j.anaerobe.2015.12.003 |
| [34] |
GHARECHAHI J, VAHIDI M F, BAHRAM M, et al. Metagenomic analysis reveals a dynamic microbiome with diversified adaptive functions to utilize high lignocellulosic forages in the cattle rumen[J]. ISME J, 2021, 15(4): 1108-1120. DOI:10.1038/s41396-020-00837-2 |
| [35] |
ORRHAGE K, NORD C E. Bifidobacteria and lactobacilli in human health[J]. Drugs Exp Clin Res, 2000, 26(3): 95-111. |
| [36] |
PARK J H, UM J I, LEE B J, et al. Encapsulated Bifidobacterium bifidum potentiates intestinal IgA production[J]. Cell Immunol, 2002, 219(1): 22-27. DOI:10.1016/S0008-8749(02)00579-8 |
| [37] |
ZE X, BEN DAVID Y, LAVERDE-GOMEZ J A, et al. Unique organization of extracellular amylases into amylosomes in the resistant starch-utilizing human colonic Firmicutes bacterium Ruminococcus bromii[J]. mBio, 2015, 6(5): e01058-15. |
| [38] |
CANTAREL B L, COUTINHO P M, RANCUREL C, et al. The carbohydrate-active enzymes database (CAZy): an expert resource for Glycogenomics[J]. Nucleic Acids Res, 2009, 37(S1): D233-D238. |
| [39] |
JIANG B W, WANG T, ZHOU Y X, et al. Effects of enzyme+bacteria treatment on growth performance, rumen bacterial diversity, KEGG pathways, and the CAZy spectrum of Tan sheep[J]. Bioengineered, 2020, 11(1): 1221-1232. DOI:10.1080/21655979.2020.1837459 |
| [40] |
PATEL D D, PATEL A K, PARMAR N R, et al. Microbial and carbohydrate active enzyme profile of buffalo rumen metagenome and their alteration in response to variation in the diet[J]. Gene, 2014, 545(1): 88-94. DOI:10.1016/j.gene.2014.05.003 |
| [41] |
FLINT H J, SCOTT K P, DUNCAN S H, et al. Microbial degradation of complex carbohydrates in the gut[J]. Gut Microbes, 2012, 3(4): 289-306. |
| [42] |
YOUSSEF N H, COUGER M B, STRUCHTEMEYER C G, et al. The genome of the anaerobic fungus Orpinomyces sp. strain C1A reveals the unique evolutionary history of a remarkable plant biomass degrader[J]. Appl Environ Microbiol, 2013, 79(15): 4620-4634. DOI:10.1128/AEM.00821-13 |
| [43] |
ZHANG H M, LI J F, WANG J Q, et al. Determinants for the improved thermostability of a mesophilic family 11 xylanase predicted by computational methods[J]. Biotechnol Biofuels, 2014, 7(1): 3. DOI:10.1186/1754-6834-7-3 |
| [44] |
LINHARDT R J, GALLIHER P M, COONEY C L. Polysaccharide lyases[J]. Appl Biochem Biotechnol, 1987, 12(2): 135-176. |
| [45] |
LYND L R, WEIMER P J, VAN ZYL W H, et al. Microbial cellulose utilization: fundamentals and biotechnology[J]. Microbiol Mol Biol Rev, 2002, 66(3): 506-577. |
(编辑 范子娟)



