济宁青山羊(Jining gray goat)属于地方羔皮用山羊品种,其嘴唇、角、被毛和蹄均为青色,前膝为黑色,具有“四青一黑”的特征。济宁青山羊是我国乃至世界上优异的种质资源,以其全年发情、多胎多羔、羔皮品质好、遗传性稳定、耐粗抗病等品种特性而著称,但其生长速度慢,加上近几十年来,猾子皮市场的下滑和肉羊市场的兴起,各地盲目引入其他品种进行改良,致使纯种数量急剧下降。目前,该品种优质种公羊主要由保种场、养殖企业和散养农户提供,然而,经过长期封闭繁殖,可能会造成近交程度的上升,对该品种的生存与保护带来严重威胁[1-2]。
动物的家系结构、亲缘关系模糊、错误的系谱记录以及随意的选育选配会降低该品种的遗传多样性,造成一定程度的近交,有效群体数量的减少,进而影响对该品种的提纯复壮、繁殖更新[3]。单核苷酸多态性(SNP)主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。SNP标记与其他DNA分子标记相比具有不可比拟的优势,SNP具有数量多、分布广、突变率低、遗传稳定性高、多态性丰富、准确性较高、成本低等优点[4]。当前全基因组SNP芯片在研究动物的遗传多样性、亲缘关系、家系结构等方面得到了广泛的应用[5]。济宁青山羊是我国珍贵的山羊遗传资源,在养羊生产和种质创新中做出了重要贡献,已实现了多点小群体活体保种,但仍需要对保种效果进行检测和评价[6-10]。为了了解济宁青山羊保种群体的保种效果,本研究利用Illumina 70 K Goat SNP芯片对40只济宁青山羊全基因组范围内的SNP进行检测,分析群体遗传多样性、近交程度,厘清家系结构、亲缘关系等,为济宁青山羊的有效保护与合理开发利用提供指导。
1 材料与方法 1.1 试验动物本研究所用40只济宁青山羊的耳组织样均采自山东省济宁青山羊原种场。所有试验羊均为成年羊,其中公羊23只(对原种场内公羊全部进行了采样,基本包含了整个保种群体的血缘),母羊17只。将采集的耳组织放置含有无水乙醇的冻存管中,记录样品的个体编号,24 h内带回实验室,-20 ℃冻存。
1.2 试验方法1.2.1 济宁青山羊全基因组DNA的提取 利用基因组DNA提取试剂盒(Vazyme)对每只山羊耳组织进行基因组DNA提取。利用超微量紫外分光光度仪Nano Drop ND-2000和琼脂糖凝胶电泳试验来检测基因组DNA的浓度和纯度,判断其质量是否合格。具体检测方法如下:1)使用量程为2.5 μL的移液枪,从提取的DNA样品中吸取1 μL加入至Nano Drop ND-2000超微量紫外分光光度计的加样孔中,再将其调至到DNA测量选项进行检测。2)配置1%的琼脂胶,在琼脂胶板滴孔中滴入4 μL PCR扩增体系,在电压为120 V的电泳槽中电泳25 min,在UV凝胶电泳成像系统下拍照检测电泳结果。
1.2.2 SNP分型及芯片数据质量控制 将提取合格的DNA样品送至纽勤生物科技(上海)有限公司,进行基因型分型,本研究所有样本都使用Illumina 70 K Goat SNP芯片进行基因型分型。该芯片由内蒙古农业大学开发,是一款中密度SNP分型芯片,利用Illumina平台定制,同时兼容了Illumina山羊52 K SNP芯片位点,最终保留了67 088个高质量SNPs,集成一款全新的山羊70 K SNP芯片。利用Plink(V1.90)软件对基因型数据进行质控,只保留分型质量最好的位点进行后续分析。质控标准为:只使用常染色体上的位点,SNP检出率大于等于90%,个体检出率大于等于90%,哈迪温伯格P值大于等于0.000 001,最小等位基因频率(minor allele frequency, MAF)大于等于0.01。
1.2.3 济宁青山羊保种群体遗传多样性分析 通过Plink(V1.90)软件计算各位点的遗传多样性参数,包括有效等位基因数、最小等位基因频率(MAF)、期望杂合度(He)、观测杂合度(Ho)、多态信息含量(PIC)和多态性比例标记(PN),以此来了解济宁青山羊群体的遗传多样性。
PN指的是在目标群体中表现为多态的位点占总位点的比例。首先使用Plink软件计算出每个位点的最小等位基因频率(MAF),然后使用R脚本计算PN,利用如下公式计算PN。
| $ P_N=\frac{M}{N} $ |
公式中,M为表现多态的位点数,N为总的位点数。
He指的是群体中任一个体的任一位点是杂合的概率;Ho指的是群体中某一位点是杂合子的个体数占总个体数的比例,用如下公式计算。
| $ \begin{gathered} H o=\frac{1}{N} \sum\nolimits_{k=1}^N \frac{H k}{n} \\ H e=\frac{2 n}{2 n-1} \frac{1}{N} \sum\nolimits_{k=1}^N\left(1-\sum p_{k i}^2\right) \end{gathered} $ |
其中,n为群体的总个体数,N为总的位点数,Hk为位点k的杂合个体数,Pki为位点k等位基因i的频率。
PIC是衡量基因变异程度高低、反映遗传信息多少的指标,PIC利用如下公式计算。
| $ P I C=1-\sum\nolimits_{i=1}^n p i^2-\sum\nolimits_{i=1}^{n-1} \sum\nolimits_{j=i+1}^n 2 p i^2 p j^2 $ |
公式中Pi和Pj分别为第i个和第j个等位基因频率,n为等位基因数。
有效等位基因数是指在理想群体中,一个基因座上产生与实际群体中相同的纯合度所需的等位基因数,它等于实际群体的纯合度的倒数。MAF是指在给定群体中的不常见的等位基因发生频率。
1.2.4 济宁青山羊保种群体亲缘关系分析 利用GCTA工具构建G矩阵,采用Plink(V1.90)软件构建状态同源(IBS)距离矩阵,分析保种群体个体间的亲缘关系,并使用R软件绘制IBS距离矩阵和G矩阵结果的热图。
1.2.5 济宁青山羊保种群体的家系结构分析 利用Plink(V1.90)软件对质控后的数据进行主成分分析,采用R语言脚本绘制主成分的二维图,分析被测济宁青山羊的群体结构,实现群体分层可视化。
1.2.6 基于ROH的近交程度分析 使用Plink(V1.90)计算得到每个样本的ROH长度,基于ROH的近交系数是通过计算个体中ROH片段的总长度占常染色体基因组总长的比例。因此,个体中ROH的总长度越长或数量越多,该个体的近交系数就越高。
| $ F_{\rm{ROH}}=\frac{\sum\nolimits_K { Length }\left({ROH}_k\right)}{L} $ |
其中,k是个体中ROH的数量,L是基因型数据覆盖的常染色体基因组的长度(山羊的常染色体基因组长约为2 468 749.279 kb)。
2 结果 2.1 济宁青山羊全基因组DNA提取质量检测结果利用超微量紫外分光光度仪Nano Drop ND-2000和凝胶电泳试验来检测基因组DNA的浓度和纯度,判断其质量是否合格,表 1为部分济宁青山羊DNA样本纯度检测结果,A260 nm/A280 nm的比值在1.8~2.0间。图 1显示,DNA条带单一,表明DNA纯度高,质量合格,可用于后续分析。
|
|
表 1 济宁青山羊DNA样本纯度检测结果 Table 1 The test results of DNA purity of Jining gray goats |
|
图 1 基因组DNA琼脂糖凝胶电泳结果 Fig. 1 Genomic DNA detected by agarose gel electrophoresis |
SNP芯片共检测到了67 088个SNPs,个体基因型检出率为98%,通过Plink(V1.90)软件对数据质控,剩余的有效SNPs有57 991个,多态性比例标记(PN)为85.5%,表明此芯片适用于济宁青山羊遗传多样性分析。经过Plink(V1.90)软件对各位点进行统计,在40个个体中共检测到了68.87个有效等位基因,平均有效等位基因数(effective number of allele)为1.697,最小等位基因频率(MAF)为0.293,各位点PIC在0~0.375之间,平均PIC为0.283;该群体平均观察杂合度(Ho)为0.409,平均期望杂合度(He)为0.418,He略高于Ho。
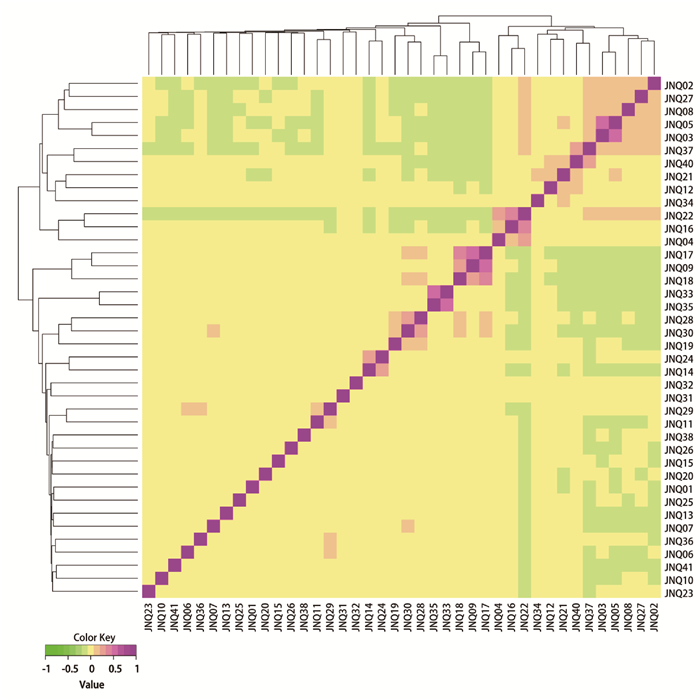
2.3 济宁青山羊保种群体亲缘关系分析通过构建G矩阵,分析被测济宁青山羊群体的亲缘关系。整个群体的G矩阵的可视化结果如图 2所示,结果表明,大部分济宁青山羊个体间的亲缘关系较远,但也有部分个体之间存在较近的亲缘关系。
|
G矩阵结果中,每个小方块代表样本两两之间的亲缘关系值,该值越大越接近红色,即两个体亲缘关系越近 In the G matrix results, each small square represents the value of the relationship between two pairs from the first sample to the last one, the larger the value, the closer it is to red, that is, the closer relationship between two individuals 图 2 G矩阵可视化结果 Fig. 2 G matrix visualization results |
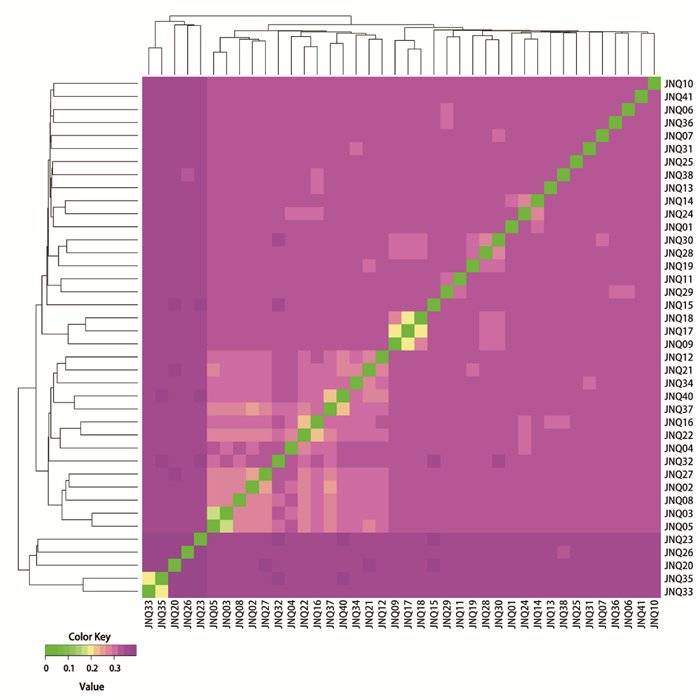
通过构建IBS距离矩阵,研究比较了40只济宁青山羊之间的遗传距离,结果显示被测济宁青山羊保种群体的IBS值在0.174~0.396之间,其平均遗传距离为0.333 4,表明济宁青山羊个体间差异较大,并存在较远的遗传距离。23只济宁青山羊公羊的IBS值在0.174 0~0.387 9之间,其平均遗传距离为0.330 3。整个群体的IBS距离矩阵的可视化结果如图 3所示,大部分被测济宁青山羊个体间的亲缘关系较远,部分个体间存在较近的亲缘关系。
|
IBS距离矩阵中,每个小方块代表样本两两之间的遗传距离值,该值越大越接近红色,即两个个体的遗传距离越大,反之亦然 Each small square in IBS distance matrix represents the genetic distance value between two pairs from the first sample to the last one, the larger the value, the closer it is to red, that is, the larger the genetic distance between two individuals, vice versa 图 3 IBS距离矩阵可视化结果 Fig. 3 The visualization results of IBS distance matrix |
从主成分分析图 4可以看出,整个济宁青山羊群体中个体间的亲缘关系不太紧密,以第一主成分为横轴,以第二主成分为纵轴,左上侧的JNQ17、JNQ18、JNQ09聚在一块,右上侧的JNQ04、JNQ12聚在一块,JNQ16、JNQ21聚在一块,JNQ22、JNQ02、JNQ08、JNQ03、JNQ05聚在一块,其他公羊聚在一块,表明该济宁青山羊保种群体可能由多个家系构成。左下侧的两个个体聚在一块,说明它们之间遗传距离较远,同时可以看出部分公羊与母羊聚集一起,表明这部分的济宁青山羊遗传距离较近。

|
图中带红色标记编号的个体为公羊,黑色圆点表示母羊 The individuals with red marks in the figure are rams, and the black dots indicate ewes 图 4 主成分分析图 Fig. 4 The results of principal component analysis |
鉴于公羊对于整个保种群体的重要性,将公羊样本提取出来单独做聚类分析,以此来判断它们之间的亲缘关系远近。以公羊间分子亲缘系数0.1为标准进行聚类,将23只公羊划分为14个家系。图中相同颜色标注的样本被评估为同一个家系,在进化树分析中被聚类在较小的分类单元中。结果如图 5所示,现有的公羊样本可以分为14个家系,分别命名为家系1、家系2、家系3……家系14,分别有1、1、1、1、1、1、1、1、1、3、1、1、2、7只公羊。

|
图中数字均为公羊耳号,进化树图中用相同颜色标注的样本被评估为同一个家系 The numbers in the figure are ear number of rams. The samples marked with the same color in the evolutionary tree are evaluated as the same family 图 5 公羊样本进化树分析结果 Fig. 5 The results of the evolutionary tree analysis of the ram samples |
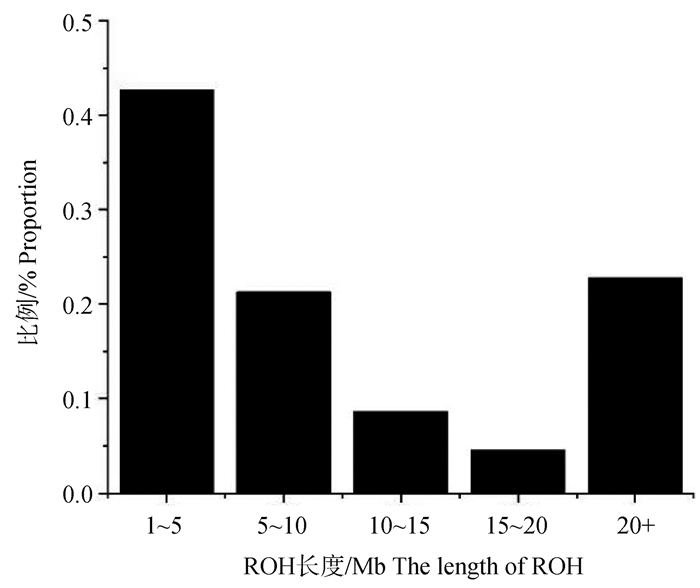
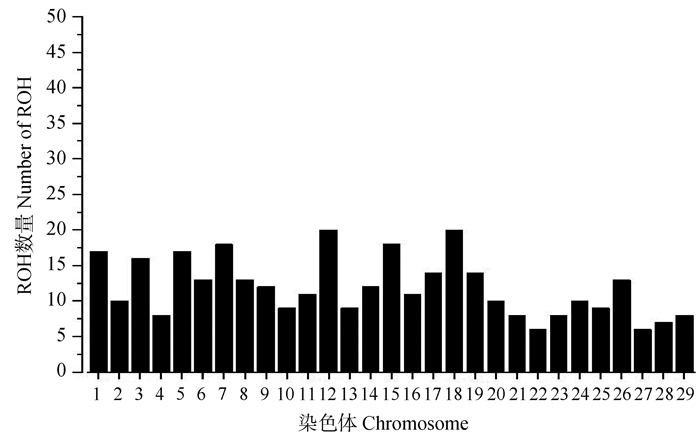
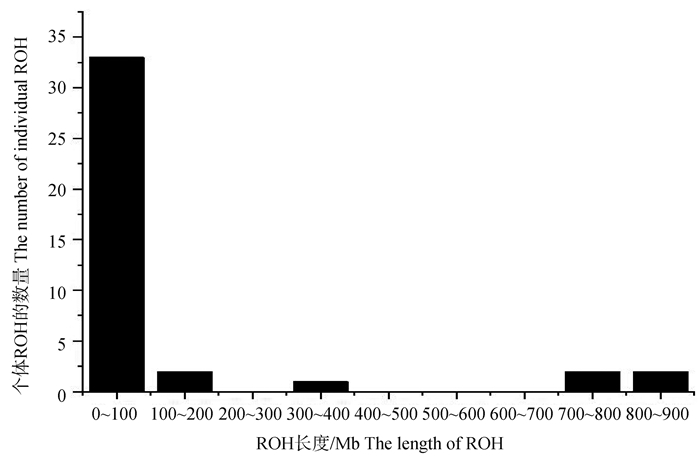
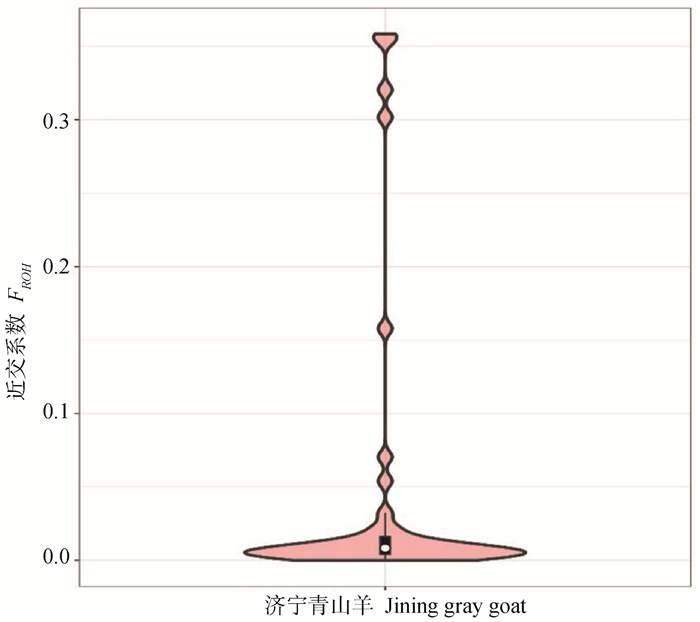
在40个样本中共鉴定出347个ROHs,其中1~5 Mb数量最多,约占42.65%(图 6),ROH片段平均覆盖基因组约11%,ROH在29条常染色体上的分布见图 7,可以看出,18号和12号染色体上的ROH数量最多,为20个,27号和22号染色体ROH数量最少,约为6个;个体ROH长度的样本数分布如图 8所示,每个济宁青山羊个体的ROH总长度在0~900 Mb,平均ROH长度为115.37 Mb,个体ROH长度在0~100 Mb内的个体数比例最大,约占33%。同时发现个体ROH长度在200~300 Mb和400~700 Mb之内的个体数很少,平均每个个体含有8.68个ROH,在没有家系记录的情况下,FROH可以作为近交估计的替代方法,该种群的平均近交系数为0.047 9,近交程度较低,群体遗传多样性丰富(图 9)。
|
图 6 济宁青山羊群体的ROH长度分布 Fig. 6 Distribution of ROH length in Jining gray goats |
|
图 7 每条染色体上的ROH数量分布 Fig. 7 The quality distribution of ROH on each chromosome |
|
图 8 济宁青山羊个体ROH长度的样本数分布 Fig. 8 Distribution of ROH length samples in Jining gray goats |
|
小提琴图主要用于展示数据的分布,中心的白色的点代表群体FROH的中位数。小提琴图的宽窄表示群体FROH的概率密度分布 The violin diagram mainly show the distribution of data, and the white point in the center represents the median of population FROH. The width of the graph indicates the probability density distribution of the population FROH 图 9 基于ROH的近交系数FROH的分布图 Fig. 9 Distribution plot of FROH based on ROH |
动物的遗传多样性研究多采用微卫星多态性(SSR)和单核苷酸多态性(SNP)的方法,但用SNP芯片分型的方法报道山羊的遗传多样性则很少[11-16]。SNP标记被证明比SSR标记有更高的准确性,已被广泛应用于动物进化研究和遗传关系鉴定等[17-22]。Stella等[23]利用山羊50 K Goat SNP芯片对来自35个国家148个群体共4 653只山羊个体,进行了种群遗传多样性研究,研究了全球山羊的种群遗传结构、群体历史选择特征、迁移路线和环境适应性等,结果表明不同品种山羊的基因组和地理环境之间存在密切联系,这为在全球范围内研究山羊适应性等奠定了基础。本研究中使用的山羊70 K SNP分型芯片由内蒙古农业大学开发,是一款中密度SNP分型芯片,利用Illumina平台定制,同时兼容了Illumina山羊52 K SNP芯片位点,最终保留67 088个高质量SNPs位点,集成一款全新的山羊70 K SNP芯片。该芯片是国内首款具有自主知识产权的山羊中密度芯片,其应用领域包括全基因组关联分析、基因组选择、群体遗传及选择进化研究、亲子鉴定、亲缘关系及种质资源鉴定等[24-26]。该款芯片在济宁青山羊群体中进行测试,结果表明基因分型成功,SNP检出率大于98%。从SNP密度分布图来看,质控前后SNP的分布较为均匀,缺失片段很少,质量合格,可用于本次试验分析。
本研究利用全基因组SNP信息从遗传多样性、亲缘关系、家系结构、近交程度等方面评估济宁青山羊保种群体的保种效果。通过杂合度、有效等位基因数、多态信息含量等参数度量群体遗传多样性。王可等[6]利用12个微卫星标记分析了济宁青山羊遗传多样性,结果发现所有微卫星标记PIC均大于0.5,平均PIC为0.73,均为高度多态标记,遗传多样性丰富;各标记的平均Ho约为0.73,平均He约为0.76;平均有效等位基因数约为4.727。在本研究中,平均Ho为0.409,平均He为0.418,各位点 PIC在0~0.375之间,平均 PIC为0.283,平均有效等位基因数约为1.697。可以看出,利用全基因组SNP信息检测的结果比利用SSR检测的结果明显降低,可能与SNP标记呈二态性有关,即SNP标记通常只能检测到两个等位基因,而SSR标记可以检测到较多的等位基因[27-31]。He略高于Ho,表明济宁青山羊保种群可能存在杂合子缺失,同时也可能有小部分引进了外来血缘,需要继续加以纯化,这一结果与王可等[6]利用微卫星方法得出的结果一致。
血统对动物遗传学和育种研究至关重要。在养殖生产中,通常根据系谱鉴定畜禽的身份信息,在养殖规模较小时,利用系谱来鉴定畜禽亲子关系准确性较好,但是随着养殖业的发展工厂化、规模化、智能化、数字化,这种传统记录方式在现代畜牧生产中存在许多弊端,无法确定亲子关系[32-34]。研究表明,可以通过遗传关系和个体间的遗传距离来构建家系[35-36],袁娇等[37]利用“中芯一号”50 K SNP芯片对通城猪的全基因组SNP进行扫描,通过IBS距离矩阵和G矩阵分析了该种群的亲缘关系、遗传结距离;通过主成分析构建了公猪的家系结构,结果表明通城猪保种群无明显的群体分层,保种效果良好。本研究采集的样本均来自同一原种场,该原种场在一段时间内一直处于封闭繁殖状态,一些个体不可避免地有近亲或远亲。IBS距离矩阵和G矩阵均表明部分个体之间存在密切的亲缘关系,因此在繁殖保护过程中,应注意公羊与母羊的交配工作,防止近亲繁殖带来的风险。主成分分析表明,整个济宁青山羊群体中个体间的亲缘关系不太紧密,表明该济宁青山羊保种群体可能由多个家系构成。考虑到公羊在整个保种群体的重要性,单独对公羊进行了聚类分析、家系构建,以公羊间分子亲缘系数0.1为标准进行聚类,将23只公羊划分为了14个家系,高于国家级保种场最低6个不同家系的要求。同时发现部分家系数量较少,因此在后续的保种利用工作中要及时关注各家系济宁青山羊的数量,对家系数量少的应进行适当扩群,控制一定的近交水平,确保家系结构维持平衡。
为了保持种群不受外来品种的影响,我国的畜禽保种群体大多采取闭锁繁殖的方式,但这可能会造成一定程度的近交,造成该品种遗传多样性降低,有效群体数量减少。长纯合片段是个体内纯合基因型的连续片段,由亲代将同源单倍型完整地传递给子代所产生的。长的ROH指示发生在较近时期的亲缘关系,越多说明家系内存在近交的可能性越高;短的ROH指示发生在较远时期的亲缘关系,现有系谱通常已不能解释这些亲缘关系[38-39]。Islam等[40]利用山羊50 K芯片分别对济宁青山羊、中卫山羊、阿尔巴斯绒山羊种公羊全基因组SNP信息进行扫描、通过亲缘关系分析和近交系数计算,结果表明中卫山羊场的保种群家系数达17个,高于国家级保种场最低6个不同家系的要求。评价进一步发现,中卫山羊品种ROH短片段较多、群体近交系数低(0.017)、有效群体数量较高,该发现表明保种群体的遗传多样性较高,为中卫山羊保种群的管理措施制定及种公羊育种计划提供新的思路。本研究中,济宁青山羊共检测到347个ROH片段,ROH数量较少,表明在历史上对济宁青山羊的选择驯化强度低。同时发现济宁青山羊群体ROH长度与频率可能成反比(图 7),济宁青山羊群体的ROH长度分布与频率的关系在0~20 Mb满足这个规律,当ROH长度大于20 Mb,可能在此区域受到了密集选择。整个群体平均近交系数为0.047 9,近交系数较小。但也发现有部分个体的近交系数较高,超过了0.3,为降低群体近交增量,同一家系的公、母羊不建议进行配种。
4 结论本研究基于70 K Goat SNP分析了济宁青山羊保种群体的遗传多样性,发现济宁青山羊保种群体近交程度较低,遗传多样性较丰富,群体内家系多,但各家系个体数较少,需要制定合理的配种计划,确保家系结构保持平衡,同时应将该品种资源的保护与开发利用相结合,不断推进该品种资源的选育,加快从资源优势向经济优势转化。
| [1] |
国家畜禽遗传资源委员会组. 中国畜禽遗传资源志: 羊志[M]. 北京: 中国农业出版社, 2011: 283-286. China National Commission of Animal Genetic Resources. Animal genetic resources in China: sheep and goats[M]. Beijing: China Agriculture Press, 2011: 283-286. (in Chinese) |
| [2] |
王可, 马建军, 张玉玉, 等. 济宁青山羊与波尔山羊肉质性能比较分析[J]. 山东农业科学, 2015, 47(9): 115-118. WANG K, MA J J, ZHANG Y Y, et al. Comparative analysis on meat quality between Jining grey goats and Boer goats[J]. Shandong Agricultural Sciences, 2015, 47(9): 115-118. (in Chinese) |
| [3] |
马丽娜, 马青. 利用高密度基因芯片开展滩羊群体亲缘关系遗传分析[J]. 黑龙江畜牧兽医, 2020(3): 65-69. MA L N, MA Q. Genetic analysis of relatedness of Tan sheep population by high-density gene chip[J]. Heilongjiang Animal Science and Veterinary Medicine, 2020(3): 65-69. (in Chinese) |
| [4] |
屠云洁, 唐燕飞, 蒋华莲, 等. 利用基因芯片开展广西麻鸡2个群体亲缘关系的遗传分析[J]. 中国畜牧杂志, 2021, 57(8): 143-146. TU Y J, TANG Y F, JIANG H L, et al. Genetic analysis of kinship of two populations of Guangxi partridge chicken by gene microarray[J]. Chinese Journal of Animal Science, 2021, 57(8): 143-146. (in Chinese) |
| [5] |
李文婷. 基于全基因组SNP对猪品种分子种质特性挖掘与保护的研究[D]. 北京: 中国农业大学, 2017. LI W T. Using genome-wide SNP dataset to reveal and conserve molecular breed characterization in pigs[D]. Beijing: China Agricultural University, 2017. (in Chinese) |
| [6] |
王可, 刘昭华, 崔绪奎, 等. 济宁青山羊微卫星标记多态性分析[J]. 山东农业科学, 2017, 49(2): 142-146. WANG K, LIU Z H, CUI X K, et al. Polymorphism analysis of microsatellite markers in Jining grey goat[J]. Shandong Agricultural Sciences, 2017, 49(2): 142-146. (in Chinese) |
| [7] |
李星星, 张钦恺, 徐君君, 等. 济宁青山羊BMPR-IB基因的多态性分析[J]. 中国草食动物科学, 2014(S1): 129-131. LI X X, ZHANG Q K, XU J J, et al. Polymorphism analysis of the BMPR-IB gene in Jining grey goat[J]. China Herbivore Science, 2014(S1): 129-131. (in Chinese) |
| [8] |
王俊杰. 济宁青山羊产羔数性状的基因组和卵泡发育的转录组研究[D]. 杨凌: 西北农林科技大学, 2020. WANG J J. Genome analysis of litter size trait and transcriptome analysis of follicular development of Jining grey goat[D]. Yangling: Northwest A&F University, 2020. (in Chinese) |
| [9] |
王可, 王金龙, 楚惠民, 等. 济宁青山羊育种核心群选育效果初步分析[J]. 中国草食动物科学, 2014(S1): 141-144. WANG K, WANG J L, CHU H M, et al. Preliminary analysis of the breeding effect in the nucleus population of Jining grey goat[J]. China Herbivore Science, 2014(S1): 141-144. (in Chinese) |
| [10] |
蒋培红, 葛仕豪, 孟冬梅, 等. 济宁青山羊高繁品系选育效果观察[J]. 中国畜禽种业, 2020, 16(9): 45-46. JIANG P H, GE S H, MENG D M, et al. Observation on the breeding effect of Jining grey goat[J]. The Chinese Livestock and Poultry Breeding, 2020, 16(9): 45-46. (in Chinese) |
| [11] |
康周才让, 刘宇, 王敏, 等. 利用微卫星分析康西草原马匹种群的遗传多样性和群体结构[J]. 畜牧兽医学报, 2022, 53(5): 1431-1441. KANG Z C R, LIU Y, WANG M, et al. Analysis of genetic diversity and population structure of horse population in Kangxi grassland by microsatellite DNA markers[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(5): 1431-1441. (in Chinese) |
| [12] |
殷彬, 岳书俭, 俞英, 等. 奶牛分子系谱构建或亲权鉴定的微卫星标记筛选[J]. 畜牧兽医学报, 2017, 48(4): 595-604. YIN B, YUE S J, YU Y, et al. Selection of microsatellite markers used for pedigree tree construction and/or paternity testing in Chinese Holstein cows[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(4): 595-604. (in Chinese) |
| [13] |
TOSSER-KLOPP G, BARDOU P, BOUCHEZ O, et al. Design and characterization of a 52K SNP chip for goats[J]. PLoS One, 2014, 9(1): e86227. DOI:10.1371/journal.pone.0086227 |
| [14] |
QIAO X, SU R, WANG Y, et al. Genome-wide target enrichment-aided chip design: a 66 K SNP chip for cashmere goat[J]. Sci Rep, 2017, 7(1): 8621. DOI:10.1038/s41598-017-09285-z |
| [15] |
KIJAS J W, ORTIZ J S, MCCULLOCH R, et al. Genetic diversity and investigation of polledness in divergent goat populations using 52 088 SNPs[J]. Anim Genet, 2013, 44(3): 325-335. DOI:10.1111/age.12011 |
| [16] |
MARTIN P M, PALHIÈRE I, RICARD A, et al. Genome wide association study identifies new loci associated with undesired coat color phenotypes in Saanen goats[J]. PLoS One, 2016, 11(3): e0152426. DOI:10.1371/journal.pone.0152426 |
| [17] |
王志刚, 吴建平, 刘丑生, 等. 用微卫星标记分析中国山羊品种的遗传多样性和群体遗传结构[J]. 农业生物技术学报, 2010, 18(5): 836-845. WANG Z G, WU J P, LIU C S, et al. Analysing genetic diversity and population genetic structure of Chinese goat breeds by microsatellite[J]. Journal of Agricultural Biotechnology, 2010, 18(5): 836-845. (in Chinese) |
| [18] |
郎侠, 吕潇潇. 兰州大尾羊微卫星DNA多态性研究[J]. 中国畜牧杂志, 2011, 47(1): 14-17. LANG X, LV X X. Study on microsatellite DNA polymorphism of Lanzhou fat-tailed sheep[J]. Chinese Journal of Animal Science, 2011, 47(1): 14-17. (in Chinese) |
| [19] |
张冬杰, 何鑫淼, 田明, 等. 民猪保种群体的遗传基础分析[J]. 黑龙江畜牧兽医, 2022(3): 64-67, 131. ZHANG D J, HE X M, TIAN M, et al. Genetic basis analysis of min pig breed preservation population[J]. Heilongjiang Animal Science and Veterinary Medicine, 2022(3): 64-67, 131. (in Chinese) |
| [20] |
冯雪燕, 刁淑琪, 刘玉强, 等. 基于SNP芯片的海南猪全基因组选择信号分析[J]. 畜牧兽医学报, 2022, 53(2): 349-359. FENG X Y, DIAO S Q, LIU Y Q, et al. Analysis of selection signatures for Hainan Pigs across the Whole Genome based on SNP BeadChips[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(2): 349-359. (in Chinese) |
| [21] |
刘彬, 沈林園, 陈映, 等. 基于SNP芯片分析青峪猪保种群体的遗传结构[J]. 畜牧兽医学报, 2020, 51(2): 260-269. LIU B, SHEN L Y, CHEN Y, et al. Analysis of genetic structure of conservation population in Qingyu Pig based on SNP Chip[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 260-269. (in Chinese) |
| [22] |
高超群, 曹然然, 杜文苹, 等. 基于全基因组SNP标记分析中国地方鸡品种的遗传多样性和种群结构[J]. 畜牧兽医学报, 2023, 54(2): 554-562. GAO C Q, CAO R R, DU W P, et al. Genetic diversity and population structure analysis of Chinese native chicken breeds using Genome-wide SNPs[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(2): 554-562. (in Chinese) |
| [23] |
STELLA A, NICOLAZZI E L, VAN TASSEL C P, et al. AdaptMap: exploring goat diversity and adaptation[J]. Genet Sel Evol, 2018, 50(1): 61. |
| [24] |
王凤红. 山羊SNP芯片设计与内蒙古绒山羊重要经济性状全基因组关联分析及基因组选择研究[D]. 呼和浩特: 内蒙古农业大学, 2021. WANG F H. Design of goat SNP chip with applications in genome-wide association study and genomic selection of important economic traits in Inner Mongolia Cashmere Goat[D]. Hohhot: Inner Mongolia Agricultural University, 2021. (in Chinese) |
| [25] |
乔贤. 绒山羊SNP芯片设计及重要经济性状全基因组关联分析研究[D]. 呼和浩特: 内蒙古农业大学, 2020. QIAO X. Study on SNP chip design and genome association analysis of important economic traits in cashmere goats[D]. Hohhot: Inner Mongolia Agricultural University, 2020. (in Chinese) |
| [26] |
王凤红, 张磊, 李晓凯, 等. 山羊基因组研究进展[J]. 遗传, 2019, 41(10): 928-938. WANG F H, ZHANG L, LI X K, et al. Progress in goat genome studies[J]. Hereditas (Beijing), 2019, 41(10): 928-938. (in Chinese) |
| [27] |
BURREN A, NEUDITSCHKO M, SIGNER-HASLER H, et al. Genetic diversity analyses reveal first insights into breed-specific selection signatures within Swiss goat breeds[J]. Anim Genet, 2016, 47(6): 727-739. |
| [28] |
李隐侠, 牙生江·那斯尔, 赛里克·都曼, 等. SNP芯片评估柯尔克孜羊群体遗传多样性和遗传结构[J]. 畜牧兽医学报, 2023, 54(2): 572-583. LI Y X, NASIER Y S N, SAI L D, et al. Evaluation of genetic diversity and genetic structure in Kirgiz Sheep population based on SNPs Chip[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(2): 572-583. (in Chinese) |
| [29] |
马丽霞, 曹国伟, 朱红芳, 等. 基于RAD-seq静原鸡保种群体的遗传变异分析[J]. 畜牧兽医学报, 2022, 53(7): 2104-2117. MA L X, CAO G W, ZHU H F, et al. Analysis of genetic variation in a conserved population of Jingyuan Chickens based on RAD-seq[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(7): 2104-2117. (in Chinese) |
| [30] |
武艳平, 魏岳, 康昭风, 等. 基于全基因组SNP分析8个地方鸡品种的遗传多样性[J]. 畜牧兽医学报, 2022, 53(2): 646-653. WU Y P, WEI Y, KANG Z F, et al. Genetic diversity analysis of 8 local Chicken Breeds based on Whole Genome SNP[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(2): 646-653. (in Chinese) |
| [31] |
胡亮, 孙伟, 马月辉. 藏系绵羊群体遗传多样性及遗传结构分析[J]. 畜牧兽医学报, 2019, 50(6): 1145-1153. HU L, SUN W, MA Y H. Study on genetic diversity and genetic structure of Tibetan Sheep populations[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(6): 1145-1153. (in Chinese) |
| [32] |
朱兰, 孙利民, 梁家充, 等. 利用微卫星标记分析云南省10个山羊品种的遗传多样性[J]. 中国畜牧杂志, 2019, 55(1): 57-63. ZHU L, SUN L M, LIANG J C, et al. Genetic diversity analysis of 10 goat breeds in Yunnan province by microsatellite markers[J]. Chinese Journal of Animal Science, 2019, 55(1): 57-63. (in Chinese) |
| [33] |
LIU B, SHEN L Y, GUO Z X, et al. Single nucleotide polymorphism-based analysis of the genetic structure of Liangshan pig population[J]. Anim Biosci, 2021, 34(7): 1105-1115. |
| [34] |
XU P, WANG X P, NI L G, et al. Genome-wide genotyping uncovers genetic diversity, phylogeny, signatures of selection, and population structure of Chinese Jiangquhai pigs in a global perspective[J]. J Anim Sci, 2019, 97(4): 1491-1500. |
| [35] |
杜胭脂, 陈玉霞, 康丽, 等. 汶上芦花鸡种质资源的遗传多样性分析[J]. 畜牧兽医学报, 2014, 45(10): 1616-1621. DU Y Z, CHEN Y X, KANG L, et al. Analysis on the genetic diversity of Wenshang Barred Chicken Breed[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(10): 1616-1621. (in Chinese) |
| [36] |
牛晓艳, 曹亮, 明世清, 等. 广灵驴分子系谱的建立及群体遗传结构分析[J]. 畜牧兽医学报, 2022, 53(2): 391-401. NIU X Y, CAO L, MING S Q, et al. Construction of molecular pedigree and analysis of population genetic structure of Guangling Donkey[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(2): 391-401. (in Chinese) |
| [37] |
袁娇, 徐国强, 周翔, 等. 基于SNP芯片监测通城猪的保种效果[J]. 畜牧兽医学报, 2022, 53(8): 2514-2523. YUAN J, XU G Q, ZHOU X, et al. SNP Chip-Based monitoring of population conservation effect of Tongcheng Pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(8): 2514-2523. (in Chinese) |
| [38] |
MASTRANGELO S, TOLONE M, SARDINA M T, et al. Genome-wide scan for runs of homozygosity identifies potential candidate genes associated with local adaptation in Valle del Belice sheep[J]. Genet Sel Evol, 2017, 49(1): 84. |
| [39] |
刘家鑫, 魏霞, 邓天宇, 等. 绵羊全基因组ROH检测及候选基因鉴定[J]. 畜牧兽医学报, 2019, 50(8): 1554-1566. LIU J X, WEI X, DENG T Y, et al. Genome-wide scan for run of homozygosity and identification of corresponding candidate genes in sheep populations[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(8): 1554-1566. (in Chinese) |
| [40] |
ISLAM R, LIU Z F, LI Y F, et al. Conservation assessment of the State Goat farms by using SNP genotyping data[J]. Genes (Basel), 2020, 11(6): 652. |
(编辑 郭云雁)



