全基因组重测序是在已知物种参考基因组的条件下,通过对比测序序列与参考基因组之间的差异,以获得单核苷酸多态性位点(single nucleotide poly-morphisms, SNP)、短插入/删除(insertion and deletion, InDel)、大片段的结构变异(structural variation, SV)和拷贝数变异(copy number variation, CNV)等遗传变异的方法。这些变异位点作为分子遗传标记,在人类复杂疾病、进化、动植物经济性状和育种研究等方面具有重大意义[1]。已有多项研究通过全基因组重测序技术获得遗传变异以解析动物基因组遗传机制[2-6]。SNPs是研究动植物品系遗传变异的重要依据[7-8],其突变可能会改变基因的功能,进而导致生物性状改变甚至致死。SVs是基因组多样性和基因表达变异的重要来源,对基因表达的影响较大[9-11]。CNV等结构变异是哺乳动物基因组变异的重要来源,覆盖更宽的染色体区域,可能导致基因结构的变化、基因调控的修饰、基因剂量的变化以及隐性等位基因的暴露,从而导致大的表型效应[12]。
皖岳黑猪以大别山腹地安徽省岳西县所产淮猪为母本,北京黑猪为第一父本,杜洛克猪为第二父本,采用杂交、回交制种及横交固定方法,正在培育的淮猪和北京黑猪血统各占37.5%,杜洛克血统占25%的优质黑猪新品种,现已完成5个世代的持续选育并初步进行了中间试验,下一步拟申请国家新品种审定。目前关于皖岳黑猪基因组结构变异方面的相关研究尚未见报道。
本研究利用皖岳黑猪全基因组重测序数据,检测基因组遗传变异,揭示皖岳黑猪的群体遗传结构,并与北京黑猪、杜洛克、大白猪、蓝塘猪、民猪、深县黑猪的SNP位点构建数据集,利用挑选最大分类能力和机器学习方法进行基因组特征SNP位点的挖掘,确定皖岳黑猪品种特征SNP位点,为皖岳黑猪进一步的选育开发提供科学依据。
1 材料与方法 1.1 样品采集与DNA提取在安徽省岳西县徽名山皖岳黑猪保种场随机选取代表全部血统且无血缘关系的24头体重达110 kg的皖岳黑猪,采集耳组织样品,并使用磁珠法进行DNA提取,用0.8%琼脂糖凝胶(25 min,170 V)和纳米滴分光光度计(NanoDrop 2000)评估DNA的质量和浓度。
1.2 全基因组重测序分析采用Illumina NovaSeq 6000平台(Illumina,圣地亚哥,加利福尼亚州,美国)对24头皖岳黑猪进行全基因组重测序。此外,从公共数据库下载22头北京黑猪、22头杜洛克、16头大白猪、5头蓝塘猪、5头民猪、39头深县黑猪的基因组数据作为对照。
使用fastp(v0.20.1)[13]过滤原始重测序数据,删除N含量超过该read碱基数的10%和低质量(Q≤5)碱基数超过该条read碱基数的50% 的paired reads。利用BWA[14]软件将原始测序数据比对到猪参考基因组(Sus Scrofa 11.1),使用GATK[15]软件检测SNP变异并对其进行过滤,保留质量得分>30,MQ RMS映射质量>20,dp>5,覆盖率>30%,最小等位基因频率(MAF)>0.01的位点。使用SnpEff[16]软件注释SNP,统计SNP各变异类型发生的比例及在各染色体上的位置;DELLY[17]进行SV检测,去除Y染色体上的变异并过滤掉检测出的低质量和<50 bp的SVs。ANNOVAR(v2019)[18]软件对过滤后的变异进行注释并统计SV在基因组各区域的分布情况。使用CNVnator和CNVcaller共同检测皖岳黑猪群体的CNV变异情况,剔除p1>0.01、Q0>0.5的CNV,将重叠至少1 bp的CNV合并为一个CNVR区域,只保留在3个及以上个体中检测到的CNVR,用CNVnator检测,再用CNVcaller直接检测得到群体的CNVR情况,取两个软件检测出的CNVR交集作为皖岳黑猪群体的真实CNVR,将两个软件检测出的CNVR重叠区域大于50%认为是同一个CNVR,并合并为新的CNVR。
1.3 皖岳黑猪群体遗传多样性分析使用Plink(v 1.90)[19]进行遗传多样性分析。群体的遗传多样性是保证物种得以生存和进化的关键因素。对皖岳黑猪保种群体遗传多样性分析主要包括有效群体大小(Ne)、群体的期望杂合度(He)和观察杂合度(Ho)、群体多态性标记比例(PN)、群体近交系数。有效群体大小反映了该群体在进化过程中受到的迁移、杂交的程度[20]。杂合度代表了群体遗传多样性的丰富度,分为期望杂合度(He)和观察杂合度(Ho),当He>Ho时,推测群体发生了近交或选择;如果He<Ho,则推测群体可能引入了其他品种的血缘[21]。R语言将所有结果可视化。
1.4 皖岳黑猪遗传距离分析基于SNP分型结果,可以计算样本间的IBS距离,IBS (identity by descent)叫做状态同源,是指两个个体中共有的等位基因序列相同。使用Plink(v 1.90)软件中的plink--file hapmap1--cluster--matrix-noweb指令计算遗传距离,所得文件在R语言中结果可视化。
1.5 皖岳黑猪ROH近交系数在Plink(V1.90)中调用--homozyg--homozyg-density 50--homozyg-gap 100--homozyg-kb 500--homozyg-snp 50--homozyg-window-het 1--homozyg-window-snp--homozyg-window-threshold 0.05-out指令计算每个样本的ROH长度,用个体ROH长度除以基因组总长计算得出每个个体的近交系数(FROH)。
1.6 基因组特征库构建及特异SNP位点功能注释本研究7个品种133个个体共检测出1 35 915个SNPs位点。将全部个体分为训练集与测试集,其中训练集共有106个个体,测试集27个个体。对测试集利用挑选最大分类能力方法[8]进行信息SNP的选择,并结合机器学习方法对挑选出的SNPs进行特征选择,以获取皖岳黑猪群体的特征位点。利用R(v 3.6.2)中的ClusterProfiler (v 3.14.0)[22]对筛选出的位点进行注释,并对所选位点注释到的基因进行GO和KEGG的富集分析,以P < 0.05作为判断差异显著的标准。
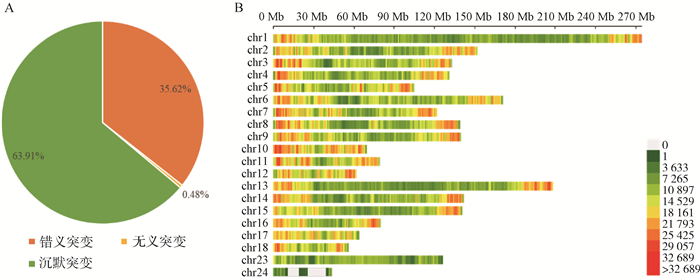
2 结果 2.1 皖岳黑猪基因组变异检测皖岳黑猪原始重测序数据质控后共获得1 463.13 Gb的原始数据,每头获得数据量平均约为60.96 Gb,平均比对深度为23.7×,平均比对率为98.4%,重复reads比率1.88%。SNP变异检测统计结果显示,共获得31 534 384个SNPs,其中12.82%不存在于dbSNP库。在外显子区域的注释中,沉默突变在所有突变类型中占比最大为63.91%,错义突变占35.62%(图 1A)。SNP在染色体上的密度分布如图 1B所示。
|
A.SNP检测中各类型突变的比例;B. SNP在染色体上的密度分布:X轴代表染色体长度,Y轴代表18条常染色体和X染色体,不同的颜色表示1 Mb内包含的SNP个数 A. Proportion of each type of mutation in SNP detection; B. Density distribution of SNPs on chromosomes: X-axis represents the chromosome length, Y-axis represents 18 autosomes and X chromosome, different colors indicate the number of SNPs contained in 1 Mb 图 1 皖岳黑猪种群的SNP特征 Fig. 1 SNP characteristics of Wanyue black pig population |
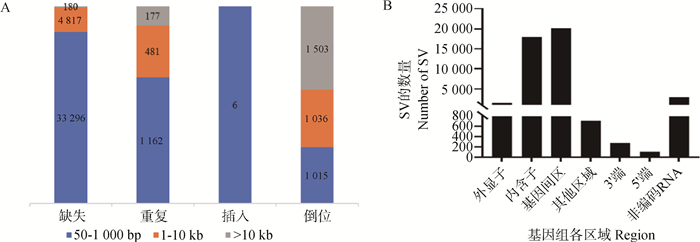
本研究共获得43 673个SVs,变异总长为354.36 Mb,覆盖了猪全基因组的14.5%,变异类型及长度所占比例表明,检出的缺失变异最多,插入变异最少(图 2A);SVs在基因组各区域的分布表明,有3.6%的SVs落在基因外显子区域(图 2B)。
|
A.SV类型的变异长度分布比例;B. 皖岳黑猪SV变异在基因组各区域分布情况 A.Proportion of variation length distribution of SV types; B. Distribution of SV variants in various regions of the genome of Wanyue black pigs 图 2 皖岳黑猪种群SV特征 Fig. 2 SV characteristics of Wanyue black pig population |
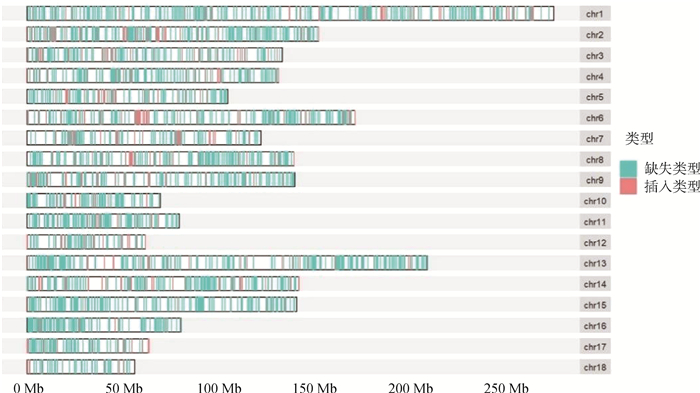
本研究共检测到3 258个CNVRs,其中“Loss”型2 427个,“Gain”型831个。检测出的CNVR在染色体上的分布如图 3所示,可以看出两种类型的CNVR在染色体上的分布都较为均匀,并且“Loss”型CNVR的基因组覆盖度远大于“Gain”型。CNVR在基因组的基因间区分布最多(47.5%),其次是基因组的内含子区域(20.5%),见图 4。
|
图 3 CNVR在染色体上的分布 Fig. 3 Distribution of CNVR on chromosomes |

|
图 4 CNVR在基因组各区域的分布 Fig. 4 Distribution of CNVR in various regions of the genome |
皖岳黑猪群体的Ne为4.2,He为0.320,Ho为0.328,PN为0.788。He与Ho近乎相等且He小于Ho, 这一结果表明皖岳黑猪群体遗传多样性丰度较低且有其他血缘的引入,这也映照了皖岳黑猪在育种过程中有杜洛克及北京黑猪血缘的加入。但其有效群体大小依旧偏低,这就提示我们,在今后的选育方案中要考虑新的特别是公猪血缘的加入。
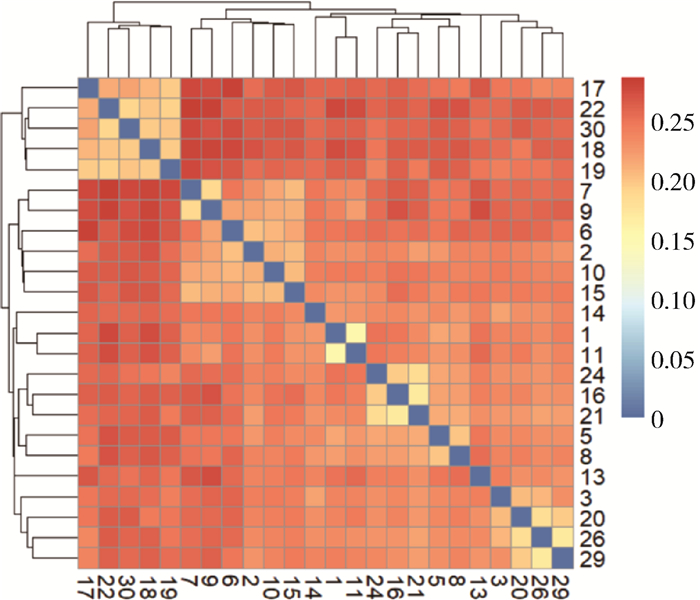
2.3 皖岳黑猪遗传距离皖岳黑猪群体IBS遗传距离在0.157 4~0.287 3之间,平均为(0.243 5±0.022 2)。皖岳黑猪群体IBS距离矩阵的结果如图 5所示。皖岳黑猪个体间的IBS遗传距离总体较远,呈中等程度的亲缘关系,部分个体间的IBS遗传距离较近,存在较高的亲缘关系,可能是皖岳黑猪在选育过程中出现了近交现象,在今后的选育中一定要多加注意,避免其发生近交衰退现象。
|
图 5 IBS遗传距离矩阵 Fig. 5 IBS genetic distance matrix |
利用Plink软件计算皖岳黑猪个体的ROH,共检测出973个ROH片段,平均每个个体中有40个ROH片段且个体总长度为14.6~178.0 Mb,平均ROH长度为(61±9.4)Mb。当前整个群体的近交系数平均值为(0.025±0.004)。基于ROH的近交系数FROH 的分布如图 6所示。
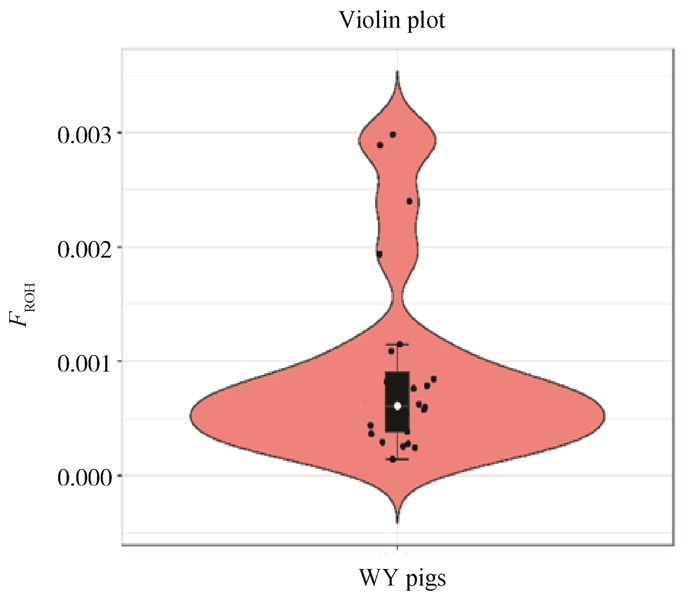
|
图中心的白色圆点代表该群体FROH的中位数,中间黑色长方形方框的上缘和下缘分别为群体FROH的上四分位数和下四分位数。小提琴图的宽窄表示群体FROH的概率密度分布,小提琴图越宽的部分表示处于该水平的样本数目越多,反之则越少 The white dot in the center of the figure represents the median of FROH in the group, and the upper and lower edges of the black rectangle in the middle are the upper and lower quartiles of the group FROH, respectively. The width of the violin plot indicates the probability density distribution of the population FROH, and the wider the violin plot, the larger the number of samples at that level, and vice versa 图 6 基于ROH的近交系数分布图 Fig. 6 Distribution of the inbreeding coefficient FROH based on ROH |
通过机器学习方法最终选出33个SNP位点作为皖岳黑猪群体的特征位点。用训练集对选出的SNP位点进行验证,其主成分分析结果如图 7所示,可以看出这33个SNPs位点可以很好的将皖岳黑猪个体与其他6个品种分开,用测试集测试这33个位点区分皖岳黑猪的准确性,准确率达到95%以上,证明这33个SNPs位点可以作为皖岳黑猪的特征库。
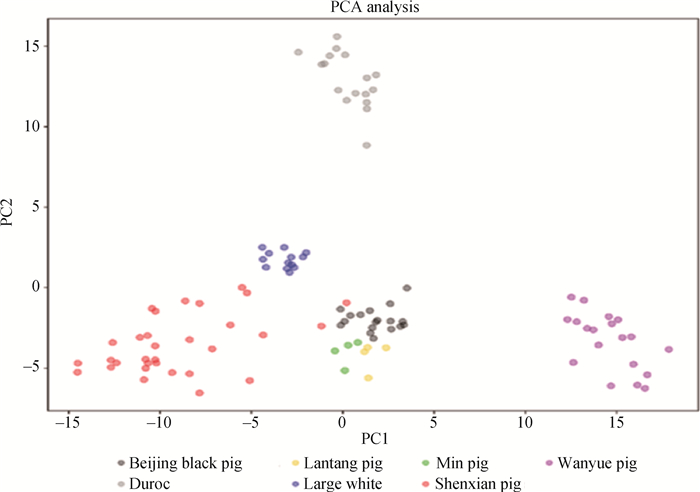
|
图 7 基于10个特征SNPs的7个猪品种的主成分分析 Fig. 7 Principal component analysis of 7 pig breeds based on 10 characteristics SNPs |
对位点进行Ensembl数据库检索,33个SNPs位点映射到15个基因Ensemble号,标记到11个基因(表 1),分别是SUSD4、GPC6、TENM2、NELL1、TMSB10、CHD1L、SLC41A2、KHDRBS2、CRISP1、NECTIN1、GRIK4。还有4个基因号未被标记,但是同样也参与生物过程。GO和KEGG进行富集分析,对这些基因进行功能评估,在84个GO项和5个KEGG途径中发现显著(FDR < 0.05)的基因富集性,GO结果显示,这些基因富集到的生物过程(biological process, BP)为39个,细胞组分(cellular component, CP)为34个,分子功能(molecular function, MF)为11个(图 8)。主要参与成骨细胞活性(NELL1)、免疫系统过程(SUSD4)、细胞膜突触(NECTIN1)等。KEGG分析结果显示,这些基因富集到了粘附连接(adherens junction,NECTIN1)、谷氨酸突触(glutamatergic synapse,GRIK4)、细胞粘附分子(cell adhesion molecules,NECTIN1)、单纯疱疹病毒1型感染(herpes simplex virus 1 infection,NECTIN1)、神经活性配体-受体相互作用(neuroactive ligand-receptor interaction,GRIK4)这5条通路(图 9)。
|
|
表 1 皖岳黑猪特征位点及注释结果 Table 1 Characteristic loci and annotation results of Wanyue black pig |
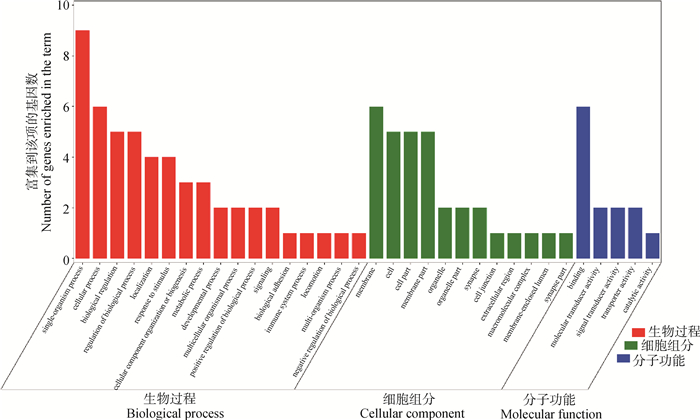
|
图 8 皖岳黑猪特定SNPs基因的GO富集结果 Fig. 8 GO terms enriched by genes of specific SNPs in Wanyue black pig |
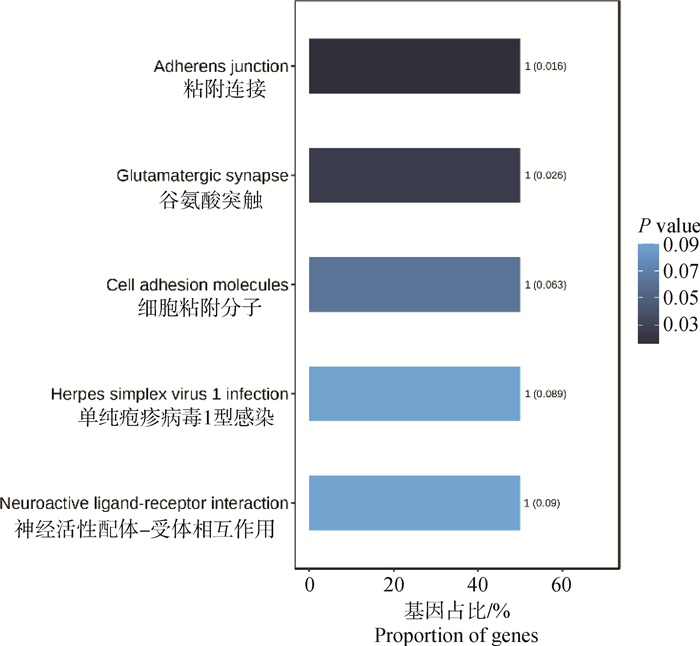
|
图 9 皖岳黑猪特定SNPs基因的KEGG pathway结果 Fig. 9 KEGG pathway enriched by genes of specific SNPs in Wanyue black pig |
本研究检测并注释了皖岳黑猪全基因组遗传变异和SNP位点。在外显子区域检测到了相对较多的沉默突变,由于密码子的简并性,沉默突变的发生对蛋白质的翻译不造成影响[23],但最近一项重要的研究表明同义突变确实会导致蛋白质折叠的改变,进而损害细胞功能[24]。本研究中有3.6%的SV发生在基因组的外显子区域,外显子区域属于基因编码区,稀有且相同的一些SV往往和疾病(包括癌症)的发生相互关联甚至还是其直接的致病诱因[25],该区域内的变异对蛋白质的翻译起重要作用。本研究结果表明,皖岳黑猪具有丰富的变异信息,这些变异信息的潜在功能对研究皖岳黑猪种质特性具有重要的作用。
当前我国绝大多数畜禽保种场都面临着群体有效大小偏低的问题,皖岳黑猪的有效群体大小相较于其他品种如北京黑猪(10.1)、民猪(8.1)[26]、撒坝猪(21)[27]要小很多,IBS遗传距离(0.157 4~0.287 3)也相对于其他品种小。造成这一结果的原因一方面是皖岳黑猪属于培育品种,在培育过程中比较封闭,且存在较严重的近交现象。该结果提示,在今后的育种过程中需要多加注意,积极引入本品种新的血缘,特别是公猪血缘。多态性标记比例(PN, MAF>0.05)为0.788,表明该品种在培育过程中保留了其亲本特异遗传基因,获得了较高的遗传多样性。
ROH是单个个体鉴定的纯合基因型片段,由一系列相同的单倍型组成。ROH提供近亲繁殖事件的年龄和起源的信息,可用于评估可靠的近亲繁殖系数[28]。本研究中,皖岳黑猪平均ROH长度为(61±9.4)Mb,低于青峪猪(131.39±121.33)Mb[29]和安庆六白猪255.19 Mb[30]的平均长度。当前整个群体的近交系数平均值为(0.025±0.004), 与报道的恩施黑猪(0.069±0.06)[31]及青峪猪(0.055)[29]的近交系数相比更低。这可能是因为皖岳黑猪在杂交培育过程中血统来源广泛。
皖岳黑猪经过多年的人工选育,集合了来自亲本的优势性状,具有瘦肉率高,繁殖性能好,抗病能力强,环境适应性强等特点。因此,其在基因组水平上一定存在相适应的变异,通过全基因组重测序技术在基因组水平上查找影响其表型和经济性状的候选基因及功能突变位点,经过统计分析,筛选其分子遗传标记。选取不同地域分布的猪种,从地理隔绝方面来证实筛选位点的可靠性,降低基因重复性,以保证筛选位点最大程度的特异性。对筛选位点进行基因注释及功能富集分析,发现这些位点大部分位于内含子区域,一般认为内含子区域突变对基因的功能不产生影响[32],但近几年的研究表明,内含子区域的变异对基因的调控作用及其致病风险要低于CDS区域和基因调控区域[33],但是位于第一个内含子的SNP比其他内含子中的SNP致病风险大。内含子中SNP主要依靠影响剪切位点活性来影响基因功能。剪切位点的失活可能会影响翻译,影响蛋白质序列[34],内含子中的SNP也有可能影响基因功能。基因功能富集结果显示,这些基因在功能上可能与皖岳黑猪生长速度快、环境适应性强的品种特征相关。GPC6和NELL1基因与生长相关。GPC6 (GLYPICAN 6)是糖基磷脂酰肌醇锚定的硫酸乙酰肝素蛋白聚糖家族的一员,与控制细胞生长和细胞分裂有关[35-36]。NELL1基因编码含有表皮生长因子(EGF)样重复序列的细胞质蛋白,是一种对骨软骨谱系高度特异性的有效生长因子,并已证明了对骨骼的强效诱导[37-38]。NECTIN1编码粘附蛋白作为伪狂犬病病毒(PRV)的糖蛋白D(gD)的受体,介导病毒进入上皮和神经元细胞[39-40]。KHDRBS2基因与大白猪[41]和梅古山羊[42]的繁殖性状有关。皖岳黑猪生长在高海拔、高坡度、高风速、高湿度、水质优良、空气负氧离子含量高的生态环境,适应当地冬季严寒、夏季酷热的气候条件,因此形成了其独特的种质特性,具有较强的环境适应能力。GRIK4基因的剪接和表达的改变有助于牦牛调节其行为认知和神经系统去适应高海拔环境[43]。CHD1L基因编码参与DNA修复的DNA解旋酶蛋白,与癌症相关[44]。该基因也被发现在早期发育阶段与细胞分裂有关[45],并通过与PARP1相互作用来调节干细胞的多能性[46],这意味着CHD1L在调节干细胞多能性及胚胎发育方面具有特定作用[47],可作为皖岳黑猪在胚胎发育方面研究的候选基因。
4 结论本研究揭示了皖岳黑猪全基因组遗传变异信息,为今后对皖岳黑猪表型性状遗传机制的研究奠定了基础。在后续选育过程中需要制定更加合理全面的育种方案以确保其遗传多样性。基于挑选最大分类能力方法初步获得皖岳黑猪33个基因组特异SNPs位点,通过注释获得这些位点的基因功能,将为未来皖岳黑猪种质特性形成的分子机制研究提供科学参考。
| [1] |
VVAN ASSELT A J, EHLI E A. Whole-genome genotyping using DNA microarrays for population genetics[M]//EYSTER K M. Estrogen Receptors: Methods and Protocols. New York: Humana, 2022: 269-287.
|
| [2] |
LI X L, YUAN L F, WANG W M, et al. Whole genome re-sequencing reveals artificial and natural selection for milk traits in East Friesian sheep[J]. Front Vet Sci, 2022, 9: 1034211. DOI:10.3389/fvets.2022.1034211 |
| [3] |
LI C Y, LIU K P, DAI J H, et al. Whole-genome resequencing to investigate the determinants of the multi-lumbar vertebrae trait in sheep[J]. Gene, 2022, 809: 146020. DOI:10.1016/j.gene.2021.146020 |
| [4] |
LI W T, ZHANG M M, LI Q G, et al. Whole-genome resequencing reveals candidate mutations for pig prolificacy[J]. Proc Biol Sci B, 2017, 284(1869): 20172437. |
| [5] |
HUANG M, ZHANG H, WU Z P, et al. Whole-genome resequencing reveals genetic structure and introgression in Pudong White pigs[J]. Animal, 2021, 15(10): 100354. DOI:10.1016/j.animal.2021.100354 |
| [6] |
ZHANG W, LI X J, JIANG Y, et al. Genetic architecture and selection of Anhui autochthonous pig population revealed by whole genome resequencing[J]. Front Genet, 2022, 13: 1022261. DOI:10.3389/fgene.2022.1022261 |
| [7] |
CHU L P, ZHONG H, ZHU Y, et al. Association of ATG7 gene polymorphisms with microscopic polyangiitis in Chinese individuals[J]. Am J Transl Res, 2022, 14(10): 7239-7251. |
| [8] |
YUAN J, ZHOU X, XU G Q, et al. Genetic diversity and population structure of Tongcheng pigs in China using whole-genome SNP chip[J]. Front Genet, 2022, 13: 910521. DOI:10.3389/fgene.2022.910521 |
| [9] |
CHIANG C, SCOTT A J, DAVIS J R, et al. The impact of structural variation on human gene expression[J]. Nat Genet, 2017, 49(5): 692-699. DOI:10.1038/ng.3834 |
| [10] |
ZHOU R, LI S T, YAO W Y, et al. The Meishan pig genome reveals structural variation-mediated gene expression and phenotypic divergence underlying Asian pig domestication[J]. Mol Ecol Resour, 2021, 21(6): 2077-2092. DOI:10.1111/1755-0998.13396 |
| [11] |
LI M Z, CHEN L, TIAN S L, et al. Comprehensive variation discovery and recovery of missing sequence in the pig genome using multiple de novo assemblies[J]. Genome Res, 2017, 27(5): 865-874. DOI:10.1101/gr.207456.116 |
| [12] |
REDON R, ISHIKAWA S, FITCH K R, et al. Global variation in copy number in the human genome[J]. Nature, 2006, 444(7118): 444-454. DOI:10.1038/nature05329 |
| [13] |
YANG W J, LIU Z, ZHAO Q Q, et al. Population genetic structure and selection signature analysis of Beijing Black pig[J]. Front Genet, 2022, 13: 860669. DOI:10.3389/fgene.2022.860669 |
| [14] |
LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler Transform[J]. Bioinformatics, 2009, 25(4): 1754-1760. |
| [15] |
VAN DER AUWERA G A, O'CONNOR B D. Genomics in the cloud: using docker GATK, and WDL in Terra[M]. Sebastopol: O'Reilly Media, 2020.
|
| [16] |
CINGOLANI P, PLATTS A, WANG L L, et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2;iso-3[J]. Fly (Austin), 2012, 6(2): 80-92. DOI:10.4161/fly.19695 |
| [17] |
RAUSCH T, ZICHNER T, SCHLATTL A, et al. DELLY: structural variant discovery by integrated paired-end and split-read analysis[J]. Bioinformatics, 2012, 28(18): i333-i339. DOI:10.1093/bioinformatics/bts378 |
| [18] |
WANG K, LI M Y, HAKONARSON H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Res, 2010, 38(16): 164. DOI:10.1093/nar/gkq603 |
| [19] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [20] |
ZHAO S L, SHI C M, MA L, et al. AIM-SNPtag: a computationally efficient approach for developing ancestry-informative SNP panels[J]. Forensic Sci Int Genet, 2019, 38: 245-253. DOI:10.1016/j.fsigen.2018.10.015 |
| [21] |
WANG J, SANTIAGO E, CABALLERO A. Prediction and estimation of effective population size[J]. Heredity, 2016, 117(4): 193-206. DOI:10.1038/hdy.2016.43 |
| [22] |
ZHANG W, ZHOU M, LIU L Q, et al. Population structure and selection signatures underlying domestication inferred from genome-wide copy number variations in Chinese indigenous pigs[J]. Genes (Basel), 2022, 13(11): 2026. DOI:10.3390/genes13112026 |
| [23] |
GE F, ZHANG Y, XU J, et al. Prediction of disease-associated nsSNPs by integrating multi-scale ResNet models with deep feature fusion[J]. Brief Bioinform, 2022, 23(1): bbab530. DOI:10.1093/bib/bbab530 |
| [24] |
WALSH I M, BOWMAN M A, SOTO SANTARRIAGA I F, et al. Synonymous codon substitutions perturb cotranslational protein folding in vivo and impair cell fitness[J]. Proc Natl Acad Sci U S A, 2020, 117(7): 3528-3534. DOI:10.1073/pnas.1907126117 |
| [25] |
KOSUGI S, MOMOZAWA Y, LIU X X, et al. Comprehensive evaluation of structural variation detection algorithms for whole genome sequencing[J]. Genome Biol, 2019, 20(1): 117. DOI:10.1186/s13059-019-1720-5 |
| [26] |
张冬杰, 何鑫淼, 王文涛, 等. 民猪全基因组序列测定与分析[J]. 东北农业大学学报, 2018, 49(11): 9-17. ZHANG D J, HE X M, WANG W T, et al. Determination and analysis of the whole genome sequence of Min pig[J]. Journal of Northeast Agricultural University, 2018, 49(11): 9-17. (in Chinese) |
| [27] |
邓俊, 刘艺端, 许文坤, 等. 基于SNP芯片撒坝猪保种群体的遗传结构分析[J]. 中国饲料, 2022(17): 7-11. DENG J, LIU Y D, XU W K, et al. Genetic structure analysis of a conserved population of Saba pigs based on SNP chips[J]. China Feed, 2022(17): 7-11. (in Chinese) |
| [28] |
CORTELLARI M, NEGRO A, BIONDA A, et al. Using pedigree and genomic data toward better management of inbreeding in Italian dairy sheep and goat breeds[J]. Animals (Basel), 2022, 12(20): 2828. |
| [29] |
刘彬, 沈林園, 陈映, 等. 基于SNP芯片分析青峪猪保种群体的遗传结构[J]. 畜牧兽医学报, 2020, 51(2): 260-269. LIU B, SHEN L Y, CHEN Y, et al. Analysis of genetic structure of conservation population in Qingyu pig based on SNP chip[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 260-269. (in Chinese) |
| [30] |
时坤鹏, 刘莹, 张志勇, 等. 基于SNP芯片分析安庆六白猪群体遗传结构[J]. 中国畜牧杂志, 2022, 58(8): 136-140. SHI K P, LIU Y, ZHANG Z Y, et al. Analysis of population genetic structure of Anqing Six-end-White pig based on SNP Chip[J]. Chinese Journal of Animal Science, 2022, 58(8): 136-140. (in Chinese) |
| [31] |
吴林慧, 孙琦, 王荔茹, 等. 恩施黑猪基因组群体遗传学参数的估计与选择信号研究[J]. 畜牧兽医学报, 2019, 50(3): 485-494. WU L H, SUN Q, WANG L R, et al. A study of the population genetics parameters and selection signatures in Enshi black pig[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(3): 485-494. (in Chinese) |
| [32] |
许超. SNP对基因表达影响的生物信息学模型[D]. 苏州: 苏州大学, 2012. XU C. SVM-based model for predicting the effect of regulatory SNPS on gene expression[D]. Suzhou: Soochow University, 2012. (in Chinese) |
| [33] |
KOMAR A A. SNPs, silent but not invisible[J]. Science, 2007, 315(5811): 466-467. DOI:10.1126/science.1138239 |
| [34] |
CHEN R, DAVYDOV E V, SIROTA M, et al. Non-synonymous and synonymous coding SNPs show similar likelihood and effect size of human disease association[J]. PLoS One, 2010, 5(10): e13574. DOI:10.1371/journal.pone.0013574 |
| [35] |
FILMUS J. The function of glypicans in the mammalian embryo[J]. Am J Physiol Cell Physiol, 2022, 322(4): C694-C698. |
| [36] |
DING R R, YANG M, QUAN J P, et al. Single-locus and multi-locus genome-wide association studies for intramuscular fat in Duroc pigs[J]. Front Genet, 2019, 10: 619. |
| [37] |
AN H J, KO K R, BAEK M, et al. Pro-angiogenic and osteogenic effects of adipose tissue-derived pericytes synergistically enhanced by nel-like protein-1[J]. Cells, 2021, 10(9): 2244. |
| [38] |
NIELSEN E Ø, CHEN L, HANSEN J O, et al. Optimizing osteogenic differentiation of ovine adipose-derived stem cells by osteogenic induction medium and FGFb, BMP2, or NELL1 in vitro[J]. Stem Cells Int, 2018, 2018: 9781393. |
| [39] |
DI GIOVINE P, SETTEMBRE E C, BHARGAVA A K, et al. Structure of herpes simplex virus glycoprotein D bound to the human receptor nectin-1[J]. PLoS Pathog, 2011, 7(9): e1002277. |
| [40] |
YANG X H, YU C Z, ZHANG Q Y, et al. A nectin1 mutant mouse model is resistant to pseudorabies virus infection[J]. Viruses, 2022, 14(5): 874. |
| [41] |
VERARDO L L, SILVA F F, LOPES M S, et al. Revealing new candidate genes for reproductive traits in pigs: combining Bayesian GWAS and functional pathways[J]. Genet Sel Evol, 2016, 48: 9. |
| [42] |
GUO J Z, TAO H X, LI P F, et al. Whole-genome sequencing reveals selection signatures associated with important traits in six goat breeds[J]. Sci Rep, 2018, 8(1): 10405. |
| [43] |
GUANG-XIN E, YANG B G, ZHU Y B, et al. Genome-wide selective sweep analysis of the high-altitude adaptability of yaks by using the copy number variant[J]. 3 Biotech, 2020, 10(6): 259. |
| [44] |
PRIGARO B J, ESQUER H, ZHOU Q, et al. Design, synthesis, and biological evaluation of the first inhibitors of oncogenic CHD1L[J]. J Med Chem, 2022, 65(5): 3943-3961. |
| [45] |
XIONG X F, LAI X D, LI A G, et al. Diversity roles of CHD1L in normal cell function and tumorigenesis[J]. Biomark Res, 2021, 9(1): 16. |
| [46] |
JIANG B H, CHEN W Y, LI H Y, et al. CHD1L regulated PARP1-driven pluripotency and chromatin remodeling during the early-stage cell reprogramming[J]. Stem Cells, 2015, 33(10): 2961-2972. |
| [47] |
LIU S S, BAI Y S, FENG L, et al. Identification of CHD1L as an important regulator for spermatogonial stem cell survival and self-renewal[J]. Stem Cells Int, 2016, 2016: 4069543. |
(编辑 郭云雁)



