mRNA疫苗是指通过体外转录(in vitro transcription, IVT)技术合成病原的mRNA,经递送系统递送至靶细胞,并在细胞核糖体内被翻译为目的抗原,进而启动机体免疫应答反应的新型核酸疫苗。mRNA疫苗是继第一代减毒/灭活疫苗和第二代亚单位疫苗基础上发展起来的第三代疫苗,具有针对病原体变异反应速度快、生产工艺简单、易规模化扩大等特点。
mRNA疫苗的研究最早可追溯到1990年,Wolff等[1]发现在被注射含有特定基因的质粒DNA或mRNA的小鼠肌肉组织局部会产生该基因编码的蛋白产物。后续的研究发现,这种被核酸免疫的动物体内会产生针对该核酸编码抗原的特异性免疫反应[2]。但由于mRNA不稳定、在组织内易被降解、细胞吸收率较低等缺陷,其研发进展缓慢。21世纪后,随着mRNA合成、修饰和递送技术的发展,上述缺陷逐渐得到克服,mRNA疫苗的研发也重新得到重视。2005年,Weissman和Karikó通过试验证明,使用修饰的核苷(假尿苷)代替尿苷可以降低体外转录mRNA的免疫原性[3],提高mRNA在机体内的稳定性,并使mRNA具有更高的翻译水平[4],这为后期研发mRNA疫苗奠定了基础。随着全球新冠肺炎疫情的暴发,mRNA疫苗再次被推上历史舞台。2020年底,BioNTech/辉瑞和Moderna的两款mRNA疫苗BNT162b2、mRNA-1273相继获批上市,在全球广泛应用,并取得了良好的免疫保护效果[2]。本文将从mRNA疫苗的类型和结构、递送系统、作用机制以及临床应用等方面对mRNA疫苗进行全面综述,并对这个新兴疫苗平台在兽医领域的发展方向进行了展望。
1 mRNA疫苗的类型和结构mRNA疫苗可以分为两种类型:非复制mRNA疫苗和自扩增RNA疫苗。虽然两者具有共同的结构,但自扩增RNA疫苗在编码区域包含用于mRNA复制的额外序列,从而实现细胞内mRNA扩增,可以在同等mRNA用量的情况下表达更多抗原[5]。mRNA疫苗由mRNA和递送系统两个部分组成,其中,mRNA的功能是为抗原合成提供遗传信息,递送系统是将mRNA转入胞内,使mRNA免受核酸酶及机体免疫系统的干扰。一个合格的mRNA疫苗在mRNA和递送系统设计上均格外考究。
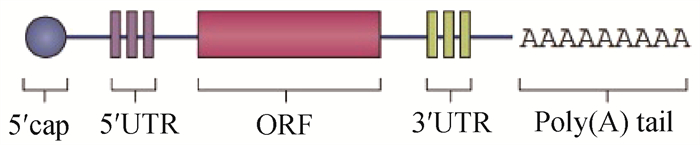
1.1 非复制mRNA疫苗结构组成信使RNA(message RNA),又称为mRNA,是以线性DNA为模板通过转录方式获得的核糖核苷酸,作为模板参与蛋白质的合成过程。用于翻译的mRNA由5′帽子结构(5′ cap)、5′非翻译区(5′untranslated region,5′UTR)、翻译区(coding sequence, CDS)、3′非翻译区(3′untranslated region,3′UTR)以及poly(A)尾组成。体外转录的RNA需要通过加帽、加尾,以模拟天然mRNA结构和功能。有些研究人员会对序列进行密码子优化,同时引入Kozak序列,以增强mRNA翻译水平。
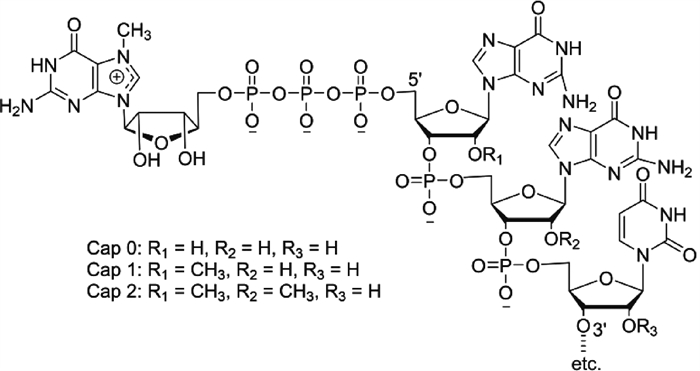
1.1.1 5′帽子结构 在真核生物中,成熟mRNA分子的5′端,有一种特殊结构:m7G5′ppp5′Np,通常被称为甲基鸟苷帽子或5′帽子,用于调节前体mRNA的剪接,提升mRNA核输出,对于mRNA的稳定性和蛋白质翻译具有重要作用[7]。此外,先天免疫系统也会通过5′帽子识别自身与非自身mRNA,因此,5′帽子可以帮助IVT mRNA逃脱免疫系统识别,使其免受外切核酸酶切割[8]。
5′帽子有3种类型,分别是cap0、cap1和cap2。鸟苷G以5′-5′焦磷酸键与初级转录本的5′端核苷酸相连,当其第7位碳原子被甲基化后,形成m7G5′ppp5′Np,即cap0。当转录本第一个核苷酸的2′-O位也发生甲基化时,形成m7GPPPN1m,此时形成的为cap1,这是多数5′帽子的存在形式。当转录本第一和第二个核苷酸的2′-O位均发生甲基化时,会形成m7GPPPN1mN2m结构,即cap2。哺乳动物细胞内5′帽子的主要形式是cap1和cap2,两者在cap0的基础上进一步提高了体外转录mRNA的翻译效率。
5′帽子通过m7G的疏水性阳离子的相互作用和翻译起始期间三磷酸桥的负电荷结合真核生物翻译起始因子4E(eukaryotic initiation factor 4E, eIF4E),eIF4E能够与支架蛋白(eIF4G)相互作用形成复合物,eIF4G还能够与poly(A)结合蛋白(poly-adenosine binding protein,PABP)以及eIF3相互作用使mRNA环化,进而启动mRNA翻译[9-11]。
目前,加帽主要是使用牛痘病毒加帽酶或通过化学共转录法进行[12]。酶法加帽的过程相对复杂,但产量较高,加帽率近100%,多用于大规模生产和实验室研究;与酶法加帽相比,化学共转录方法最大程度上减少了反应步骤和酶的用量,过程简单,但产量和加帽率较低,不能满足大规模生产使用。在使用标准帽类似物进行化学共转录加帽时,会同时产生m7GpppGN和Gpppm7GN两种结构。为此,设计了抗反向帽类似物(ARCA,3′-O-Me-m7G5′ppp5′G),其内包含一个5′-5′三磷酸桥,可以有效解决方向错误的问题[13]。下一代共转录加帽技术CleanCap在ARCA的基础上进一步提升了产量,加帽率升至94%[14]。但目前没有研究证明何种加帽方式更具优势。
1.1.2 非翻译区 在天然mRNA分子编码区的5′端和3′端,有两个不被翻译成多肽的区域,称为非翻译区(untranslated regions, UTRs)。UTRs上存在多个调控元件来控制mRNA的稳定性和翻译效率,除此之外,UTRs还与mRNA二级结构以及核糖体对mRNA的识别有关[16]。许多促进mRNA翻译的UTRs序列已经被从天然存在的序列中鉴定出来,如源自α-珠蛋白和β-珠蛋白的UTRs序列,目前已被广泛用于构建IVT mRNA[17]。
5′UTR主要参与启动翻译过程,作为蛋白质翻译的预起始复合物的起始结合位点,在控制蛋白质翻译效率方面起着重要作用[10]。eIF4A与5′UTR的结合可以协助在蛋白质翻译发生之前解开mRNA的二级结构[10],这对于eIF1A与mRNA的结合十分重要[18]。目前在进行mRNA疫苗设计时,通常会在5′UTR下游引入Kozak(GCCACC)序列,提高核糖体识别翻译起始位点的准确性,提升蛋白的表达效率。
3′UTR中包含mRNA降解信号,优化3′UTR序列,可以有效提高mRNA的稳定性,延长半衰期,进而提升蛋白的表达效率。如mRNA的3′UTR中富含A、U的序列参与了mRNA降解过程中poly(A)尾的去除,因此,改变3′UTR中的A、U含量可以延长mRNA半衰期,提高稳定性[19]。
1.1.3 编码区 编码区序列(coding sequence, CDS)是用以编码目的蛋白的基因序列,在进行mRNA疫苗设计时,CDS序列的设计对mRNA的翻译效率和免疫原性具有重要影响[20]。CDS的设计需要考虑以下几个因素:首先,不同宿主细胞的密码子偏好性不同,针对mRNA疫苗的目标物种进行密码子优化,使用常见密码子替代稀有密码子,可以显著增强mRNA在细胞内的翻译水平[21],但有些蛋白质的部分密码子需要低翻译效率才能保证蛋白的正确折叠,因此需要对靶抗原进行抗原表位预测和蛋白结构预测;其次,研究表明,适当提高序列中鸟嘌呤和胞嘧啶(G和C)含量可以增加mRNA的稳定性和翻译效率,提高体外稳态mRNA含量和体内蛋白质表达水平[21-22];再次,在设计mRNA序列时,增加其二级结构的含量,可以有效减少mRNA的降解。最近,一种专门为最大碱基堆叠区域设计最佳mRNA序列的算法也被证明可以显著提高mRNA稳定性[23]。
1.1.4 poly(A)尾 在真核生物mRNA的3′端,有一个聚腺苷酸化区域,被称为poly(A)尾,其作用是与PABP[poly(A)-binding protein]结合,后者随后募集eIF4G并增加对mRNA 5′帽子的亲和力以形成环状mRNA[24]。除少数个例(例如组蛋白)外,所有编码mRNA的细胞蛋白都具有poly(A)尾,其对于增强mRNA稳定性、提高翻译效率至关重要。在哺乳动物细胞中,天然mRNA分子的poly(A)尾部长度约为250 nt[25]。通常,翻译效率与poly(A) 尾中的腺苷数量成正比[26]。对于mRNA药物而言,需要至少含有20 nt poly(A)才能有效翻译[27]。曾有研究表明,120 nt的poly(A)尾能够提高mRNA的稳定性和翻译效率[28],但进行mRNA疫苗设计时添加poly(A)尾的具体长度目前尚无定论。
1.1.5 修饰核苷酸 研究表明,IVT RNA通过刺激Toll样受体(TLR),特别是TLR3、TLR7和TLR8来激活先天免疫系统的免疫细胞。然而,将修饰核苷如假尿苷(Ψ)、5-甲基胞苷(m5C)、N6-甲基腺苷(m6A)、5-甲基尿嘧啶(m5U)或2-硫代胺(S2U)用于体外转录时,大多数TLR不再被激活[4, 29]。Karikó等[4]率先开发了用于产生IVT mRNA的修饰核苷酸,并声称将IVT mRNA中的尿苷替换为假尿苷不仅会降低针对mRNA的先天免疫反应,也能增强蛋白质翻译水平。然而,修饰核苷对TLR非依赖性免疫反应的影响尚不清楚。
1.2 自扩增RNA疫苗自扩增RNA(self-assembled RNA, saRNA)疫苗除包含上述结构外,还引入了来自RNA病毒的促进RNA复制的其他结构:非结构蛋白基因nsP1、nsP2、nsP3和nsP4。nsP1~4分别编码负责mRNA加帽的蛋白、NTP酶/解旋酶/蛋白酶、macrodomain蛋白和RNA聚合酶[30]。自扩增RNA进入宿主细胞质后,会通过核糖体中的内源性翻译机制,表达nsP1~4前体多聚蛋白,这些非结构蛋白重新组装成RNA依赖性RNA聚合酶转录酶复合物,以IVT RNA为底物分子供翻译为抗原的mRNA[31]。因此,一个saRNA可以产生多个mRNA,进而提高抗原表达量。自扩增RNA疫苗单剂所需RNA的量更低,可以进一步降低疫苗成本[32]。但自扩增RNA疫苗也存在着一定局限性,比如其庞大而复杂的saRNA序列,通常nsP1~4序列的长度约为7 000 nt,这使saRNA疫苗的全长超过9 000 nt[5],过大的体积会限制RNA进入细胞的效率。目前,saRNA疫苗已经表现出了良好的应用前景,Arcturus、CureVac公司设计的SARS-CoV-2 saRNA疫苗已进入临床前研究阶段,Imperial College London公司设计的SARS-CoV-2 saRNA疫苗率先进入Ⅰ期临床试验。
2 递送系统IVT mRNA相对分子质量为104~106 u,表面带有负电荷,因此难以通过胞吞等方式进入细胞[33]。外源性mRNA进入机体时,易被机体先天免疫系统捕获,也易被核酸酶降解[34]。合适的递送系统对mRNA进入细胞、抗原表达水平、免疫持续时间以及保护性免疫反应效力具有重要作用。目前,常用的递送系统包括脂质纳米颗粒(LNP)、脂质体、脂质复合物、高分子材料、胶束、多肽、鱼精蛋白、电穿孔等[33, 35-37],其中LNP因其表现出的更高的转染效率已成为当下最常用的递送系统之一[38-40]。
LNP具有磷脂单分子层结构,带正电荷的LNP与带负电荷的mRNA通过静电作用,将mRNA包裹在LNP内[33, 41]。LNP含有多种成分,以Moderna公司Spikevax疫苗为例,该疫苗中LNP的组成成分包括阳离子脂质(SM-102)、辅助脂质磷脂二甲酰磷脂胆碱(DSPC)、胆固醇(cholesterol)和聚乙二醇化脂质(PEG2000-DMG)[42],这四种成分按50 ∶38.5 ∶1.5 ∶10的比例混合,形成LNP复合物。这四种成分的功能各不相同,阳离子脂质可以根据不同内环境的pH,通过电荷相互作用调节LNP的自组装和mRNA的释放[43];辅助脂质通常是饱和磷脂,可以提高整体相变温度和稳定性[44];胆固醇具有很强的膜融合能力,可以促进细胞对mRNA的摄入[45];聚乙二醇化磷脂位于LNP表面,可以提高LNP亲水性,避免其被免疫系统快速清除,防止颗粒聚集,调节颗粒大小,并提高稳定性[46]。
目前脂质纳米颗粒的制备方法主要有薄膜水化法、挤出法、均质法和反相蒸发等传统方法[47]。与这些方法相比,采用微流控混合技术来制备LNP,相对简便快速,条件温和,粒径均匀,转染效率高,容易实现生产放大[48]。
在过去几年里,大量研究表明,使用LNP技术可以极大地促进mRNA的递送并增强抗原表达[49]。这些研究阐述了LNP对mRNA疫苗诱导有效的免疫反应起到重要作用,但其机制尚不明晰[50-51],未来需要作出更多努力,来阐明LNP递送mRNA的作用机制[52]。
3 mRNA疫苗诱导免疫应答的机制非复制mRNA疫苗若要引发针对抗原的适应性免疫,必须通过宿主细胞的翻译机制将mRNA翻译成内源性抗原,内源性抗原在细胞内被蛋白酶体和溶酶体降解为小分子抗原片段后,分别激活MHC Ⅰ类和Ⅱ类途径,引起机体产生有效的细胞免疫和体液免疫。
mRNA疫苗的免疫效果与接种途径有关,人们通常选择用皮内、肌肉和皮下注射,注射后mRNA可以转染至注射部位附近的非免疫细胞和组织驻留免疫细胞中[53]。在非免疫细胞中,经翻译得到的抗原最初位于宿主细胞的胞质中,由于合成的抗原蛋白不用于细胞,因此它们在泛素化后作为内源性抗原被蛋白酶体降解[2]。降解后的抗原片段与MHC Ⅰ类分子形成复合物,随后抗原片段被呈递于细胞表面,供细胞毒性T淋巴细胞(CD8+T细胞)识别,进而激活CD8+T细胞介导的免疫应答反应[54]。对于组织驻留免疫细胞,主要是针对抗原递呈细胞(antigen-presenting cell, APC),如树突细胞(dendritic cells, DC)和巨噬细胞(macrophages)[55]。与非免疫细胞不同的是,mRNA转染至组织驻留免疫细胞后,除可以激活MHCⅠ类途径,还能通过MHCⅡ类途径处理抗原,进而激活CD4+T细胞介导的体液免疫应答[54]。通常,MHC Ⅱ类途径由外源性抗原激活,但也存在交叉递呈途径,部分内源性抗原可以通过自噬作用激活MHCⅡ类途径[56],还有一部分内源性抗原会分泌出胞外,重新作为外源性抗原被内吞入胞内,进而激活MHC Ⅱ类途径[5]。该过程是内源性抗原被分泌至胞外后,重新进入细胞,并被溶酶体降解为小分子抗原片段,随后进入内质网中,与MHCⅡ类分子形成复合物,再经高尔基体转运至细胞表面,供CD4+T细胞识别[57]。CD4+T细胞受抗原刺激后,通过分泌细胞因子等方式使B细胞成熟并分化为浆细胞,进而激活体液免疫应答[58-59]。
由此可见,若想使mRNA引起有效的细胞和体液免疫反应,需要依靠内源性抗原同时激活MHCⅠ类和MHCⅡ类途径,其中能够激活MHC Ⅱ类途径是成功激活体液免疫的关键。通常,MHCⅡ类途径由外源性抗原激活,如果引入分泌型信号肽,通过基因工程改造将抗原引导至胞外[60],便可以将内源性抗原转换为外源性抗原,进而更有效激活MHCⅡ类途径[61]。
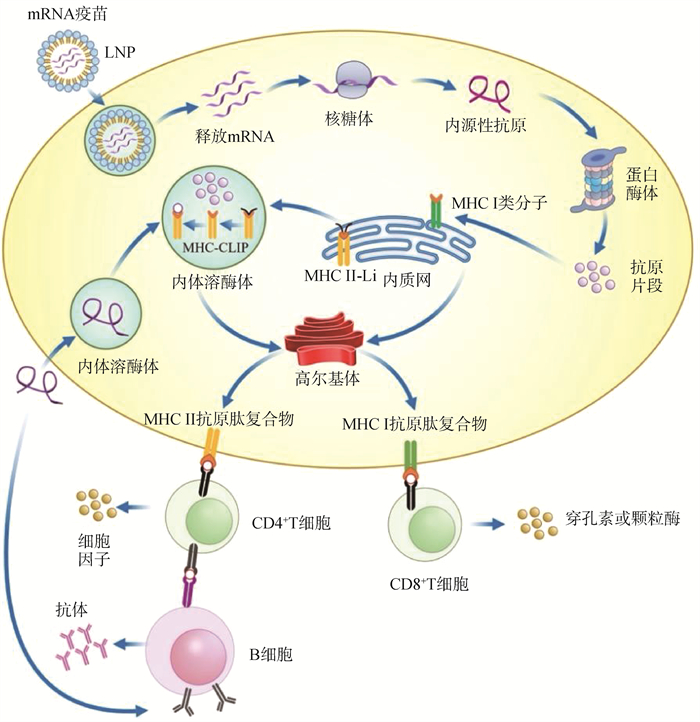
|
图 3 mRNA疫苗作用机制 Fig. 3 Mechanism of action of mRNA vaccine |
mRNA疫苗因其相较于传统疫苗更快的研发速度和良好的保护效力在新冠肺炎疫情防控中脱颖而出。2021年8月23日,由BioNTech设计的mRNA疫苗(商品名为Comirnaty)正式获批上市,用于预防16岁及以上人群的新冠病毒感染,临床试验结果显示,该疫苗预防新冠的有效率为91%[62]。当前,Moderna、NIAID、Canadian Immunization Research Network、BioNTech和CureVac等多家公司研发的针对新冠的mRNA疫苗已进入临床阶段。此外,还有多种针对不同病原的mRNA疫苗(如人副流感病毒、呼吸道合胞病毒、巨细胞病毒、艾滋病毒等)也已经进入临床研究阶段,但目前没有更多数据得到披露[63-64]。
针对人畜共患病的mRNA疫苗也已有多种进入临床试验。2015年12月—2017年8月,德国和美国开展了H10N8和H7N9流感病毒mRNA疫苗的Ⅰ期临床试验,评估了流感病毒mRNA疫苗的安全性和免疫原性。结果表明,疫苗没有导致严重的不良反应,并能引起强烈的体液免疫反应[65]。2021年11月23日,CureVac公司完成了对狂犬病病毒mRNA疫苗CV7202的Ⅰ期临床试验,用以评估在成人中的安全性、反应原性和免疫原性,受试者分别接种了1、2和5 μg mRNA,结果表明1和2 μg剂量的耐受性更好,二者均诱导产生了中和抗体,提高了IgG水平,没有引起相关的安全问题,与商品化狂犬病疫苗相当[66]。表 1中汇总了进入临床试验的人畜共患病mRNA疫苗。
|
|
表 1 进入临床试验的人畜共患病mRNA疫苗(截至2022年12月) Table 1 List of mRNA vaccines against zoonoses in clinical trials(till December 2022) |
与减毒活疫苗和灭活疫苗相比,mRNA疫苗以IVT mRNA为主要成分,排除了在传统疫苗中常见的内毒素和其他等感染风险。此外,mRNA疫苗进入细胞质即可表达,无需进入细胞核,不会产生DNA疫苗存在的基因组突变的风险,其安全性优于DNA疫苗和病毒活载体疫苗。且不同于传统疫苗动辄数年甚至数十年的研发周期,mRNA疫苗的研发速度也远超于其他疫苗,这种极短的研发周期对攻克具有高突变率特点的病毒来说,具有重要意义。但是,mRNA疫苗的研发也存在一些问题。由于知识产权分散,mRNA疫苗通常是由不同部门的许多参与者共同完成的,这种多头合作会增加沟通成本,降低mRNA疫苗研发效率[67]。此外,虽然使用LNP可以提高mRNA递送效率,但LNP的不稳定性要求mRNA疫苗的储存和运输需满足苛刻的条件。有研究显示,基于LNP的mRNA疫苗冻干化处理后可以在室温下长期储存,这为mRNA疫苗的储存和运输提供了一个潜在的解决方案[68]。随着全球合作的进一步深化,mRNA疫苗的专利限制和技术瓶颈等问题或许有望得到解决。
目前,虽然没有兽用mRNA疫苗进入临床试验阶段,但新冠mRNA疫苗的成功上市依旧点燃了人们对研发兽用mRNA疫苗的热情。我国是畜牧业大国,动物传染病是制约我国畜牧业发展的重要原因,其中人畜共患病也会对人类的健康和生命财产安全产生重大影响,而接种有效的疫苗是预防和控制传染病最有效的公共卫生干预措施之一[69]。当前影响畜牧业生产和发展的主要动物传染病如非洲猪瘟病毒(African swine fever virus, ASFV)、猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus, PRRSV)、猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)、口蹄疫病毒(foot and mouth disease virus, FMDV)、牛病毒性腹泻病毒(bovine viral diarrhea virus, BVDV)等,其疫苗保护效力均不理想,mRNA疫苗已在以上动物传染病疫苗研发中展现出了巨大的潜力。在此,作者将以动物冠状病毒和非洲猪瘟病毒为例展望mRNA疫苗在兽医领域的应用。
5.1 基于动物冠状病毒RBD区的mRNA疫苗冠状病毒的宿主谱广泛,除了人类之外,还能够感染猪、牛等家畜以及宠物犬、猫等多种动物[70]。感染不同物种的冠状病毒具有遗传结构相似性,刺突蛋白(S蛋白)是冠状病毒主要的结构蛋白,也是诱发细胞免疫和体液免疫的重要组成成分。S蛋白由S1和S2两个亚基组成,S1亚基包含一个受体结合域,即RBD区,负责与特定的受体结合,而S2亚基则介导病毒和宿主细胞膜的融合。各种证据表明,RBD区是诱导机体产生中和抗体的主要靶点,因此,这一区域也成为疫苗研发的重点目标[31]。新冠肺炎大流行期间,多个公司基于新冠病毒RBD区域设计开发了多款mRNA疫苗,数据显示,基于RBD区的候选疫苗,如BNT162b1、mRNA-1273等均具有良好的免疫原性[71]。
人类mRNA疫苗的成功为mRNA疫苗在兽医学中的应用展示了希望。猪流行性腹泻、猪传染性胃肠炎及新发的猪急性腹泻综合征均是由冠状病毒引发的严重危害养猪业的重要胃肠道传染病[70],常用的灭活疫苗、减毒活疫苗和基因工程疫苗虽然对于防控猪冠状病毒感染起到了一定的效果,但由于冠状病毒易发生重组或突变,常规疫苗在防控方面往往不尽如人意,研发基于猪冠状病毒RBD区的mRNA疫苗或许将为疾病防控提供新思路。在宠物疫病方面,猫传染性腹膜炎病毒(feline infectious peritonitis virus, FIPV)是目前发生最普遍的一种由冠状病毒变异株引起的高度致死性传染病,目前没有商品化的疫苗上市[72]。随着对动物冠状病毒RBD和细胞表面受体认识的进一步深入,基于RBD的mRNA疫苗将成为一种对抗新发和再发动物冠状病毒非常有前途的策略。
5.2 T细胞靶向的非洲猪瘟mRNA疫苗非洲猪瘟(ASF)是由非洲猪瘟病毒(ASFV)引起的猪的一种急性、热性、高度接触性传染病,所有品种和年龄的猪均可感染,发病率和死亡率最高可达100%。由于缺乏有效的疫苗,非洲猪瘟已经蔓延到许多国家并造成了严重的经济损失。我国2019年至今非洲猪瘟已造成近700万头猪死亡[73]。国内外科研人员虽然已经开发出多种删除了毒力基因的ASFV减毒活疫苗候选疫苗株,但这些减毒疫苗的大规模生产严重依赖于原代猪肺泡巨噬细胞或骨髓细胞,既耗时又费钱[74-76];基于结构蛋白设计的重组蛋白疫苗,虽会诱导出相应中和抗体,并延迟临床症状,但中和抗体无法提供充分的保护。研究证明T细胞在ASFV的抗病毒免疫和保护中发挥重要作用。近期,科研人员利用iVAX计算疫苗设计平台,在ASFV编码的蛋白质中鉴定出高度保守的细胞毒性T细胞表位[77-78],开发了一种含有预测的猪MHCⅠ类和Ⅱ类表位的T细胞靶向ASF DNA疫苗,并正在评估这种新型疫苗的免疫原性[79-81]。考虑到DNA疫苗存在整合到宿主基因组的潜在风险,基于mRNA的疫苗方法有可能促进安全有效ASF疫苗的改进。ASFV为大型双链DNA病毒,基因组大小为170~194 kb,含有150~167个开放阅读框,编码150~200种蛋白质,目前仍有一半以上的ASFV编码蛋白功能尚不清楚。随着研究的逐渐深入,更多潜在的T细胞表位将被挖掘,为开发具有更大表位范围的T细胞靶向mRNA疫苗提供了广阔前景。
| [1] |
WOLFF J, MALONE R, WILLIAMS P, et al. Direct gene transfer into mouse muscle in vivo[J]. Science, 1990, 247(4949): 1465-1468. DOI:10.1126/science.1690918 |
| [2] |
KWON S, KWON M, IM S, et al. mRNA vaccines: the most recent clinical applications of synthetic mRNA[J]. Arch Pharm Res, 2022, 45(4): 245-262. DOI:10.1007/s12272-022-01381-7 |
| [3] |
KARIKÓ K, BUCKSTEIN M, NI H, et al. Suppression of RNA recognition by Toll-like receptors: the impact of nucleoside modification and the evolutionary origin of RNA[J]. Immunity, 2005, 23(2): 165-175. DOI:10.1016/j.immuni.2005.06.008 |
| [4] |
KARIKÓ K, MURAMATSU H, WELSH F A, et al. Incorporation of pseudouridine into mRNA yields superior nonimmunogenic vector with increased translational capacity and biological stability[J]. Mol Ther, 2008, 16(11): 1833-1840. DOI:10.1038/mt.2008.200 |
| [5] |
KIM J, EYGERIS Y, GUPTA M, et al. Self-assembled mRNA vaccines[J]. Adv Drug Delivery Rev, 2021, 170: 83-112. DOI:10.1016/j.addr.2020.12.014 |
| [6] |
CHAUDHARY N, WEISSMAN D, WHITEHEAD K A. mRNA vaccines for infectious diseases: principles, delivery and clinical translation[J]. Nat Rev Drug Discov, 2021, 20(11): 817-838. DOI:10.1038/s41573-021-00283-5 |
| [7] |
RAMANATHAN A, ROBB G B, CHAN S H. mRNA capping: biological functions and applications[J]. Nucleic Acids Res, 2016, 44(16): 7511-7526. DOI:10.1093/nar/gkw551 |
| [8] |
DAFFIS S, SZRETTER K J, SCHRIEWER J, et al. 2'-O methylation of the viral mRNA cap evades host restriction by IFIT family members[J]. Nature, 2010, 468(7322): 452-456. DOI:10.1038/nature09489 |
| [9] |
QIN S G, TANG X S, CHEN Y T, et al. mRNA-based therapeutics: powerful and versatile tools to combat diseases[J]. Sig Transduct Target Ther, 2022, 7(1): 166. DOI:10.1038/s41392-022-01007-w |
| [10] |
HINNEBUSCH A G, IVANOV I P, SONENBERG N. Translational control by 5'-untranslated regions of eukaryotic mRNAs[J]. Science, 2016, 352(6292): 1413-1416. DOI:10.1126/science.aad9868 |
| [11] |
ZOHRA F T, CHOWDHURY E H, TADA S, et al. Effective delivery with enhanced translational activity synergistically accelerates mRNA-based transfection[J]. Biochem Biophys Res Commun, 2007, 358(1): 373-378. DOI:10.1016/j.bbrc.2007.04.059 |
| [12] |
LADAK R J, HE A J, HUANG Y H, et al. The current landscape of mRNA vaccines against viruses and cancer-A mini review[J]. Front Immunol, 2022, 13: 885371. DOI:10.3389/fimmu.2022.885371 |
| [13] |
MUTTACH F, MUTHMANN N, RENTMEISTER A. Synthetic mRNA capping[J]. Beilstein J Org Chem, 2017, 13: 2819-2832. DOI:10.3762/bjoc.13.274 |
| [14] |
HENDERSON J M, UJITA A, HILL E, et al. Cap 1 messenger RNA synthesis with Co-transcriptional CleanCapⓇ analog by in vitro transcription[J]. Curr Protoc, 2021, 1(2): e39. DOI:10.1002/cpz1.39 |
| [15] |
SHANMUGASUNDARAM M, SENTHILVELAN A, KORE A R. Recent advances in modified cap analogs: synthesis, biochemical properties, and mRNA based vaccines[J]. Chem Rec, 2022, 22(8): e202200005. |
| [16] |
CHATTERJEE S, PAL J K. Role of 5'-and 3'-untranslated regions of mRNAs in human diseases[J]. Biol Cell, 2009, 101(5): 251-262. DOI:10.1042/BC20080104 |
| [17] |
SCHROM E, HUBER M, ANEJA M, et al. Translation of angiotensin-converting enzyme 2 upon liver-and lung-targeted delivery of optimized chemically modified mRNA[J]. Mol Ther Nucleic Acids, 2017, 7: 350-365. DOI:10.1016/j.omtn.2017.04.006 |
| [18] |
SVITKIN Y V, PAUSE A, HAGHIGHAT A, et al. The requirement for eukaryotic initiation factor 4A (eIF4A) in translation is in direct proportion to the degree of mRNA 5' secondary structure[J]. RNA, 2001, 7(3): 382-394. DOI:10.1017/S135583820100108X |
| [19] |
BARREAU C, PAILLARD L, OSBORNE H B. AU-rich elements and associated factors: are there unifying principles?[J]. Nucleic Acids Res, 2005, 33(22): 7138-7150. DOI:10.1093/nar/gki1012 |
| [20] |
DE JONGH R P H, VAN DIJK A D J, JULSING M K, et al. Designing eukaryotic gene expression regulation using machine learning[J]. Trends Biotechnol, 2020, 38(2): 191-201. DOI:10.1016/j.tibtech.2019.07.007 |
| [21] |
THESS A, GRUND S, MUI B L, et al. Sequence-engineered mRNA without chemical nucleoside modifications enables an effective protein therapy in large animals[J]. Mol Ther, 2015, 23(9): 1456-1464. DOI:10.1038/mt.2015.103 |
| [22] |
KUDLA G, LIPINSKI L, CAFFIN F, et al. High guanine and cytosine content increases mRNA levels in mammalian cells[J]. PLoS Biol, 2006, 4(6): e180. DOI:10.1371/journal.pbio.0040180 |
| [23] |
WAYMENT-STEELE H K, KIM D S, CHOE C A, et al. Theoretical basis for stabilizing messenger RNA through secondary structure design[J]. Nucleic Acids Res, 2021, 49(18): 10604-10617. DOI:10.1093/nar/gkab764 |
| [24] |
YU S, KIM V N. A tale of non-canonical tails: gene regulation by post-transcriptional RNA tailing[J]. Nat Rev Mol Cell Biol, 2020, 21(9): 542-556. DOI:10.1038/s41580-020-0246-8 |
| [25] |
TANG T T L, PASSMORE L A. Recognition of poly(A) RNA through its intrinsic helical structure[J]. Cold Spring Harb Symp Quant Biol, 2019, 84: 21-30. DOI:10.1101/sqb.2019.84.039818 |
| [26] |
OH S, KESSLER J A. Design, assembly, production, and transfection of synthetic modified mRNA[J]. Methods, 2018, 133: 29-43. DOI:10.1016/j.ymeth.2017.10.008 |
| [27] |
ELANGO N, ELANGO S, SHIVSHANKAR P, et al. Optimized transfection of mRNA transcribed from a d(A/T)100 tail-containing vector[J]. Biochem Biophys Res Commun, 2005, 330(3): 958-966. DOI:10.1016/j.bbrc.2005.03.067 |
| [28] |
ECKMANN C R, RAMMELT C, WAHLE E. Control of poly(A) tail length[J]. Wiley Interdiscip Rev RNA, 2011, 2(3): 348-361. DOI:10.1002/wrna.56 |
| [29] |
LACZKÓ D, HOGAN M J, TOULMIN S A, et al. A single immunization with nucleoside-modified mRNA vaccines elicits strong cellular and humoral immune responses against SARS-CoV-2 in mice[J]. Immunity, 2020, 53(4): 724-732. DOI:10.1016/j.immuni.2020.07.019 |
| [30] |
ABU BAKAR F, NG L F P. Nonstructural proteins of alphavirus-potential targets for drug development[J]. Viruses, 2018, 10(2): 71. DOI:10.3390/v10020071 |
| [31] |
HAAKE C, COOK S, PUSTERLA N, et al. Coronavirus infections in companion animals: virology, epidemiology, clinical and pathologic features[J]. Viruses, 2020, 12(9): 1023. DOI:10.3390/v12091023 |
| [32] |
COHEN J. First self-copying mRNA vaccine proves itself in pandemic trial[J]. Science, 2022, 376(6592): 446. DOI:10.1126/science.abq7232 |
| [33] |
LI M Y, LI Y, LI S Q, et al. The nano delivery systems and applications of mRNA[J]. Eur J Med Chem, 2022, 227: 113910. DOI:10.1016/j.ejmech.2021.113910 |
| [34] |
KNUDSON C J, ALVES-PEIXOTO P, MURAMATSU H, et al. Lipid-nanoparticle-encapsulated mRNA vaccines induce protective memory CD8 T cells against a lethal viral infection[J]. Mol Ther, 2021, 29(9): 2769-2781. DOI:10.1016/j.ymthe.2021.05.011 |
| [35] |
JARZEBSKA N T, LAUCHLI S, ISELIN C, et al. Functional differences between protamine preparations for the transfection of mRNA[J]. Drug Deliv, 2020, 27(1): 1231-1235. DOI:10.1080/10717544.2020.1790692 |
| [36] |
COPPIN L, LECLERC J, VINCENT A, et al. Messenger RNA life-cycle in cancer cells: emerging role of conventional and non-conventional RNA-binding proteins?[J]. Int J Mol Sci, 2018, 19(3): 650. DOI:10.3390/ijms19030650 |
| [37] |
LI B, ZHANG X F, DONG Y Z. Nanoscale platforms for messenger RNA delivery[J]. Wiley Interdiscip Rev Nanomed Nanobiotechnol, 2019, 11(2): e1530. |
| [38] |
EYGERIS Y, PATEL S, JOZIC A, et al. Deconvoluting lipid nanoparticle structure for messenger RNA delivery[J]. Nano Lett, 2020, 20(6): 4543-4549. DOI:10.1021/acs.nanolett.0c01386 |
| [39] |
ALAMEH M G, TOMBÁCZ I, BETTINI E, et al. Lipid nanoparticles enhance the efficacy of mRNA and protein subunit vaccines by inducing robust T follicular helper cell and humoral responses[J]. Immunity, 2021, 54(12): 2877-2892. DOI:10.1016/j.immuni.2021.11.001 |
| [40] |
KOWALSKI P S, RUDRA A, MIAO L, et al. Delivering the messenger: advances in technologies for therapeutic mRNA delivery[J]. Mol Ther, 2019, 27(4): 710-728. DOI:10.1016/j.ymthe.2019.02.012 |
| [41] |
HOU X C, ZAKS T, LANGER R, et al. Lipid nanoparticles for mRNA delivery[J]. Nat Rev Mater, 2021, 6(12): 1078-1094. DOI:10.1038/s41578-021-00358-0 |
| [42] |
BADEN L R, EL SAHLY H M, ESSINK B, et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine[J]. N Engl J Med, 2021, 384(5): 403-416. DOI:10.1056/NEJMoa2035389 |
| [43] |
CUI S H, WANG Y Y, GONG Y, et al. Correlation of the cytotoxic effects of cationic lipids with their headgroups[J]. Toxicol Res (Camb), 2018, 7(3): 473-479. DOI:10.1039/C8TX00005K |
| [44] |
GUIMARAES P P G, ZHANG R, SPEKTOR R, et al. Ionizable lipid nanoparticles encapsulating barcoded mRNA for accelerated in vivo delivery screening[J]. J Control Release, 2019, 316: 404-417. DOI:10.1016/j.jconrel.2019.10.028 |
| [45] |
HORIUCHI Y, LAI S J, YAMAZAKI A, et al. Validation and application of a novel cholesterol efflux assay using immobilized liposomes as a substitute for cultured cells[J]. Biosci Rep, 2018, 38(2): BSR20180144. DOI:10.1042/BSR20180144 |
| [46] |
BASKARARAJ S, PANNEERSELVAM T, GOVINDARAJ S, et al. Formulation and characterization of folate receptor-targeted PEGylated liposome encapsulating bioactive compounds from Kappaphycus alvarezii for cancer therapy[J]. 3 Biotech, 2020, 10(3): 136. DOI:10.1007/s13205-020-2132-7 |
| [47] |
ERASMUS J H, KHANDHAR A P, GUDERIAN J, et al. A nanostructured lipid carrier for delivery of a replicating viral RNA provides single, low-dose protection against Zika[J]. Mol Ther, 2018, 26(10): 2507-2522. DOI:10.1016/j.ymthe.2018.07.010 |
| [48] |
TO K K W, CHO W C S. An overview of rational design of mRNA-based therapeutics and vaccines[J]. Expert Opin Drug Discov, 2021, 16(11): 1307-1317. DOI:10.1080/17460441.2021.1935859 |
| [49] |
ZHANG C L, MARUGGI G, SHAN H, et al. Advances in mRNA vaccines for infectious diseases[J]. Front Immunol, 2019, 10: 594. DOI:10.3389/fimmu.2019.00594 |
| [50] |
SCHOENMAKER L, WITZIGMANN D, KULKARNI J A, et al. mRNA-lipid nanoparticle COVID-19 vaccines: structure and stability[J]. Int J Pharm, 2021, 601: 120586. DOI:10.1016/j.ijpharm.2021.120586 |
| [51] |
VERBEKE R, HOGAN M J, LORÉ K, et al. Innate immune mechanisms of mRNA vaccines[J]. Immunity, 2022, 55(11): 1993-2005. DOI:10.1016/j.immuni.2022.10.014 |
| [52] |
COOLEN A L, LACROIX C, MERCIER-GOUY P, et al. Poly(lactic acid) nanoparticles and cell-penetrating peptide potentiate mRNA-based vaccine expression in dendritic cells triggering their activation[J]. Biomaterials, 2019, 195: 23-37. DOI:10.1016/j.biomaterials.2018.12.019 |
| [53] |
PROBST J, WEIDE B, SCHEEL B, et al. Spontaneous cellular uptake of exogenous messenger RNA in vivo is nucleic acid-specific, saturable and ion dependent[J]. Gene Ther, 2007, 14(15): 1175-1180. DOI:10.1038/sj.gt.3302964 |
| [54] |
PARDI N, HOGAN M J, PORTER F W, et al. mRNA vaccines-a new era in vaccinology[J]. Nat Rev Drug Discov, 2018, 17(4): 261-279. DOI:10.1038/nrd.2017.243 |
| [55] |
LINDSAY K E, BHOSLE S M, ZURLA C, et al. Visualization of early events in mRNA vaccine delivery in non-human primates via PET-CT and near-infrared imaging[J]. Nat Biomed Eng, 2019, 3(5): 371-380. DOI:10.1038/s41551-019-0378-3 |
| [56] |
MVNZ C. Antigen processing for MHC class ii presentation via autophagy[J]. Front Immunol, 2012, 3: 9. |
| [57] |
BELL G D, YANG Y, LEUNG E, et al. mRNA transfection by a Xentry-protamine cell-penetrating peptide is enhanced by TLR antagonist E6446[J]. PLoS One, 2018, 13(7): e0201464. DOI:10.1371/journal.pone.0201464 |
| [58] |
BAEZA GARCIA A, SIU E, SUN T, et al. Neutralization of the Plasmodium-encoded MIF ortholog confers protective immunity against malaria infection[J]. Nat Commun, 2018, 9(1): 2714. DOI:10.1038/s41467-018-05041-7 |
| [59] |
PARDI N, HOGAN M J, NARADIKIAN M S, et al. Nucleoside-modified mRNA vaccines induce potent T follicular helper and germinal center B cell responses[J]. J Exp Med, 2018, 215(6): 1571-1588. DOI:10.1084/jem.20171450 |
| [60] |
MARUGGI G, CHIAROT E, GIOVANI C, et al. Immunogenicity and protective efficacy induced by self-amplifying mRNA vaccines encoding bacterial antigens[J]. Vaccine, 2017, 35(2): 361-368. DOI:10.1016/j.vaccine.2016.11.040 |
| [61] |
CORBETT K S, EDWARDS D K, LEIST S R, et al. SARS-CoV-2 mRNA vaccine design enabled by prototype pathogen preparedness[J]. Nature, 2020, 586(7830): 567-571. DOI:10.1038/s41586-020-2622-0 |
| [62] |
THOMAS S J, MOREIRA E D Jr, KITCHIN N, et al. Safety and efficacy of the BNT162b2 mRNA Covid-19 vaccine through 6 months[J]. N Engl J Med, 2021, 385(19): 1761-1773. DOI:10.1056/NEJMoa2110345 |
| [63] |
KADAMBARI S, EVANS C, LYALL H. Congenital infections: priorities and possibilities for resource-limited settings[J]. Pediatr Infect Dis J, 2023, 42(2): e45-e47. DOI:10.1097/INF.0000000000003710 |
| [64] |
ABBASI J. Moderna 's mRNA vaccine for seasonal flu enters clinical trials[J]. JAMA, 2021, 326(14): 1365. |
| [65] |
FELDMAN R A, FUHR R, SMOLENOV I, et al. mRNA vaccines against H10N8 and H7N9 influenza viruses of pandemic potential are immunogenic and well tolerated in healthy adults in phase 1 randomized clinical trials[J]. Vaccine, 2019, 37(25): 3326-3334. DOI:10.1016/j.vaccine.2019.04.074 |
| [66] |
ALDRICH C, LEROUX-ROELS I, HUANG K B, et al. Proof-of-concept of a low-dose unmodified mRNA-based rabies vaccine formulated with lipid nanoparticles in human volunteers: a phase 1 trial[J]. Vaccine, 2021, 39(8): 1310-1318. DOI:10.1016/j.vaccine.2020.12.070 |
| [67] |
KUMAR A, BLUM J, THANH LE T, et al. The mRNA vaccine development landscape for infectious diseases[J]. Nat Rev Drug Discov, 2022, 21(5): 333-334. DOI:10.1038/d41573-022-00035-z |
| [68] |
MURAMATSU H, LAM K, BAJUSZ C, et al. Lyophilization provides long-term stability for a lipid nanoparticle-formulated, nucleoside-modified mRNA vaccine[J]. Mol Ther, 2022, 30(5): 1941-1951. DOI:10.1016/j.ymthe.2022.02.001 |
| [69] |
RAHMAN T, SOBUR A, ISLAM S, et al. Zoonotic diseases: etiology, impact, and control[J]. Microorganisms, 2020, 8(9): 1405. DOI:10.3390/microorganisms8091405 |
| [70] |
WOO P C Y, LAU S K P, HUANG Y, et al. Coronavirus diversity, phylogeny and interspecies jumping[J]. Exp Biol Med (Maywood), 2009, 234(10): 1117-1127. DOI:10.3181/0903-MR-94 |
| [71] |
GREANEY A J, LOES A N, GENTLES L E, et al. Antibodies elicited by mRNA-1273 vaccination bind more broadly to the receptor binding domain than do those from SARS-CoV-2 infection[J]. Sci Transl Med, 2021, 13(600): eabi9915. DOI:10.1126/scitranslmed.abi9915 |
| [72] |
IZES A M, YU J, NORRIS J M, et al. Current status on treatment options for feline infectious peritonitis and SARS-CoV-2 positive cats[J]. Vet Quart, 2020, 40(1): 322-330. DOI:10.1080/01652176.2020.1845917 |
| [73] |
ZHAO D M, LIU R Q, ZHANG X F, et al. Replication and virulence in pigs of the first African swine fever virus isolated in China[J]. Emerg Microbes Infect, 2019, 8(1): 438-447. DOI:10.1080/22221751.2019.1590128 |
| [74] |
CHEN W Y, ZHAO D M, HE X J, et al. A seven-gene-deleted African swine fever virus is safe and effective as a live attenuated vaccine in pigs[J]. Sci China Life Sci, 2020, 63(5): 623-634. DOI:10.1007/s11427-020-1657-9 |
| [75] |
BORCA M V, RAMIREZ-MEDINA E, SILVA E, et al. ASFV-G-ΔI177L as an effective oral nasal vaccine against the Eurasia strain of Africa swine fever[J]. Viruses, 2021, 13(5): 765. DOI:10.3390/v13050765 |
| [76] |
BORCA M V, RAMIREZ-MEDINA E, SILVA E, et al. Development of a highly effective African swine fever virus vaccine by deletion of the I177L gene results in sterile immunity against the current epidemic Eurasia strain[J]. J Virol, 2020, 94(7): e02017-19. |
| [77] |
ROS-LUCAS A, CORREA-FIZ F, BOSCH-CAMÓS L, et al. Computational analysis of African swine fever virus protein space for the design of an epitope-based vaccine ensemble[J]. Pathogens, 2020, 9(12): 1078. DOI:10.3390/pathogens9121078 |
| [78] |
BOSCH-CAMÓS L, LÓPEZ E, NAVAS M J, et al. Identification of promiscuous African swine fever virus T-cell determinants using a multiple technical approach[J]. Vaccines (Basel), 2021, 9(1): 29. DOI:10.3390/vaccines9010029 |
| [79] |
BURMAKINA G, MALOGOLOVKIN A, TULMAN E R, et al. Identification of T-cell epitopes in African swine fever virus CD2v and C-type lectin proteins[J]. J Gen Virol, 2019, 100(2): 259-265. DOI:10.1099/jgv.0.001195 |
| [80] |
MOISE L, GUTIÉRREZ A H, KHAN S, et al. New immunoinformatics tools for swine: designing epitope-driven vaccines, predicting vaccine efficacy, and making vaccines on demand[J]. Front Immunol, 2020, 11: 563362. DOI:10.3389/fimmu.2020.563362 |
| [81] |
PÉREZ-NÚÑEZ D, SUNWOO S Y, SÁNCHEZ E G, et al. Evaluation of a viral DNA-protein immunization strategy against African swine fever in domestic pigs[J]. Vet Immunol Immunopathol, 2019, 208: 34-43. DOI:10.1016/j.vetimm.2018.11.018 |
(编辑 白永平)