2. 甘孜县农牧农村和科技局, 甘孜 626700
2. Ganzi County Agriculture, Animal Husbandry, Rural and Technology Bureau, Ganzi Tibetan Autonomous Prefecture, SichuanGanzi 626700, China
仔猪腹泻已成为世界各地养猪业的常见疾病之一,是导致仔猪死亡率高的主要原因,尤其在秋冬季节具有较高的发病率。腹泻造成仔猪成活率下降、生长缓慢、发育停滞成为僵猪、饲料回报率低、甚至死亡,给养猪业带来巨大的经济损失。导致仔猪腹泻的原因有细菌、病毒、寄生虫和饲养管理等因素[1],其中仔猪病毒性腹泻造成的危害最为严重。目前已知能导致仔猪腹泻的病原有猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)、猪传染性胃肠炎病毒(transmissible gastroenteritis virus,TGEV)和猪轮状病毒(porcine rotavirus,PoRV)。随着高通量测序技术的发展,一些导致仔猪腹泻的新型病原陆续被发现,如猪丁型冠状病毒(porcine deltacoronavirus,PDCoV)[2-3]、猪急性腹泻综合征冠状病毒(swine acute diarrhea syndrome coronavirus,SADS-CoV)[4]。这些新病原的出现对仔猪病毒性腹泻防控带来一定的挑战,而近年来是否有一些能导致仔猪腹泻的新型病毒的出现有待进一步调查。
病毒宏基因组学(viral metagenomics)是利用二代高通量测序技术来研究特定环境中病毒群落的一项技术方法[5]。该技术突破了以往鉴定病毒的技术局限,具有从混合感染样本中识别全部病毒种类的优势,并可用于检测难以分离培养的病毒和发现新的病原体,以及监测病毒的遗传变异[6]。如2019年武汉出现的SARS-CoV-2,Lu等[7]利用高通量测序技术迅速确定引起该病的病原为新型冠状病毒,并获得全基因组序列。本团队2012年利用病毒宏基因组学的技术对四川地区腹泻仔猪粪便中的病毒种群进行研究,发现四川地区腹泻仔猪粪便中的病毒群落复杂多样[8]。为了进一步研究四川地区腹泻仔猪粪便中病毒种群的变化,2019年采集了四川地区15个集约化猪场中腹泻仔猪粪便并运用病毒宏基因组学技术进行分析,进而为仔猪腹泻的防控提供一定的参考依据。
1 材料与方法 1.1 材料1.1.1 样品采集 2019年从四川不同区域的15个集约化猪场中各采集腹泻仔猪粪便样本共156份,仔猪品种均为外三元,仔猪日龄为20~30 d,采样信息如表 1,以上猪场均免疫商品化的仔猪腹泻相关疫苗。粪便样本冰上运输, 并冻存于-80 ℃冰箱。
|
|
表 1 四川部分地区猪场采样信息 Table 1 The sample information of some pig farms in Sichuan |
1.1.2 主要试剂 0.22 μm细菌滤器,Millipore公司产品;PBS,赛默飞公司产品;RNA/DNA Shield(核酸液体保护剂),复申生物科技有限公司产品;QIAamp Viral RNA Mini试剂盒,QIAGEN有限公司产品;反转录试剂,成都市蓉为基因生物科技有限公司产品;DNase、RNase,大连宝生物工程有限公司产品。
1.2 方法1.2.1 样本处理及总核酸提取 在15个集约化猪场中各取3份腹泻样本,大约3 g重悬在10 mL PBS中涡旋混匀,将来自15个集约化猪场中的粪便悬浮液以每个100 μL混合成一个pool样,并以8 000 r ·min-1 4 ℃离心15 min。0.22 μm过滤器(Millipore)过滤上清液以去除完整的细菌和宿主细胞碎片。含有病毒颗粒的滤液在37℃下用DNase和RNase(TaKaRa)的混合物处理90 min以去除未受保护的核酸。使用QIAamp Viral RNA Mini试剂盒(QIAGEN)从样品中提取总RNA。使用Qubit分光光度计(Invitrogen,Carlsbad,CA,USA)测量提取的RNA的浓度和纯度,并对RNA进行反转录得到cDNA,将获得的cDNA用Qubit分光光度计来评估是否可以用于构建文库。
1.2.2 TruSeq Illumina测序、建库及数据分析 cDNA送到华大基因有限公司进行测序。使用超声波对cDNA(500 ng)进行机械破碎。测序在Illumina HiSeq 4000上进行。对原始reads进行预处理包括修剪和去除短于150 bp读长的reads并进行文库制备。对得到的原始数据在http://www.virome-swun.cn/网站进行病毒序列分析。
1.2.3 序列组装及系统发育分析 对在Illumina HiSeq 4000的测序结果进行筛选,对原始数据进行质控并去除低质量的raw reads数据,只有经过质控的clean reads才能进行下游分析。针对clean reads中的序列,通过使用NCBI refseq数据库比较得到的宿主、细菌或真菌等序列被删除。并将剩余的clean reads与病毒基因组数据库比对,然后使用diamond将未比对上的reads与来自NCBI Refseq DB的病毒蛋白数据库比对,按科属种等信息对已知病毒进行注释分类。对于与所有数据库都比对不上的reads,使用megahit进行组装,丰度前1000的重叠群序列(contigs)被用来进行新病毒的预测。使用SOAP denovo组装软件对reads丰度较高的病毒重叠群序列(contigs)进行从头组装拼接成更大的重叠群或基因组序列。利用MEGA 7.0软件构建了组装好的病毒基因组序列与NCBI数据库的核酸序列比对和氨基酸序列比对。使用Megalign对多序列比对结果进行分析以计算它们的同源性。最后利用MEGA 7.0软件中的Neighbor-Joining(Bootstrap值设为1 000)法建立系统进化树。对组装好的序列和高通量测序结果已全部提交到GenBank中,(BioProject:PRJNA831587、SRA:SRR18908183)。
2 结果 2.1 腹泻仔猪粪便样本的Illumina测序结果通过高通量测序从本次采集的腹泻仔猪粪便中共获得了2 492 650条raw reads,在经过NCBI对比后去除宿主、真菌、细菌和噬菌体的reads,获得与哺乳动物病毒相关的reads数共531 741条,占总reads数的21.332%。使用BLASTX软件分析发现该腹泻仔猪粪便病毒种群包括12个病毒科(如图 1a),并对序列进行详细比对后发现与此对应的有20种病毒(如图 1b),各病毒科序列按reads数占比顺序如下:冠状病毒科(Coronaviridae)、微小RNA病毒科(Picornaviridae)、杯状病毒科(Caliciviridae)、星状病毒科(Astroviridae)、细小病毒科(Parvoviridae)、腺病毒科(Adenoviridae)、呼肠孤病毒科(Reoviridae)、疱疹病毒科(Herpesviridae)、基因病毒科(Genomoviridae)、鳞状病毒科(Smacoviridae)、环曲病毒亚科(Torovirinae)、小双节RNA病毒科(Picobirnaviridae)。经序列比对得到的20种病毒按照reads数占比依次是:猪流行性腹泻病毒(PEDV)、猪嵴病毒(porcine kobuvirus,PKV)、猪传染性胃肠炎病毒(TGEV)、猪札幌病毒(porcine sapovirus,PoSaV)、猪星状病毒2型(PAstV-2)、波萨病毒1型(porcine stool-associated RNA virus 1,Posavirus 1)、猪腺病毒3型(porcine adenovirus 3,PADV-3)、猪细小病毒4型(PPV-4)、猪细小病毒5型(porcine parvovirus 5,PPV-5)、猪捷申病毒(porcine teschovirus,PTV)、猪萨佩罗病毒(porcine sapelovirus,PSV)、猪博卡病毒3型(porcine bocavirus 3,PBoV-3)、猪轮状病毒(PoRV)、猪伪狂犬病病毒(porcine pseudorabies virus,PRV)、猪粪便相关巨环病毒(porcine feces-associated gemycircularvirus,PFGV)、猪血凝性脑脊髓炎病毒(porcine hemagglutinating encephalomyelitis virus,PHEV)、猪粪便相关病毒7型(porcine stool-associated virus 7,PoSCV-7)、猪环曲病毒(porcine torovirus,PToV)、(swine pasivirus 1,SPaV-1)、(porcine picobirnavirus,PBV)。
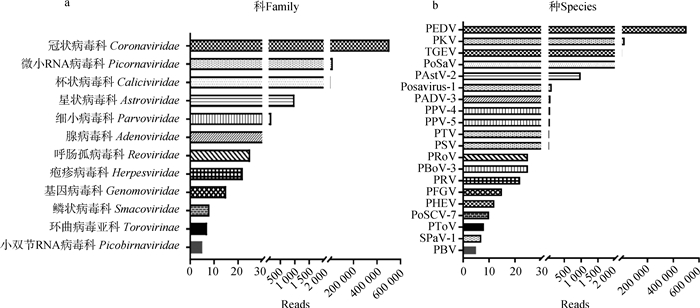
|
图 1 腹泻仔猪粪便样本中检测到的病毒科(a)和病毒种(b)的序列分类 Fig. 1 Sequence classification of virus families (a) and virus species (b) detected in fecal samples from piglets with diarrhea |
本团队曾在2012年从四川省集约化猪场中采集仔猪粪便样本,仔猪为20~30日龄,采集了12个猪场的27份腹泻仔猪粪便样本和4个猪场的29份健康仔猪粪便样本并通过病毒宏基因组学技术鉴定了15种病毒。如图 2所示, 与2012年数据相比有9种病毒在2019年被持续检测到,按照reads数占比分别是PEDV、PKV、PoSaV、PAstV-2、Posavirus-1、PSV、PoSCV-7、PToV和PBV。有趣的是多达11种病毒是2012年未被监测到的,按照reads数占比分别是TGEV、PADV-3、PPV-4、PPV-5、PTV、PRoV、PBoV-3、PRV、PFGV、PHEV和SPaV-1。
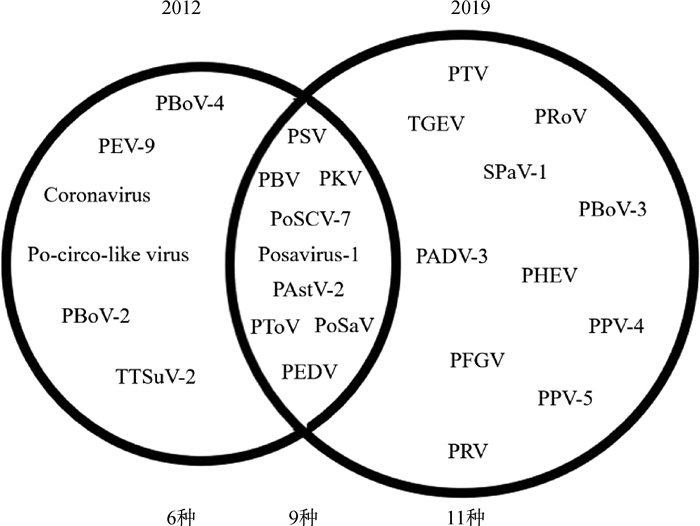
|
图 2 四川省2012与2019年仔猪粪便中病毒群落 Fig. 2 Piglet fecal virus populations in Sichuan Province, 2012 and 2019 |
使用SOAP denovo组装软件对20种病毒的contigs进行组装,组装出5个病毒基因组的部分序列(表 2),包括PEDV完整的N蛋白基因序列,以及TGEV ORF1a部分基因序列、PAstV-2 ORF1a部分基因序列、PPV-4 NS1部分基因序列、以及Posavirus 1的部分基因组,并对组装出的序列进行遗传进化分析。
|
|
表 2 通过深度测序在仔猪腹泻粪便库中进行病毒组装 Table 2 Viruses assembled in piglet feces samples with diarrhea libraries by deep sequencing |
2.3.1 PEDV序列组装分析 在腹泻仔猪粪便样本中共检测到502 757个对应于PEDV序列一致性的reads,并组装出PEDV完整的N基因序列,其组装出的N基因序列长度为1 325 bp,覆盖PEDV毒株N基因序列100%,GC含量为36.00%,与33株PEDV GenBank参考病毒序列相比,本研究中的PEDV N 基因与GⅡ亚群N基因的核苷酸相似性为96.1%~99.8%,氨基酸相似性为97.3%~99.8%;与GⅠ亚群N基因核苷酸相似性为94.0%~95.8%,氨基酸相似性为96.0%~97.1%。基于PEDV N基因建立的系统发育树分析表明(图 2),本研究中的PEDV与OL762 458.1/SDLY/China/2020和OL762 457.1/HK/China/2021聚为一支,提示本研究中PEDV有可能为近些年流行的新毒株。
2.3.2 TGEV序列组装分析 在腹泻仔猪粪便样本中共检测到2 688条具有对应于TGEV序列同一性的reads,并组装出TGEV位于ORF1a的部分序列,其长度为2 315 bp,覆盖TGEV参考毒株ORF1a基因序列18.77%,与27株TGEV GenBank参考病毒序列相比,本研究中TGEV ORF1a基因与其它TGEV ORF1a的核苷酸相似性为96.5%~99.6%,氨基酸相似性为96.9%~99.6%。基于TGEV ORF1a基因建立的系统发育树分析结果表明(图 3a),本研究中的TGEV与KT696 544.1 JS2012/China/2012聚为一支,跟EU074 218.2 TGEV attenuated H/China/2007经典疫苗株同属于Miller群。
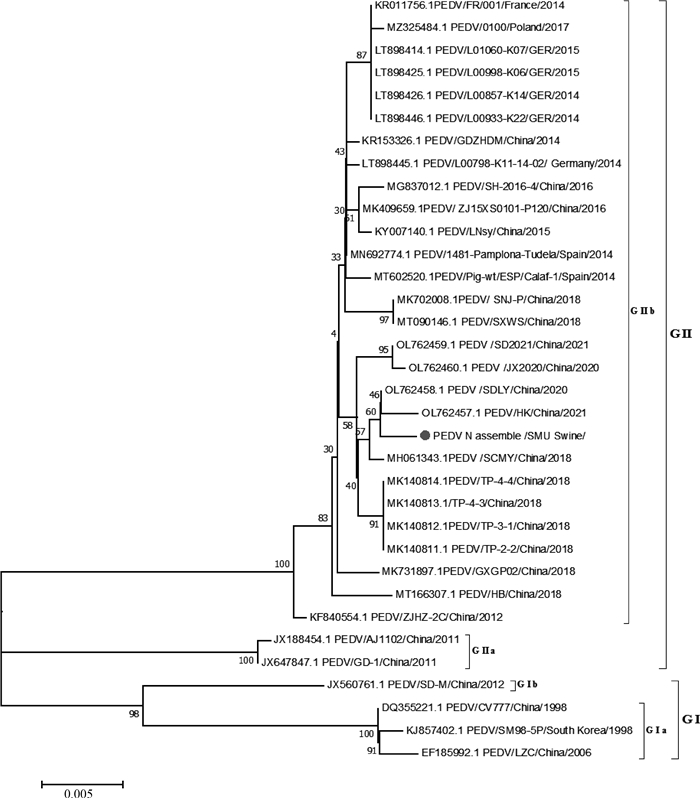
|
图 3 PEDV N基因拼接序列遗传进化分析(本研究病毒序列用●标记) Fig. 3 Genetic evolutionary analysis of PEDV N gene splicing sequences (viral sequences in this study were marked with ●) |
2.3.3 PAstV-2序列组装分析 在腹泻仔猪粪便样本中共检测到987条具有对应于PAstV-2序列同一性的reads,并组装出PAstV-2 ORF1a的部分序列,其组装出来ORF1a的长度为713 bp,覆盖PAstV-2参考毒株ORF1a基因序列28.84%,与25株ORF1a GenBank参考病毒序列相比,PAstV-2 ORF1a基因的核苷酸相似性为80.6%~93.4%。基于PAstV-2 ORF1a基因建立的系统发育树分析结果表明(图 3b),本研究中的PAstV-2与KJ495 986.1 PAstV-2/ExpPig-36/USA/2012和MW784 075.1 PAstV-2/HB-85/China/2015聚为一支。
2.3.4 Posavirus 1序列组装分析 在腹泻仔猪粪便样本中共检测到127条具有对应于Posavirus 1序列同一性的reads,并组装出Posavirus 1的部分序列,其中长度为814 bp,覆盖Posavirus 1参考毒株全基因序列的8.58%,与26株Posavirus 1 GenBank参考病毒序列相比,Posavirus 1的核苷酸相似性为84.1%~97.1%。基于组装出的Posavirus 1部分基因序列建立的系统发育树分析结果表明(图 3c),本研究中的Posavirus 1与KU981 058.1/HBTS-11/China/2015聚为一支。
2.3.5 PPV-4序列组装分析 在仔猪粪便样本中共检测到80条具有对应于PPV-4序列同一性的reads,并组装完整的PPV-4 NS1基因序列,其中组装出的非结构蛋白NS1长度为2 246 bp,覆盖PPV-4参考毒株NS1基因序列100%,与23株PPV-4 GenBank参考病毒序列相比,PPV-4 NS1基因的核苷酸相似性为96.2%~99.9%。基于PPV-4 NS1基因建立的系统发育树分析结果表明(图 3d),本研究中的PPV-4与MK378 269.1 HuN02 C1/China/2018聚为一支。
3 讨论自从高通量测序和病毒宏基因组学分析方法出现以来,病毒宏基因组学在研究未知疾病和鉴定新病毒方面显示出巨大的潜力,如今已经成为探索新病毒的主流工具,并被学者们广泛使用[9]。自2007年以来,已有大量通过宏病毒组测序在人类和动物中发现新病毒群落的研究报道[10]。近年来,该方法在分析病毒组和鉴定血清、血浆、粪便、呼吸道分泌物和组织样本中的病毒方面取得了成功[11]。如今已经对各种动物腹泻粪便中的病毒群落组进行了表征。如2015年Chen等[12]利用病毒宏基因组学技术在腹泻牦牛粪便中发现了一种新型星状病毒。2019年Fahsbender等[13]通过病毒宏基因组学技术分析犬腹泻粪便样本, 并发现了一种新型的细小病毒。2021年阿比克哈莫(Abi)等[14]使用病毒宏基因组学方法在藏绵羊的腹泻粪便样本中发现了一种新型嵴病毒。并且也有学者通过病毒宏基因学技术在猪粪便中发现了一些与腹泻相关的病毒,包括猪肠道α冠状病毒(swine enteric alphacoronavirus,SeACoV)[15-16]、猪杯状病毒(porcine calicivirus)[17]、猪捷申病毒(porcine teschoviruses,PTV)[18]、Bufavirus[19]等。与猪腹泻相关的新发病毒的存在值得继续研究。
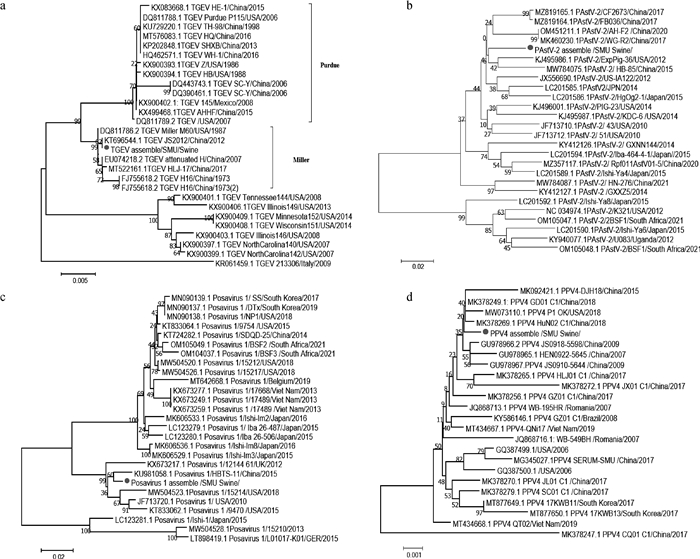
|
a. TGEV;b. PAstV-2;c. Posavirus 1;d. PPV-4的遗传进化分析 a. TGEV; b. PAstV-2; c. Posavirus 1; d. PPV-4 phylogenetic analysis 图 4 拼接序列遗传进化分析(本研究病毒序列用●标记) Fig. 4 Phylogenetic analysis of assembled sequences (viral sequences in this study were marked with ●) |
本研究通过病毒宏基因组学手段,在2019年对四川省15个集约化猪场中腹泻仔猪粪便的病毒群落进行分析,数据分析结果表明该地区腹泻仔猪粪便样本中共包含12个病毒科的20种病毒。与本团队在2012年鉴定的15种病毒相比病毒种类更为复杂多样,有多达11种病毒是2012年未被监测到的[8]。而且这两次的研究中冠状病毒科成员持续存在,且在本次研究中的含量更高reads含量达到95.05%,包括了α冠状病毒属的猪流行性腹泻病毒(PEDV)、猪传染胃肠炎病毒(TGEV)和β冠状病毒属的猪血凝性脑脊髓炎病毒(PHEV)[20],它们在临床上都能引起仔猪的水样腹泻和呕吐等症状。冠状病毒具有胃肠道、呼吸道、中枢神经系统的嗜性,而且表现出很高的突变率,使它们之间频繁发生重组突变克服宿主物种的障碍,从而实现跨种间传播[21]。例如,从一名腹泻儿童身上分离出的一种β冠状病毒HECoV4408被认为起源于牛类[22]。Woo等[23]发现禽类德尔塔冠状病毒可以感染猪。Zhou等[4]在中国广东省腹泻仔猪粪便中发现了一种新的蝙蝠源猪急性腹泻综合征冠状病毒(SADS-CoV),在中国广东省已导致4个猪场的24 693头仔猪死亡。关于冠状病毒的跨种间传播,引起了人们的高度重视。因此,有必要对猪体内的冠状病毒进行持续和密集的监测其是否发生变异和跨种间传播。此外猪嵴病毒(PKV)和猪札幌病毒(PoSaV)也检测到较高的reads含量,但因本次建库获得的拼接序列较短并未对该病毒进行遗传进化分析;大量流行病学和组织病理学等研究表明这两种病毒与仔猪腹泻高度相关,应在临床中引起高度重视[24-25]。
本次研究中通过病毒宏基因组学首次在四川的集约化猪场腹泻仔猪粪便中发现一种新发病毒SPaV-1。SPaV-1属于微小RNA病毒科(Picornaviridae)成员,在法国健康猪粪便中被首次发现[26]。Zaulet等[27]在罗马尼亚东部7个密集养猪场的不同生产阶段的健康猪身上采集了564个猪的样本,包括82个粪便样本和482个器官样本,在所调查的7个地区中的6个地区发现了该病毒,在分析的564份样本中,218份SPaV-1呈阳性,器官匀浆中病毒的流行率最高(39% 阳性),其次是粪便(37% 阳性)。Chen等[28]在2015—2016年从美国17个州收集了217个PEDV阳性的仔猪腹泻样本,其中SPaV-1的检出率为(9%,20/217)。胡承哲等[6]在腹泻藏猪粪便中也曾发现SPaV-1的存在。但该病毒是否能导致仔猪腹泻还需进一步研究。以上结果表明,上述SPaV-1可能与仔猪腹泻有一定联系,因此对于SPaV-1在四川集约化猪场中流行情况有待一步调查。在利用SOAP denovo组装软件得到的5种病毒的部分基因序列分析得出,本研究中根据PEDV完整的N基因建立的系统发育树发现其与OL762 458.1/SDLY/China/2020和OL762 457.1/HK/China/2021聚为一支且属于GⅡ b亚群,与经典疫苗毒株CV777和变异毒株AJ1102遗传关系较远,为近几年流行的新型变异毒株;近些年PEDV变异毒株在国内不断循环,这可能是导致经典毒株疫苗不能对变异毒株产生保护的原因。根据TGEV ORF1a基因建立的系统发育树分析结果表明本研究中的TGEV与KT696 544.1 JS2012/China/2012聚为一支, 且相似性达到99.6%,跟国内经典疫苗株EU074 218.2 TGEV attenuated H/China/2007同属于Miller群。以上结果有助于为四川省集约化猪场中仔猪腹泻的防控提供参考。
4 结论利用宏病毒基因组学方法获得了2019年四川省15个集约化猪场中腹泻仔猪粪便的病毒群落信息,鉴定了20种病毒,病毒种群相较于2012年更为复杂多样;其冠状病毒的含量较高,是否存在与其他物种相互感染的情况,值得高度重视;此外还在腹泻仔猪粪便中发现一种新发病毒,为四川省仔猪腹泻的防控提供一定的数据支撑。
| [1] |
王红宁. 仔猪腹泻成因及综合防治技术措施[J]. 中国畜牧杂志, 2006, 42(6): 58-60. WANG H N. The cause and integrated control of piglet diarrhea[J]. Chinese Journal of Animal Science, 2006, 42(6): 58-60. DOI:10.3969/j.issn.0258-7033.2006.06.020 (in Chinese) |
| [2] |
LI G, ZHAI S L, ZHOU X, et al. Phylogeography and evolutionary dynamics analysis of porcine delta-coronavirus with host expansion to humans[J]. Transbound Emerg Dis, 2022, 69(5): e1670-e1681. |
| [3] |
PÉREZ-RIVERA C, RAMÍREZ-MENDOZA H, MENDOZA-ELVIRA S, et al. First report and phylogenetic analysis of porcine deltacoronavirus in Mexico[J]. Transbound Emerg Dis, 2019, 66(4): 1436-1441. |
| [4] |
ZHOU P, FAN H, LAN T, et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin[J]. Nature, 2018, 556(7700): 255-258. DOI:10.1038/s41586-018-0010-9 |
| [5] |
韩文, 罗玉子, 赵碧波, 等. 基于宏基因组学的猪群样本病毒探测方法的建立[J]. 微生物学报, 2013, 53(2): 197-203. HAN W, LUO Y Z, ZHAO B B, et al. Metagenomics-based detection of swine viruses[J]. Acta Microbiologica Sinica, 2013, 53(2): 197-203. DOI:10.13343/j.cnki.wsxb.2013.02.005 (in Chinese) |
| [6] |
胡承哲, 周群, 李玉, 等. 利用宏基因组学鉴定藏猪腹泻粪便病毒种群[J]. 畜牧兽医学报, 2019, 50(12): 2479-2487. HU C Z, ZHOU Q, LI Y, et al. Identification of viral community in diarrheal feces of Tibetan pig by metagenomics[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(12): 2479-2487. DOI:10.11843/j.issn.0366-6964.2019.12.012 (in Chinese) |
| [7] |
LU R J, ZHAO X, LI J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding[J]. Lancet, 2020, 395(10224): 565-574. DOI:10.1016/S0140-6736(20)30251-8 |
| [8] |
ZHANG B, TANG C, YUE H, et al. Viral metagenomics analysis demonstrates the diversity of viral flora in piglet diarrhoeic faeces in China[J]. J General Virol, 2014, 95(7): 1603-1611. DOI:10.1099/vir.0.063743-0 |
| [9] |
CHENG W X, LI J S, HUANG C P, et al. Identification and nearly full-length genome characterization of novel porcine bocaviruses[J]. PLoS One, 2010, 5(10): e13583. DOI:10.1371/journal.pone.0013583 |
| [10] |
DELWART E L. Viral metagenomics[J]. Rev Med Virol, 2007, 17(2): 115-131. DOI:10.1002/rmv.532 |
| [11] |
何彪, 涂长春. 病毒宏基因组学的研究现状及应用[J]. 畜牧兽医学报, 2012, 43(12): 1865-1870. HE B, TU C C. The advances and applications of viral metagenomics[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(12): 1865-1870. (in Chinese) |
| [12] |
CHEN X, ZHANG B, YUE H, et al. A novel astrovirus species in the gut of yaks with diarrhoea in the Qinghai-Tibetan Plateau, 2013[J]. J General Virol, 2015, 96(12): 3672-3680. DOI:10.1099/jgv.0.000303 |
| [13] |
FAHSBENDER E, ALTAN E, SEGUIN M A, et al. Chapparvovirus DNA found in 4% of dogs with diarrhea[J]. Viruses, 2019, 11(5): 398. DOI:10.3390/v11050398 |
| [14] |
ABI K M, YU Z H, JING Z Z, et al. Identification of a novel Aichivirus D in sheep[J]. Infect Genet Evolut, 2021, 91: 104810. DOI:10.1016/j.meegid.2021.104810 |
| [15] |
PAN Y F, TIAN X Y, QIN P, et al. Discovery of a novel swine enteric alphacoronavirus (SeACoV) in southern China[J]. Vet Microbiol, 2017, 211: 15-21. DOI:10.1016/j.vetmic.2017.09.020 |
| [16] |
GONG L, LI J, ZHOU Q F, et al. A New Bat-HKU2-like coronavirus in Swine, China, 2017[J]. Emerg Infect Dis, 2017, 23(9): 1607-1609. DOI:10.3201/eid2309.170915 |
| [17] |
FAN K Z, WANG R. Complete genome of a porcine calicivirus strain in Anhui province, China, is significantly shorter than that of the other Chinese strain[J]. J Virol, 2012, 86(24): 13823. DOI:10.1128/JVI.02613-12 |
| [18] |
OBA M, NAOI Y, ITO M, et al. Metagenomic identification and sequence analysis of a Teschovirus A-related virus in porcine feces in Japan, 2014-2016[J]. Infect Genet Evolut, 2018, 66: 210-216. DOI:10.1016/j.meegid.2018.10.004 |
| [19] |
ZHOU L, HU C Z, ZHOU Q, et al. Viral communities associated with porcine diarrhoeal disease and genetic characterization of a bufavirus in Tibetan pigs in China[J]. Arch Virol, 2021, 166(2): 613-617. DOI:10.1007/s00705-020-04932-9 |
| [20] |
徐囡, 吴长德, 张英俊, 等. 猪血凝性脑脊髓炎研究进展[J]. 动物医学进展, 2016, 37(11): 94-97. XU N, WU C D, ZHANG Y J, et al. Progress on porcine hemagglutinating encephalomyelitis[J]. Progress in Veterinary Medicine, 2016, 37(11): 94-97. DOI:10.3969/j.issn.1007-5038.2016.11.021 (in Chinese) |
| [21] |
沈小娟, 李晶娇, 边疆, 等. 腹泻仔猪肠道病毒组学分析[J]. 畜牧兽医学报, 2019, 50(3): 611-619. SHEN X J, LI J J, BIAN J, et al. Metagenomics analysis of feces from diarrhea piglets reveals the viral composition and epidemic characteristics of coronavirus[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(3): 611-619. (in Chinese) |
| [22] |
ZHANG X M, HERBST W, KOUSOULAS K G, et al. Biological and genetic characterization of a hemagglutinating coronavirus isolated from a diarrhoeic child[J]. J Med Virol, 1994, 44(2): 152-161. DOI:10.1002/jmv.1890440207 |
| [23] |
WOO P C Y, LAU S K P, LAM C S F, et al. Discovery of seven novel Mammalian and avian coronaviruses in the genus Deltacoronavirus supports bat coronaviruses as the gene source of Alphacoronavirus and Betacoronavirus and avian coronaviruses as the gene source of Gammacoronavirus and Deltacoronavirus[J]. J Virol, 2012, 86(7): 3995-4008. DOI:10.1128/JVI.06540-11 |
| [24] |
SHEN H G, ZHANG J F, GAUGER P C, et al. Genetic characterization of porcine sapoviruses identified from pigs during a diarrhoea outbreak in Iowa, 2019[J]. Transbound Emerg Dis, 2022, 69(3): 1246-1255. DOI:10.1111/tbed.14087 |
| [25] |
YANG F, LIU X W, ZHOU Y C, et al. Histopathology of Porcine kobuvirus in Chinese piglets[J]. Virol Sin, 2015, 30(5): 396-399. DOI:10.1007/s12250-015-3608-1 |
| [26] |
SAUVAGE V, AR GOUILH M, CHEVAL J, et al. A member of a new Picornaviridae genus is shed in pig feces[J]. J Virol, 2012, 86(18): 10036-10046. DOI:10.1128/JVI.00046-12 |
| [27] |
ZAULET M, PETROVAN V, BIRLADEANU A M, et al. Identification and prevalence of swine pasivirus 1 in eastern Romanian pig farms[J]. J Vet Diagnost Investigat, 2017, 29(3): 305-311. DOI:10.1177/1040638717696044 |
| [28] |
CHEN Q, WANG L Y, ZHENG Y, et al. Metagenomic analysis of the RNA fraction of the fecal virome indicates high diversity in pigs infected by porcine endemic diarrhea virus in the United States[J]. Virol J, 2018, 15(1): 95. DOI:10.1186/s12985-018-1001-z |
(编辑 白永平)



