2. 四川农业大学“畜禽遗传资源发掘与创新利用四川省重点实验室”,成都 611130
2. Farm Animal Genetic Resources Exploration and Innovation Key Laboratory of Sichuan Province, Sichuan Agricultural University, Chengdu 611130, China
内江猪是我国著名的优良地方猪种,2000年被列入《国家畜禽品种资源保护名录》,原产于四川盆地中部的内江、资阳一带,属于西南型猪种。内江猪体型较大,全身被黑毛,体躯宽而深,背腰宽且长,腹部较大略微下垂,臀部较宽略微后倾,头短额宽,额上有较深的横向褶皱,有“狮子头”和“二方头”之分,四肢坚实,皮较厚[1]。内江猪肉质优良,性成熟早、抗逆性强、耐粗饲,但瘦肉率低、屠宰率不高且背膘较厚[2]。内江猪拥有极佳的遗传配合力,在各类二元杂交试验中,杂交后代均显示出了明显的杂种优势,因而曾被国内各地区大量引种,一度成为国内引种最多的地方品种[3]。随着市场经济发展和外种猪迅速占领国内市场,内江猪群体迅速减少,内江猪保种成为了重点工作,近年来受非洲猪瘟的影响,内江猪保种工作面临严峻挑战[4]。
SNP(single-nucleotide polymorphism)即单核苷酸多态性,具有数量大、分布广、覆盖密度大、遗传稳定性高等特点。通过高密度SNP芯片和重测序方法可以直接获得全基因组水平的SNP,在猪[5-7]、牛[8-11]、羊[12-15]、鸡[16-18]等各类畜禽研究和全基因组育种中被广泛应用。随着高通量测序技术的发展,测序成本急剧下降,但高深度全基因组测序相比SNP芯片仍然较为昂贵。低深度全基因组重测序结合基因填充技术,能够获得覆盖全基因组的高质量SNP标记[19-20],同时通过降低测序深度有效降低测序成本,已被广泛用于各类研究[21-23]。通过全基因组模拟数据或SNP芯片全基因组填充数据评估基因组选育效果,研究发现全基因组数据可以进一步提高选择准确性,基于基因组填充或全基因组重测序能够将选择准确性提高30%~40%[24-25]。
本研究利用1×低深度重测序技术获取全基因组SNP,通过不同的SNP质控策略获取了两组数据,全面分析了133头内江猪群体结构和遗传多样性,为内江猪遗传资源保护提供依据。
1 材料与方法 1.1 试验材料本试验选择内江市国家内江猪保种备份场的内江猪为研究对象,共采集各家系具有代表性的133头种猪样品,其中公猪12头,母猪121头。耳组织样品采集后置于含无水乙醇的离心管中,迅速放入-20 ℃冰箱保存备用。
1.2 试验方法1.2.1 DNA提取及测序 使用CTAB法提取133份样品的基因组DNA。在1%琼脂糖凝胶上检测DNA降解和污染程度,采用分光光度计检测DNA纯度,采用QubitⓇ2.0荧光计(Life Technologies, CA, USA)中的QubitⓇDNA测定试剂盒测定DNA浓度。
获得高质量的基因组DNA后,使用制造商建议的Truseq Nano DNA HT试剂盒构建测序文库,并添加barcode以区分不同样品。利用Illumina NovaSeq 6000平台对上述文库进行测序,测序模式PE 150 bp,插入片段大小约为350 bp,测序深度为1×。
1.2.2 测序数据处理及比对 测序原始数据需要进行严格的质控以保证后续分析的准确性,质控包括:1)去除含有≥10%未知碱基(N)的序列;2)去除含50%以上碱基质量值小于5的序列;3)去除>10个碱基比对到接头的序列,允许≤10%的不匹配;4)去除文库构建过程中PCR扩增的重复序列。随后通过BWA(Burrows-Wheeler Aligner)将每个样本的clean read与参考基因组进行比对,采用GATK默认参数进行SNP检测。
1.2.3 SNP数据质控 将获取的SNP数据通过Plink(v1.90)软件进行质控。质控筛选包括:1)按照SNP检出率大于90%和95%获得两组SNP数据,分别编号为NJ90和NJ95;2)分别剔除两组数据中次等位基因频率小于5%的SNP;3)根据哈代-温伯格定律,分别剔除两组数据中不符合哈温平衡的偏离位点(P < 1×10-6);4)去除性染色体SNP。后续分析使用质控后的NJ90和NJ95两组SNP数据。
1.2.4 遗传多样性分析
杂合度是反映群体遗传多样性的重要指标,包括观测杂合度和期望杂合度。观测杂合度(observed heterozygosity, Ho)是指群体中杂合子所占比例,期望杂合度(expected heterozygsity, He)是指群体内随机抽取两个等位基因各不相同的概率,其计算公式为He=
1.2.5 群体结构分析 主成分分析(principal component analysis, PCA)常用于高纬数据降维,使具有相同数据特征的数据聚集在一起,简化复杂变异。本研究使用Plink(v1.90)软件利用SNP标记构建亲缘关系G矩阵计算个体间的亲缘系数,并计算前2个主成分,结果通过Rstudio进行可视化。
随后利用Plink(v1.90)软件进行连锁不平衡分析,以50个SNPs为一个窗口,每次步移5个SNPs,r2阈值为0.6,去除强连锁位点。再基于状态同源(idendity of state, IBS)计算个体间的遗传距离(D),计算公式为D=1-DST,DST=
1.2.6 群体近交系数
基因组近交系数计算常用方法包括基于SNP纯合性计算的Fhom和基于连续性纯合片段(runs of homozygosity,ROH)计算的FROH。Fhom可利用Plink(v1.90)软件计算,其公式为Fhom
测序数据经过整体质控后,按照SNP检出率高于90%和95%分为NJ90和NJ95两组数据用于后续分析。遗传多样性评估结果如表 1所示。NJ90中133个个体包含135 760个SNPs标记,其中等位基因频率为0.87,有效等位基因数为1.27,多态性标记比为0.76,观测杂合度和期望杂合度分别为0.15和0.21,整体来看内江猪该群体拥有较为丰富的遗传多样性。NJ95中133个个体包含32 266个SNPs标记,等位基因频率为0.79,有效等位基因数为1.44,多态性标记比为0.74,观测杂合度和期望杂合度分别为0.30和0.31。相较两组数据结果,NJ95的SNPs位点与NJ90完全重合且NJ90包含更多的SNPs位点,各类指标计算结果有差异。NJ90等位基因频率高于NJ95,有效等位基因数和杂合度低于NJ95,且杂合度差异较大。
|
|
表 1 内江猪遗传多样性分析 Table 1 Genetic diversity analysis of Neijiang pigs |
对内江猪133个个体进行主成分分析,结果如图 1所示。NJ90主成分分析结果(图 1A)表明, 群体内个体较为分散,主成分1能解释38.65%的变异,主成分2能解释32.31%的变异,且被分为较远的3个群。NJ95主成分分析结果(图 1B)同样说明, 群体内个体较为分散,主成分1能解释36.00%的变异,主成分2能解释32.57%的变异,群体内分层不明显。

|
两图分别为NJ90(A)和NJ95(B)的主成分分析结果 The two figures display the PCA analysis results of NJ90(A) and NJ95(B) 图 1 内江猪主成分分析 Fig. 1 PCA analysis of Neijiang pigs |
基于IBS遗传距离和亲缘关系矩阵分析群体结构,NJ90结果显示内江猪群体内遗传距离平均值为0.20,最大值和最小值分别为0.21和0.15,亲缘系数平均为0.9%(图 2A)。NJ95结果显示群体遗传距离平均值为0.25,最大值和最小值分别为0.27和0.20,亲缘系数平均为0.7%(图 2B)。NJ90和NJ95结果比较,群体平均亲缘系数差异不大,NJ90评估遗传距离较NJ95更小,NJ90和NJ95的群体分析结果表明内江猪群体大部分个体间亲缘关系较远。

|
两图分别为NJ90(A)和NJ95(B)的亲缘关系矩阵结果。图中每一个小方格代表个体之间的亲缘系数,该值越大越接近橙色,该值越小越接近蓝色 The two figures display the genetic relationship matrix results of NJ90(A) and NJ95(B). Each small square in the figure represents the relationship coefficient value between individuals, the larger the value, the color is closer to orange, the smaller the value, the color is closer to blue 图 2 内江猪亲缘关系G矩阵 Fig. 2 Genetic relationship G matrix of Neijiang pigs |
由于公猪在保种群中具有重要地位,因而采用邻接法首先对12头公猪进行聚类分析从而划分家系。NJ90结果显示,结合亲缘系数,12头公猪可以分为6个家系(图 3A)。NJ95聚类结果与NJ90差异较大(图 3B),结合亲缘系数可以分为5个家系,且每个家系中的个体与NJ90不同。

|
两图分别为NJ90(A)和NJ95(B)的公猪聚类分析结果。图中右侧数字表示内江猪个体编号,相同颜色代表同一家系 The two figures display the cluster analysis results of boars of NJ90(A) and NJ95(B).The numbers on the right of the figures represent the number of Neijiang pigs, the same color represents the same family 图 3 公猪聚类分析 Fig. 3 Cluster analysis of boars |
随后基于遗传距离构建群体进化树。NJ90结果显示根部将133个个体分为3个主要分枝,结果与PCA分析一致(图 4A)。NJ95结果有所不同,根部同样将个体分为3枝,但其中有两枝个体较少,个体聚类结果与NJ90相比差异较大(图 4B)。例如公猪129在NJ90中与母猪94、112、106、30、29、16聚为一枝,而在NJ95中却与112、106、119、10、6聚为一枝,只有112、106、119三个个体一致。聚类结果表明,NJ90的公猪聚类结果和群体聚类结果一致性更高,对群体分群和保种工作更具有参考价值。

|
两图分别为NJ90(A)和NJ95(B)的群体聚类结果。图中外圈数字代表个体号,相同颜色代表相同家系。黑点标记公猪 The two figures display the population cluster analysis results of NJ90(A) and NJ95(B). The outer numbers represent the individual number, and the same color represents the same family. The black dots represent boars 图 4 内江猪群体聚类分析 Fig. 4 Population cluster analysis of Neijiang pigs |
根据NJ90公猪聚类结果和亲缘关系,可以将内江猪群体划分为6个家系。家系1包含公猪1头,母猪13头;家系2包含公猪3头,母猪15头;家系3包含公猪1头,母猪6头;家系4包含公猪3头,母猪34头;家系5包含公猪3头,母猪34头;家系6包含公猪1头,母猪4头。剩余15头母猪与公猪亲缘关系较远, 因此可以单独分类(表 2)。
|
|
表 2 内江猪(NJ90)群体家系划分 Table 2 Family classification of Neijiang pigs(NJ90) |
运用全基因组方法利用Plink(v1.90)软件计算群体近交系数,NJ90结果显示Fhom平均近交系数为0.27,其中最大Fhom为0.67,最小Fhom为0.05。NJ95结果显示Fhom平均近交系数为0.01,其中最大Fhom为0.55,最小Fhom为-0.23。两组数据近交系数评估结果差异较大,但结果显示该群体存在一定程度的近交。
通过Plink(v1.90)软件对全基因组连续性纯合片段(ROH)进行检测,NJ90共发现11 062个ROHs,平均ROH长度为1 959.29 kb,长度范围在1 000.05~12 498.5 kb之间,主要集中在1~2.5 Mb之间,1~1.5 Mb之间占比39.44%,1.5~2.5 Mb之间占比40.39%(图 5A)。从染色体上分布来看,1号染色体上ROH数量最多(1 207个),12号染色体最少(261个),染色体2、3、4、5、6、7、8、9、13、14、15上较多且长度差异较小(图 5B)。基于该ROH结果评估群体近交系数FROH为6.65%。
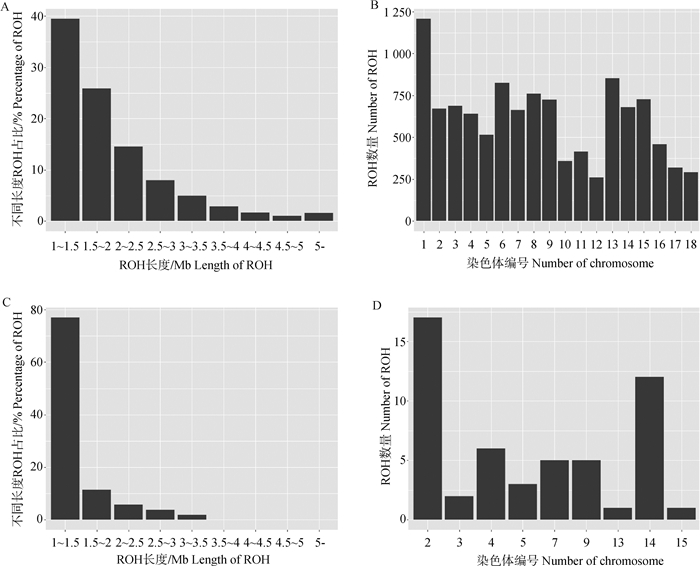
|
A.不同长度ROH数量占比(NJ90);B.不同染色体上ROH数量(NJ90);C.不同长度ROH数量占比(NJ95);D.不同染色体上ROH数量(NJ95) A. Proportion of ROH with different lengths(NJ90); B.ROH numbers on different chromosomes (NJ90); C. Proportion of ROH with different lengths(NJ95); D. ROH numbers on different chromosomes (NJ95) 图 5 内江猪ROH分布 Fig. 5 ROH distribution of Neijiang pigs |
NJ95采用同样的参数检测ROH,共发现52个ROHs,平均ROH长度为1 382.76 kb,长度范围在1 005.04~3 104.19 kb之间,主要集中在1~1.5 Mb之间占比76.92%(图 5C)。从染色体分布上来看,2号染色图上ROH最多(17个),1、6、8、10、11、12、16、17、18号染色体上均未检测到ROH,同时部分个体也未检测到ROH(图 5D)。基于该ROH结果评估群体近交系数FROH为0.02%。
3 讨论内江猪作为西南特色的地方猪种,以极佳的遗传配合力闻名,是非常宝贵的遗传资源。同其他许多地方品种一样,自20世纪80年代开始,内江猪在政府支持下开始了研究、开发和选育工作,其规模从3万余头一度发展到10万头,生长性能、繁殖性能、肉品质等多种性状研究也取得了一定成果[2]。然而在多种因素影响下,如今的内江猪保种工作仍然面临诸多挑战。一方面,内江猪经济性状相比外种猪不具竞争力,加之饲养环境复杂,使得内江猪种群规模得不到有效保障;另一方面,对于内江猪的开发和利用进展缓慢,虽然具有一定的市场前景但目前仍不能有效发挥其种质优势。本研究利用全基因组SNPs信息,对内江猪保种群体情况做了较为全面的分析,为内江猪保种工作开展提供了依据。
基于全基因组SNPs数据分析群体遗传结构和遗传多样性,是调查评估地方品种群体情况的有效手段。青峪猪[27]、淳安花猪[28]、通城猪[29]、安庆六白猪[30]、里岔黑猪[31]、枣庄黑盖猪[32]等地方品种群体遗传研究均使用商业化SNP芯片,而低深度重测序技术在牛的群体结构和遗传多样性分析中报道较多[9-10]。高深度测序可以通过位点的多层覆盖大大提高基因分型的准确性,并检测到真实的SNP变异,而低深度测序主要依靠基因型填充技术来实现。目前,研究者已提出多种基因型填充的方法,都可以对低覆盖率基因组测序数据进行填充并获得较好的准确性[33-34]。同时研究发现,使用低深度测序数据填充后进行育种值估计相较SNP芯片能够有效提高准确性,其准确度也与遗传力高低相关[11]。因此,低深度重测序相比SNP芯片能更加真实全面的反映基因组SNP变异,可以更准确的进行群体结构和遗传多样性分析。
SNP质控往往首先需要根据检出率剔除部分检出率低的SNP和个体。本研究SNP位点质控中,对SNP检出率按照90%和95%分别过滤,得到NJ90和NJ95两组数据,NJ90总位点数是NJ95的4倍,包含了NJ95全部位点,可以发现低深度重测序获取的SNP检出率在90%~95%之间的位点数量非常多,而这部分数据在NJ95的分析中并未有效利用,造成群体结构和遗传多样性分析结果相比NJ90有较大差异,尤其对群体遗传距离、亲缘关系和近交系数的估计影响较大。此外,商业化SNP芯片检测位点经过筛选后数量有限,常用猪SNP芯片获取SNP数据与NJ95位点总数相当,因此本研究通过数据分组处理可以模拟低深度重测序和SNP芯片两种技术获取的SNP数据,比较其在群体遗传结构和遗传多样性分析中的差别。本研究由于样本量较少,未对个体检出率进行过滤,经统计发现NJ90个体SNP平均缺失率为2.4%,最大缺失率为10.3%;NJ95个体SNP平均缺失率为8.4%,最大缺失率为23%。显然,NJ95个体SNP缺失率更大,这可能也是后续各项分析结果差异较大的原因之一。
本研究分别利用NJ90和NJ95两组数据分析了内江猪遗传多样性,其中等位基因频率、有效等位基因数和多态性标记比结果差别不大,但杂合度估计中NJ90的观测杂合度和期望杂合度都远低于NJ95,这可能是因为NJ90包含更多的SNPs位点,其中杂合位点的比例相对来说低于NJ95。NJ95的结果与其他通过SNP芯片分析地方猪种遗传多样性的结果更加相似,但两组结果都显示出了内江猪较为丰富的遗传多样性。在亲缘关系分析中,NJ90和NJ95两组数据结果差异不大,都说明了群体内大部分个体亲缘关系较远。NJ90和NJ95的主成分分析和遗传距离聚类分析结果分别相互印证,但两相比较结果差异较大,尤其是聚类分析。在公猪遗传距离聚类分析中,个体129、125、133、124虽然都被聚到一起,但NJ90能够更准确的评估其中的差异,126、122、132聚类结果也不相同,相较而言NJ90可能更能反映基因组的真实情况,更具有参考价值。
近交系数用于衡量个体的近交水平,表示个体随机选择两个等位基因是血缘同源的概率。在有限的群体规模中,近交水平增加会导致有害纯合子增加,从而产生近交衰退,后代生产性能降低。传统近交系数计算基于完整准确的系谱资料,但实际生产中很难保证系谱资料完整、准确,地方猪保种工作中情况可能更加严峻。基于基因组数据计算群体近交系数能有效规避系谱资料缺失错误的问题,同时也能较为准确的反映基因组的真实情况。常用方法主要包括基于SNP纯合位点的方法和基于连续性纯合片段的方法。基于SNP纯合位点的方法计算结果可能出现负值[35],其结果区间为-∞~1,如NJ95结果显示最小近交系数为-0.23,负值的出现可能是因为当前研究样本群体具有密切的相关性或较高水平的近交,则远交亲本后代的近交系数将远低于参考群体,出现负值[36]。连续性纯合片段(ROH)是亲代将来自共同祖先的同一段单倍型传递给后代形成,基于ROH估算近交系数也被广泛应用[37-38],其结果一般在0~1区间内,且结果跟ROH的长度具有一定的相关性[39-40]。本研究针对两组数据同时使用了上述两种方法,结果差异较大。NJ90和NJ95计算Fhom平均值分别为0.27和0.01,结果说明群体具有一定程度的近交。而FROH分别为0.066 5和0.000 2,该结果又显示群体近交程度较低。两组数据不同结果可能是由于质控后SNP数量差异较大,覆盖基因组程度不同,导致相同方法检测的ROH数量和长度不同,从而导结果差异。同时也可能受基因型填充的影响而不能反映基因组真实情况[38]。有研究人员提出,在有效规模较小、群体关系更紧密、选择相对不太强烈的群体中,基于SNP纯合估计近交系数出现明显的高估,基于ROH估计近交系数更加准确[41]。因而本研究中基于NJ90,通过ROH评估近交系数更具参考价值。
4 结论本研究基于低深度全基因组测序,分析了133头内江猪的遗传多样性和群体结构,结果表明内江猪群体具有较为丰富的遗传多样性。同时根据公猪聚类分析和亲缘系数可以将该群体划分为6个家系。该群体整体亲缘关系较远,近交程度较低,但仍需要严格规划选配,控制群体近交。同时低深度重测序能够更大范围覆盖基因组信息,对群体遗传多样性分析和遗传结构分析更具参考价值。
| [1] |
陈帅. 内江猪品种特性及生态养殖[J]. 四川畜牧兽医, 2014, 41(9): 45. CHEN S. Neijiang pig breed characteristics and ecological breeding[J]. Sichuan Animal & Veterinary Sciences, 2014, 41(9): 45. DOI:10.3969/j.issn.1001-8964.2014.09.023 (in Chinese) |
| [2] |
段诚中, 唐显作, 李宴群. 内江猪的选育及利用[J]. 猪业科学, 2008, 25(12): 92-94. DUAN C Z, TANG X Z, LI Y Q. Selection and utilization of Neijiang pigs[J]. Swine Industry Science, 2008, 25(12): 92-94. DOI:10.3969/j.issn.1673-5358.2008.12.025 (in Chinese) |
| [3] |
黄礼光, 王希龙, 王峰, 等. 内江猪在海南的引种繁育与杂交利用[J]. 四川畜牧兽医, 2003, 30(4): 23-24. HUANG L G, WANG X L, WANG F, et al. Study on the introducing reproduction and crossbreeding of Neijiang pig in Hainan[J]. Sichuan Animal & Veterinary Sciences, 2003, 30(4): 23-24. (in Chinese) |
| [4] |
夏运红, 胡红文, 黄跃成, 等. 浅析内江猪种质资源保护与利用现状及发展对策[J]. 中国猪业, 2021, 16(6): 90-92. XIA Y H, HU H W, HUANG Y C, et al. The current situation of the protection, utilization and development of Neijiang pigs[J]. China Swine Industry, 2021, 16(6): 90-92. (in Chinese) |
| [5] |
JOAQUIM L B, CHUD T C S, MARCHESI J A P, et al. Genomic structure of a crossbred Landrace pig population[J]. PLoS One, 2019, 14(2): e0212266. DOI:10.1371/journal.pone.0212266 |
| [6] |
CAI Z X, SARUP P, OSTERSEN T, et al. Genomic diversity revealed by whole-genome sequencing in three Danish commercial pig breeds[J]. J Anim Sci, 2020, 98(7): skaa229. DOI:10.1093/jas/skaa229 |
| [7] |
DELPUECH E, ALIAKBARI A, LABRUNE Y, et al. Identification of genomic regions affecting production traits in pigs divergently selected for feed efficiency[J]. Genet Sel Evol, 2021, 53(1): 49. DOI:10.1186/s12711-021-00642-1 |
| [8] |
LIU Z, SUN H, LAI W, et al. Genome-wide re-sequencing reveals population structure and genetic diversity of Bohai Black cattle[J]. Anim Genet, 2022, 53(1): 133-136. DOI:10.1111/age.13155 |
| [9] |
MCKAY S D, SCHNABEL R D, MURDOCH B M, et al. An assessment of population structure in eight breeds of cattle using a whole genome SNP panel[J]. BMC Genet, 2008, 9: 37. |
| [10] |
XIA X T, ZHANG S J, ZHANG H J, et al. Assessing genomic diversity and signatures of selection in Jiaxian Red cattle using whole-genome sequencing data[J]. BMC Genomics, 2021, 22(1): 43. DOI:10.1186/s12864-020-07340-0 |
| [11] |
IHESHIULOR O O M, WOOLLIAMS J A, YU X J, et al. Within-and across-breed genomic prediction using whole-genome sequence and single nucleotide polymorphism panels[J]. Genet Sel Evol, 2016, 48: 15. DOI:10.1186/s12711-016-0193-1 |
| [12] |
AL KALALDEH M, GIBSON J, DUIJVESTEIJN N, et al. Using imputed whole-genome sequence data to improve the accuracy of genomic prediction for parasite resistance in Australian sheep[J]. Genet Sel Evol, 2019, 51(1): 32. DOI:10.1186/s12711-019-0476-4 |
| [13] |
AL-MAMUN H A, CLARK S A, KWAN P, et al. Genome-wide linkage disequilibrium and genetic diversity in five populations of Australian domestic sheep[J]. Genet Sel Evol, 2015, 47: 90. DOI:10.1186/s12711-015-0169-6 |
| [14] |
ZHOU M L, WANG G F, CHEN M H, et al. Genetic diversity and population structure of sheep (Ovis aries) in Sichuan, China[J]. PLoS One, 2021, 16(9): e0257974. DOI:10.1371/journal.pone.0257974 |
| [15] |
刘家鑫, 魏霞, 邓天宇, 等. 绵羊全基因组ROH检测及候选基因鉴定[J]. 畜牧兽医学报, 2019, 50(8): 1554-1566. LIU J X, WEI X, DENG T Y, et al. Genome-wide scan for run of homozygosity and identification of corresponding candidate genes in sheep populations[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(8): 1554-1566. (in Chinese) |
| [16] |
CENDRON F, MASTRANGELO S, TOLONE M, et al. Genome-wide analysis reveals the patterns of genetic diversity and population structure of 8 Italian local chicken breeds[J]. Poult Sci, 2021, 100(2): 441-451. DOI:10.1016/j.psj.2020.10.023 |
| [17] |
LIU R R, XING S Y, WANG J, et al. A new chicken 55K SNP genotyping array[J]. BMC Genomics, 2019, 20(1): 410. DOI:10.1186/s12864-019-5736-8 |
| [18] |
RAJKUMAR U, PRINCE L L L, RAJARAVINDRA K S, et al. Analysis of (co) variance components and estimation of breeding value of growth and production traits in Dahlem Red chicken using pedigree relationship in an animal model[J]. PLoS One, 2021, 16(3): e0247779. DOI:10.1371/journal.pone.0247779 |
| [19] |
CHAT V, FERGUSON R, MORALES L, et al. Ultra low-coverage whole-genome sequencing as an alternative to genotyping arrays in genome-wide association studies[J]. Front Genet, 2022, 12: 790445. DOI:10.3389/fgene.2021.790445 |
| [20] |
LOU R N, JACOBS A, WILDER A P, et al. A beginner′s guide to low-coverage whole genome sequencing for population genomics[J]. Mol Ecol, 2021, 30(23): 5966-5993. DOI:10.1111/mec.16077 |
| [21] |
ZHOU B, HO S S, ZHANG X L, et al. Whole-genome sequencing analysis of CNV using low-coverage and paired-end strategies is efficient and outperforms array-based CNV analysis[J]. J Med Genet, 2018, 55(11): 735-743. DOI:10.1136/jmedgenet-2018-105272 |
| [22] |
RUSTAGI N, ZHOU A B, WATKINS W S, et al. Extremely low-coverage whole genome sequencing in South Asians captures population genomics information[J]. BMC Genomics, 2017, 18(1): 396. DOI:10.1186/s12864-017-3767-6 |
| [23] |
ZAN Y J, PAYEN T, LILLIE M, et al. Genotyping by low-coverage whole-genome sequencing in intercross pedigrees from outbred founders: a cost-efficient approach[J]. Genet Sel Evol, 2019, 51(1): 44. DOI:10.1186/s12711-019-0487-1 |
| [24] |
DRUET T, MACLEOD I M, HAYES B J. Toward genomic prediction from whole-genome sequence data: impact of sequencing design on genotype imputation and accuracy of predictions[J]. Heredity (Edinb), 2014, 112(1): 39-47. DOI:10.1038/hdy.2013.13 |
| [25] |
MEUWISSEN T, GODDARD M. Accurate prediction of genetic values for complex traits by whole-genome resequencing[J]. Genetics, 2010, 185(2): 623-631. DOI:10.1534/genetics.110.116590 |
| [26] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. |
| [27] |
刘彬, 沈林園, 陈映, 等. 基于SNP芯片分析青峪猪保种群体的遗传结构[J]. 畜牧兽医学报, 2020, 51(2): 260-269. LIU B, SHEN L Y, CHEN Y, et al. Analysis of genetic structure of conservation population in Qingyu pig based on SNP Chip[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 260-269. (in Chinese) |
| [28] |
戴丽荷, 褚晓红, 陈晓宇, 等. 利用SNP标记分析淳安花猪群体遗传多样性和群体结构[J]. 养猪, 2021(6): 59-64. DAI L H, CHU X H, CHEN X Y, et al. Analysis of genetic diversity and genetic structure in Chun′an spotted pigs conserved population based on SNP Chip[J]. Swine Production, 2021(6): 59-64. (in Chinese) |
| [29] |
袁娇, 徐国强, 周翔, 等. 基于SNP芯片监测通城猪的保种效果[J]. 畜牧兽医学报, 2022, 53(8): 2514-2523. YUAN J, XU G Q, ZHOU X, et al. SNP Chip-based monitoring of population conservation effect of Tongcheng pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(8): 2514-2523. (in Chinese) |
| [30] |
时坤鹏, 刘莹, 张志勇, 等. 基于SNP芯片分析安庆六白猪群体遗传结构[J]. 中国畜牧杂志, 2022, 58(8): 136-140. SHI K P, LIU Y, ZHANG Z Y, et al. Analasis of genetic structure of Anqing Liubai pig based on SNP chip[J]. Chinese Journal of Animal Science, 2022, 58(8): 136-140. (in Chinese) |
| [31] |
李晓, 崔超, 王源, 等. 基于SNP芯片对里岔黑猪进行遗传多样性与遗传结构分析的研究[J]. 中国畜牧杂志, 2022, 58(11): 117-122. LI X, CUI C, WANG Y, et al. Research on genetic diversity and genetic structure analysis of Licha black pig based on SNP chip[J]. Chinese Journal of Animal Science, 2022, 58(11): 117-122. (in Chinese) |
| [32] |
王余北, 肖莲梅, 何帅涵, 等. 基于50K SNP芯片信息的枣庄黑盖猪群体遗传结构分析[J]. 中国畜牧杂志, 2022, 58(9): 168-171, 178. WANG Y B, XIAO L M, HE S H, et al. Genetic structure analysis of Zaozhuang black-capped pigs population based on 50K SNP chip[J]. Chinese Journal of Animal Science, 2022, 58(9): 168-171, 178. (in Chinese) |
| [33] |
DAVIES R W, KUCKA M, SU D W, et al. Rapid genotype imputation from sequence with reference panels[J]. Nat Genet, 2021, 53(7): 1104-1111. |
| [34] |
BROWNING B L, BROWNING S R. A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals[J]. Am J Hum Genet, 2009, 84(2): 210-223. |
| [35] |
MCGIVNEY B A, HAN H G, CORDUFF L R, et al. Genomic inbreeding trends, influential sire lines and selection in the global Thoroughbred horse population[J]. Sci Rep, 2020, 10(1): 466. |
| [36] |
WANG J. Marker-based estimates of relatedness and inbreeding coefficients: an assessment of current methods[J]. J Evol Biol, 2014, 27(3): 518-530. |
| [37] |
GOMEZ-RAYA L, RODRÍGUEZ C, BARRAGÁN C, et al. Genomic inbreeding coefficients based on the distribution of the length of runs of homozygosity in a closed line of Iberian pigs[J]. Genet Sel Evol, 2015, 47: 81. |
| [38] |
DADOUSIS C, ABLONDI M, CIPOLAT-GOTET C, et al. Genomic inbreeding coefficients using imputed genotypes: assessing different estimators in Holstein-Friesian dairy cows[J]. J Dairy Sci, 2022, 105(7): 5926-5945. |
| [39] |
GURGUL A, SZMATOŁA T, TOPOLSKI P, et al. The use of runs of homozygosity for estimation of recent inbreeding in Holstein cattle[J]. J Appl Genet, 2016, 57(4): 527-530. |
| [40] |
刘刚, 孙飞舟, 朱芳贤, 等. 连续性纯合片段在畜禽基因组研究中的应用[J]. 遗传, 2019, 41(4): 304-317. LIU G, SUN F Z, ZHU F X, et al. Runs of homozygosity and its application on livestock genome study[J]. Hereditas (Beiijng), 2019, 41(4): 304-317. (in Chinese) |
| [41] |
CABALLERO A, VILLANUEVA B, DRUET T. On the estimation of inbreeding depression using different measures of inbreeding from molecular markers[J]. Evol Appl, 2021, 14(2): 416-428. |
(编辑 郭云雁)



