流感病毒能够引起人类、鸟类和猪等多种宿主发生呼吸道疾病,每年造成巨大的经济损失[1]。在众多流感病毒中,甲型流感病毒(influenza A virus,IAV)是在全球范围内引发大规模疫情的主要毒株。IAV属正黏病毒科,有包膜,基因组由8股单链负义RNA片段组成[2]。根据病毒表面蛋白的抗原性,IAV分为18种血凝素(HA)亚型(H1~H18)和11种神经氨酸酶(NA)亚型(N1~N11)[3]。其中,H1N1 IAV是在人群中最广泛传播的病毒亚型之一,它在1918年和2009年先后造成了两次大流行,对人类健康造成了重大威胁[4-5]。
在IAV感染过程中,需要调节宿主细胞的状态,利用细胞的底物和合成系统来进行病毒的生物合成。病毒对细胞的感染还可以通过模式识别受体(pattern recognition receptor, PRR)依赖性信号通路激活宿主的免疫应答,导致细胞产生干扰素(interferon, IFN)和其他细胞因子,激活干扰素刺激基因(IFN-stimulated gene, ISG)的表达[6-7]。先天免疫的激活能限制病毒复制并导致病毒清除;相应的,IAV也会进化出多种策略来拮抗宿主先天免疫反应,来实现病毒的自我复制[8-9]。了解宿主因子及其在IAV感染中的作用,对于全面揭示IAV的感染调控机制、控制该病毒感染引起的疾病具有重要的意义。已有的研究表明,流感病毒的感染可以改变大量的宿主细胞基因的转录水平。但是相关研究大部分都集中在蛋白质编码基因上,只有少量报道涉及了长链非编码RNA和环状RNA等非编码RNA在流感病毒感染中的作用[10-12]。环状RNA具有共价闭合的单链环状结构,没有5′和3′末端,因此对外切核糖核酸酶的降解具有较强的抵抗力,比线性RNA更稳定[13]。已有研究证明环状RNA在病毒感染中能够影响病毒复制,因此越来越多的研究开始关注环状RNA在病毒感染中的作用[14-15]。尽管病毒感染对环状RNA表达的影响尚不清楚,但考虑到环状RNA的高度稳定性以及其在抗病毒免疫应答中的调节作用,环状RNA也被认为是诊断和治疗病毒性疾病的潜在靶标[16-17]。近年来,有研究在流感病毒感染的细胞中发现了大量差异表达的环状RNA。例如,在H7N9流感病毒感染的小鼠肺部鉴定出数千个环状RNA的表达,其中186个在感染PB2突变病毒时差异表达[18];另一项研究在H3N2和H5N1流感病毒感染的MDCK细胞中分别发现了262和189个差异表达的环状RNA[19];最近的一项研究在H9N2流感病毒感染的A549细胞中发现了411种差异表达的环状RNA,其中一种新发现的环状RNA AIVR被鉴定为一种抗病毒因子,它可以通过吸收miRNA来增强CREBBP的表达,以此促进IFN-β的产生[20]。此外,研究发现环状RNA GATAD2A可以通过抑制自噬来促进H1N1复制,circ-0050463可以作为miR-33b-5p的海绵来上调真核翻译延伸因子1A1的表达,从而促进IAV复制[21-22]。这些研究结果表明,环状RNA在IAV的病毒感染中可能发挥着重要的调节作用。
本研究中,作者通过RNAseq对H1N1 AIV感染和未感染的A549细胞中环状RNA的表达谱进行了分析,对病毒感染前后差异表达的环状RNA进行了筛选和功能富集分析,为深入研究环状RNA在H1N1病毒感染过程中的调控作用奠定了基础。
1 材料与方法 1.1 细胞培养和病毒感染人非小细胞肺癌细胞(A549)购自中国科学院上海生物化学与细胞生物学研究所。甲型流感病毒Influenza A virus (strain A/Wilson-Smith/1933 H1N1)毒株由湖北医药学院刘龙博士惠赠。A549细胞在37 ℃,50 mL ·L-1 CO2培养条件下,使用含有10 mL ·L-1胎牛血清、100 U ·mL-1青霉素和100 μg ·mL-1链霉素的Ham ’s F-12K培养基培养。将A549细胞接种在100 mm细胞培养皿中,当细胞汇合度达到80%~90%时,使用MOI为0.1的A/WSN/33(H1N1)流感病毒感染细胞。在37 ℃下孵育2 h后将培养基更换为含有1 mL ·L-1牛血清蛋白(BSA)和0.2 μg ·mL-1 TPCK胰酶的F12K培养基。细胞经磷酸盐缓冲液(PBS)处理后用作对照。在感染36 h后收集细胞并提取RNA。
1.2 主要试剂Trizol RNA提取试剂盒(北京天根生化科技有限公司),核糖体RNA去除试剂盒(New England Biolabs, Inc., Massachusetts, USA),NEBNext Ⓡ Ultra Ⅱ Directional RNA建库试剂盒(New England Biolabs),Ham ’s F-12K培养基,TPCK胰酶(Sigma), 青链霉素(Sangon Biotech),牛血清蛋白(BSA, Solarbio),磷酸盐缓冲液,RNase R(500 U ·mL-1, GENESEED),HiFiScript gDNA Removal cDNA Synthesis Kit (Cwbio, China)反转录试剂盒,ChamQ Universal SYBR qPCR Master Mix荧光定量PCR Mix(Vazyme, China)。
1.3 主要仪器核酸电泳仪,NanoDrop ND-1000分光光度计(Thermo Fisher Scientific),BioAnalyzer 2100 system(Agilent Technologies),illumina Hiseq测序仪,荧光定量PCR仪。
1.4 全转录组测序1.4.1 RNA提取和样品检测 使用Trizol法提取RNA,通过琼脂糖凝胶电泳检测RNA完整性及是否存在基因组DNA污染。利用分光光度计测定总RNA的浓度。通过OD260 nm/OD280 nm比值来评估RNA的质量,并将比值在1.8~2.1的样品视为合格。
1.4.2 文库建库及测序 高通量测序服务由上海云序生物技术有限公司提供。具体步骤包括:使用核糖体RNA去除试剂盒,从总RNA中去除核糖体RNA。将处理后的总RNA经超声波处理,随机打断至200~300 bp。使用建库试剂盒将RNA反转录为cDNA,并添加测序接头和引物,最终生成测序文库。通过BioAnalyzer 2100系统评估文库的长度分布和浓度。长度主要分布在100~600 bp且浓度大于2 ng ·μL-1的文库质量视为合格,并使用illumina Hiseq测序仪进行150 bp双端测序。
1.5 转录组测序数据分析1.5.1 数据质控 使用fastqc软件对原始测序fastq文件进行质量评估。使用Cutadapt(v1.9.3)软件去除低质量和测序接头污染的reads,最终得到高质量测序数据[23]。
1.5.2 参考基因组比对和环状RNA鉴定 使用STAR(v2.5.1b)软件将得到的高质量测序数据比对到hg38参考基因组后使用DCC(v0.4.4)软件鉴定环状RNA[24-25]。DCC软件能够利用STAR软件的比对结果并结合hg38注释文件来识别剪接位点,最终特异性地检测和定量环状RNA。
1.5.3 差异表达环状RNA的筛选及功能分析 使用EdgeR(v3.16.5)包,以丨log2FC丨≥1、P value < 0.05为条件筛选出流感病毒感染后差异表达的环状RNA[26]。使用R包pheatmap对差异表达的环状RNA进行聚类分析[27]。使用R包ClusterProfiler对差异表达环状RNA的来源基因进行GO(Gene Ontology)富集分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路分析[28]。
1.6 环状RNA验证1.6.1 荧光定量PCR验证环状RNA差异表达 使用HiFiScript gDNA Removal cDNA Synthesis Kit试剂盒将H1N1亚型流感病毒感染和未感染细胞的总RNA逆转录为cDNA。以GAPDH为标准化对照,使用ChamQ Universal SYBR qPCR Master Mix进行实时定量PCR。每个样本设置3个重复,通过2-ΔΔCt方法计算每个环状RNA的相对表达水平。引物序列如表 1所示。荧光定量检测的程序为预变性: 95 ℃ 30 s; 40个循环反应: 95 ℃ 10 s, 60 ℃ 30 s;熔解曲线: 95 ℃ 15 s, 60 ℃ 60 s, 95 ℃ 15 s。
|
|
表 1 用于环状RNA PCR扩增的引物序列 Table 1 Primer sequences for PCR amplification of circRNAs |
1.6.2 RNase R消化和Sanger测序验证环状RNA表达 使用RNase R将RNA样品在37 ℃下消化30 min,然后在70 ℃下灭活10 min。未用RNase R处理的RNA用作对照。使用HiFiScript gDNA Removal cDNA Synthesis Kit试剂盒将RNA样品反转录为cDNA。使用特异性引物(表 1),对环状RNA hsa_circ_0104558、hsa_circ_0077717和hsa_circ_0000025的反向剪接位点进行2~3轮PCR扩增。对PCR产物进行琼脂糖凝胶电泳并进行Sanger测序。
2 结果 2.1 总RNA的提取和质量检验将提取的总RNA在1%的琼脂糖凝胶中,150 V电压下电泳分离20 min, 如图 1所示, RNA条带清晰完整。经分光光度计测定,样品的OD260 nm/OD280 nm比值在1.8~2.0(表 2),质量合格,可以用于测序。
|
C1~C3为未感染组RNA;E1~E3为病毒感染组RNA C1-C3 are RNA samples from uninfected cells; E1-E3 are RNA samples from IAV infected cells 图 1 琼脂糖凝胶电泳检测从A549细胞中提取的RNA样品 Fig. 1 Agarose gel electrophoresis examination of RNA samples extracted from A549 cells |
|
|
表 2 RNA质量检验 Table 2 RNA quality inspection |
转录组测序获得的原始数据情况如表 3所示。感染组(E)和未感染组(C)分别获得了246 438 632和276 572 244条原始读数。将得到的原始数据进行质控和过滤后,最终分别得到239 048 836和272 234 451条高质量读数。这些高质量读数的碱基Q30值均高于92.77%,GC含量趋近于55%,表明测序数据质量高,数据准确可靠,能够进行后续分析。
|
|
表 3 测序数据质量分析 Table 3 Sequencing data quality analysis |
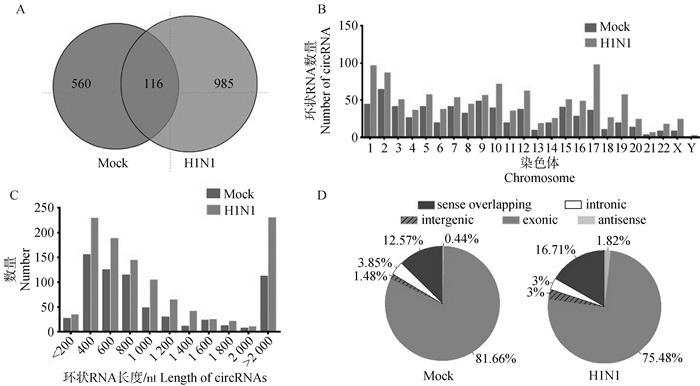
经过DCC软件鉴定,在感染组(H1N1)和未感染组(Mock)中分别检测到1 101个和676个环状RNA表达,其中只有116个环状RNA在病毒感染前后均表达。这表明流感病毒感染显著改变了宿主细胞中环状RNA的表达模式(图 2A)。统计病毒感染前后环状RNA的染色体分布,发现在所有的染色体上均有环状RNA表达,并且在感染组中每条染色体上检测到的环状RNA数量比未感染组更多(图 2B)。环状RNA的长度分析表明,大约60%的环状RNA长度在300~1 000 nt,约20%的环状RNA长度超过2 000 nt。未感染组中环状RNA的平均长度为3 567 nt,在感染组中为3 556 nt(图 2C)。根据来源,将环状RNA分为5类,分别为外显子型(exonic)、内含子型(intronic)、基因间型(intergenic)、反义链转录本(antisense)、正义链重叠型(sense overlapping)。流感病毒感染前后,大部分(75%~82%)环状RNA都来源于外显子,少部分(1%~4%)来源于内含子型、基因间型和反义链转录本(图 2D)。
|
A. 环状RNA在H1N1感染组和Mock未感染组A549细胞中的表达数;B. 环状RNA在H1N1感染组和Mock未感染组A549细胞中的染色体分布;C. 环状RNA在H1N1感染组和Mock未感染组A549细胞中的长度统计;D. 环状RNA在H1N1感染组和Mock未感染组A549细胞中的来源类型。扫描文章首页OSID码可查看彩图 A. The number of circRNAs identified in uninfected and H1N1-infected A549 cells; B. Distribution of circRNAs on chromosomes in uninfected and H1N1-infected cells; C. Length statistics of circRNAs in uninfected and H1N1-infected cells; D. Classification of circRNAs in uninfected and H1N1-infected cells.The color pictures can be found by scanning the OSID code on the front page of the article 图 2 环状RNA在A549细胞中的表达概况 Fig. 2 The expression profile of circRNAs in A549 cells |
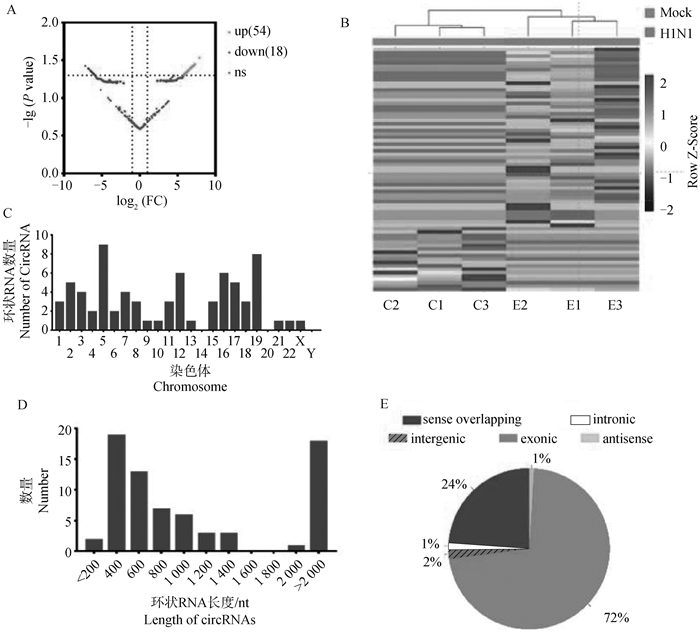
使用EdgeR包对流感病毒感染前后表达的环状RNA进行差异表达分析, 共筛选出54个表达显著上调和18个表达显著下调的环状RNA(丨log2FC丨≥1,P value < 0.05) (图 3A)。对这72个差异表达环状RNA进行层次聚类分析,表明这些环状RNA的表达水平在组内具有良好的重复性,并且在病毒感染前后表达水平有明显差异(图 3B)。差异表达环状RNA主要分布在5、12、16和19号染色体,而在14、20和Y染色体上未检测到差异表达的环状RNA(图 3C)。根据长度统计显示,约62%的差异表达环状RNA长度在300~1 000 nt,约25%的环状RNA长度超过2 000 nt(图 3D)。分析差异表达环状RNA的来源,72%的差异表达环状RNA属于外显子型,24%属于正链重叠型,剩下的4%来源于内含子型、基因间型和反义链转录本(图 3E)。
|
A. 差异表达环状RNA火山图;B. 差异表达环状RNA表达量聚类热图;C. 差异表达环状RNA染色体分布;D. 差异表达环状RNA长度统计;E. 差异表达环状RNA分类。扫描文章首页OSID码可查看彩图 A. Volcano plot of differentially expressed circRNAs; B. Cluster heatmap of the expression levels of differentially expressed circRNAs; C. Distribution of differentially expressed circRNAs on chromosomes; D. Length statistics of differentially expressed circRNAs; E. Classification of differentially expressed circRNAs.The color pictures can be found by scanning the OSID code on the front page of the article 图 3 环状RNA差异表达分析 Fig. 3 Differential expression analysis of circular RNA |
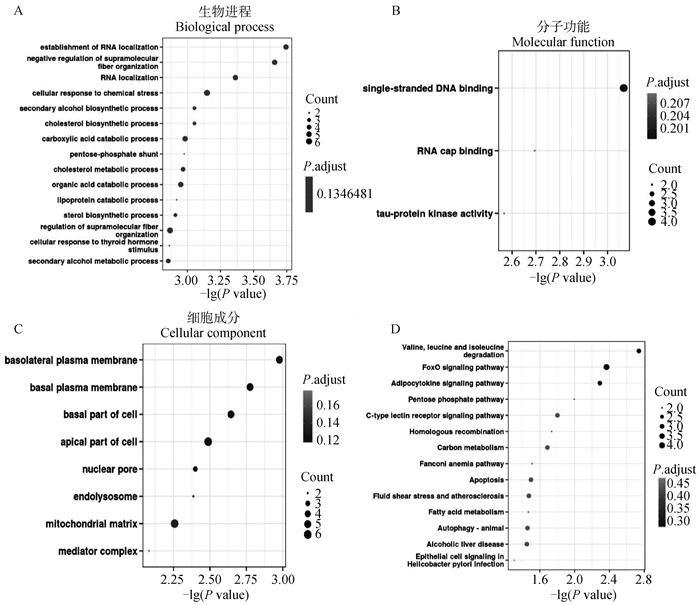
为了研究环状RNA的潜在功能,对差异表达环状RNA的来源基因进行了GO富集分析和KEGG通路分析,并以P value < 0.05为标准,筛选出显著富集的GO条目和KEGG通路。在生物进程方面,差异表达环状RNA来源基因主要富集到RNA定位、超分子纤维组织的负调控、细胞对化学刺激的响应和胆固醇合成等条目。在分子功能方面,来源基因主要富集在单链DNA结合、RNA帽子结合和Tau蛋白激酶活性等条目。在细胞组分方面,其主要富集到细胞质膜和细胞基底等条目(图 4A)。KEGG通路分析显示,环状RNA主要富集到缬氨酸,亮氨酸和异亮氨酸降解通路、FoxO信号通路、脂肪细胞因子信号通路等(图 4B)。
|
A~C. GO富集分析结果;D. KEGG通路富集结果。扫描文章首页OSID码可查看彩图 A-C. The result of GO enrichment; D. The result of KEGG pathway analysis.The color pictures can be found by scanning the OSID code on the front page of the article 图 4 差异表达环状RNA来源基因富集分析 Fig. 4 Enrichment analysis of parent gene of differentially expressed circRNAs |
为了验证环状RNA鉴定的准确性,用外切核糖核酸酶RNase R对提取的RNA进行了消化处理,然后逆转录,用于PCR扩增。结果显示,经RNase R消化后,在流感病毒感染和未感染的细胞中无法检测到GAPDH mRNA的表达,但是可以检测到三种环状RNA的表达,这证实了这三种环状RNA具有抵抗外切核糖核酸酶消化的能力(图 5A)。经Sanger测序证实了三种环状RNA的反向剪接位点(图 5B)。
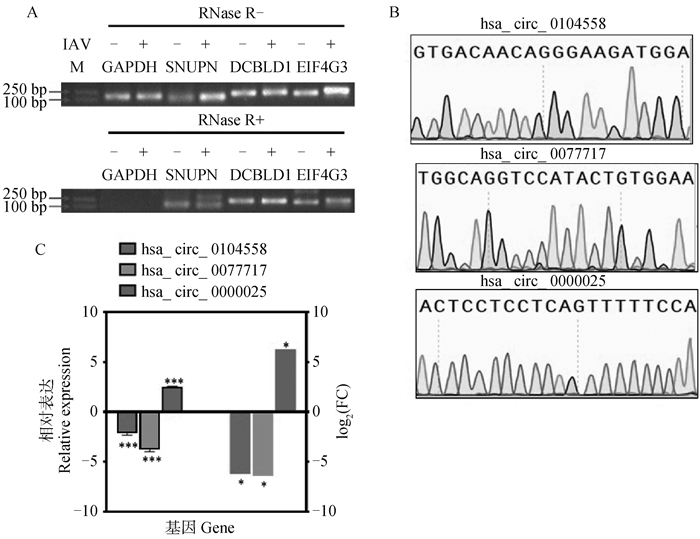
|
A. 环状RNA抗RNase R降解能力的检测。上排和下排胶图分别展示了RNA未使用RNase R处理和使用RNase R处理后的检测结果。GAPDH mRNA作为线性RNA对照;B. 三种环状RNA反向剪接位点邻近序列的Sanger测序结果;C. 以GAPDH为内参, 通过RT-qPCR验证IAV感染前后环状RNA的差异表达。左侧柱状图为RT-qPCR结果,右侧为转录组测序差异倍数。*. P < 0.05, **. P < 0.01, ***. P < 0.001。扫描文章首页OSID码可查看彩图 A. The resistance analysis of circRNAs to RNase R digestion. Upper and lower gel images show the RNA detection results before and after RNase R treatment, respectively. GAPDH mRNA was used as a linear RNA control; B. The adjacent sequence of back-spliced junction regions of the selected circRNAs examined by Sanger sequencing; C. Relative expression levels of circRNAs before and after IAV infection detected by RT-qPCR, using GAPDH as the internal reference. The left three bars represent the RT-qPCR results, and the right three bars represent RNA-seq results. *. P < 0.05, **. P < 0.01 and ***. P < 0.001. The color pictures can be found by scanning the OSID code on the front page of the article 图 5 差异环状RNA验证 Fig. 5 Validation of differentially expressed circRNAs |
通过实时荧光定量PCR来检验感染前后环状RNA的表达水平,结果表明:在流感病毒感染细胞后,环状RNA SNUPN(has_circ_0104558)和DCBLD1(hsa_circ_0077717) 的表达显著下调;EIF4G3(hsa_circ_0000025)的表达显著上调,与转录组测序的结果一致(图 5C)。综上所述,这些结果对转录组测序和环状RNA鉴定的准确性进行了验证,证实了三种环状RNA在病毒感染前后的差异表达。
3 讨论环状RNA是一类不具有5′帽子和3′尾巴结构,由RNA反向剪接而形成的闭环非编码RNA,能够参与调控多种生物进程[29]。随着高通量测序技术的发展,越来越多的环状RNA被发现,对环状RNA的研究也越来越深入。已有研究结果显示,环状RNA在疾病发展的进程中起着重要调控作用,有可能从中筛选出用于疾病诊断或预测的生物标志物[30-31]。研究发现,环状RNA能以不同的方式调控病毒感染。例如,RIG-Ⅰ能够识别外源环状RNA,从而激活免疫反应,保护宿主以抵抗病毒感染[14];dsRNA结合蛋白NF90和NF110能够调控胞内环状RNA的表达,从而影响病毒复制[15];卡波西肉瘤疱疹病毒能够诱导细胞中circ_0001400的表达,以维持病毒在细胞内的潜伏感染状态[32]。最近的研究表明,由IAV的H3N2、H5N1、H7N9和H9N2毒株引起的感染能够导致数百个环状RNA的差异表达[17, 19-20]。本研究中,作者通过转录组测序对H1N1亚型流感病毒感染前后的A549细胞中环状RNA的表达谱进行了分析,在感染组和未感染组中分别检测到了1 101和676个环状RNA表达。相比于未感染组,在感染组的各个染色体上检测到了更多的环状RNA表达,这表明H1N1亚型流感病毒感染能够全面提高细胞中环状RNA的表达丰度。这些环状RNA的表达水平是由于转录或者剪接活性的改变而上调,还是由于病毒感染导致细胞生长速度变慢,从而选择性地降解线性RNA分子,导致环状RNA的积累?还需要进一步的深入研究来回答这个问题。
差异表达分析结果显示,与未感染的细胞相比,H1N1病毒感染的A549细胞中发现了72个显著差异表达的环状RNA,其中54个表达上调和18个表达下调。这些差异表达环状RNA大多分布在5、12、16和19号染色体,而在14、20和Y染色体上未检测到。虽然H1N1亚型流感病毒感染能够引起环状RNA的差异表达,但是目前尚不清楚差异表达的环状RNA在H1N1亚型病毒感染期间的作用,需要进一步研究来揭示它们之间的关联。
研究表明,环状RNA可以通过多种方式调节病毒感染,如作为microRNA的海绵、与RNA结合蛋白相互作用、作为线性RNA的竞争者,以及调控亲本基因的表达等[17, 33]。然而,关于环状RNA在流感病毒感染中的作用还需深入研究。目前,只有环状RNA Circ-GATAD2A被报道能够通过抑制细胞自噬来促进H1N1亚型病毒复制[22]。本研究中,通过RNase R去除线性RNA,PCR扩增、Sanger测序和RT-qPCR验证了环状RNA SNUPN(has_circ_0104558)、DCBLD1(hsa_circ_0077717)和EIF4G3(hsa_circ_0000025)在IAV感染前后的差异表达。SNUPN蛋白能够作为snRNP的特异性核输入受体而发挥作用[34];DCBLD1蛋白能够通过与酪氨酸激酶受体EGFR、VEGFR2、PDGFRβ和INSR结合,从而影响下游MAPKs或Akt的激活,导致细胞增殖和迁移速率的变化[35];真核翻译起始因子4G(EIF4G3)是翻译起始复合物中重要的支架蛋白[36]。本研究中鉴定出来的环状RNA是否识别这几种亲本基因,调控这些宿主细胞蛋白的表达和功能还有待深入研究。
通过对H1N1亚型流感病毒感染后差异表达环状RNA的亲本基因进行GO和KEGG分析,发现差异表达的环状RNA主要富集到生物进程中的RNA定位、超分子纤维组织的负调控、细胞对化学刺激的响应以及胆固醇合成过程等条目;分子功能中的单链DNA结合、RNA帽子结合和Tau蛋白激酶活性等条目;细胞组分中的细胞质膜和细胞基底等条目。此外,富集到的KEGG通路包括缬氨酸、亮氨酸和异亮氨酸降解通路,FoxO信号通路,脂肪细胞因子信号通路等。这些差异表达的环状RNA是否参与IAV感染过程的调控及其调控机制,还有待进一步的研究。
4 结论本研究通过转录组测序,获得了在H1N1亚型流感病毒感染的A549细胞中的环状RNA表达谱。在感染组和未感染组中分别检测到了1 101和676个环状RNA表达。H1N1亚型流感病毒感染后,环状RNA丰度整体上调,表明环状RNA与病毒感染之间存在某种关联。通过差异分析,筛选出72个显著差异表达的环状RNA。GO和KEGG分析预测,这些环状RNA可能与RNA定位、氨基酸代谢等相关。这些数据可以为进一步探索环状RNA在流感病毒-宿主中的功能及病毒感染标志物的筛选提供参考。
| [1] |
SALOMON R, WEBSTER R G. The influenza virus enigma[J]. Cell, 2009, 136(3): 402-410. DOI:10.1016/j.cell.2009.01.029 |
| [2] |
PLESCHKA S. Overview of influenza viruses[J]. Curr Top Microbiol Immunol, 2013, 370: 1-20. |
| [3] |
DE VRIES E, DU W J, GUO H B, et al. Influenza A virus hemagglutinin-neuraminidase-receptor balance: preserving virus motility[J]. Trends Microbiol, 2020, 28(1): 57-67. DOI:10.1016/j.tim.2019.08.010 |
| [4] |
RUSSELL C J, WEBSTER R G. The genesis of a pandemic influenza virus[J]. Cell, 2005, 123(3): 368-371. DOI:10.1016/j.cell.2005.10.019 |
| [5] |
KRAMMER F, SMITH G J D, FOUCHIER R A M, et al. Influenza[J]. Nat Rev Dis Primers, 2018, 4(1): 3. DOI:10.1038/s41572-018-0002-y |
| [6] |
ACHDOUT H, ARNON T I, MARKEL G, et al. Enhanced recognition of human NK receptors after influenza virus infection[J]. J Immunol, 2003, 171(2): 915-923. DOI:10.4049/jimmunol.171.2.915 |
| [7] |
GORAYA M U, WANG S, MUNIR M, et al. Induction of innate immunity and its perturbation by influenza viruses[J]. Protein Cell, 2015, 6(10): 712-721. DOI:10.1007/s13238-015-0191-z |
| [8] |
BOHANNON C D, ENDE Z, CAO W P, et al. Influenza virus infects and depletes activated adaptive immune responders[J]. Adv Sci (Weinh), 2021, 8(16): e2100693. DOI:10.1002/advs.202100693 |
| [9] |
TOPHAM D J, DEDIEGO M L, NOGALES A, et al. Immunity to Influenza Infection in Humans[J]. Cold Spring Harb Perspect Med, 2021, 11(3): a038729. DOI:10.1101/cshperspect.a038729 |
| [10] |
MA Y M, OUYANG J, WEI J Y, et al. Involvement of host non-coding RNAs in the pathogenesis of the influenza virus[J]. Int J Mol Sci, 2016, 18(1): 39. DOI:10.3390/ijms18010039 |
| [11] |
WANG J, CEN S. Roles of lncRNAs in influenza virus infection[J]. Emerg Microbes Infect, 2020, 9(1): 1407-1414. DOI:10.1080/22221751.2020.1778429 |
| [12] |
WINTERLING C, KOCH M, KOEPPEL M, et al. Evidence for a crucial role of a host non-coding RNA in influenza A virus replication[J]. RNA Biol, 2014, 11(1): 66-75. DOI:10.4161/rna.27504 |
| [13] |
JECK W R, SHARPLESS N E. Detecting and characterizing circular RNAs[J]. Nat Biotechnol, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890 |
| [14] |
CHEN Y G, KIM M V, CHEN X Q, et al. Sensing self and foreign circular RNAs by intron identity[J]. Mol Cell, 2017, 67(2): 228-238. e5. DOI:10.1016/j.molcel.2017.05.022 |
| [15] |
LI X, LIU C X, XUE W, et al. Coordinated circRNA biogenesis and function with NF90/NF110 in viral infection[J]. Mol Cell, 2017, 67(2): 214-227. e7. DOI:10.1016/j.molcel.2017.05.023 |
| [16] |
AWAN F M, YANG B B, NAZ A, et al. The emerging role and significance of circular RNAs in viral infections and antiviral immune responses: possible implication as theranostic agents[J]. RNA Biol, 2021, 18(1): 1-15. DOI:10.1080/15476286.2020.1790198 |
| [17] |
MO Y Y, LIU Y Z, LU A N, et al. Role of circRNAs in viral infection and their significance for diagnosis and treatment (Review)[J]. Int J Mol Med, 2021, 47(5): 88. DOI:10.3892/ijmm.2021.4921 |
| [18] |
LIU Z Y, GUO Y N, ZHAO L C, et al. Analysis of the circRNAs expression profile in mouse lung with H7N9 influenza A virus infection[J]. Genomics, 2021, 113(1 Pt 2): 716-727. |
| [19] |
TAO P, NING Z Y, HAO X Q, et al. Comparative analysis of whole-transcriptome RNA expression in MDCK cells infected with the H3N2 and H5N1 canine influenza viruses[J]. Front Cell Infect Microbiol, 2019, 9: 76. DOI:10.3389/fcimb.2019.00076 |
| [20] |
QU Z Y, MENG F, SHI J Z, et al. A novel Intronic circular RNA antagonizes influenza virus by absorbing a microRNA that degrades CREBBP and accelerating IFN-β production[J]. mBio, 2021, 12(4): e0101721. DOI:10.1128/mBio.01017-21 |
| [21] |
SHI N, ZHANG S, GUO Y D, et al. CircRNA_0050463 promotes influenza A virus replication by sponging miR-33b-5p to regulate EEF1A1[J]. Vet Microbiol, 2021, 254: 108995. DOI:10.1016/j.vetmic.2021.108995 |
| [22] |
YU T Q, DING Y N, ZHANG Y N, et al. Circular RNA GATAD2A promotes H1N1 replication through inhibiting autophagy[J]. Vet Microbiol, 2019, 231: 238-245. DOI:10.1016/j.vetmic.2019.03.012 |
| [23] |
MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet J, 2011, 17(1): 3. DOI:10.14806/ej.17.1.202 |
| [24] |
DOBIN A, DAVIS C A, SCHLESINGER F, et al. STAR: ultrafast universal RNA-seq aligner[J]. Bioinformatics, 2013, 29(1): 15-21. DOI:10.1093/bioinformatics/bts635 |
| [25] |
JAKOBI T, CZAJA-HASSE L F, REINHARDT R, et al. Profiling and validation of the circular RNA repertoire in adult murine hearts[J]. Genom Proteom Bioinformat, 2016, 14(4): 216-223. DOI:10.1016/j.gpb.2016.02.003 |
| [26] |
ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 |
| [27] |
KOLDE R. Pheatmap: pretty heatmaps[CP/OL]. 2015. [2023-03-01] https://cran.r-project.org/web/packages/pheatmap/index.html.
|
| [28] |
WU T Z, HU E Q, XU S B, et al. clusterProfiler 4. 0: A universal enrichment tool for interpreting omics data[J]. Innovation (Camb), 2021, 2(3): 100141. |
| [29] |
KRISTENSEN L S, ANDERSEN M S, STAGSTED L V W, et al. The biogenesis, biology and characterization of circular RNAs[J]. Nat Rev Genet, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7 |
| [30] |
XIA X, TANG X Y, WANG S J. Roles of CircRNAs in autoimmune diseases[J]. Front Immunol, 2019, 10: 639. DOI:10.3389/fimmu.2019.00639 |
| [31] |
YU X H, LIU H Y, CHANG N, et al. Circular RNAs: New players involved in the regulation of cognition and cognitive diseases[J]. Front Neurosci, 2023, 17: 1097878. DOI:10.3389/fnins.2023.1097878 |
| [32] |
TAGAWA T, OH D, DREMEL S, et al. A virus-induced circular RNA maintains latent infection of Kaposi 's sarcoma herpesvirus[J]. Proc Natl Acad Sci USA, 2023, 120(6): e2212864120. DOI:10.1073/pnas.2212864120 |
| [33] |
ZHOU W Y, CAI Z R, LIU J, et al. Circular RNA: metabolism, functions and interactions with proteins[J]. Mol Cancer, 2020, 19(1): 172. DOI:10.1186/s12943-020-01286-3 |
| [34] |
STRASSER A, DICKMANNS A, LVHRMANN R, et al. Structural basis for m3G-cap-mediated nuclear import of spliceosomal UsnRNPs by snurportin1[J]. EMBO J, 2005, 24(13): 2235-2243. DOI:10.1038/sj.emboj.7600701 |
| [35] |
SCHMOKER A M, WEINERT J L, MARKWOOD J M, et al. FYN and ABL regulate the interaction networks of the DCBLD receptor family[J]. Mol Cell Proteomics, 2020, 19(10): 1586-1601. DOI:10.1074/mcp.RA120.002163 |
| [36] |
HU J J, SUN F Y, HANDEL M A. Nuclear localization of EIF4G3 suggests a role for the XY body in translational regulation during spermatogenesis in mice[J]. Biol Reprod, 2018, 98(1): 102-114. DOI:10.1093/biolre/iox150 |
(编辑 孟培)



