2. 广东省兽医临床重大疾病综合防控重点实验室, 广州 510642;
3. 广东省宠物工程技术研究中心, 广州 510642
2. Guangdong Provincial Key Laboratory of Prevention and Control for Severe Clinical Animal Diseases, Guangzhou 510642, China;
3. Guangdong Pet Engineering Technology Research Center, Guangzhou 510642, China
肺炎克雷伯菌(Klebsiella pneumonia)是肠杆菌科克雷伯菌属的一种常见的条件性致病菌[1-2],存在于人和动物的肠道和呼吸道,可引起肺炎、支气管炎、泌尿道系统和创伤的感染,甚至脑膜炎、腹膜炎和败血症等[3]。近年来,肺炎克雷伯菌引起的动物群体性细菌性感染的疾病不断增多[4-6]。此外,由于抗菌药物的广泛使用,导致该菌对多种抗菌药物产生了耐药性,从而使该菌所致疾病的预防和治疗难度增大,因而其发病率和死亡率均明显升高[7],给养殖业造成的损失亦日趋严重。
羊感染肺炎克雷伯菌主要引起肺炎以及上呼吸道感染等,有些病羊表现为流鼻涕、咳嗽和呼吸急促等症状[8]。由于肺炎克雷伯菌病死率较高,会造成一定的经济损失,因此对它的关注日渐增加,羊感染肺炎克雷伯菌的报道也越来越多。本试验从广东省各个羊场采集了150份有呼吸道疾病羊的样品,利用PCR方法分离鉴定出肺炎克雷伯菌,并对其进行了毒力、耐药基因和ERIC-PCR基因分析,为羊源肺炎克雷伯菌的更深入研究奠定了理论基础,同时为广东羊场的临床用药提供了参考。
1 材料与方法 1.1 病料采集采集广东省3个不同市羊场的150份患呼吸道疾病病羊鼻拭子样品,进行细菌分离培养。
1.2 试剂与培养基营养琼脂、营养肉汤培养基均购自青岛海博试剂有限公司,DL2000 Marker、Ex Taq DNA聚合酶均购自TaKaRa公司;革兰氏染色试剂、药敏纸片均购自杭州滨和微生物试剂有限公司,细菌基因组DNA提取试剂盒购自天根生化科技有限公司;DL2000 DNA Marker、100 bp DNA Ladder(Dye Plus)、Premix Taq (Ex Taq version 2.0 plus dye) 等购自宝生物(大连)工程有限公司。
1.3 细菌分离培养将采集的150份鼻拭子样品分别接种于LB琼脂平板上,37 ℃需氧培养24 h后进行纯化培养,挑取单菌落于LB液体培养基中进行扩增培养。
1.4 染色镜检挑取纯化后的单菌落,按常规方法进行革兰染色,观察菌体形态。
1.5 PCR鉴定肺炎克雷伯菌根据细菌基因组DNA提取试剂盒说明书提取细菌DNA。肺炎克雷伯菌特异性(khe溶血酵素基因[9])引物序列如下:5′-ATGAAACGACCTGATTGCATTCGC-3′;5′-TTACTTTTTCCGCGGCTTACCGTC-3′,预期扩增片段长度496 bp。扩增体系(25 μL):2×Taq Mix Buffer 12.5 μL,上、下游引物各1 μL,模板DNA 2 μL,ddH2O 8.5 μL。扩增程序:95 ℃预变性3 min;94 ℃变性1 min,55 ℃退火45 s,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min,PCR产物用1.5%琼脂糖凝胶电泳进行检测,电泳时间为45 min,电压90V,电泳结束采用凝胶成像系统分析。
1.6 药敏试验依据美国临床和实验室标准协会(CLSI)手册对包括头孢菌素类、大环内酯类等在内的21种常用抗菌药物进行药敏试验[10],测定分离菌对抗菌药物的抑菌圈直径,判定分离菌对抗菌药物的敏感性。
1.7 肺炎克雷伯菌毒力基因检测参照文献[11, 12],并根据GenBank数据库公布的毒力基因相关序列设计相关引物(表 1)。分别扩增分离菌的相关毒力基因。反应体系为25 μL:上、下游引物各1 μL,基因组DNA 2 μL,2×PCR Mix 12.5 μL,ddH2O 8.5 μL。反应条件:95 ℃ 5 min;95 ℃ 1 min,Tm(50~58 ℃)45 s,72 ℃ 90 s,30个循环后;72 ℃延伸10 min。
|
|
表 1 肺炎克雷伯菌毒力基因引物序列 Table 1 Virulence gene sequence of Klebsiella pneumoniae |
参照文献[13, 14],并根据GenBank数据库公布的耐药基因相关序列设计相关引物(表 2), 以提取的细菌基因组DNA为模板,进行耐药基因PCR扩增。反应体系为20 μL:2×Taq PCR StarMix 10 μL、上下游引物各1 μL、模板2 μL,剩余用ddH2O补齐。反应条件:95 ℃ 5 min;95 ℃ 30 s,相应Tm 30 s,72 ℃ 1 min,35个循环,72 ℃终延伸10 min。
|
|
表 2 肺炎克雷伯菌耐药基因序列 Table 2 Klebsiella pneumoniae resistance gene sequence |
1.9.1 引物设计 参照文献[15],并根据GenBank数据库公布的ERIC-PCR相关序列设计相关引物,引物序列如下:5′-ATGTAAGCTCCTGGGGATTCAC-3′;5′-AAGTAAGTGACTGGGGTGAGCG-3′,引物由天一辉远生物科技有限公司合成。
1.9.2 ERIC-PCR扩增 以提取细菌基因组DNA为模板,进行ERIC-PCR扩增,采用20 μL反应体系:2×Taq PCR StarMix 10 μL,上、下游引物(10 μmol·L-1)各1 μL,模板DNA 2 μL,ddH2O 6 μL。
PCR反应程序为94 ℃预变性5 min; 94 ℃变性45 s,52 ℃退火1 min,72 ℃延伸5 min,35个循环;72 ℃延伸10 min; 4 ℃保存,PCR产物通过1.5%琼脂糖凝胶电泳进行检测。
1.9.3 ERIC-PCR聚类图谱分析 ERIC-PCR电泳图谱首先采用Quantity One(Bio-Rad,USA)软件进行扩增条带分析,识别结果记录方法:针对某一条带,软件自动识别后“有”记作“1”,“无”记作“0”,并辅助相应的人工校对;再采用NTSYSpc 2.1软件计算得到遗传相似性系数矩阵和遗传距离矩阵,随后利用遗传距离矩阵采用非加权配对算术平均法对分离株进行遗传聚类分析;最后根据聚类结果进行系统进化树的构建,比较不同来源菌株间的亲缘关系。
2 结果 2.1 肺炎克雷伯菌分离鉴定本试验通过革兰染色及PCR鉴定的方法,在150份样品中共分离出42株肺炎克雷伯菌,总分离率为28%(42/150),该菌在LB固体培养基上呈现白色不透明型菌落,其边缘光滑,中央略微突起(图 1)。
|
图 1 肺炎克雷伯菌在LB固体平板上的菌落形态 Fig. 1 Colony morphology of Klebsiella pneumoniae on LB plate |
革兰染色镜检结果如图 2A显示,该菌呈现红色,形态表现为较短粗的杆菌,单独、成双或短链状排列,无芽孢和鞭毛。PCR结果显示(图 2B),扩增条带大小为496 bp,分离得到细菌均携带溶血酵素基因khe,鉴定为肺炎克雷伯菌。
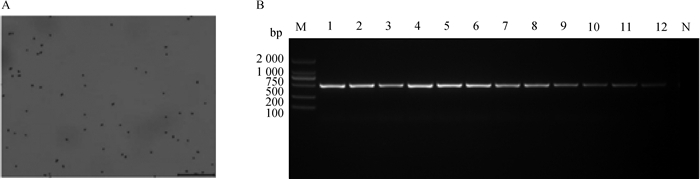
|
A.革兰染色结果(1 000×);B. 部分菌株PCR鉴定(M. DL2000 DNA相对分子质量标准;1~12. 肺炎克雷伯菌分离菌kp1、kp7、kp13、kp15、kp194-7、kp17-13、kp131、kp268、kp2、kp11、kp6、kp9;N. 阴性对照) A. Gram-negative staining results (1 000×); B. PCR identification results of some isolates (M. DL2000 DNA marker; 1-12. Klebsiella pneumonia: kp1, kp7, kp13, kp15, kp194-7, kp17-13, kp131, kp268, kp2, kp11, kp6, kp9; N. Negative control) 图 2 肺炎克雷伯菌分离株的鉴定 Fig. 2 Identification results of Klebsiella pneumonia isolates |
通过对42株肺炎克雷伯菌进行21种常用抗生素的药敏试验,结果显示,肺炎克雷伯菌分离株对其中16种抗生素有不同程度的耐药性,对阿莫西林、氧氟沙星、青霉素等耐药率达100%,对恩诺沙星、阿奇霉素耐药率达30%以上,而对头孢吡肟、复方新诺明、氨苄西林/舒巴坦的敏感性达100%,具体详见图 3。
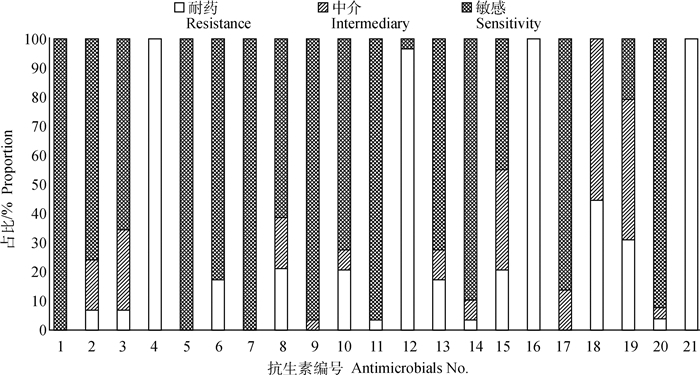
|
1.头孢吡肟;2.头孢曲松;3.头孢呋辛;4.阿莫西林;5.氨苄西林/舒巴坦;6.氯霉素;7.复方新诺明;8.庆大霉素;9.多黏菌素B;10.四环素;11.美罗培南;12.苯唑西林;13.阿米卡星;14.诺氟沙星;15.强力霉素;16.青霉素G;17.环丙沙星;18.恩诺沙星;19.阿奇霉素;20.麦迪霉素;21.氧氟沙星 1. Cefepime; 2. Ceftriaxone; 3. Cefuroxime; 4. Amoxicillin; 5. Ampicillin/sulbactam; 6. Chloramphenicol; 7. Complex sulfamethoxazole; 8. Gentamycin; 9. Polymyxin B; 10. Tetracyclin; 11. Meropenem; 12. Oxacillin; 13. Amikacin; 14. Norfloxacin; 15. Doxycycline; 16. Penicillin G; 17. Ciprofloxacin; 18. Enrofloxacin; 19. Azithromycin; 20. Medemcyin; 21. Ofloxacin 图 3 肺炎克雷伯菌药敏结果 Fig. 3 Drug sensitivity of Klebsiella pneumoniae |
采用PCR方法对肺炎克雷伯菌12种毒力基因rmpA、aerobactin、alls、kfuBC、wcaG、iucB、iroNB、ureA、wabG、uge、fim、ybtA进行检测,共检测到10种毒力基因,其中,黏液表型基因rmpA、Fe3+摄取基因kfuBC与细菌尿素酶基因ureA检出率均>80%,分别为88.0%和85.7%、83.3%,而iroNB和ybtA没有被检出,这表明ureA、rmpA、kfuBC毒力基因在广东地区感染肺炎克雷伯菌中占主导地位。
2.4 耐药基因检测结果采用PCR的检测方法对42株肺炎克雷伯菌分离株进行耐药基因检测,结果显示,42株肺炎克雷伯菌分离株均可检测出多种耐药基因,其中,氨基糖苷类耐药基因aphA、aacC4、aacC2的检出率分别为42.9%(18/42)、28.6%(12/42)、23.8%(10/42),喹诺酮类耐药基因qnrA的检出率为40.5%(17/42),β-内酰胺类耐药基因SHV的检出率为35.7%(15/42),大环内酯类耐药基因ermC的检出率为16.7%(7/42),而磺胺类以及碳青霉烯类的耐药基因未被检出,分离所得的肺炎克雷伯菌均含有2种及2种以上的耐药基因。
2.5 ERIC-PCR分型结果采用ERIC-PCR对细菌进行分型,42株肺炎克雷伯菌ERIC-PCR分型结果见图 4。结果显示,运用ERIC-PCR法能扩增出较为清晰的ERIC图谱,每个分离株的ERIC条带大小均为100~4 000 bp,扩增条带数为5~8。

|
M. DL5000 DNA相对分子质量标准;1~20. 肺炎克雷伯菌分离菌;N. 阴性对照 M. DL5000 DNA marker; 1-20. Klebsiella pneumonia isolates; N. Negative control 图 4 部分肺炎克雷伯菌ERIC-PCR电泳图 Fig. 4 ERIC-PCR electrophoresis of part of Klebsiella pneumonia |
运用NTSYSpc2.1软件对上述ERIC电泳图谱进行聚类分析,使用非加权配对平均法(UPGMA)计算得到聚类树状图(图 5),从图中可以看到42株肺炎克雷伯菌具有较高的相似性,根据聚类图谱亲缘远近关系,当相似值处于80.0%以上, 可认定为同一类群,结果显示,42株肺炎克雷伯菌分离株可分为7个不同的类群(表 3),其中,遗传多样性优势群为群体Ⅰ,菌株所占比例为66.6%.

|
图 5 42株肺炎克雷伯菌的ERIC-PCR聚类图 Fig. 5 ERIC-PCR clustering of 42 strains of Klebsiella pneumoniae |
|
|
表 3 42株肺炎克雷伯菌菌的ERIC-PCR聚类分析表 Table 3 ERIC-PCR cluster analysis of 42 strains of Klebsiella pneumoniae |
肺炎克雷伯菌是常见的条件致病菌,易感染人和动物,而且广泛分布于动物的呼吸道及肠道中,发病死亡率高,给防治增加了一定的难度,感染率仅次于大肠杆菌,必须要严加控制[16]。在2018年,新疆曾报道1例绵羊感染肺炎克雷伯菌,同时做了部分生物学特性研究[17]。2019年,朱利霞等[18]以肺炎克雷伯菌流行病学,耐药现状等方面进行综述。随后,2020年对内蒙古通辽市一羊场的病死羊进行采样,分离鉴定得出致死菌株是肺炎克雷伯菌,并且发现菌株出现耐药情况[19]。而在广东省有关羊源肺炎克雷伯菌的报道不是很多,本研究从广东不同规模化的羊场采集150份呼吸道样品,共分离到42株肺炎克雷伯菌分离株,具有较高分离率。王哲红[20]从新疆地区495份牛鼻拭子样品中分离得到60株肺炎克雷伯菌,分离率为11.95%。贡嘎等[21]从西藏地区109份牦牛呼吸道样品中分离得到8株肺炎克雷伯菌分离株,分离率为7.76%。表明在不同省市由于南北气候环境以及饲养管理模式的不同,导致肺炎克雷伯菌分离率具有较大差异。
由于抗生素的大范围使用以及抗感染治疗手法的单一,导致肺炎克雷伯菌对多种抗生素产生了耐药性,给养殖业造成的损失亦日趋严重,尤其是超广谱β-内酰胺酶(Extended Spectrum β-lactamases,ESBLs)和耐碳青霉烯肺炎克雷伯菌(Carbapenem resistant Klebsiella pneumoniae,CRKP)的出现增加了对肺炎克雷伯菌感染的难度,此外本次分离出的42株肺炎克雷伯菌对多种抗生素耐药,其中,阿莫西林、苯唑西林及氧氟沙星耐药率高达100%,相对的头孢吡肟和氨苄西林舒巴坦类药物的敏感性达100%,其余药物呈现不同程度的耐药性,可以采用β-内酰胺酶抑制剂克拉维酸、舒巴坦、三唑巴坦进行预防控制。耐药基因检测结果显示,分离株对氨基糖苷类、喹诺酮类以及β-内酰胺类耐药基因SHV的检出率都处于较高水平,其余耐药基因检出率较低甚至零检出,表明对氨基糖苷类、氟喹诺酮类以及β-内酰胺类抗生素呈现不同程度的耐药,与庆大霉素、氧氟沙星以及阿莫西林的药敏结果一致。王林峰和王选锭[22]研究发现,产SHV-1型酶的肺炎克雷伯氏菌菌株对阿莫西林耐药,对替卡西林和哌拉西林的敏感性降低,但对头孢菌素类抗生素敏感,本次研究结果显示,SHV基因检出率为35.7%,且分离株对阿莫西林耐药,对头孢菌素类抗生素敏感,与上述试验结果相符。
本研究对分离到的42株肺炎克雷伯菌,检测12种常见的毒力基因,毒力基因检测结果显示,rmpA基因的检出率为88.0%,表明所分离到的肺炎克雷伯菌菌体可表达荚膜多糖,荚膜多糖是肺炎克雷伯菌最重要最基本的毒力因子之一[23]。rmpA基因存在于毒力菌株的大质粒上,负责调控细菌荚膜的合成,增强细菌毒力[24]。Sun等[25]研究表明,在导致肝脓肿的hvKP中,92.1%(35/38)为rmpA阳性。kfuBC基因的检出率为85.7%,kfuBC基因可介导Fe3+的摄取,多存在于高毒力肺炎克雷伯菌菌株中,有研究发现几乎所有K1型高毒力肺炎克雷伯菌均携带kfuBC基因[26]。基因urea的检出率为83.3%,基因urea与allS均可调控细菌尿素酶,allS基因可使KP在有氧和无氧条件下都能利用尿素囊而获得碳源、氮源等能量来源[27]。不同毒力的组合使分离株产生不同的特性,增加了其致病性,虽然毒力基因常见于高毒力肺炎克雷伯菌菌株中,不代表该细菌会产生高致病力,而探究高致病力原因是未来研究高毒力肺炎克雷伯菌感染的方向。
综上所述,肺炎克雷伯菌在广东省羊场中存在流行,且耐药率比较严重,需要在实际养殖过程中合理使用抗生素,建议通过药敏试验确定给药种类,可以联合应用抗生素,以减少耐药现象的发生,同时能够达到最好的治疗效果。
4 结论广东省羊养殖场中肺炎克雷伯菌分离率较高,同时分离所得菌株大部分为多重耐药菌株,对喹诺酮类及β-内酰胺类抗生素具有较高的耐药性,且含有多种毒力基因,具有较高的致病性,在治疗肺炎克雷伯菌引起的呼吸道疾病时,可选用多种抗生素进行周期性交替使用,避免细菌耐药性的产生,同时做好日常防护清洁工作,切断传播途径,减少感染量。
| [1] |
MARONCLE N, BALESTRINO D, RICH C, et al. Identification of Klebsiella pneumoniae genes involved in intestinal colonization and adhesion using signature-tagged mutagenesis[J]. Infect Immun, 2002, 70(8): 4729-4734. DOI:10.1128/IAI.70.8.4729-4734.2002 |
| [2] |
PODSCHUN R, ULLMANN U. Klebsiella spp. as nosocomial pathogens: epidemiology, taxonomy, typing methods, and pathogenicity factors[J]. Clin Microbiol Rev, 1998, 11(4): 589-603. DOI:10.1128/CMR.11.4.589 |
| [3] |
姜绍全. 毛皮动物肺炎克雷伯氏菌的研究现状[J]. 山东畜牧兽医, 2016, 37(1): 42-45. JIANG S Q. Research status of Klebsiella pneumoniae in fur-bearing animals[J]. Shandong Journal of Animal Science and Veterinary Medicine, 2016, 37(1): 42-45. (in Chinese) |
| [4] |
徐海圣, 舒妙安. 中华鳖肺炎克雷伯氏菌病的病原研究[J]. 浙江大学学报: 理学版, 2002, 29(6): 702-706. XU H S, SHU M A. Studies on the pathogens of the Klebsiella pneumoniae disease of Trionyx sinensis[J]. Journal of Zhejiang University: Science Edition, 2002, 29(6): 702-706. (in Chinese) |
| [5] |
SIU L K, FUNG C P, CHANG F Y, et al. Molecular typing and virulence analysis of serotype K1 Klebsiella pneumoniae strains isolated from liver abscess patients and stool samples from noninfectious subjects in Hong Kong, Singapore, and Taiwan[J]. J Clin Microbiol, 2011, 49(11): 3761-3765. DOI:10.1128/JCM.00977-11 |
| [6] |
钟世勋, 王迪, 曲亭合, 等. 不同动物种源肺炎克雷伯氏菌分离株23S rRNA序列分析[J]. 中国预防兽医学报, 2013, 35(11): 937-939. ZHONG S X, WANG D, QU T H, et al. Sequence analysis of 23S rRNA genes of different animal species Klebsiella pneumoniae[J]. Chinese Journal of Preventive Veterinary Medicine, 2013, 35(11): 937-939. DOI:10.3969/j.issn.1008-0589.2013.11.18 (in Chinese) |
| [7] |
PADILLA E, LLOBET E, DOMEÉNECH-SÁNCHEZ A, et al. Klebsiella pneumoniae AcrAB efflux pump contributes to antimicrobial resistance and virulence[J]. Antimicrob Agents Chemother, 2010, 54(1): 177-183. DOI:10.1128/AAC.00715-09 |
| [8] |
黄旭俊, 王鹏勇, 陈素娟, 等. 一株湖羊肺炎克雷伯菌的分离与鉴定[J]. 上海畜牧兽医通讯, 2018(3): 12-15. HUANG X J, WANG P Y, CHEN S J, et al. Isolation and identification of Klebsiella pneumoniae strain from Hu sheep[J]. Shanghai Journal of Animal Husbandry and Veterinary Medicine, 2018(3): 12-15. (in Chinese) |
| [9] |
冯娜. 一株牛源肺炎克雷伯菌的分离鉴定及其基因组初步分析[D]. 兰州: 甘肃农业大学, 2017. FENG N. Isolation and identification of a bovine Klebsiella pneumoniae and preliminary analysis of its genome[D]. Lanzhou: Gansu Agricultural University, 2017. (in Chinese) |
| [10] |
马越, 李景云, 金少鸿. 美国临床实验室标准委员会推荐药敏试验操作方法和判断标准(2005年修订版)[J]. 中华医学杂志, 2005, 85(17): 1182-1184. MA Y, LI J Y, JIN S H. American clinical laboratory standards committee recommends the methods and criteria for drug susceptibility testing (2005 revision)[J]. National Medical Journal of China, 2005, 85(17): 1182-1184. DOI:10.3760/j:issn:0376-2491.2005.17.009 (in Chinese) |
| [11] |
戴玥. 赣南地区耐碳青霉烯类肺炎克雷伯菌血清分型、毒力及分子机制研究[D]. 南昌: 南昌大学, 2020. DAI Y. Study on the haematological classification, virulence and molecular mechanism of Carbapene-resistant Klebsiella pneumoniae in Gannan area[D]. Nanchang: Nanchang University, 2020. (in Chinese) |
| [12] |
刘康军, 曹菲菲, 孙莹慧, 等. 奶牛乳房炎金黄色葡萄球菌的分离鉴定及毒力基因检测[J]. 中国兽医学报, 2019, 39(2): 323-327. LIU K J, CAO F F, SUN Y H, et al. Identification and investigation on virulence determinants of Staphylococcus aureus isolated from bovine mastitis[J]. Chinese Journal of Veterinary Science, 2019, 39(2): 323-327. (in Chinese) |
| [13] |
陈莲子. 葡萄球菌对常用大环内酯类、克林霉素的耐药性及耐药基因和分子流行病学研究[D]. 合肥: 安徽医科大学, 2008. CHEN L Z. Study of macrolide, lincosamide resistance in staphylococcal isolates[D]. Hefei: Anhui Medical University, 2008. (in Chinese) |
| [14] |
杨帆, 邓保国, 魏纪东, 等. 医源与动物源肺炎克雷伯菌耐药性和分子特性研究[J]. 中国人兽共患病学报, 2017, 33(10): 888-892, 902. YANG F, DENG B G, WEI J D, et al. Drug-resistant and molecular characteristic of Klebsiella pneumoniae isolated from nosocomial and animal origins[J]. Chinese Journal of Zoonoses, 2017, 33(10): 888-892, 902. DOI:10.3969/j.issn.1002-2694.2017.10.007 (in Chinese) |
| [15] |
张海强, 安伟, 肖雨. 虾源副溶血弧菌致病基因检测与ERIC-PCR分型[J]. 上海海洋大学学报, 2019, 28(6): 848-856. ZHANG H Q, AN W, XIAO Y. Pathogenic gene detection of Vibrio parahaemolyticus isolated from shrimp and genotyping by ERIC-PCR[J]. Journal of Shanghai Ocean University, 2019, 28(6): 848-856. (in Chinese) |
| [16] |
刘海月, 齐丽娜, 孙弋雅, 等. 鹅源肺炎克雷伯菌分离鉴定及其致病性分析[J]. 现代畜牧兽医, 2020(4): 20-25. LIU H Y, QI L N, SUN Y Y, et al. Isolation, identification and pathogenicity analysis of Klebsiella pneumoniae from goose[J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2020(4): 20-25. (in Chinese) |
| [17] |
赵洁雅, 陈世军, 杨会国, 等. 新疆绵羊肺炎克雷伯菌分离鉴定及部分生物学特性研究[J]. 黑龙江农业科学, 2018(4): 57-60. ZHAO J Y, CHEN S J, YANG H G, et al. Isolation and identification of sheep Klebsiella pneumoniae in Xinjiang and the research of its partial biological characteristics[J]. Heilongjiang Agricultural Sciences, 2018(4): 57-60. (in Chinese) |
| [18] |
朱利霞, 王洪彬, 赵希艳, 等. 肺炎克雷伯菌研究进展[J]. 黑龙江畜牧兽医, 2019(13): 42-45. ZHU L X, WANG H B, ZHAO X Y, et al. Research progress of Klebsiella pneumoniae[J]. Heilongjiang Animal Science and Veterinary Medicine, 2019(13): 42-45. (in Chinese) |
| [19] |
范培超, 史量全, 温树波, 等. 羊源肺炎克雷伯菌的分离鉴定及毒力基因检测[J]. 中国兽医学报, 2021, 41(1): 96-101. FAN P C, SHI L Q, WEN S B, et al. Isolation, identification and virulence gene detection of Klebsiella pneumoniae from sheep[J]. Chinese Journal of Veterinary Science, 2021, 41(1): 96-101. (in Chinese) |
| [20] |
王哲红. 新疆集约化牛场肺炎克雷伯菌分子流行病学调查及耐药特性研究[D]. 阿拉尔: 塔里木大学, 2021. WANG Z H. Molecular epidemiological investigation and drug resistance characteristics on Klebsiella pneumoniae of intensive cattle farms in Xinjiang[D]. Alar: Tarim University, 2021. (in Chinese) |
| [21] |
贡嘎, 格桑卓玛, 左伟, 等. 西藏部分地区牦牛源肺炎克雷伯菌的分离、鉴定、毒力及耐药基因分析[J]. 中国兽医学报, 2021, 41(1): 102-109. GONG G, GE S Z M, ZUO W, et al. Isolation, identification, virulence and drug resistance genes detection of Klebsiella pneumoniae in Tibet yak[J]. Chinese Journal of Veterinary Science, 2021, 41(1): 102-109. (in Chinese) |
| [22] |
王林峰, 王选锭. 肺炎克雷伯氏菌耐药机制研究进展[J]. 中国抗生素杂志, 2004, 29(6): 324-328. WANG L F, WANG X D. Advances in the mechanism of drug resistance of Klebsiella pneumoniae[J]. Chinese Journal of Antibiotics, 2004, 29(6): 324-328. (in Chinese) |
| [23] |
DOMENICO P, SALO R J, CROSS A S, et al. Polysaccharide capsule-mediated resistance to opsonophagocytosis in Klebsiella pneumoniae[J]. Infect Immun, 1994, 62(10): 4495-4499. |
| [24] |
STRUVE C, ROE C C, STEGGER M, et al. Mapping the evolution of hypervirulent Klebsiella pneumoniae[J]. mBio, 2015, 6(4): e00630. |
| [25] |
SUN Y, WU H, SHEN D. Clinical and molecular analysis of Klebsiella pneumoniae causing liver abscess in China[J]. J Mol Microbiol Biotechnol, 2016, 26(4): 245-251. |
| [26] |
FANG C T, LAI S Y, YI W C, et al. Klebsiella pneumoniae genotype K1: an emerging pathogen that causes septic ocular or central nervous system complications from pyogenic liver abscess[J]. Clin Infect Dis, 2007, 45(3): 284-293. |
| [27] |
CHOU H C, LEE C Z, MA L C, et al. Isolation of a chromosomal region of Klebsiella pneumoniae associated with allantoin metabolism and liver infection[J]. Infect Immun, 2004, 72(7): 3783-3792. |
(编辑 白永平)



