2. 国家生猪技术创新中心, 荣昌 402460
2. National Center of Technology Innovation for Pigs, Rongchang 402460, China
荣昌猪是我国著名的地方良种猪,享有“华夏之宝”美誉,具有毛色独特、肉质优良、耐粗饲、杂交配合力高及繁殖性能好等突出优点。繁殖性状是养猪生产中重要的经济性状,荣昌猪初产仔数为10头左右,经产仔数在12头左右。近年来,荣昌猪的各项生产性能缺乏持续的选育,在产仔数、瘦肉率、料肉比等生产性能指标上与外种猪差距逐渐拉大。繁殖力是由微效多基因控制的数量性状,遗传力低,通过传统的选育手段,遗传评估准确性低,遗传进展十分缓慢[1]。因此,迫切需要通过联合常规育种与分子育种技术开展科学选育,提升荣昌猪遗传改良速度,振兴荣昌猪种业。荣昌猪繁殖性状的遗传解析能为分子育种提供可靠的基因资源和分子标记,有助于提升分子育种的准确性和育种进程。但是,目前有关荣昌猪繁殖性状特异性功能基因和位点的研究鲜有报道。
猪繁殖性状功能基因的鉴定一直是猪育种领域的研究热点,也是一个极具挑战性的研究难点。随着全基因组基因分型技术和功能基因鉴定方法的发展,全基因组关联分析已经成为鉴定动物疾病和数量性状遗传变异的重要手段。近年来,国内外众多研究已经鉴定出大量影响猪繁殖性状的重要基因和分子标记。Rothschild等[2]最早发现ESR基因是影响猪产仔数的因果基因,BB型母猪每窝的产仔数比AA型母猪多1.5~2.3头,并且发现ESR在不同胎次中对产仔数的影响明显不同。大量研究报道证实,AHR基因与母猪繁殖性状有紧密联系[3-5]。Bosse等[6]通过基因组分析发现,AHR基因的非同义突变能显著增加母猪的产仔数。研究表明,FSHβ基因对不同品种母猪繁殖性能的影响差异较大,大白猪BB型母猪比其他基因型母猪每窝多产0.41~1.49头仔猪[7];长白猪AB型产仔数高于其他基因型;中国地方猪AA基因型母猪的产仔数高于BB基因型母猪[8]。此外,对外种猪繁殖性状的研究已鉴定出大量与繁殖性状紧密相关的变异位点[9],如RFRP基因g.45859759 C>T位点的突变显著影响母猪产仔数[10],NR4A1基因g3952 A>G突变与总产仔数、活产仔数和窝重显著相关[11],GNB2L1基因g2373 T>C突变显著影响仔猪的初生重[11]。根据猪PigQTL数据库统计显示,已经鉴定到384个与总产仔数相关的QTLs;228个与活产仔数相关的QTLs;85个与窝重相关的QTLs;138个与死产仔数相关的QTLs。
然而,绝大部分重要基因和位点是通过外种猪群体筛选到的,对荣昌猪产仔性状的特异性基因和位点的研究较少,在一定程度上限制了荣昌猪繁殖性状分子选育的实施。本研究基于纯种荣昌猪群体,统计总产仔数、活产仔数、死产仔数以及初生窝重等繁殖表型数据,通过猪50K基因芯片对群体进行基因分型,利用GWAS方法鉴定繁殖性状相关的候选基因和位点,为荣昌猪本品种定向化持续选育以及新品种培育提供重要的分子标记,为荣昌猪种源“卡脖子”技术攻关奠定理论基础。
1 材料与方法 1.1 试验动物与繁殖性状本试验所用的429头纯种荣昌猪均来源于国家生猪核心育种场-重庆琪泰佳牧畜禽养殖有限公司。所有猪只均为初产母猪,在相同的饲养环境、营养水平和饲养管理等条件下饲养。收集2021年1月至2022年6月共429条初产繁殖记录,包括总产仔数(total number born, TNB)、活产仔数(total number born alive, NBA)、死胎数(number of stillborn, NS)和初生窝重(litter weight born alive, LWB)。采集所有母猪的耳组织样品于离心管中,加入75%乙醇,-20 ℃保存。
1.2 基因组DNA提取与质检利用氯仿抽提法提取猪耳组织样的基因组DNA,通过紫外分光光度计(Nanodrop 2000)和1%的琼脂糖凝胶电泳进行DNA质量检测,当浓度>100 ng·μL-1 OD260 nm/OD280 nm介于1.8~2.0之间,条带清晰,无拖尾降解,则DNA质检合格。
1.3 基因分型与基因型填充质检合格的基因组DNA样本利用“中芯一号”芯片(北京康普森农业科技有限公司,北京)进行SNP基因分型,获得的基因分型数据使用Plink(V1.90)软件[12]进行质量控制,剔除无染色体位置信息以及Y染色体上的位点、最小等位基因频率(minor allele frequency,MAF)小于0.01的位点、个体的SNP检出率小于90%的位点、哈迪-温伯格平衡检验P < 10-6的位点。基于质控后的基因型数据,采用Beagle(V5.0)软件[13]对有缺失的基因型数据进行填充,填充后的数据用于后续分析。
1.4 群体结构分析群体分层会造成全基因组关联分析出现假阳性结果[14]。因此采用Plink和GCTA软件[15]对填充后的基因型数据进行遗传距离分析和主成分分析,计算基于状态同源(idengtical by state, IBS)的遗传距离,构建基因组亲缘关系矩阵,以研究荣昌猪群体的亲缘关系和群体分布。采用R软件的ggplot2包绘制热图,对解释方差最大的前3个主成分进行散点图绘制。
1.5 全基因组关联分析首先,采用R软件GenABEL包[16]将繁殖数据校正至标准正态分布。然后,采用GEMMA软件[17]的单变量线性混合模型,将出生年、出生月作为固定因子,主成分分析的前3个主成分作为协变量,进行荣昌猪繁殖性状的关联分析。单变量线性混合模型如下:
| $ y = \gamma Cov + \mathit{\boldsymbol{X}}\beta + \mathit{\boldsymbol{Z}}\alpha + \mathit{\boldsymbol{W}}\mu + e $ | (1) |
其中,y为表型值向量;γ是回归系数;Cov是协变量向量;β为出生年、出生月固定效应向量;α为SNP效应值向量;μ为剩余多基因效应值向量,服从(a~MVN(0, Aσa2))分布,A表示分子亲缘关系矩阵;e为残差效应值向量,服从(e~MVN(0, Iσe2))分布,I表示单位向量;X、Z、W分别是β、α、μ的关联矩阵。
多重检验后,采用Bonferroni校正法来设定显著阈值,基因组水平显著阈值设置为0.05/N=0.05/35 046=1.43×10-6;基因组潜在显著阈值设置为1/N=1/35 046=2.85×10-5,其中N表示参与分析的SNPs数量。使用R软件的qqman包[18]绘制GWAS结果的曼哈顿图和Q-Q图。
1.6 候选基因筛选本研究以显著位点上、下游0.5 Mb范围内的基因组区域作为QTLs区域,通过Ensembl网站(http://ensembl.org/Sus_scrofa/Info/Index)检索QTLs区域内的候选基因,通过NCBI(https://www.ncbi.nlm.nih.gov/)和Genecards在线网站(https://www.genecards.org/)以及已报道文献进一步查找基因的功能,利用Animal QTL Database下载影响猪产仔数、死胎数和初生窝重的QTLs信息,与本研究鉴定出的QTLs进行比对,综合分析所有候选基因的生物学功能和QTLs信息,初步筛选出影响荣昌猪繁殖性状的重要候选基因。
2 结果 2.1 表型数据分析表 1展示了荣昌猪初产繁殖性状的平均值、标准差和变异系数等信息。表 1结果显示,总产仔数的平均值和变异系数分别是10.18头和25.57%;活产仔数的平均值和变异系数分别是8.89头和28.71%;死胎数的平均值和变异系数分别是0.38头和264.49%;初生窝重的平均值和变异系数分别是8.20 kg和28.77%。该结果表明荣昌猪繁殖性状的变异系数较高,具有较大的遗传改良潜力。
|
|
表 1 荣昌猪繁殖性状表型数据统计分析 Table 1 Descriptive statistics of reproductive traits in Rongchang pig population |
利用中芯一号50K SNP芯片对429头纯种荣昌母猪进行基因分型检测,质量控制后,得到35 046个有效的SNPs,其中1号染色体上的位点数最多,有3 815个;18号染色体上的位点数最少,有879个。质控后的SNPs分布密度图见图 1,同一条染色体上相邻SNPs间的平均距离最大为0.089 Mb,位于11号染色体上;最小为0.055 Mb,位于X染色体上。
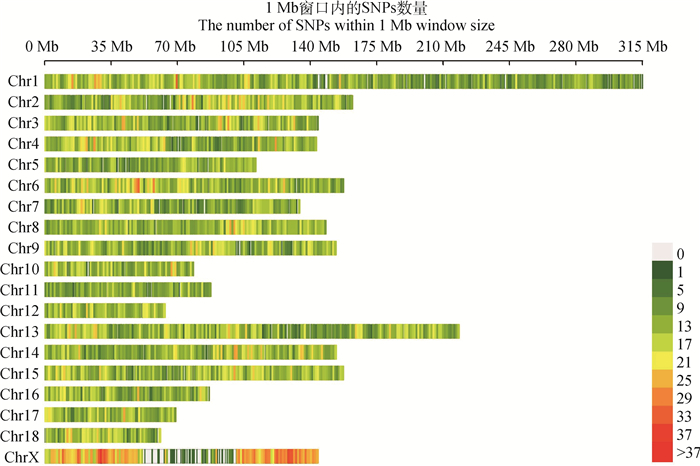
|
图 1 SNP分布密度图 Fig. 1 SNP distribution density map |
图 2为429头荣昌母猪基于IBS的亲缘关系矩阵热图,亲缘关系矩阵热图的颜色越红表示个体间遗传距离越远。结果显示,荣昌母猪群体的IBS距离值为0.000 1~0.343,平均遗传距离为0.242 7,绝大部分个体的遗传距离都大于0.1,表明荣昌猪群体个体间的平均遗传距离较远。

|
横坐标和纵坐标都代表样本个体编号;图中每个小方格代表其所连接的两个个体之间的IBS遗传距离,遗传距离越远越接近红色,反之亦然 The x-axis and y-axis indicate the sample number. Each small square shows the IBS genetic distance value between tow connected individuals, the farther genetic distance, the color is closer to red, and vice versa 图 2 亲缘关系矩阵热图 Fig. 2 Kinship matrix heatmap |
图 3是荣昌猪群体结构分布情况,从图中可以看出试验群体分成了3个部分,说明该群体在人工选育过程中引起群体结构差异,但是各部分之间存在一定的联系,说明各部分间存在基因交流。因此,在后续的全基因组关联分析中将群体分层情况作为协变量,对群体结构进行校正,以降低群体分层带来的影响。
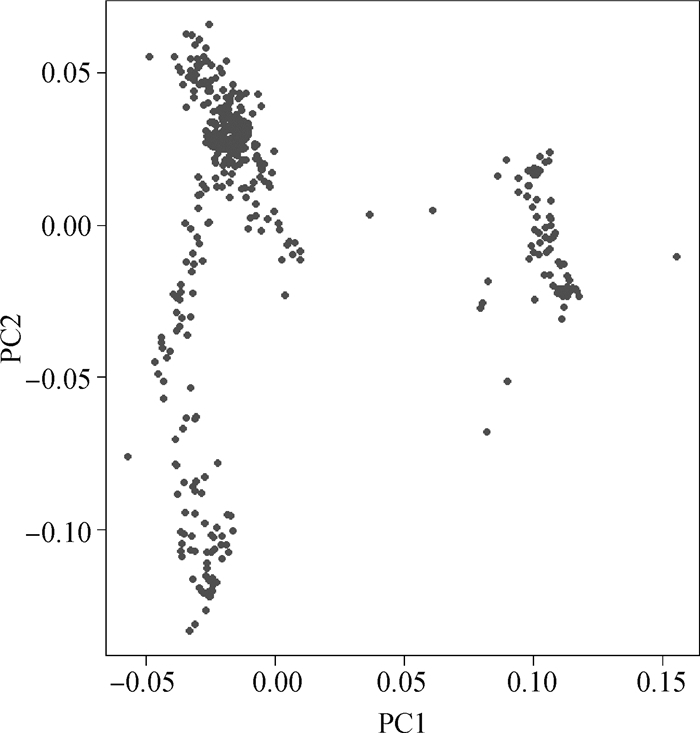
|
图 3 PCA群体结构图 Fig. 3 Principal components analysis of population structure |
本研究采用单标记关联分析对荣昌猪初产的总产仔数、活产仔数、死胎数和初生窝重进行全基因组关联分析,获得与目标性状显著关联的位点和候选基因,结果见图 4和表 2。总产仔数的关联分析结果显示,共检测到5个潜在关联的SNPs(P < 2.85×10-5),分别位于1、8、9、14、17号染色体上,其中最显著的SNPs位于SSC1: 279 214 647 bp(P=5.94×10-6),但是在其上、下游500 kb范围内没有发现候选基因。

|
左图为繁殖性状全基因组关联分析的曼哈顿图,图中红色实线代表全基因组水平显著阈值(1.43×10-6),蓝色实线代表全基因组水平潜在显著阈值(2.85×10-5);右图为繁殖性状全基因组关联分析的Q-Q图 The figures at left represent Manhattan plots of genome-wide association study for reproductive traits, the red line in each figure shows genome-wide significant threshold: 1.43×10-6, and the blue line shows the suggestive significant threshold: 2.85×10-5. The figures at right represent quantile-quantile (Q-Q) plots of genome-wide association study for reproductive traits 图 4 繁殖性状全基因组关联分析的曼哈顿图和Q-Q图 Fig. 4 Manhattan and Q-Q plots of genome-wide association study for reproductive traits in Rongchang pig population |
|
|
表 2 荣昌猪繁殖性状的全基因组关联分析结果 Table 2 The genome-wide association study results for reproductive traits in Rongchang pigs |
活产仔数关联分析结果显示,共检测出3个潜在关联的SNPs(P < 2.85×10-5),分别位于1、5、17号染色体上,其中最显著的SNPs SSC17: 57 315 180 bp(P = 6.02×10-6)位于BMP7、TFAP2C基因附近。
死胎数关联分析结果显示,在1、2、12、13、15号染色体上共检测到11个显著的SNPs(P < 2.85×10-5),其中有1个SNPs(SSC2: 89 287 506 bp,P=8.72×10-8)达基因组显著水平,并且在其上、下游500 kb内检测到一个候选基因MSH3。在13号染色体上检测到3个连续的显著SNPs,位于CBLB基因内。
初生窝重关联分析结果显示,在5和17号染色体上共检测到2个基因组水平显著的SNPs(P < 1.43×10-6)。其中最显著的SNPs(SSC17: 57 315 180 bp,P=5.61×10-8)位于BMP7、TFAP2C、RBM38基因附近。
2.4 显著的共享位点本研究中鉴定到2个共享SNP位点与多个性状存在显著关联,其中SNP位点SSC1: 279 214 647 bp为总产仔数和活产仔数的共享位点;SNP位点SSC17: 57 315 180 bp同时与总产仔数、活产仔数、初生窝重存在潜在关联(表 2),暗示该位点存在一因多效性,可能同时影响荣昌猪总产仔数、活产仔数和初生窝重等繁殖性状。
3 讨论总产仔数、活产仔数和初生窝重是衡量母猪繁殖力的重要指标,死胎数是直接衡量母猪繁殖力损失的重要特征。荣昌猪作为地方猪品种之一,由于缺乏长期的持续选育,繁殖性能的改良仍在原地踏步。为了提高荣昌猪的生产效率,育种研究者正在积极探索针对繁殖性能的育种策略。随着分子育种技术的发展,利用基因组选择等分子技术有望快速提高荣昌猪繁殖性能的遗传改良速度。目前,与荣昌猪繁殖性能相关的特征性遗传变异位点和基因的挖掘鉴定仍然十分缺乏,迫切需要加快繁殖性状特征遗传变异位点的鉴定与利用。全基因组关联研究是解析表型变异的强有力方法,为数量性状的遗传解析提供了许多关键的遗传信息,在畜禽育种中做出了重大贡献。但是,荣昌猪繁殖性状的全基因组关联研究鲜有研究报道。因此,本研究基于50K芯片数据对荣昌猪第一胎次的总产仔数、活产仔数、初生窝重和死胎数进行全基因组关联研究,以期鉴定出影响荣昌猪繁殖性状的重要变异位点和基因,为荣昌猪多个繁殖性状的基因组协同选择提供基础数据。
3.1 群体分层与胎次效应群体分层是直接导致假阳性关联分析结果的主要因素,也是衡量关联分析结果可靠性的决定因素。本研究中,主成分分析结果说明荣昌猪群体存在一定的群体分离情况。为了减少因群体分层带来的假阳性结果,本研究在线性混合模型中将群体主成分作为协变量,出生年、出生月作为固定因子,以消除群体分层造成的影响。理想模型中,当基因组膨胀系数为1.00时,说明群体分层对关联分析没有影响。在实际的研究中,当基因组膨胀系数小于1.05时,说明不存在群体分层的影响。本研究中Q-Q图分析结果说明该关联分析很好的控制了群体分层带来的影响。
猪的繁殖性状是数量性状,受微效多基因的综合调控,并且因母猪个体生理、饲养环境等因素的影响,不同胎次间同一基因的表达模式不同,直接造成不同胎次间产仔性能的差异[19]。不同胎次的繁殖性状之间遗传相关较低[20],说明不同胎次的遗传机制存在明显差异,母猪胎次效应可能是影响繁殖性能的重要因素,不同胎次可定义为不同的性状进行分析[19]。为了避免胎次效应带来的假阳性或假阴性结果,提升繁殖性状候选基因鉴定的准确性,本研究仅对荣昌猪第一胎次的繁殖性状进行了关联分析,挖掘出一系列影响荣昌猪初产繁殖性能的遗传变异。
3.2 候选基因功能分析本研究发现17号染色体上的位点CNC10171044同时与总产仔数、活产仔数和初生窝重显著相关,并且在该位点附近筛选出BMP7基因。BMP7基因是骨形态发生蛋白,属于BMP亚家族,通过调节激素生成、颗粒细胞状态和卵泡发育来影响雌性动物的生殖力,并且在生长发育过程中发挥着关键作用[21]。有研究表明,母羊生殖器官中BMP7基因表达丰度与繁殖力呈正相关,繁殖力高的个体卵巢中BMP7基因表达丰度比繁殖力低的个体高[22],并且发现BMP7基因g.58171886位点变异直接影响母羊第一胎次的产仔数[23]。Li等[24]基于全基因组重测序,通过选择信号分析等方法鉴定了高、低繁殖力大白母猪基因组差异,结果发现,BMP7是影响母猪排卵率和窝产仔数性状的潜在候选基因。此外,有研究表明,BMP7基因的c.2256 G>C、c.1569 A>G、g.35161 T>C都与母猪总产仔数、死胎数以及初生窝重显著相关[25-26],是影响母猪繁殖力的重要候选基因。BMP7基因可能通过调节母猪激素生成和卵泡发育进而影响母猪的繁殖力。因此,该基因可能是影响荣昌母猪产仔数、活产仔数和初生窝重的重要候选基因。
与荣昌母猪死胎数显著相关的位点位于2号染色体89 287 506 bp,其邻近基因为MSH3基因。通过与猪QTL数据库比对发现,该位点接近于已报道的大白猪死胎数相关的QTLs(87.4~87.4 Mb)[27]。MSH3基因位于染色体5q11~q13,编码1 137个氨基酸残基的蛋白质,在DNA错配识别、修复以及维持基因组稳定中发挥着关键作用。MSH3基因表达降低和缺失都会引起生殖相关疾病的发生。有研究表明,MSH3基因的表达与精子活力显著相关,并且与卵巢癌、乳腺癌等繁殖相关的疾病直接相关,进而影响生殖力[28-29]。有研究发现,DNA损伤修复基因表达在胚胎发育过程中发挥着关键作用[30],暗示MSH3基因可能通过调节DNA错配识别和修复进而影响胚胎的发育。母体妊娠期疾病(如:肾病、糖尿病以及心血管疾病等)是妊娠期胎儿死亡的重要原因,有研究发现,与母体肾病、糖尿病以及心血管疾病相关的基因可能直接引发妊娠期胎儿致死[31]。本研究在13号染色体上检测到3个连续的SNPs影响荣昌母猪死胎数,并且在其附近发现CBLB基因。CBLB基因与多种疾病相关,有研究表明,CBLB基因突变是先天免疫性疾病和Ⅰ型糖尿病发生的重要原因[32]。CBLB基因SNP位点的错义突变可能通过改变蛋白质的结构进而影响牛炎症相关疾病的发生[33]。综上,MSH3和CBLB基因可能通过诱发妊娠期母体疾病的发生,进而影响胎儿的正常生长发育。
4 结论本研究基于50K基因芯片对荣昌猪总产仔数、活产仔数和初生窝重进行全基因组关联分析,挖掘影响荣昌猪繁殖性状的重要遗传变异信息。BMP7基因可能是影响荣昌猪总产仔数、活产仔数和初生窝重的重要功能位点;MSH3和CBLB基因可能是影响荣昌猪死胎数性状的重要候选基因。研究结果为荣昌猪繁殖性状提供了重要的遗传变异位点和候选基因,也为荣昌猪繁殖性状的基因组选择提供了重要的理论基础。
| [1] |
YU G, WANG C, WANG Y. Genetic parameter analysis of reproductive traits in Large White pigs[J]. Anim Biosci, 2022, 35(11): 1649-1655. DOI:10.5713/ab.22.0119 |
| [2] |
ROTHSCHILD M, JACOBSON C, VASKE D, et al. The estrogen receptor locus is associated with a major gene influencing litter size in pigs[J]. Proc Natl Acad Sci U S A, 1996, 93(1): 201-205. DOI:10.1073/pnas.93.1.201 |
| [3] |
BABA T, MIMURA J, NAKAMURA N, et al. Intrinsic function of the aryl hydrocarbon (Dioxin) receptor as a key factor in female reproduction[J]. Mol Cell Biol, 2005, 25(22): 10040-10051. DOI:10.1128/MCB.25.22.10040-10051.2005 |
| [4] |
POCAR P, FISCHER B, KLONISCH T, et al. Molecular interactions of the aryl hydrocarbon receptor and its biological and toxicological relevance for reproduction[J]. Reproduction, 2005, 129(4): 379-389. DOI:10.1530/rep.1.00294 |
| [5] |
JABLONSKA O, PIASECKA J, OSTROWSKA M, et al. The expression of the aryl hydrocarbon receptor in reproductive and neuroendocrine tissues during the estrous cycle in the pig[J]. Anim Reprod Sci, 2011, 126(3-4): 221-228. DOI:10.1016/j.anireprosci.2011.05.010 |
| [6] |
BOSSE M, MEGENS H J, FRANTZ L A F, et al. Genomic analysis reveals selection for Asian genes in European pigs following human-mediated introgression[J]. Nat Commun, 2014, 5(1): 4392. DOI:10.1038/ncomms5392 |
| [7] |
PANG P F, LI Z Z, HU H, et al. Genetic effect and combined genotype effect of ESR, FSHβ, CTNNAL1 and miR-27a loci on litter size in a Large White population[J]. Anim Biotechnol, 2019, 30(4): 287-292. DOI:10.1080/10495398.2018.1486322 |
| [8] |
戴丽荷, 胡锦平, 褚晓红, 等. FSHβ基因多态性及对不同品种猪繁殖性状的影响[J]. 浙江农业学报, 2013, 25(3): 461-466. DAI L H, HU J P, CHU X H, et al. Polymorphism of FSHβ gene and its association with the reproductive traits in different breeds of pigs[J]. Acta Agriculturae Zhejiangensis, 2013, 25(3): 461-466. (in Chinese) |
| [9] |
彭雅鑫, 刘军, 赵诗瑜, 等. 猪RXRB基因SNPs检测及其与生长育肥和繁殖性状的关联分析[J]. 畜牧兽医学报, 2021, 52(3): 596-609. PENG Y X, LIU J, ZHAO S Y, et al. Detection of SNPs in porcine RXRB gene and their association analysis with growth, fattening and reproduction traits[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(3): 596-609. (in Chinese) |
| [10] |
FANG M X, HUANG Y S, YE J, et al. Identification and characterization of RFRP gene in pigs and its association with reproductive traits[J]. Genet Mol Res, 2014, 13(1): 1661-1671. DOI:10.4238/2014.January.14.8 |
| [11] |
KUMCHOO T, MEKCHAY S. Association of NR4A1 and GNB2L1 genes with reproductive traits in commercial pig breeds[J]. Genet Mol Res, 2015, 14(4): 16276-16284. DOI:10.4238/2015.December.8.18 |
| [12] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [13] |
BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels[J]. Am J Hum Genet, 2018, 103(3): 338-348. DOI:10.1016/j.ajhg.2018.07.015 |
| [14] |
郝兴杰, 胡林, 张淑君. 全基因组关联分析方法的研究进展[J]. 畜牧兽医学报, 2016, 47(2): 213-217. HAO X J, HU L, ZHANG S J. Progresses in research of genome-wide association study methods[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(2): 213-217. (in Chinese) |
| [15] |
YANG J, LEE S H, GODDARD M E, et al. GCTA: a tool for genome-wide complex trait analysis[J]. Am J Human Genet, 2011, 88(1): 76-82. DOI:10.1016/j.ajhg.2010.11.011 |
| [16] |
AULCHENKO Y S, RIPKE S, ISAACS A, et al. GenABEL: an R library for genome-wide association analysis[J]. Bioinformatics, 2007, 23(10): 1294-1296. DOI:10.1093/bioinformatics/btm108 |
| [17] |
ZHOU X, STEPHENS M. Genome-wide efficient mixed-model analysis for association studies[J]. Nat Genet, 2012, 44(7): 821-824. DOI:10.1038/ng.2310 |
| [18] |
TURNER S D. qqman: an R package for visualizing GWAS results using Q-Q and manhattan plots[J]. J Open Source Softw, 2018, 3(25): 731. DOI:10.21105/joss.00731 |
| [19] |
ONTERU S K, FAN B, DU Z Q, et al. A whole‐genome association study for pig reproductive traits[J]. Anim Genet, 2012, 43(1): 18-26. DOI:10.1111/j.1365-2052.2011.02213.x |
| [20] |
NOGUERA J L, VARONA L, BABOT D, et al. Multivariate analysis of litter size for multiple parities with production traits in pigs: I.Bayesian variance component estimation[J]. J Anim Sci, 2002, 80(10): 2540-2547. |
| [21] |
MONSIVAIS D, CLEMENTI C, PENG J, et al. BMP7 induces uterine receptivity and blastocyst attachment[J]. Endocrinology, 2017, 158(4): 979-992. DOI:10.1210/en.2016-1629 |
| [22] |
BAHIRE S V, RAJPUT P K, KUMAR V, et al. Quantitative expression of mRNA encoding BMP/SMAD signalling genes in the ovaries of Booroola carrier and non-carrier GMM sheep[J]. Reprod Domest Anim, 2019, 54(10): 1375-1383. DOI:10.1111/rda.13535 |
| [23] |
ZHANG Z B, LIU Q Y, DI R, et al. Single nucleotide polymorphisms in BMP2 and BMP7 and the association with litter size in Small Tail Han sheep[J]. Anim Reprod Sci, 2019, 204: 183-192. DOI:10.1016/j.anireprosci.2019.04.001 |
| [24] |
LI X J, YE J W, HAN X L, et al. Whole-genome sequencing identifies potential candidate genes for reproductive traits in pigs[J]. Genomics, 2020, 112(1): 199-206. DOI:10.1016/j.ygeno.2019.01.014 |
| [25] |
DU X, YIN H, PAN Z X, et al. BMP7 is a candidate gene for reproductive traits in Yorkshire sows[J]. Anim Reprod Sci, 2020, 221: 106598. DOI:10.1016/j.anireprosci.2020.106598 |
| [26] |
FENG X, XIE S Y, ZHOU J S, et al. Polymorphisms of the bone morphogenetic protein 7 gene (BMP7) and association analysis with sow productive traits[J]. Anim Reprod Sci, 2013, 142(1-2): 56-62. DOI:10.1016/j.anireprosci.2013.08.011 |
| [27] |
VERARDO L L, SILVA F F, LOPES M S, et al. Revealing new candidate genes for reproductive traits in pigs: combining Bayesian GWAS and functional pathways[J]. Genet Sel Evol, 2016, 48: 9. DOI:10.1186/s12711-016-0189-x |
| [28] |
ZHAO X Y, MU C L, MA J, et al. The association of four SNPs in DNA mismatch repair genes with idiopathic male infertility in Northwest China[J]. Int J Immunogenet, 2019, 46(6): 451-458. DOI:10.1111/iji.12448 |
| [29] |
SONG H L, RAMUS S J, QUAYE L, et al. Common variants in mismatch repair genes and risk of invasive ovarian cancer[J]. Carcinogenesis, 2006, 27(11): 2235-2242. DOI:10.1093/carcin/bgl089 |
| [30] |
HU M H, LOU Y Y, LIU S Y, et al. Altered expression of DNA damage repair genes in the brain tissue of mice conceived by in vitro fertilization[J]. Mol Hum Reprod, 2020, 26(3): 141-153. DOI:10.1093/molehr/gaaa010 |
| [31] |
LYNCH T A, WESTEN E, LI D M, et al. Stillbirth in women with diabetes: a retrospective analysis of fetal autopsy reports[J]. J Matern Fetal Neonatal Med, 2022, 35(11): 2091-2098. DOI:10.1080/14767058.2020.1779213 |
| [32] |
YOKOI N, FUJIWARA Y, WANG H Y, et al. Identification and functional analysis of CBLB mutations in type 1 diabetes[J]. Biochem Biophys Res Commun, 2008, 368(1): 37-42. DOI:10.1016/j.bbrc.2008.01.032 |
| [33] |
刘娟, 韩硕, 邹紫雯, 等. 中国荷斯坦奶牛TLR1和CBLB基因SNP位点筛选及分析[J]. 中国生物制品学杂志, 2021, 34(5): 539-546. LIU J, HAN S, ZOU Z W, et al. Screening and analysis of SNP loci of TLR1 and CBLB genes in Chinese Holstein cows[J]. Chinese Journal of Biologicals, 2021, 34(5): 539-546. (in Chinese) |
(编辑 郭云雁)



