捻转血矛线虫(Haemonchus contortus)属毛圆科(Trichostrongylidae)血矛属(Haemonchus),是一种寄生性线虫[1]。由于其寄生于牛、羊等反刍动物的皱胃中,吸食宿主动物血液,而使虫体肠管呈红色,雌虫白色的生殖管和红色肠管相互缠绕,使虫体常呈红白相间的麻花状外观,因此又被称为捻转胃虫或麻花虫[2]。捻转血矛线虫生活史简单,产卵量大,极易造成环境严重污染,宿主动物常因大量感染而引起严重贫血、高度营养不良,以及虫体在发育过程中产生的代谢物质和分泌物质对宿主动物造成毒害作用,最终常因宿主动物极度衰竭而死亡[3-4]。死亡多发于春季,是牛羊春乏死亡的主要因素之一,给牛羊健康养殖造成严重威胁[5]。
捻转血矛线虫的种类鉴定,主要以雌性成虫红白相间的麻花状外观,以及在显微镜下观察雌虫阴门处发达的阴门盖形态为依据,但曾报道捻转血矛线虫雌虫阴门盖形态存在多态性现象[6-7]。近些年,人们利用分子生物学技术对捻转血矛线虫进行遗传进化分析,Yin等[8]利用线粒体烟酰胺脱氢酶亚基4(mitochondrial nicotinamide dehydrogenase subunit 4,nad4)基因作为遗传标记首次对中国广西、云南、陕西、辽宁、黑龙江以及湖北随州和宜都7个地区捻转血矛线虫的遗传多样性进行研究,结果表明我国境内捻转血矛线虫种群内部发生高度的遗传变异,种群遗传分化结构不明显,种群间存在较高的基因流动。Shen等[9]对贺兰山地区同域内的野生蓝羊和绵羊体内捻转血矛线虫分离株的nad4、第二内转录间隔区(internal transcribed spacer 2,ITS-2)序列进行了分析,结果表明不同宿主来源的虫株间存在一定的基因变异,但无明显的宿主特异性。Dey等[10]研究了孟加拉国七个地形带捻转血矛线虫种群内部及种群间的遗传变异,分析发现不同宿主和地理位置的捻转血矛线虫分离株共享一个基因库,种群内部发生高度的遗传变异,但种群之间的遗传差异不明显。Sallé等[11]采用单个虫体全基因组测序法对来自五大洲19个分离株的223个个体进行研究分析,阐述了气候及人类活动与全球范围内捻转血矛线虫的种群遗传进化关系之间的关联性。Hunt等[7]通过虫体表型变化和微卫星作为遗传标记对来自澳大利亚不同地理来源的捻转血矛线虫种群遗传结构进行解析,结果显示,不同地理位置的捻转血矛线虫在种群结构及表型上均存在差异性。
本研究通过对山羊捻转血矛线虫临床样本进行形态学观察,结果表明,捻转血矛线虫雌虫的阴门盖具有舌瓣形、亚球形和舌-球混合形三种形态,但鲜有基于形态学差异性并结合遗传进化差异进行研究的相关报道[12-15]。本研究基于在捻转血矛线虫雌虫阴门盖形态学差异的基础上,进一步筛选ITS-1、ITS-2及nad4三个基因位点进行PCR扩增和序列分析,探究不同阴门盖形态的捻转血矛线虫的遗传进化差异,以期为捻转血矛线虫的种类鉴定和分子流行病学研究提供参考数据。
1 材料与方法 1.1 材料本试验所用71条捻转血矛线虫雌虫虫体样本由西北农林科技大学动物医学院兽医寄生虫学教研室保存,均分离自陕西关中地区养殖场山羊的皱胃内,采集时间为2019年9月。本试验在西北农林科技大学动物医学院畜禽重要寄生虫致病机制与防控技术研究实验室完成。
1.2 标本制作及雌虫阴门盖形态学观察采集到的虫体样本清洗干净后,放入0.85% NaCl溶液中,置于4 ℃使虫体自然松弛,待死亡后,再移入加热至70 ℃左右的70%酒精中,使虫体充分伸展固定。最后将固定后的虫体保存于70%酒精中,置于4 ℃备用。
进行形态学观察时将虫体从保存液中取出,蒸馏水冲洗后置于乳酸透明液中透明虫体5 min。将透明后的虫体置于载玻片上进行显微观察,并利用测微尺测量虫体全长和阴门至虫体末端长度,试验数据统计使用SPSS26.0进行T检验。
1.3 虫体DNA的提取不同阴门盖形态的捻转血矛线虫雌虫各3条,蒸馏水洗净虫体后,将虫体放入1.5 mL离心管中(1条·管-1),加入150 μL蛋白酶K溶液(20 mg·mL-1),56 ℃过夜放置14 h以上,取出震碎混匀后,根据TIANamp Genomic DNA Kit组织基因组提取试剂盒说明书,从不同阴门盖形态的捻转血矛线虫虫体样品中分别提取基因组DNA,将提取的DNA置-20 ℃保存备用。
1.4 PCR扩增参照严若峰等[16]文献中的正向引物P18S和反向引物P5.8SDW扩增捻转血矛线虫ITS-1基因的部分片段,PCR反应体系总体系为25 μL,包括15.1 μL双蒸水、2.5 μL10×Ex Taq Buffer(不含Mg2+)、0.9 μL MgCl2(2.5 mmol·L-1)、2 μL dNTPs(2.5 mmol·L-1)、0.5 μL Ex Taq酶(5 U·μL-1)、上下游引物(25 pmol·μL-1)各1 μL、2 μL模板DNA。PCR扩增程序:94 ℃预变性3 min;94 ℃变性40 s,46 ℃退火40 s,72 ℃延伸40 s,共30个循环;72 ℃延伸10 min,降至16 ℃结束。
参照Stevenson等[17]的通用正向引物NC1和反向引物NC2扩增ITS-2基因,PCR反应体系总体系为25 μL,包括14.25 μL双蒸水、2.5 μL 10×Ex Taq Buffer(不含Mg2+)、2 μL MgCl2(2.5 mmol·L-1)、2 μL dNTPs(2.5 mmol·L-1)、0.25 μL Ex Taq酶(5 U·μL-1)、上下游引物(25 pmol·μL-1)各1 μL、2 μL模板DNA。PCR扩增程序:94 ℃预变性5 min;94 ℃变性2 min,46 ℃退火45 s,72 ℃延伸2 min,共35个循环;72 ℃延伸10 min,降至16 ℃结束。
参照Troell等[18]文献中的正向引物NAD4-F和反向引物NAD4-R扩增线粒体nad4基因的部分片段。PCR反应体系总体系为25 μL,包括15.1 μL双蒸水、2.5 μL10×Ex Taq Buffer(不含Mg2+)、0.9 μL MgCl2(2.5 mmol·L-1)、2 μL dNTPs (2.5 mmol·L-1)、0.5 μL Ex Taq酶(5 U·μL-1)、上下游引物(25 pmol·μL-1)各1 μL、2 μL模板DNA。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃延伸7 min,降至16 ℃结束。引物序列见表 1,所有引物均由生工生物工程(上海)有限公司合成。
|
|
表 1 引物序列 Table 1 Primer sequence |
对PCR产物进行琼脂糖凝胶电泳,观察结果后,利用DNA纯化试剂盒对PCR产物进行纯化回收,然后将纯化产物连接至pMD-19T载体,并转化至大肠杆菌JM109感受态细胞进行克隆,克隆菌液经PCR鉴定后,将阳性菌液送生工生物工程(上海)有限公司测序。
1.5 序列分析和构建遗传进化树将测得的基因序列使用Clustal X 1.83软件进行比对,并参考图谱校准,将校准后的序列上传至NCBI使用BLAST进行比对,下载匹配度较高的虫种序列作为参考序列,最后采用MEGA6.06中的Neighbor-Joining (N-J)法评估序列之间的系统发育关系[19-20]。采用Kimura 2-parameter参数模型,引导分析使用1 000个重复/树进行,序列的比对分析使用DNAMAN软件进行分析,并利用软件DnaSP v6分析变异位点,核苷酸多样性指数,基因型多样性,单倍型多样性[21]。
2 结果 2.1 捻转血矛线虫雌虫阴门盖形态学观察71条捻转血矛线虫雌虫经显微镜观察发现,其阴门盖存在3种形态,分别为舌瓣形、亚球形和舌-球混合形(图 1),所占比例分别为45.07%(32/71)、50.70%(36/71)和4.23%(3/71)。

|
A. 亚球形阴门盖;B. 舌瓣形阴门盖;C. 舌-球混合形阴门盖 A. Spheroidal type vulval flap; B. Linguiform type vulval flap; C. Ligule-spheroid mixed type vulval flap 图 1 捻转血矛线虫雌虫阴门盖的不同形态特征(100×) Fig. 1 Morphological characteristics of the vulval flap of female H. contortus (100×) |
利用测微尺在光学显微镜下测量挑选出的43条捻转血矛线虫虫体的全长和阴门至虫体末端长度,其中20条舌瓣形阴门盖虫体的平均全长及阴门至虫体末端长度分别为25.47和4.47 mm;20条亚球形阴门盖虫体的平均全长及阴门至虫体末端长度分别为24.80和3.96 mm;3条舌-球混合形阴门盖虫体的平均全长及阴门至虫体末端长度分别为26.66和4.64 mm。3种不同形态阴门盖的捻转血矛线虫的全长统计学差异不显著(P>0.05),而亚球形阴门盖的捻转血矛线虫的阴门至虫体末端长度与其他两种阴门盖的捻转血矛线虫的阴门至虫体末端长度统计学差异显著(P<0.05)。
2.3 不同形态阴门盖捻转血矛线虫PCR扩增基于ITS-1、TIS-2和nad4基因,分别对3种不同形态阴门盖的捻转血矛线虫DNA样品进行PCR扩增,均成功扩增出目的条带,其中,ITS-1扩增出大小约为500 bp的目的条带(图 2A),ITS-2扩增出大小约为321 bp的目的条带(图 2B),nad4扩增出大小约为800 bp的目的条带(图 2C)。

|
A. ITS-1基因PCR扩增结果;B. ITS-2基因PCR扩增结果;C. nad4基因PCR扩增结果;M. DL2000 DNA相对分子质量标准;1. 舌瓣形阴门盖;2. 亚球形阴门盖;3. 舌-球混合形阴门盖;4. 阴性对照 A. PCR amplification results of ITS-1; B. PCR amplification results of ITS-2; C.PCR amplification results of nad4; M. DL2000 DNA marker; 1. Spheroidal type vulval flap; 2. Linguiform type vulval flap; 3. Ligule-spheroid mixed type vulval flap; 4. Negative control 图 2 不同形态阴门盖捻转血矛线虫的3个基因位点PCR扩增结果 Fig. 2 PCR amplification results of three gene of H. contortus with different vulval flap |
|
|
表 2 不同形态阴门盖的捻转血矛线虫全长及阴门至虫体末端长度的长度范围和平均值 Table 2 The range and average value of the body length and the length from the vulva to the posterior of H. contortus with different vulval flap |
对具有不同形态阴门盖捻转血矛线虫的ITS-1、ITS-2及nad4基因片段PCR扩增产物分别进行克隆及测序,去除两端模糊序列后,获得约404 bp的ITS-1基因序列、231 bp的ITS-2基因序列以及730 bp的nad4基因序列。基于ITS-1基因序列的分析显示,3种阴门盖形态捻转血矛线虫的9个样品存在7个基因型,它们彼此之间的相似性均达97.3%以上,与GenBank上H. contortus的相应参考序列(AF04492)一致性为97.5%~100%(图 3A);本研究中ITS-1的7个基因型中共发现10个多态性位点,分别在68、69、87、125、137、157、225、252、318和333,其中,简约型位点有4个,分别在69、137、225和318;此外,有5个位点存在碱基缺失,ITS1-S3序列在305位缺失1个碱基,ITS1-S1、ITS1-S2、ITS1-Q1、ITS1-Q2和ITS1-SQ1在270至273位连续缺失4个碱基;核苷酸多态性为0.85%。ITS-2基因序列的分析显示9个样品存在6个基因型,它们彼此之间的相似性均达95.7%以上,与GenBank上H. contortus的相应参考序列(KX534104)一致性为97.0%~100%,其中, 3个亚球形样品的ITS-2基因序列一致性均达100%(图 3B);6个基因型中共有13个多态性位点,分别在18、21、22、28、29、59、63、115、123、139、146、196和219,无简约型位点,没有碱基缺失或插入现象,核苷酸多态性为1.34%。而基于nad4基因序列的分析结果表明,9个捻转血矛线虫样品共存在9个单倍型,各单倍型之间的相似性为97.3%~98.9%,与GenBank上H. contortus的相应参考序列(KJ724441)一致性为97.8%~99.2%(图 3C);9个单倍型中共有43个多态性位点,19个简约型位点,核苷酸多态性为1.97%。

|
A. ITS-1基因; B. ITS-2基因; C. nad4基因; S1-3. 舌瓣形阴门盖虫体;Q1-3. 亚球形阴门盖虫体;SQ1-3. 舌-球混合形阴门盖虫体 A. ITS-1 gene; B. ITS-2 gene; C. nad4 gene; S1-3. Nematodes with linguiform vulval flap; Q1-3. Nematodes with spheroid-shaped vulval flap; SQ1-3. Nematodes with ligule-spheroid form vulval flap 图 3 不同形态阴门盖捻转血矛线虫ITS-1、ITS-2和nad4基因序列比对分析 Fig. 3 Sequence analysis of ITS-1, ITS-2 and nad4 gene of H. contortus with different vulval flap |
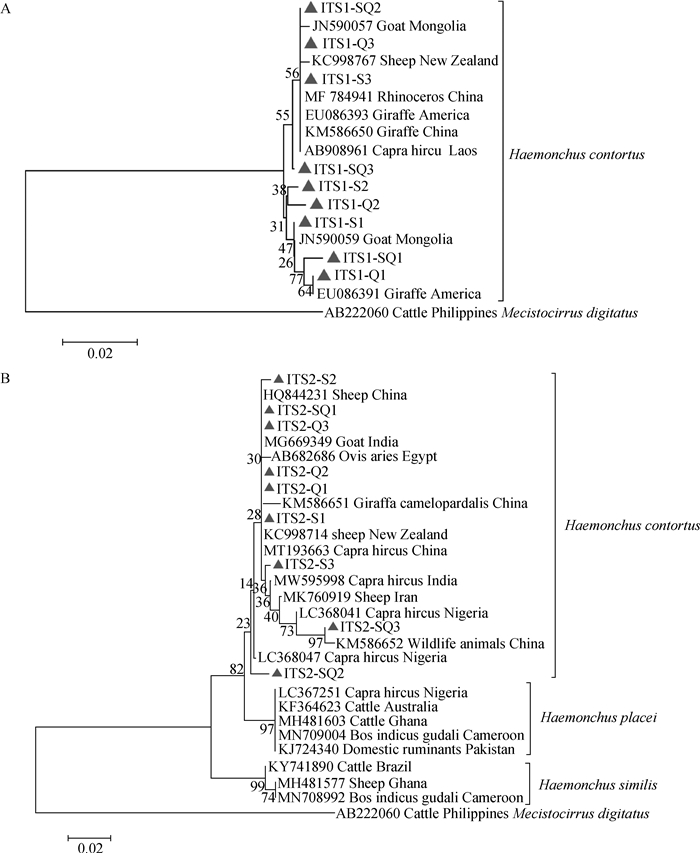
分别利用ITS-1和ITS-2基因位点构建遗传进化树,并以指形长刺线虫(AB222060)作为外群。基于ITS-1基因序列构建的N-J进化树显示,本研究中分离的3种不同形态阴门盖的捻转血矛线虫与NCBI的GenBank数据库中捻转血矛线虫相应基因序列均处于同一进化支,基于ITS-2基因序列构建的N-J进化树显示相似的结果,本研究中分离的3种不同形态阴门盖的捻转血矛线虫与NCBI的GenBank数据库中捻转血矛线虫相应基因序列均处于同一进化支,而与柏氏血矛线虫(H. placei)和似血矛线虫(H. similis)分属不同的进化支(图 4)。
|
A. ITS-1基因; B. ITS-2基因; S1-3. 舌瓣形阴门盖虫体;Q1-3. 亚球形阴门盖虫体;SQ1-3. 舌-球混合形阴门盖虫体 A. ITS-1 gene; B. ITS-2 gene; S1-3. Nematodes with linguiform vulval flap; Q1-3. Nematodes with spheroid-shaped vulval flap; SQ1-3. Nematodes with ligule-spheroid form vulval flap 图 4 捻转血矛线虫分离株ITS-1和ITS-2基因位点系统进化树(Neighbor-Joining法) Fig. 4 The phylogenetic tree based on ITS-1 and ITS-2 loci for isolations of H. contortus (Neighbor-Joining method) |
捻转血矛线虫病是一种呈世界性分布的牛羊等反刍动物寄生虫病,对畜牧养殖行业造成了极大的经济损失[5]。世界范围内,有关捻转血矛线虫遗传多样性的报道较多,但基于形态学差异结合分子生物学进行序列分析的研究报道尚较少[2-3]。在我国,Yin等[8]对广西、云南、陕西、辽宁、黑龙江以及湖北随州和宜都7个地区的捻转血矛线虫虫体进行的研究结果显示,除云南地区部分标本外,其他地区的捻转血矛线虫均未表现出与地理位置相关的遗传多样性。Shen等[9]对贺兰山地区同域内的野生蓝羊和绵羊捻转血矛线虫的遗传多样性进行了报道,显示了相似的结果,表明来自不同地区的群体之间有较高的基因流动,但却没有明确的地理障碍。Dey等[10]对来自孟加拉国7个地形带不同宿主(山羊和绵羊)的捻转血矛线虫种群进行系统进化分析,进化树显示了两个主要的簇,表明孟加拉国存在两个捻转血矛线虫分离株种群,但簇既不是由地理来源形成的,也不是由宿主来源形成的。Kandil等[22]对来自埃及3个地理区域绵羊源的捻转血矛线虫雄虫的遗传多样性进行了报道,结果显示,源自埃及的序列彼此之间的差异很小,并且显示出与源自其他国家序列的巨大差异。本研究基于形态学观察结果分析和统计分析结果表明,捻转血矛线虫雌虫阴门盖存在舌瓣状、亚球形和舌-球混合形3种不同形态,且不形阴门盖的捻转血矛线虫的虫体全长差异不显著(P>0.05),而亚球形阴门盖的捻转血矛线虫与其他两种虫体阴门到虫体末端的长度存在显著差异性(P<0.05)。
此外,基因序列比对分析结果表明,本研究中捻转血矛线虫的ITS-1、ITS-2和nad4基因序列与GenBank上H. contortus的参考序列相似性为均在97%以上。基于ITS-1和ITS-2两个位点构建的遗传进化树显示,本研究所得基因序列与GenBank上H. contortus的参考序列处于同一进化支,提示本研究所分离到的虫体均属捻转血矛线虫。进一步对ITS-2序列进行分析,结果表明,本研究中9个捻转血矛线虫分离株ITS-2共存在13个多态性位点,个体间的遗传差异达5.6%,高于中国其他不同地理区域(2.6%)、孟加拉国(3.1%)以及澳大利亚等7个国家(5.2%)地理源的分离株,同时也高于我国贺兰山地区野生蓝羊源和绵羊源捻转血矛线虫ITS-2的序列差异(4.8%,3.5%)[8-10, 23]。相较于地理及宿主动物源的分离株,本研究中基于形态学差异获得的分离株样品在ITS-2基因序列中存在更高的差异程度以及更多的多态性位点,但本研究中3个亚球形样品间的ITS-2基因序列却具有高度一致性,均达100%(图 4)。此外,本研究中分离株的nad4基因核苷酸多态性(1.97%)与中国其他地理区域(1.8%~3.7%),贺兰山野生蓝羊源(2.1%)和绵羊源(2.0%)的捻转血矛线虫核苷酸多态性相近,但本研究中9个分离株的多态性位点和简约性位点少于前者[8-9]。综上所述,本研究初步解析了基于不同形态阴门盖捻转血矛线虫在多个基因位点上的序列差异性,这为捻转血矛线虫的种类鉴定和分子流行病学研究提供了参考数据,但要深入阐明其形态多样性与遗传进化的关系,需要进行更加深入的研究。
4 结论本研究基于形态学方法,发现捻转血矛线虫雌虫成虫阴门盖存在舌瓣形、亚球形和舌-球混合形3种形态,并进一步对不同形态阴门盖的捻转血矛线虫ITS-1、ITS-2和nad4 3个基因位点的序列进行比对分析,发现不同形态阴门盖虫体在3个基因位点上均存在不同程度的碱基差异。而且相较于其他地理源及宿主动物源的分离株,本研究中的分离株在ITS-2基因序列中的差异程度更高,多态性位点更丰富。
| [1] |
BESIER R B, KAHN L P, SARGISON N D, et al. Diagnosis, treatment and management of Haemonchus contortus in small ruminants[J]. Adv Parasitol, 2016, 93: 181-238. |
| [2] |
ZHOU J R, BU D R, ZHAO X F, et al. Hc-hrg-2, a glutathione transferase gene, regulates heme homeostasis in the blood-feeding parasitic nematode Haemonchus contortus[J]. Parasit Vectors, 2020, 13(1): 40. DOI:10.1186/s13071-020-3911-z |
| [3] |
ESTRADA-REYES Z M, TSUKAHARA Y, AMADEU R R, et al. Signatures of selection for resistance to Haemonchus contortus in sheep and goats[J]. BMC Genomics, 2019, 20(1): 735. DOI:10.1186/s12864-019-6150-y |
| [4] |
BESIER R B, KAHN L P, SARGISON N D, et al. The pathophysiology, ecology and epidemiology of Haemonchus contortus infection in small ruminants[J]. Adv Parasitol, 2016, 93: 95-143. |
| [5] |
PALEVICH N, MACLEAN P H, CHOI Y J, et al. Characterization of the complete mitochondrial genomes of two sibling species of parasitic roundworms, Haemonchus contortus and Teladorsagia circumcincta[J]. Front Genet, 2020, 11: 573395. DOI:10.3389/fgene.2020.573395 |
| [6] |
刘阳. 捻转血矛线虫转录组和蛋白组学分析及耐IVM候选基因功能研究[D]. 呼和浩特: 内蒙古农业大学, 2020. LIU Y. Analysis of transcriptomics and proteomics and functional reseach of IVM-resistance candidate genes in Haemonchus contortus[D]. Hohhot: Inner Mongolia Agricultural University, 2020. (in Chinese) |
| [7] |
HUNT P W, KNOX M R, LE JAMBRE L F, et al. Genetic and phenotypic differences between isolates of Haemonchus contortus in Australia[J]. Int J Parasitol, 2008, 38(8-9): 885-900. DOI:10.1016/j.ijpara.2007.11.001 |
| [8] |
YIN F Y, GASSER R B, LI F C, et al. Genetic variability within and among Haemonchus contortus isolates from goats and sheep in China[J]. Parasit Vectors, 2013, 6(1): 279. DOI:10.1186/1756-3305-6-279 |
| [9] |
SHEN D D, WANG J F, ZHANG D Y, et al. Genetic diversity of Haemonchus contortus isolated from sympatric wild blue sheep (Pseudois nayaur) and sheep in Helan Mountains, China[J]. Parasit Vectors, 2017, 10(1): 437. DOI:10.1186/s13071-017-2377-0 |
| [10] |
DEY A R, ZHANG Z Z, BEGUM N, et al. Genetic diversity patterns of Haemonchus contortus isolated from sheep and goats in Bangladesh[J]. Infect Genet Evol, 2019, 68: 177-184. DOI:10.1016/j.meegid.2018.12.021 |
| [11] |
SALLÉ G, DOYLE S R, CORTET J, et al. The global diversity of Haemonchus contortus is shaped by human intervention and climate[J]. Nat Commun, 2019, 10(1): 4811. DOI:10.1038/s41467-019-12695-4 |
| [12] |
AKKARI H, JEBALI J, GHARBI M, et al. Epidemiological study of sympatric Haemonchus species and genetic characterization of Haemonchus contortus in domestic ruminants in Tunisia[J]. Vet Parasitol, 2013, 193(1-3): 118-125. DOI:10.1016/j.vetpar.2012.12.014 |
| [13] |
GHARAMAH A A, AZIZAH M N S, RAHMAN W A. Genetic variation of Haemonchus contortus (Trichostrongylidae) in sheep and goats from Malaysia and Yemen[J]. Vet Parasitol, 2012, 188(3-4): 268-276. DOI:10.1016/j.vetpar.2012.04.003 |
| [14] |
MA G X, WANG T, KORHONEN P K, et al. Dauer signalling pathway model for Haemonchus contortus[J]. Parasit Vectors, 2019, 12(1): 187. DOI:10.1186/s13071-019-3419-6 |
| [15] |
GILLEARD J S, REDMAN E. Genetic diversity and population structure of Haemonchus contortus[J]. Adv Parasitol, 2016, 93: 31-68. |
| [16] |
严若峰, 宋小凯, 徐立新, 等. 基于ITS序列的捻转血矛线虫系统进化分析[J]. 畜牧兽医学报, 2012, 43(7): 1117-1122. YAN R F, SONG X K, XU L X, et al. Phylogenetic analysis of Haemonchus contortus based on ITS sequence[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(7): 1117-1122. (in Chinese) |
| [17] |
STEVENSON L A, CHILTON N B, GASSER R B. Differentiation of Haemonchus placei from H. contortus (Nematoda: Trichostrongylidae) by the ribosomal DNA second internal transcribed spacer[J]. Int J Parasitol, 1995, 25(4): 483-488. DOI:10.1016/0020-7519(94)00156-I |
| [18] |
TROELL K, ENGSTR? M A, MORRISON D A, et al. Global patterns reveal strong population structure in Haemonchus contortus, a nematode parasite of domesticated ruminants[J]. Int J Parasitol, 2006, 36(12): 1305-1316. DOI:10.1016/j.ijpara.2006.06.015 |
| [19] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [20] |
TAMURA K, STECHER G, PETERSON D, et al. MEGA6:Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [21] |
KIMURA M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [22] |
KANDIL O M, ABDELRAHMAN K A, FAHMY H A, et al. Phylogenetic patterns of Haemonchus contortus and related trichostrongylid nematodes isolated from Egyptian sheep[J]. J Helminthol, 2017, 91(5): 583-588. DOI:10.1017/S0022149X16000687 |
| [23] |
GASSER R B, ZHU X W, CHILTON N B, et al. Analysis of sequence homogenisation in rDNA arrays of Haemonchus contortus by denaturing gradient gel electrophoresis[J]. Electrophoresis, 1998, 19(14): 2391-2395. |
(编辑 白永平)



