2. 丹麦奥胡斯大学,切勒 DK-8830;
3. 北京奶牛中心,北京 100192;
4. 北京首农畜牧发展有限公司,北京 100176;
5. 山东农业大学动物科技学院,泰安 271018
2. Arhus University, Tjele DK-8830, Denmark;
3. Beijing Dairy Cattle Center, Beijing 100192, China;
4. Beijing Sunlon Livestock Development Co. Ltd., Beijing 100176, China;
5. College of Animal Science and Technology, Shandong Agricultural University, Tai 'an 271018, China
实际生产中,牛群的繁殖效率至关重要,对牛场的利润会产生直接影响,体现在兽医、繁殖管理以及牛群结构(淘汰率)等方面[1-2]。奶牛繁殖性状具有重要的经济价值,而其与生产性状有一定的拮抗关系[3-4]。因此,繁殖性状在实际育种指数中占有一定权重。繁殖性状为低遗传力性状,由微效多基因调控,在使用常规遗传分析手段时难以获得较好的选择效果[5]。
自基因组选择(genomic selection,GS)的概念[6]提出以来,其逐渐被广泛应用于我国各种动物实际育种过程中[7-8],也成为奶牛育种策略的重要组成部分[9]。GS是利用覆盖于个体全基因组的单核苷酸多态性(single nucleotide polymorphism,SNP)标记代表个体的基因组信息,结合常规遗传评估信息来计算个体的基因组估计育种值(genomic estimated breeding value,GEBV)。随着GS理论的发展,人们使用高密度标记构建个体间的亲缘关系矩阵,用于替代传统育种中由系谱构建的亲缘关系矩阵。其中,VanRaden[10]基于SNP信息构建了G矩阵,结合最佳线性无偏估计(best linear unbiased prediction,BLUP)方法用于估计个体GEBV,称为GBLUP。Aguilar等[11]提出,将系谱信息及基因组信息同时利用,构建了混合亲缘关系矩阵H阵用于遗传评估,该方法称为一步法GBLUP(single-step GBLUP,ssGBLUP)。在ssGBLUP方法中,可选择不同的参数进行H阵的构建,不同参数组合(α、β、τ和ω; 定义详见材料方法部分)可能会影响最终的预测效果[12]。Misztal等[13]对这些参数进行梯度赋值,评估了参数变化对荷斯坦牛体型性状基因组预测效果的影响。研究表明,当τ的取值线性升高时,基因组预测准确性提高了0.14左右,且各性状无偏性(unbiasedness,b)取值也会有不同程度的增加; 当ω的取值线性升高时,基因组预测准确性上升了0.17左右,但各性状b值会有较大程度的下降。因此,寻找ssGBLUP的最佳参数组合能够保证基因组预测时无偏性最优的同时达到较高的准确性。
GS较传统奶牛育种的优势在于节省后裔测定成本、进行早期选择以减少世代间隔,提高育种值估计的准确性,并加速遗传进展。很多研究表明,将基因组信息纳入育种过程后,能够显著提高估计育种值的可靠性,对低遗传力性状(例如繁殖性状)更加有效[14-15]。但是,目前国内尚未有成熟的奶牛繁殖性状GS体系,且各类GS方法对中国荷斯坦群体繁殖性状的基因组预测效果,以及预测模型不同参数选择对GS效果的影响都是未知的。
本研究基于BLUP、GBLUP和ssGBLUP方法对北京地区中国荷斯坦牛群的部分繁殖性状进行了基因组预测,并比较了系谱信息和基因组的权重对ssGBLUP预测的影响,旨在填补我国奶牛繁殖性状GS体系的空白,并为后续选种及育种策略改良提供理论依据。
1 材料与方法 1.1 试验数据1.1.1 表型数据 原始繁殖记录来源于北京首农畜牧有限公司33个规模化奶牛场阿菲金管理系统。数据收集时间为1990年1月至2020年12月,共保留出生记录381 640条,产犊记录509 054条,配种记录1 789 650条和妊检记录663 759条。系谱数据来源于中国奶牛数据中心(https://www.holstein.org.cn),通过对牛只出生记录和产犊记录进行整理追溯,去除信息不明和错误的个体,最终包含211 131头个体,其中包括6 171头公牛。根据繁殖性状的研究报告及生产实际,本研究主要对3个重要繁殖性状进行分析:1)产犊至首次配种间隔(interval from calving to first service,ICF,d),即产犊日期与该胎次首次配种日期的间隔天数; 2)青年牛配种次数(number of services for heifers,NSH),即青年牛第一次产犊前的配种次数; 3)成母牛配种次数(number of services for cows,NSC),即成母牛每个胎次的配种次数。其中,成母牛的胎次范围为1~3胎。
各繁殖性状表型质量控制范围参考北欧遗传评估中心(Nordic cattle genetic evaluation,NAV)制定的标准[16]。各性状经过最终质控后的描述性统计结果如表 1所示。
|
|
表 1 各性状描述性统计 Table 1 Descriptive statistics for each trait |
1.1.2 基因组数据 芯片测定数据来自于课题组数据库,共有12 195头,包括8 718头母牛和3 477头公牛。所有芯片测定牛中,有3 119头母牛和81头公牛采用GeneSeek公司牛150K芯片(Illumina Bovine 150K SNP),剩余牛只采用50K芯片(Illumina Bovine 50K SNP),后续通过Beagle v5.0软件[17]填充至150K用于本研究。基因组数据通过PLINK1.9软件[18]质控,标准如下:1)个体检出率大于90%;2)剔除最小等位基因频率(minor allele frequency,MAF)小于5%的位点; 3)设定哈迪-温伯格平衡检验(Hardy-Weinberg equilibrium test)阈值为10-5。最终保留了111 181个SNPs位点。
1.2 逆回归育种值计算为了计算繁殖性状的逆回归育种值(De-regressed proof,DRP),首先使用动物(重复力)模型BLUP估计各性状的个体EBVs,然后基于VanRaden等[19]和Guarini等[20]提出的方法计算DRPs:
| $ \begin{gathered} E R C_P=\left[\lambda \frac{E B V_{r e l}}{\left(1-E B V_{r e l}\right)}\right]-E R C_{P A} \\ E R C_{P A}=\lambda \frac{P A_{r e l}}{\left(1-P A_{r e l}\right)} \\ D R P=P A+\left[\frac{E B V-P A}{E R C_P /\left(E R C_P+E R C_{P A}+1\right)}\right] \end{gathered} $ |
其中,DRP为逆回归育种值; ERCPA为父母均值(parent average,PA)的有效记录贡献值(effective record contribution,ERC); ERCP为个体的ERC; PArel为PA的可靠性; EBVrel为EBV的可靠性; λ=(1-h2)/h2。个体DRP的可靠性(rDRP2)则由该公式[21]估计:ERCP/(ERCP+(1-h2)/h2)。
1.3 统计模型本研究所使用的BLUP、GBLUP和ssGBLUP方法的数学模型形式相同:
| $y=\boldsymbol{X} b+\boldsymbol{Z} a+\left(\boldsymbol{W}_c\right)+e $ |
其中,y是表型值(BLUP)或DRP(GBLUP和ssGBLUP)向量; X、Z和W分别是固定效应、加性遗传效应和永久环境效应的关联矩阵; b是固定效应向量,包括配种场-年-月、配种员、性控精液、首配月龄以及胎次(NSH无此效应); a是随机加性遗传效应向量; c是随机永久环境遗传效应向量; e是随机残差向量。在BLUP方法中,a服从正态分布
| $ \boldsymbol{H}^{-1}=\boldsymbol{A}^{-1}+\left[\begin{array}{cc} 0 & 0 \\ 0 & \left(\tau \boldsymbol{G}^{-1}-\omega \boldsymbol{A}_{22}^{-1}\right) \end{array}\right] $ |
式中,G矩阵的构造为:
本研究所有计算均基于BLUPF90软件[24]。先使用AIREML90模块,平均信息约束最大似然法(average information restricted maximum likelihood,AIREML)估计各性状的方差组分,再使用BLUPF90模块构建G和H矩阵进行基因组预测。
1.4 基因组预测验证群对于每个性状,本研究将使用3套数据集进行基因组预测及验证:1)总数据集①:经过NAV标准质控后的数据,用于估计EBVs和计算DRPs,后者作为基因组育种值预测反应变量和预测模型验证的及基准值; 2)母牛验证数据集②:基于数据集①和母牛出生时间划分参考群和验证群,验证群为母牛; 3)公牛验证数据集③:在数据集①的基础上剔除最近4年的母牛(女儿)记录,再按个体出生时间划分参考群和验证群,验证群为公牛。
在数据集②中,根据Su等[25]的方法剔除了验证群母牛在参考群中的近亲,避免信息二次利用。在数据集③中,验证公牛需满足在参考群内无女儿和近亲,但在数据集①中至少有10个女儿。各数据集的参考群都包括了公牛和母牛,不同性状参考验证群划分情况具体见表 2和表 3。
|
|
表 2 GBLUP方法参考验证群划分情况 Table 2 Structure of training and validation data sets for GBLUP method |
|
|
表 3 BLUP和ssGBLUP方法参考验证群划分情况 Table 3 Structure of training and validation data sets for BLUP and ssGBLUP |
验证群体通过各方法所预测的(基因组)估计育种值((G)EBVs)和基准值(DRPs)的皮尔逊相关系数(Pearson correlation coefficient)作为准确性,(G)EBVs对DRPs的回归系数作为无偏性。以这两个参数作为评价基因组预测效果的标准。准确性越高,说明预测值与基准值越接近; 无偏性越接近于1,说明(G)EBVs的尺度与DRPs越接近[26]。
2 结果 2.1 中国荷斯坦牛繁殖性状遗传参数通过AIREML估计得3个繁殖性状的遗传力分别为0.03、0.05和0.08。其中NSC的遗传力最高,NSH次之,ICF的遗传力最低。所有性状皆为低遗传力性状,所估计的方差组分及遗传力估计标准误见表 4。
|
|
表 4 各性状遗传力和方差组分估计结果 Table 4 Heritabilities and variance components for each trait |
针对研究所选3个繁殖性状,在不同的验证群下筛选ssGBLUP方法的最优权重组合(τ和ω),预测准确性结果如图 1所示。对于所有性状,不同验证群下的预测结果有一定差异。当τ为0.5时,所有性状使用任意验证群皆可获得较高的预测准确性,且随着τ的增大,所有性状的预测准确性皆有不同程度的下降。对于ω最优取值,除了基于公牛验证群的NSH外(图 1e,ω=1准确性最高),其余预测皆在ω=0时达到最高预测准确性。
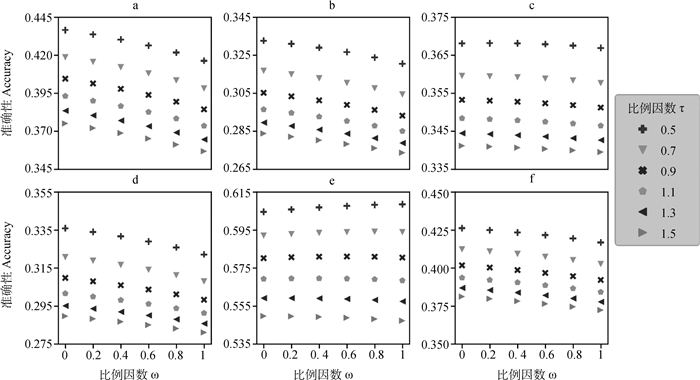
|
a.母牛验证群下ssGBLUP对ICF的预测准确性; b.母牛验证群下ssGBLUP对NSH的预测准确性; c.母牛验证群下ssGBLUP对NSC的预测准确性; d.公牛验证群下ssGBLUP对ICF的预测准确性; e.公牛验证群下ssGBLUP对NSH的预测准确性; f.公牛验证群下ssGBLUP对NSC的预测准确性 a.Prediction accuracies of ssGBLUP for ICF of validation cows; b. Prediction accuracies of ssGBLUP for NSH of validation cows; c. Prediction accuracies of ssGBLUP for NSC of validation cows; d. Prediction accuracies of ssGBLUP for ICF of validation bulls; e. Prediction accuracies of ssGBLUP for NSH of validation bulls; f. Prediction accuracies of ssGBLUP for NSC of validation bulls 图 1 不同权重组合及验证群下各繁殖性状ssGBLUP预测准确性 Fig. 1 Prediction accuracies of ssGBLUP for each fertility trait using various combinations of weights and validation populations |
各性状在不同验证群及权重组合下,ssGBLUP的无偏性(b)结果如图 2所示。对于ICF和NSC两个经产牛性状,母牛验证的b值相比公牛验证更加接近1,而NSH在两种验证群下b值均在1附近波动。当τ减小时,所有性状母牛验证下的b值呈增加趋势。其中,NSH的增加幅度最大,ICF的增加幅度最小,且b值最大可能超过1(图 2a和2b)。对于公牛验证群,各性状b值随τ增加的变化趋势不一致(图 2d-2f)。当τ取值最小时,NSH和NSC的b值最接近1,而ICF则完全相反。对于ω,无论验证群和性状,b值随增大皆呈下降趋势。基于b值,ICF、NSH和NSC在母牛验证群下最优权重组合分别为:τ=1.3和ω=0,τ=0.5和ω=0.4以及τ=0.5和ω=0;在公牛验证群下最优权重组合分别为:τ=1.5和ω=0,τ=1.3和ω=0.8以及τ=0.5和ω=0。
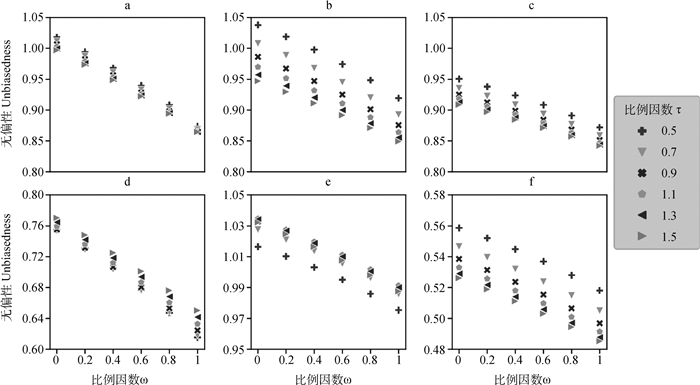
|
a.母牛验证群下ssGBLUP对ICF的预测无偏性; b.母牛验证群下ssGBLUP对NSH的预测无偏性; c.母牛验证群下ssGBLUP对NSC的预测无偏性; d.公牛验证群下ssGBLUP对ICF的预测无偏性; e.公牛验证群下ssGBLUP对NSH的预测无偏性; f.公牛验证群下ssGBLUP对NSC的预测无偏性 a.Unbiasedness of ssGBLUP for ICF of validation cows; b. Unbiasedness of ssGBLUP for NSH of validation cows; c. Unbiasedness of ssGBLUP for NSC of validation cows; d. Unbiasedness of ssGBLUP for ICF of validation bulls; e. Unbiasedness of ssGBLUP for NSH of validation bulls; f. Unbiasedness of ssGBLUP for NSC of validation bulls 图 2 不同权重组合及验证群下各繁殖性状ssGBLUP预测无偏性 Fig. 2 Unbiasedness of ssGBLUP for each fertility trait using various combinations of weights and validation populations |
通过不同验证群比较传统BLUP、GBLUP和ssGBLUP三种方法对不同繁殖性状的基因组预测准确性和无偏性的结果见表 5,其中ssGBLUP选择了使无偏性最接近1的权重组合。母牛验证群下,BLUP、GBLUP和ssGBLUP对3个繁殖性状的平均估计准确性分别为0.25、0.24和0.36,平均无偏性离差分别为0.263、0.267和0.017。对于各性状,ssGBLUP较BLUP和GBLUP方法准确性分别提升了35.7%~65.2%和27.6%~65.2%。ssGBLUP的平均b值与1相差0.017,优于BLUP(0.263)和GBLUP(0.267)。对于ICF,GBLUP的准确性低于BLUP,但b值更接近1;对于NSH,BLUP与GBLUP的准确性相同,且BLUP的b值更优; 对于NSC,GBLUP的预测准确性及无偏性都优于BLUP。公牛验证群下的结果也类似,GBLUP和ssGBLUP的预测准确性和无偏性均优于BLUP,且ssGBLUP的准确性较GBLUP提升24.4%~70.6%。除了ICF外,ssGBLUP的无偏性离差均较GBLUP有所下降。
|
|
表 5 各性状基因组预测准确性和无偏性 Table 5 Accuracies and unbiasedness of genomic prediction for each trait |
对于BLUP方法的结果,使用母牛验证群时准确性高于使用公牛验证群的准确性,且无偏性更加接近于1。基于公牛验证群的GBLUP和ssGBLUP所得平均准确性与基于母牛验证群的平均准确性都相差0.07。除了ICF外,NSH和NSC的准确性都是在使用公牛验证群时较高。就无偏性而言,母牛验证下GBLUP和ssGBLUP方法的平均b值较公牛验证群更接近于1。
3 讨论 3.1 中国荷斯坦牛繁殖性状遗传评估分析本研究系统地分析了不同验证群及预测方法下,荷斯坦牛3个繁殖性状的基因组预测效力。首先,使用动物模型和重复力模型BLUP对各性状的遗传参数进行了估计,结果表明遗传力估计值在0.03~0.08之间,符合该类性状的正常范围[20, 27-29]。对于配种次数性状,本研究中青年牛群体(NSH)的遗传力估计值要低于经产牛群体(NSC)。Guarini等[20]对加拿大荷斯坦牛群繁殖性状的研究也表明NSC的遗传力(0.08)要高于NSH的遗传力(0.02),与本研究结果一致。
3.2 权重取值对基因组预测结果的影响使用ssGBLUP方法构建H阵时,使用不同参数组合改变G阵和A阵的组合,能够影响基因组预测的准确性,目前主要有3种构造H阵的方法。其一为本研究所评估的方法,参数包括α、β、τ和ω(本研究中参数α和β取值为0.95和0.05);方法二为方法一的特殊形式,即将τ和ω的取值都定为1,仅考虑参数α和β的取值[13]; 方法三则进一步对G阵进行调整[30]。构造H阵时,不同的参数取值会影响基因组预测结果的准确性和无偏性,甚至影响算法的收敛容易度[12]。针对τ和ω的取值,本研究结果表明,尽管各性状最佳权重取值不同,但整体而言τ越小,所有性状的基因组预测准确性和b值都越高,且大部分性状在ω=0时能获得更高的准确性和b值(图 1、图 2)。其中,ω对准确性和b值的变化规律与前人研究相同,但τ的变化趋势则与之相反[13]。这可能是由于该先前研究的性状为产奶性状,与本研究的繁殖性状不同,同时其系谱和表型记录数量(超过百万)远高于本研究。另外,其使用的SNP芯片密度为50K,而本研究中的150K芯片大部分是由50K芯片填充所得,部分SNP标记填充错误可能会影响G矩阵的构造[31],进而影响最佳权重系数的取值。Guarini等[20]使用公牛验证群对加拿大荷斯坦群体繁殖性状的GS研究表明,当τ=1且ω=0.6或0.7时,ICF、NSH和NSC的基因组预测效果最好。导致结果不一致的部分原因可能是其研究所使用的芯片密度为50K,且参考群体中拥有系谱个体与基因型数据个体的比例远高于本研究。由于H阵是由基因组和系谱构造,所以研究中参考群体的数量、基因测定个体的数量、系谱深度和SNP芯片密度皆会影响权重的取值。综上,使用ssGBLUP方法之前,确定τ和ω的取值能够在一定程度上提升基因组预测的效力,但该权重的取值会因研究群体和所用SNP芯片的不同而产生差异。
3.3 不同验证群及方法对基因组预测结果的影响实际生产中,GS一般用于提前选择青年公牛以减少育种的世代间隔,进而加快遗传进展。以此为前提,一般会对具有后裔测定信息的公牛和青年公牛进行基因组测定,进而预测青年公牛的基因组育种值,达到提前选种的目的。但近年来,随着芯片测定技术的发展,许多国家也对母牛进行基因组测定,并纳入GS的参考群体来扩充参考群,同时利用基因组育种值选择优秀母牛或者公牛母亲[25]。本研究采用验证群,公牛验证群体个体数较少,但DRP可靠性较高; 而验证母牛群体个体数远远大于公牛验证群体,但DRP可靠性远低于公牛群体(表 2)。验证结果显示, 预测准确性和无偏性在两种验证群体有差别,最主要原因是DRP的准确性。本研究用(G)EBV和DRP的相关系数表示预测准确性,但该相关系数又与DRPs本身的估计准确性相关,而公牛验证群体和母牛验证群体DRPs准确性差别很大。实际上,本研究所使用的预测准确性仅解释了对表型(DRPs)的预测效果,若要计算对估计育种值的预测效果,还需进一步除以表型值(例如DRPs)的准确性[29]。然而,对于低遗传力性状(如繁殖性状),DRPs准确性较小(表 2),作为被除数时可能会因为DRPs准确性估计误差导致异常结果(例如超过1)。因此,对低遗传力性状基因组预测的相关研究中,经常使用表型值与预测值的皮尔逊相关系数作为准确性参数[20, 32-34]。因此,本研究两种验证群结果不能直接比较,但可以在同一验证群体比较不同模型的预测能力,以及不同模型预测能力在两种验证群体的排序是否一致。
在奶牛实际育种中,受到成本限制,一般仅有少部分个体会进行基因组测定。使用GBLUP方法时,需要被评估个体有基因组信息,因此大量的表型数据及系谱信息无法利用。而ssGBLUP则可有效地将基因组、系谱和表型信息同时利用,无基因组信息个体也可以借助基因型信息进行评估。本研究结果表明,ssGBLUP方法的基因组预测效力最好,表现为较高的预测准确性及更好的无偏性。在部分性状中,GBLUP方法的准确性和无偏性可能略逊于BLUP方法(ICF和NSH),其原因可能是GBLUP仅利用少部分基因组测定个体,预测值误差较大(特别是针对低遗传力性状)[35-36]。该结果反映出ssGBLUP方法在GS体系中具有一定的优势。在不同群体及不同物种的研究中,ssGBLUP较BLUP和GBLUP方法都得到了较高的预测准确性和无偏性[20, 37-38]。因此,在本研究比较的3种方法中,ssGBLUP是对中国荷斯坦牛繁殖性状进行基因组选择的最佳方法。
4 结论本研究以中国荷斯坦牛为对象,利用BLUP、GBLUP和ssGBLUP方法对部分重要繁殖性状进行了基因组预测效果分析。研究发现构建H阵时,权重组合对ssGBLUP的预测效果存在影响。因此,正式进行预测前尝试不同权重参数取值,并选择最优的权重组合能够在一定程度上提升ssGBLUP方法的预测性能。另外,研究结果表明所使用的3种方法里,ssGBLUP的预测结果具有较高准确性和无偏性,因此是中国荷斯坦牛繁殖性状基因组选择的最佳方法之一。
致谢: 感谢中国农业大学动物科技学院奶牛分子数量遗传学团队提供部分芯片数据,以及团队老师在性状数据整理分析及构建基因组预测模型等方面提供的帮助。感谢中国农业大学动物科技学院459实验室同学胡丽蓉、张海亮、娄文琦、常瑶、竹磊和安涛等人以及新疆农业大学动物科技学院魏趁和张梦华博士参与血样采样,并在文章撰写过程中提出宝贵的意见。
| [1] |
SAMARAWEERA A M, VAN DER WERF J H J, BOERNER V, et al. Economic values for production, fertility and mastitis traits for temperate dairy cattle breeds in tropical Sri Lanka[J]. J Anim Breed Genet, 2022, 139(3): 330-341. DOI:10.1111/jbg.12667 |
| [2] |
SCHMIDTMANN C, THALLER G, KARGO M, et al. Derivation of economic values for German dairy breeds by means of a bio-economic model—with special emphasis on functional traits[J]. J Dairy Sci, 2021, 104(3): 3144-3157. DOI:10.3168/jds.2019-17635 |
| [3] |
LIU A X, LUND M S, WANG Y C, et al. Variance components and correlations of female fertility traits in Chinese Holstein population[J]. J Anim Sci Biotechnol, 2017, 8(1): 56. DOI:10.1186/s40104-017-0189-x |
| [4] |
SUN C, MADSEN P, LUND M S, et al. Improvement in genetic evaluation of female fertility in dairy cattle using multiple-trait models including milk production traits[J]. J Anim Sci, 2010, 88(3): 871-878. DOI:10.2527/jas.2009-1912 |
| [5] |
VEERKAMP R F, BEERDA B. Genetics and genomics to improve fertility in high producing dairy cows[J]. Theriogenology, 2007, 68 Suppl 1: S266-S273. |
| [6] |
MEUWISSEN T H E, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819-1829. DOI:10.1093/genetics/157.4.1819 |
| [7] |
李森, 杜永旺, 文杰, 等. 快速型黄羽肉鸡饲料利用效率性状的基因组选择研究[J]. 畜牧兽医学报, 2021, 52(8): 2151-2161. LI S, DU Y W, WEN J, et al. A study of genomic selection for feed efficiency traits in fast-growing yellow-feathered broilers[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(8): 2151-2161. (in Chinese) |
| [8] |
彭潇, 尹立林, 梅全顺, 等. 猪主要经济性状的基因组选择研究[J]. 畜牧兽医学报, 2019, 50(2): 439-445. PENG X, YIN L L, MEI Q S, et al. A study of genome selection based on the porcine major economic traits[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 439-445. (in Chinese) |
| [9] |
苏丁然, 彭朋, 闫青霞, 等. 我国荷斯坦青年公牛基因组选择效果分析[J]. 畜牧兽医学报, 2021, 52(6): 1550-1562. SU D R, PENG P, YAN Q X, et al. Analysis of the effects for genomic selection in Holstein young bulls in China[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(6): 1550-1562. (in Chinese) |
| [10] |
VANRADEN P M. Efficient methods to compute genomic predictions[J]. J Dairy Sci, 2008, 91(11): 4414-4423. DOI:10.3168/jds.2007-0980 |
| [11] |
AGUILAR I, MISZTAL I, JOHNSON D L, et al. Hot topic: a unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final score[J]. J Dairy Sci, 2010, 93(2): 743-752. DOI:10.3168/jds.2009-2730 |
| [12] |
MISZTAL I, AGGREY S E, MUIR W M. Experiences with a single-step genome evaluation[J]. Poult Sci, 2013, 92(9): 2530-2534. DOI:10.3382/ps.2012-02739 |
| [13] |
MISZTAL I, AGUILAR I, LEGARRA A, et al. Choice of parameters for single-step genomic evaluation for type[C]//61 st Annual Meeting of the European Association for Animal Production. 2010: 357.
|
| [14] |
GARCÍA-RUIZ A, COLE J B, VANRADEN P M, et al. Changes in genetic selection differentials and generation intervals in us Holstein dairy cattle as a result of genomic selection[J]. Proc Natl Acad Sci U S A, 2016, 113(28): E3995-E4004. |
| [15] |
WIGGANS G R, COLE J B, HUBBARD S M, et al. Genomic selection in dairy cattle: the USDA experience[J]. Annu Rev Anim Biosci, 2017, 5(1): 309-327. DOI:10.1146/annurev-animal-021815-111422 |
| [16] |
NAV (Nordic Cattle Genetic Evaluation). NAV routine genetic evaluation of dairy cattle—Data and genetic models (6th)[EB/OL]. (2018-11-06)[2022-02-25]. https://www/nordicebv.info/wp-content/uploads/2018/11/2018.11.06-NTM-2018-report-Full.pdf.
|
| [17] |
BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels[J]. Am J Hum Genet, 2018, 103(3): 338-348. DOI:10.1016/j.ajhg.2018.07.015 |
| [18] |
CHANG C C, CHOW C C, TELLIER L C, et al. Second-generation PLINK: rising to the challenge of larger and richer datasets[J]. GigaScience, 2015, 4: 7. DOI:10.1186/s13742-015-0047-8 |
| [19] |
VANRADEN P M, VAN TASSELL C P, WIGGANS G R, et al. Invited review: reliability of genomic predictions for north American Holstein bulls[J]. J Dairy Sci, 2009, 92(1): 16-24. DOI:10.3168/jds.2008-1514 |
| [20] |
GUARINI A R, LOURENCO D A L, BRITO L F, et al. Comparison of genomic predictions for lowly heritable traits using multi-step and single-step genomic best linear unbiased predictor in Holstein cattle[J]. J Dairy Sci, 2018, 101(9): 8076-8086. DOI:10.3168/jds.2017-14193 |
| [21] |
MA P, LUND M S, NIELSEN U S, et al. Single-step genomic model improved reliability and reduced the bias of genomic predictions in Danish Jersey[J]. J Dairy Sci, 2015, 98(12): 9026-9034. DOI:10.3168/jds.2015-9703 |
| [22] |
CHRISTENSEN O F, LUND M S. Genomic prediction when some animals are not genotyped[J]. Genet Sel Evol, 2010, 42(1): 2. DOI:10.1186/1297-9686-42-2 |
| [23] |
NILFOROOSHAN M A. Application of single-step GBLUP in New Zealand Romney sheep[J]. Anim Prod Sci, 2020, 60(9): 1136-1144. DOI:10.1071/AN19315 |
| [24] |
MISZTAL I. Complex models, more data: simpler programming?[J]. IEEE Int Comput, 1999, 20: 33-42. |
| [25] |
SU G, MA P, NIELSEN U S, et al. Sharing reference data and including cows in the reference population improve genomic predictions in Danish jersey[J]. Animal, 2016, 10(6): 1067-1075. DOI:10.1017/S1751731115001792 |
| [26] |
WIGGANS G R, COOPER T A, VANRADEN P M, et al. Technical note: adjustment of traditional cow evaluations to improve accuracy of genomic predictions[J]. J Dairy Sci, 2011, 94(12): 6188-6193. DOI:10.3168/jds.2011-4481 |
| [27] |
陈紫薇, 师睿, 罗汉鹏, 等. 宁夏地区荷斯坦牛青年牛繁殖性状遗传参数估计[J]. 畜牧兽医学报, 2021, 52(2): 344-351. CHEN Z W, SHI R, LUO H P, et al. Estimation of genetic parameters of reproductive traits of Holstein heifers in Ningxia[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(2): 344-351. (in Chinese) |
| [28] |
SHI R, BRITO L F, LIU A X, et al. Genotype-by-environment interaction in Holstein heifer fertility traits using single-step genomic reaction norm models[J]. BMC Genomics, 2021, 22(1): 193. DOI:10.1186/s12864-021-07496-3 |
| [29] |
CHEN Z W, BRITO L F, LUO H P, et al. Genetic and genomic analyses of service sire effect on female reproductive traits in Holstein Cattle[J]. Front Genet, 2021, 12: 713575. DOI:10.3389/fgene.2021.713575 |
| [30] |
CHRISTENSEN O F, MADSEN P, NIELSEN B, et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012, 6(10): 1565-1571. DOI:10.1017/S1751731112000742 |
| [31] |
ERTL J, EDEL C, EMMERLING R, et al. On the limited increase in validation reliability using high-density genotypes in genomic best linear unbiased prediction: observations from fleckvieh cattle[J]. J Dairy Sci, 2014, 97(1): 487-496. DOI:10.3168/jds.2013-6855 |
| [32] |
SU G, MADSEN P, NIELSEN U S, et al. Genomic prediction for Nordic red cattle using one-step and selection index blending[J]. J Dairy Sci, 2012, 95(2): 909-917. DOI:10.3168/jds.2011-4804 |
| [33] |
REZENDE F M, HAILE-MARIAM M, PRYCE J E, et al. Across-country genomic prediction of bull fertility in Jersey dairy cattle[J]. J Dairy Sci, 2020, 103(12): 11618-11627. DOI:10.3168/jds.2020-18910 |
| [34] |
SONG H, ZHANG J, JIANG Y, et al. Genomic prediction for growth and reproduction traits in pig using an admixed reference population[J]. J Anim Sci, 2017, 95(8): 3415-3424. |
| [35] |
ALVES A A C, DA COSTA R M, BRESOLIN T, et al. Genome-wide prediction for complex traits under the presence of dominance effects in simulated populations using GBLUP and machine learning methods[J]. J Anim Sci, 2020, 98(6): skaa179. DOI:10.1093/jas/skaa179 |
| [36] |
MOGHADDAR N, VAN DER WERF J H J. Genomic estimation of additive and dominance effects and impact of accounting for dominance on accuracy of genomic evaluation in sheep populations[J]. J Anim Breed Genet, 2017, 134(6): 453-462. DOI:10.1111/jbg.12287 |
| [37] |
GUO X, CHRISTENSEN O F, OSTERSEN T, et al. Improving genetic evaluation of litter size and piglet mortality for both genotyped and nongenotyped individuals using a single-step method[J]. J Anim Sci, 2015, 93(2): 503-512. DOI:10.2527/jas.2014-8331 |
| [38] |
MANCIN E, TULIOZI B, SARTORI C, et al. Genomic prediction in local breeds: the rendena cattle as a case study[J]. Animals, 2021, 11(6): 1815. |
(编辑 郭云雁)



