2. 中国农业科学院北京畜牧兽医研究所,北京 100193;
3. 宁夏农林科学院动物科学研究所,银川 750002;
4. 东营职业学院,东营 257091
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China;
3. Institute of Animal Science, Ningxia Academy of Agriculture and Forestry Sciences, Yinchuan 750002, China;
4. Dongying Vocational Institute, Dongying 257091, China
滩羊是我国优良的地方绵羊种质资源之一,原属蒙古羊,具有个体大、增重快、耐干旱、耐热、耐低营养水平、抗逆性强、抗病力强、适应性强等优良特性[1],其肉质鲜美,裘皮更是中外闻名。然而,由于存在饲养规模小且分散等问题,滩羊的生存空间不断减少[2]。另外,养殖户对滩羊的资源保护意识非常薄弱,甚至受到某些利益驱使,对滩羊进行盲目杂交,致使纯种受到威胁[3]。因此,亟待进行准确遗传评估和科学化管理,以提高滩羊的总体生产水平。系谱对动物遗传及育种研究至关重要,但在散户甚至场区的养殖过程中,难免会出现不可避免的失误,导致系谱记录不全或错误。家畜育种工作中,亲子鉴定能够补全一部分缺失的亲子代关系,对优良种畜的保护和繁衍具有重要意义。
近年来,在动物领域中,分子标记已然成为亲子鉴定的首选遗传标记,其由来已久。此前出现的传统标记方法,如形态标记、细胞标记和生化标记,由于极易受到外界环境的干扰,准确率低,无法成功判断,因此在实际应用中受到了诸多限制[4]。而利用DNA多态性进行亲子鉴定技术具有更灵敏、稳定、准确可靠等优点,以DNA多态性为代表的新型分子标记有限制性片段长度多态性(restriction fragment length polymorphism,RFLP)、随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、简单重复序列(simple sequence repeat,SSR)和单核苷酸多态性(single nucleotide polymorphism,SNP)等。SNP具有二态性,它包括碱基的替换、颠倒、插入和缺失4种情况,在生物体中数量庞大,一些常见的哺乳动物全基因组中,SNP的频率约为每500~1 000个碱基出现一次[5],在基因组中数量多、分布广,具有检测简单,变异率低且遗传稳定性高等众多优点。迄今为止,在亲缘鉴定领域,尽管基于微卫星的研究依然占据绝大多数,但是随着下一代测序技术的兴起,微卫星标记的使用量急剧下降[6],特别是在群体遗传学和局部适应的研究中。SNP作为典型的下一代测序标记,目前在亲缘分析中仍未得到充分利用,但正在获得动力,是最具发展潜力的分子标记[7]。许多研究比较了SNP和微卫星在亲子关系中的效力。早在21世纪初,Hauser等[8]就证明了SNP比STR更加敏捷,之后周磊等[9]模拟了10 000对含微卫星或SNP标记信息的非亲子关系的个体,发现当SNP和微卫星标记的平均杂合度分别为0.363和0.566时,需要的SNP标记数约是微卫星标记的2倍。余国春[10]在猪亲子鉴定中发现,30个SNPs就能达到12个微卫星同样的鉴定效力,并且随着SNP数目的增加,亲缘概率逐渐达到了1。由于SNP标记在不同品种和群体中具有很高的差异性,因此大量研究在不同动物中开展,例如牛[11-16]、羊[17-19]、水产动物[20-24]等,但对滩羊的研究尚未见报道。鉴于此,本研究特异性针对我国宁夏地区滩羊进行亲子鉴定,补充场内现有的系谱,寻找适合的部分SNP组合,为亲子鉴定提供了便利,对盐池地区滩羊的保种与利用具有重要意义。
1 材料与方法 1.1 试验材料本研究试验材料为我国宁夏滩羊场的159只滩羊,包括106个子代个体,以及有系谱记录的4个父本个体和49个疑似父本个体。颈静脉采集它们的血液并置于EDTA抗凝管中,过程中为防止凝固,边采血边摇匀,最后置于-80 ℃冰箱中长期保存。
1.2 试验方法对所有血样均采用Promega试剂盒提取基因组DNA,Nanodrop1000(Thermo Fisher Scientific, DE)进行基因组DNA的质量和浓度检测,检测结果合格后交由北京康普森生物技术有限公司采用600K绵羊芯片进行SNP基因分型。
1.3 遗传结构分析根据全基因重测序数据,利用PLINK(V1.90)软件对所获得的159个滩羊个体的SNPs进行严格的质量控制,标准为:个体检出率大于0.9、位点检出率大于0.9、最小等位基因频率(MAF)大于0.05、位点要符合哈代-温伯格平衡(HWE)。
SNP经Prune后(-indep-pairwise 1 000 5 0.5),使用Rstudio(V4.1.2)软件进行主成分分析(principal component analysis,PCA)。PCA是一种降维的分析方法,它把多个变量通过线性变换选出较少个数重要变量,以得到样本的聚类情况,从而揭示滩羊样本的遗传背景,辅助后续的分析。
系统进化树是以类似树状分支的图形分析各种(类)生物之间的亲缘关系,基于SNP频率构建遗传距离矩阵,利用MEGA(V7.0.26)软件,采用邻接法(Neighbour Joining,NJ)绘制,依据样本的聚类数目划分各个家系。
1.4 亲子鉴定对质控后的SNP及个体信息导入Cervus(V3.0.7)软件[25]进行分析,用于亲子鉴定研究的SNP筛选标准为:个体检出率大于0.95、位点检出率大于0.9、MAF大于0.48、位点符合哈代-温伯格平衡、每条染色体上相邻SNP间距大于10 Mb。
亲子鉴定常用的方法有两种,分别是排除法和似然法。排除法是根据孟德尔的基因分离和自由组合定律,子代的等位基因须分别来自父本和母本同一位点的基因型[26],确定子代与候选亲本是否存在亲子关系,适用于位点较多的分析,本研究选用似然法进行亲子关系推断。Cervus(V3.0.7)软件首先通过Allele Frequency Analysis模块计算各位点的基因频率、杂合度、多态信息含量和排除概率,之后在Simulation模拟分析中估计各置信水平下候选亲本的LOD与对应的Delta临界值,依据此值进行正式亲子鉴定分析,最终找到真实亲本。其中,LOD值大于0表示候选亲本比无关亲本更可能成为子代的真实亲本,且LOD值越大可靠性越高。当出现多个LOD>0的候选亲本时,再通过Delta值(Δ,候选父母亲权可靠性的统计值)分配最可能的亲本(n≥2,Δ=LOD1-LOD2;n=1,Δ=LOD;n=0,Δ无意义)。
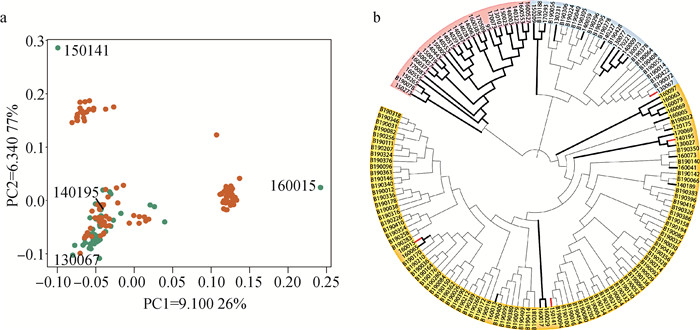
2 结果 2.1 遗传结构分析2.1.1 154个个体遗传结构分析 基因型文件经质控后,剔除5个子代个体,因此剩余154只滩羊和234 337个独立的SNPs位点用于后续分析。PCA散点图(图 1a)中绿色圆点代表父本,红色圆点代表子代,可以看出,样本整体分成3部分,在PC1的正值方向,最右边的家系很明显的与其他两个区分开;除图上标注的系谱记录的4个父本外,其余疑似父本都集中在左下角部位,表明他们之间的遗传背景非常接近。NJ树显示(图 1b),滩羊群体聚集成3个独立的大分支,图中用红色、蓝色和黄色加以区分,这与PCA结果一致;加粗树枝的个体代表父本,多数聚集在红色大分支上,遗传距离相近,少数分布在另外几个分支上,遗传距离较远。
|
a.绿色圆点代表父本,红色圆点代表子代,150141、160015、130067、140195为系谱记录的父本;b.加粗黑色分支为候选父本,加粗红色分支为系谱记录的父本 a.The green dots represent the male parent, the red dots represent the offspring, and 150141, 160015, 130067, 140195 are the male parents recorded in the pedigree; b. The bold black branch is the candidate male parent, and the bold red branch is the male parent recorded in the pedigree 图 1 154个个体PCA与NJ树 Fig. 1 154 individuals PCA and NJ tree |
2.1.2 100个个体遗传结构分析 为进一步观察黄色大分支(图 1b)内个体的聚类情况,本研究提取了这100个样本进行分析,PCA(图 2a)和NJ树(图 2b)将它们划分成3个分支。因此,综合上述研究结果,154只滩羊可分为5个大家系。

|
a. 绿色圆点代表父本,红色圆点代表子代,150141、160015、140195为系谱记录的父本;b.加粗黑色分支为候选父本,加粗红色分支为系谱记录的父本 a. The green dots represent the male parent, the red dots represent the offspring, and 150141, 160015, 140195 are the male parents recorded in the pedigree; b. The bold black branch is the candidate male parent, and the bold red branch is the male parent recorded in the pedigree 图 2 100个个体PCA与NJ树 Fig. 2 100 individuals PCA and NJ tree |
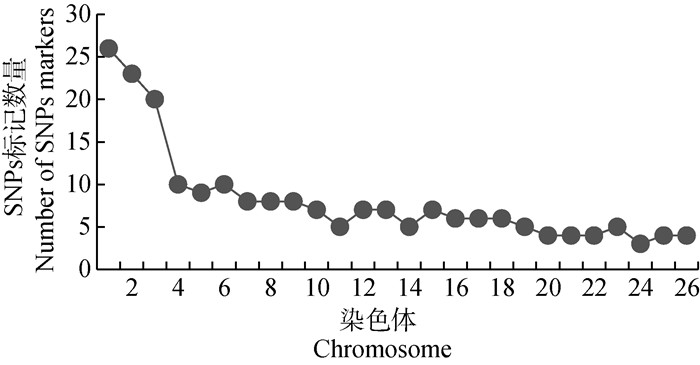
2.2.1 SNP标记分布情况 在PLINK的严格质控下,筛选得到211个可用于亲子鉴定的高质量SNPs,在染色体上的具体的分布情况如图 3所示,它们分布于26条染色上,平均间距为10.81 Mb,其中,1号染色体上的标记数最多,有26个,平均间隔是10.75 Mb。Allele Frequency Analysis计算结果可知(表 1),该标记组合的平均期望杂合度(He)是0.502;平均多态信息含量(PIC)为0.375,属于中度多态性位点;母本未知时,单亲累积父权排除概率高于99.99%,具有很高的亲子鉴定准确性和可靠性。
|
图 3 SNPs标记在26条染色体上的分布情况 Fig. 3 Distribution of SNPs markers on 26 chromosomes |
|
|
表 1 SNP标记信息 Table 1 SNP markers information |
2.2.2 父权鉴定结果 Simulation模拟分析10 000个子代,结果见表 2。在80%的置信水平下,模拟LOD临界值为-39,72%的子代被分配了亲子关系,2 779个子代未找到真实亲本;95%置信度下,分配率为63%。在此模拟环境下,导入质控后剩余的101个子代个体做父权鉴定分析,结果发现(表 3),80%置信度下,有88个子代被分配了亲子关系,13个未找到真实父本;95%置信水平下,观测鉴定率为79%;与原有系谱对照得知,仅12个子代的父本与系谱记录一致,其余在疑似父本中找到。
|
|
表 2 父权鉴定模拟-LOD Table 2 Paternity identification simulation-LOD |
|
|
表 3 父权鉴定参数 Table 3 Paternity authentication parameters |
SNP携带着大量的遗传信息,已经应用于人类[27-30]和动物[31-35]的亲子鉴定中,而在滩羊中的研究尚未见报道。Strucken等[35]对3个牛群和2个羊群研究后,发现仅已知一个亲本基因型且基因分型误差为1%的情况下,至少需要200个SNPs标记进行亲本检验。王悦等[36]在新疆绵羊SNP亲子鉴定的研究中,发现不同MAF梯度和不同数目的SNP对亲子鉴定准确性的影响不同,MAF越大,累积排除概率(CPE)增长速度越快。郭刚等[37]在对北京地区荷斯坦牛进行亲子鉴定研究时,采用了最小等位基因频率(MAF)大于0.45、同一染色体上的标记之间大于10 Mb的筛选标准,本研究在郭刚等[37]筛选的基础上,又缩小MAF范围为大于0.48,故该标记组合具有很高的鉴定可靠性。
除以上影响因素外,殷彬[38]在奶牛的系谱分析中发现,亲子鉴定的排除概率还与位点的期望杂合度和多态信息含量呈显著正相关,根据Botstein等[39]提出的当0.25 < PIC < 0.5时,标记为中度多态位点,PIC>0.5时为高度多态位点,PIC < 0.25为低多态位点,本研究筛选出的标记平均多态信息含量为0.375,属于中度多态性。郭立平[40]的试验证明了45个左右平均期望杂合度为0.5的SNP能够抵消75个平均期望杂合度为0.3的SNP位点。Zhang等[41]在中国西门塔尔牛的SNP开发中,选出了50个高信息含量的SNP标记,它们的平均期望杂合度、平均多态信息含量分别为0.499 8和0.374 8,MAF在0.440 4~0.496 0之间,累积排除概率为99.89%,而本研究筛选到的SNP数量远高于50个,且标记的平均期望杂合度为0.502,累积排除概率超过了99.99%,综合所有参数,理论上,这211个SNPs标记具有很高的亲子鉴定准确效力,能够满足滩羊亲缘关系推断的条件。
3.2 亲子关系推断经推断,被分配亲子关系的个体中,补充了一部分缺失的亲子关系,同时一部分子代的真实亲本也得到了纠正。刘峻宇等[42]研究凡纳滨对虾SNP的亲缘关系验证分析得到,双亲鉴定,实际鉴定率很高,这可能是由于本试验采样的亲本均为父本,缺少母本材料,双亲较单亲检测亲子鉴定效果更好。此外,PCA和NJ树表明,采集的疑似父本间遗传距离很接近,Cervus(V3.0.7)软件很难判断亲子关系,所以导致本研究的鉴定率较低。在父权鉴定模拟中,需要确定抽样父本所占比例,一般设置小于1,除非真实亲本全部在采样个体中,本研究设置为0.6,再加上系谱记录不完全,这种情况下应该增加SNP的数量,以期达到更高的鉴定率。但本研究筛选出的标记质量极高,信息含量丰富,211个完全满足亲子鉴定的要求。另外,在有结果的子代中,很大部分真实亲本在疑似父本中被找到,说明系谱记录有一定的错误,在实际生产中养殖场还应该加强对系谱的管理,预防错误的发生。
4 结论本研究将宁夏滩羊场采集的样本划分为5个家系,并在滩羊中筛选出可用于亲子鉴定的211个高质量SNPs,该组合的平均期望杂合度为0.502,平均多态信息含量为0.375,属于中度多态性位点,单亲累积排除概率大于99.99%,具有很高的准确性和可靠性,为滩羊的保护和繁育提供宝贵的参考价值。
| [1] |
杨秀芳. 宁夏盐池滩羊品种资源的保护现状及发展建议[J]. 中国草食动物, 2006, 26(6): 36-37. YANG X F. Conservation status and development suggestions of sheep breed resources in Yanchitan, Ningxia[J]. China Herbivore Science, 2006, 26(6): 36-37. DOI:10.3969/j.issn.2095-3887.2006.06.015 (in Chinese) |
| [2] |
田黛君, 陈亮, 牛文智. 宁夏滩羊发展现状与对策[C]//全国养羊生产与学术研讨会议论文集(2007-2008). 榆林: 中国畜牧兽医学会养羊学分会, 2008: 37-39. TIAN D J, CHEN L, NIU W Z. Ningxia Tan sheep development status and countermeasures[C]//Discussion Papers of the National Sheep Breeding Production and Academic Symposium (2007-2008). Yulin: Sheep Raising Branch of Chinese Society of Animal Husbandry and Veterinary Medicine, 2008: 37-39. (in Chinese) |
| [3] |
高彦玲. 滩羊种质资源现状及保种研究[J]. 甘肃畜牧兽医, 2022, 52(5): 9-11. GAO Y L. Study on the present situation of Tan sheep germplasm resources and conservation[J]. Gansu Animal Husbandry and Veterinary Medicine, 2022, 52(5): 9-11. (in Chinese) |
| [4] |
VISSCHER P M, WOOLLIAMS J A, SMITH D, et al. Estimation of pedigree errors in the UK dairy population using microsatellite markers and the impact on selection[J]. J Dairy Sci, 2002, 85(9): 2368-2375. DOI:10.3168/jds.S0022-0302(02)74317-8 |
| [5] |
O'BRIEN S J, MENOTTI-RAYMOND M, MURPHY W J, et al. The promise of comparative genomics in mammals[J]. Science, 1999, 286(5439): 458-481. DOI:10.1126/science.286.5439.458 |
| [6] |
FLANAGAN S P, JONES A G. The future of parentage analysis: From microsatellites to SNPs and beyond[J]. Mol Ecol, 2019, 28(3): 544-567. DOI:10.1111/mec.14988 |
| [7] |
余国春. SSR与SNP标记在动物亲缘关系鉴定中的应用[J]. 畜禽业, 2020, 31(12): 23-25. YU G C. Application of SSP and SNP molecular marker in animal parentage analysis[J]. Livestock and Poultry Industry, 2020, 31(12): 23-25. (in Chinese) |
| [8] |
HAUSER L, BAIRD M, HILBORN R, et al. An empirical comparison of SNPs and microsatellites for parentage and kinship assignment in a wild sockeye salmon (Oncorhynchus nerka) population[J]. Mol Ecol Resour, 2015, 11(Suppl): 150-161. |
| [9] |
周磊, 初芹, 刘林, 等. 利用微卫星和SNP标记信息进行奶牛亲子鉴定的模拟研究[J]. 畜牧兽医学报, 2011, 42(2): 169-176. ZHOU L, CHU Q, LIU L, et al. Simulation study on paternity identification in dairy cattle with microsatellite and SNP markers[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(2): 169-176. (in Chinese) |
| [10] |
余国春. 微卫星与SNP标记技术在猪亲子鉴定中的有效性研究[D]. 雅安: 四川农业大学, 2014. YU G C. The effectiveness of microsatellite and SNP labeling technology in pig paternity testing[D]. Ya'an: Sichuan Agricultural University, 2014. (in Chinese) |
| [11] |
ALLEN A R, TAYLOR M, MCKEOWN B, et al. Compilation of a panel of informative single nucleotide polymorphisms for bovine identification in the northern Irish cattle population[J]. BMC Genet, 2010, 11: 5. |
| [12] |
HEATON M P, HARHAY G P, BENNETT G L, et al. Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle[J]. Mamm Genome, 2002, 13(5): 271-281. |
| [13] |
WERNER F A O, DURSTEWITZ G, HABERMANN F A, et al. Detection and characterization of SNPs useful for identity control and parentage testing in major European dairy breeds[J]. Anim Genet, 2004, 35(1): 44-49. DOI:10.1046/j.1365-2052.2003.01071.x |
| [14] |
REVERTER A, HUDSON N J, MCWILLIAM S, et al. A low-density SNP genotyping panel for the accurate prediction of cattle breeds[J]. J Anim Sci, 2020, 98(11): skaa337. DOI:10.1093/jas/skaa337 |
| [15] |
YANG T F, MILLER M, FORGACS D, et al. Development of SNP-based genomic tools for the Canadian bison industry: Parentage verification and subspecies composition[J]. Front Genet, 2020, 11: 585999. DOI:10.3389/fgene.2020.585999 |
| [16] |
GEBREHIWOT N Z, STRUCKEN E M, MARSHALL K, et al. SNP panels for the estimation of dairy breed proportion and parentage assignment in African crossbred dairy cattle[J]. Genet Sel Evol, 2021, 53(1): 21. DOI:10.1186/s12711-021-00615-4 |
| [17] |
CLARKE S M, HENRY H M, DODDS K G, et al. A high throughput single nucleotide polymorphism multiplex assay for parentage assignment in New Zealand sheep[J]. PLoS One, 2013, 9(4): e93392. |
| [18] |
HEATON M P, LEYMASTER K A, KALBFLEISCH T S, et al. SNPs for parentage testing and traceability in globally diverse breeds of sheep[J]. PLoS One, 2014, 9(4): e94851. DOI:10.1371/journal.pone.0094851 |
| [19] |
TORTEREAU F, MORENO C R, TOSSER-KLOPP G, et al. Development of a SNP panel dedicated to parentage assignment in French sheep populations[J]. BMC Genet, 2017, 18(1): 50. DOI:10.1186/s12863-017-0518-2 |
| [20] |
马群群. 拟穴青蟹SNP和SSR标记开发及系谱认证技术的建立研究[D]. 上海: 上海海洋大学, 2012. MA Q Q. Development of SNP and SSR markers and establishment of parentage analysis technique in Scylla paramamosain[D]. Shanghai: Shanghai Ocean University, 2012. (in Chinese) |
| [21] |
葛会争. 德国镜鲤与黑龙江野鲤杂交组合的亲子鉴定及鲤鱼免疫功能基因SNP位点研究[D]. 上海: 上海海洋大学, 2012. GE H Z. Parentage identification of Heilongjiang wild carp and the German mirror carp cross combinations on microsatellite DNA markers and carp immune function gene SNP research[D]. Shanghai: Shanghai Ocean University, 2012. (in Chinese) |
| [22] |
BEACHAM T D, WALLACE C, MACCONNACHIE C, et al. Population and individual identification of chinook salmon in British Columbia through parentage-based tagging and genetic stock identification with single nucleotide polymorphisms[J]. Can J Fish Aquat Sci, 2018, 75(7): 1096-1105. DOI:10.1139/cjfas-2017-0168 |
| [23] |
THONGDA W, ZHAO H G, ZHANG D D, et al. Development of SNP panels as a new tool to assess the genetic diversity, population structure, and parentage analysis of the eastern oyster (Crassostrea virginica)[J]. Mar Biotechnol (NY), 2018, 20(3): 385-395. DOI:10.1007/s10126-018-9803-y |
| [24] |
WENG Z Y, YANG Y, WANG X, et al. Parentage analysis in giant grouper (Epinephelus lanceolatus) using microsatellite and SNP markers from genotyping-by-sequencing data[J]. Genes (Basel), 2021, 12(7): 1042. DOI:10.3390/genes12071042 |
| [25] |
KALINOWSKI S T, TAPER M L, MARSHALL T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J]. Mol Ecol, 2007, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [26] |
李冰. DNA检测用于亲子鉴定的基本原理[J]. 中国工作犬业, 2006(10): 39. LI B. The principle of DNA paternity testing[J]. China Working Dog, 2006(10): 39. (in Chinese) |
| [27] |
CHANG L, YU H Y, MIAO X Y, et al. Evaluation of a custom SNP panel for identifying and rectifying of misjudged paternity in deficiency cases[J]. Front Genet, 2021, 12: 602429. DOI:10.3389/fgene.2021.602429 |
| [28] |
布蕾楠. 法医个体识别多等位基因SNP的筛选及NGS-SNP分型体系的建立[D]. 石家庄: 河北医科大学, 2020. BU L N. Genome-wide screening of multi-allelic SNPs for forensic individual identification and development of NGS-SNP panel[D]. Shijiazhuang: Hebei Medical University, 2020. (in Chinese) |
| [29] |
LONG J. Parentage analysis using genome-wide high-density SNP microarray[J]. Gene, 2021, 785: 145605. |
| [30] |
MO S K, LIU Y C, WANG S Q, et al. Exploring the efficacy of paternity and kinship testing based on single nucleotide polymorphisms[J]. Forensic Sci Int Genet, 2016, 22: 161-168. |
| [31] |
HU L R, LI D, CHU Q, et al. Selection and implementation of single nucleotide polymorphism markers for parentage analysis in crossbred cattle population[J]. Animal, 2021, 15(1): 100066. |
| [32] |
MCCLURE M C, MCCARTHY J, FLYNN P, et al. SNP data quality control in a national beef and dairy cattle system and highly accurate SNP based parentage verification and identification[J]. Front Genet, 2018, 9: 84. |
| [33] |
SERMYAGIN A A, DOTSEV A V, GLADYR E A, et al. Whole-genome SNP analysis elucidates the genetic structure of Russian cattle and its relationship with Eurasian taurine breeds[J]. Genet Sel Evol, 2018, 50(1): 37. |
| [34] |
VAN DER ZWAN H, VISSER C, SCHOONEN M, et al. Development of an SNP-based parentage verification panel for lovebirds[J]. Anim Genet, 2019, 50(6): 764-767. |
| [35] |
STRUCKEN E M, LEE S H, LEE H K, et al. How many markers are enough?Factors influencing parentage testing in different livestock populations[J]. J Anim Breed Genet, 2016, 133(1): 13-23. |
| [36] |
王悦, 覃锡忠, 贺三刚, 等. SNP标记用于新疆5个绵羊品种亲子鉴定准确性的研究[J]. 中国科技论文, 2017, 12(24): 2867-2872. WANG Y, QIN X Z, HE S G, et al. Accuracy of parentage identification for 5 sheep breeds in Xinjiang based on SNPs[J]. China Sciencepaper, 2017, 12(24): 2867-2872. (in Chinese) |
| [37] |
郭刚, 周磊, 刘林, 等. 利用SNP标记进行北京地区中国荷斯坦牛亲子推断的研究[J]. 畜牧兽医学报, 2012, 43(1): 44-49. GUO G, ZHOU L, LIU L, et al. Parentage inference with single nucleotide polymorphism markers in the Chinese Holstein in Beijing[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(1): 44-49. (in Chinese) |
| [38] |
殷彬. 奶牛分子系谱鉴定的微卫星标记筛选及其与生产性能的关联分析[D]. 泰安: 山东农业大学, 2016. YIN B. Selection of micro-satellite markers for molecular genealogical confirmation and their association with production traits in Holstein dairy cows[D]. Tai'an: Shandong Agricultural University, 2016. (in Chinese) |
| [39] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314-331. |
| [40] |
郭立平. 利用微卫星和SNP标记对西门塔尔牛进行亲子推断的研究[D]. 北京: 中国农业科学院, 2013. GUO L P. Parentage testing with microsatellite and SNP markers in Simmental[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. (in Chinese) |
| [41] |
ZHANG T L, GUO L P, SHI M Y, et al. Selection and effectiveness of informative SNPs for paternity in Chinese Simmental cattle based on a high-density SNP array[J]. Gene, 2018, 673: 211-216. |
| [42] |
刘峻宇, 刘均辉, 孔杰, 等. 凡纳滨对虾SNP标记开发与家系亲缘关系验证分析[J]. 渔业科学进展, 2021, 42(1): 108-116. LIU J Y, LIU J H, KONG J, et al. Development of SNP markers and verification analysis of relationship on family in Litopenaeus vannamei[J]. Progress in Fishery Sciences, 2021, 42(1): 108-116. (in Chinese) |
(编辑 郭云雁)



