2. 河南省鸡种质资源创新与利用重点实验室,郑州 450046;
3. 河南省家禽育种国际联合实验室,郑州 450002
2. Henan Key Laboratory for Innovation and Utilization of Chicken Germplasm Resources, Zhengzhou 450046, China;
3. International Joint Research Laboratory for Poultry Breeding of Henan, Zhengzhou 450002, China
DNA甲基化是指DNA序列上特定的碱基在DNA甲基转移酶(DNMT)的催化作用下,通过共价键结合的方式获得一个甲基基团的化学修饰过程。其中CpG二核苷酸中胞嘧啶(C)上第5位碳原子的甲基化(5 mC)是动、植物等真核生物DNA甲基化的主要形式。基因特定功能元件的甲基化会抑制基因表达,去甲基化则可以使得基因重新表达[1]。研究表明,DNA甲基化参与诸多生物学过程,包括基因印迹、X染色体的失活、基因组稳定性、转座子及逆转录转座子沉默及组织特异性基因沉默等[2-3]。
肝是动物体内重要的代谢器官,在脂类分解、合成及运输等过程中均发挥重要作用。已有的研究表明,DNA甲基化是哺乳动物肝脂质代谢的重要调控机制之一。高脂饮食(HFD)饲喂的小鼠,其肝促炎性半胱天冬酶(CASP1)基因和与能量代谢相关的NADH脱氢酶1β亚基复合物9(NDUFB9)基因启动子区甲基化水平分别显著降低和升高,从而改变了各自的表达水平[4]。高脂肪蔗糖(HFS)饮食可诱导大鼠肝脂质积累,但如果在高脂肪蔗糖饮食中补充甲基供体,会影响小鼠肝脂肪酸合成酶(FASN)基因启动子区域甲基化水平,进而预防高脂肪蔗糖饮食诱导的肝甘油三酯积累[5]。然而,在一些特定的生理过程中,肝基因表达水平的变化与其甲基化水平并无必然的联系。低剂量(1.5 μg·kg-1) 丙二酚S(BPS)处理小鼠可导致其肝374个基因表达水平显著变化,同时引起58.5%的不同甲基化区域(DMR)发生低甲基化。进一步的分析发现,包括涉及脂质代谢途径的4个基因(FASN、HMGCR、ELOVL6、LPIN1)在内的18个基因的表达水平受甲基化状态影响,但并未发现这些基因的表达谱和甲基化谱之间存在显著的关联[6]。可见,DNA甲基化并不是影响基因转录的唯一表观遗传修饰途径,还受到其他表观遗传修饰的影响[7-9]。
与哺乳动物相比,鸡肝在脂质代谢过程中的作用更为重要,超过90%脂肪酸的从头合成在肝中进行[10]。尤其在产蛋期,在雌激素等生殖激素的协同作用下,肝脂质代谢会更加旺盛,合成大量的甘油三酯、胆固醇和其他脂类,并组装为极低密度脂蛋白,然后通过血液循环运输到生长中的卵母细胞用于蛋黄的形成[11]。本研究对产蛋前期(20周龄)和产蛋高峰期(30周龄)鸡肝转录组的比较分析表明,与产蛋前期相比,2 567个基因的表达水平在产蛋高峰期发生了显著的变化,这些差异表达基因主要富集于脂质代谢生物学过程和代谢通路[12]。但这些基因的表达差异是否与基因组甲基化修饰的改变有关,需要进一步研究。
本研究利用全基因组重亚硫酸盐测序法(whole genome bisulfite sequencing,WGBS)分析产蛋前期(20周龄)及产蛋高峰期(30周龄)卢氏绿壳蛋母鸡基因组甲基化模式,结合肝转录组数据,探讨两个生理阶段基因组甲基化与基因转录调控的关系,为深入研究蛋鸡不同生理阶段肝代谢的分子调控机制提供理论依据。
1 材料与方法 1.1 试验动物和样品采集试验动物和样品采集方法参照文献[12]。以河南农业大学家禽种质资源场饲养的地方品种卢氏绿壳蛋鸡为试验材料,所有试验鸡饲养管理条件一致,自由采食和饮水。根据卢氏绿壳蛋鸡开产日龄及产蛋曲线分析,平均21周龄开始产蛋,28~32周龄达到产蛋高峰[13]。因此,选取产蛋前期(20周龄)及产蛋高峰期(30周龄)健康卢氏绿壳蛋母鸡各3只,颈部静脉放血处死后,迅速采集肝组织样品,液氮速冻后,-80 ℃保存备用。
1.2 肝全基因组重亚硫酸盐测序(WGBS)1.2.1 DNA提取及样品检测 利用标准的苯酚-氯仿法提取基因组DNA,琼脂糖凝胶电泳分析DNA降解程度,Nanodrop(Life Technologies,美国)检测DNA的纯度(OD260 nm/OD280 nm比值),Qubit 2.0(Life Technologies,美国)检测DNA浓度。
1.2.2 文库构建 基因组DNA检测合格后,分别将产蛋前期及产蛋高峰期3个个体的DNA样品等量混合,加入24 ng阴性对照(λDNA),使用Covaris S220(Covaris,美国)将基因组DNA随机打断至200~300 bp;对打断后的DNA片段进行末端修复、加A尾,并连接上所有胞嘧啶均经过甲基化修饰的测序接头;随后进行Bisulfite处理(EZ DNA Methylation Gold Kit,Zymo Research)。未发生甲基化的C变成U(PCR扩增后变为T),而甲基化的C保持不变,最后进行PCR扩增,得到最终的DNA文库。
1.2.3 文库质检 文库构建完成后,将DNA浓度稀释至1 ng·μL-1,随后使用Agilent 2100(Agilent,美国)对文库的插入片段长度进行检测,符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度>2 nmol·L-1),以保证文库质量。
1.2.4 上机测序 库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后进行Hiseq测序,测序方式为双末端测序,由Novogene(北京)完成测序。
1.3 全基因组甲基化测序数据分析1.3.1 参考序列比对分析 参照Bolger等[14]的方法对测序原始数据进行过滤,然后利用Bismark软件(version 0.16.1)将测序的结果和参考基因组都进行了C→T和G→A(反向互补)的转化,将转化后的测序结果和基因组分别进行两两比对,计算并输出每个样品比对率和Bisulfite(BS)转化率等结果。
1.3.2 测序深度统计、覆盖度和胞嘧啶甲基化(mC)分析 每个碱基位点比对到染色体上的读数即为该位点的测序深度,并对基因组上每个单碱基位点的覆盖深度(即支持该位点的reads数)进行计算,据此得到覆盖度统计结果。计算CG、CHH、CHG(H代表A、C或T碱基)3种序列环境下C位点的测序深度。单个位点甲基化水平计算公式:单个位点甲基化水平=(覆盖到mC的序列数/有效覆盖序列总数)×100。
1.3.3 差异甲基化区域(DMR)分析 针对无生物学重复的试验,采用swDMR软件(http://122.228.158.106/swDMR/)鉴定差异甲基化区域(differentially methylated region,DMR)。该软件基于每个位点的甲基化信息(设定reads coverage≥5),采取滑窗的方法在基因组上扫描,鉴定出甲基化程度存在差异的区域。基于DMR的重要生物学意义,利用DMR所在的基因组位置与基因组结构注释信息,对其进行结构注释。不同区域发生甲基化其调控基因表达的方式有所区别,统计不同功能区域DMR的数量。为了更好地了解DNA甲基化与基因表达之间的关系,将基因组划分为几个功能区域,即启动子(promoter,转录起始位点(TSS)上游2 kb区域),基因体(gene body,5′非翻译区(5′UTR)、外显子(exon)、内含子(intron)和3′非翻译区(3′UTR))和重复序列(repeat,转座子和其他重复元件)[15]。
1.3.4 差异甲基化基因(differentially methylated gene,DMG)的GO和KEGG富集分析 当DMR所在区域与特定基因功能元件有重叠时,将相应的基因挑选出来,称为DMR相关基因(DMGs)。利用GOseq R软件包[16]对DMGs进行基因本体(GO)富集分析,并对基因长度偏差进行校正,校正后P < 0.05的GO是DMGs显著富集项。进一步使用KOBAS软件[17]检验在KEGG(Kyoto Encyclopedia of Genes and Genomes)通路中显著富集的DMGs。
1.3.5 WGBS和转录组测序(RNA-seq)数据整合分析 将与WGBS同一批次的卢氏绿壳蛋鸡产蛋前期和产蛋高峰期转录组测序原始数据已上传国家生物技术信息中心(National Center for Biotechnology Information,NCBI),登记号为GSE70010[12]。对产蛋前期和产蛋高峰期差异表达基因CG、CHG和CHH序列环境下C位点的甲基化水平进行统计,并根据转录组基因表达量的上下四分位数将基因分为4组:none(FPKM<1);low(1<FPKM<上四分位数);medium(下四分位数<FPKM<上四分位数);high(FPKM>上四分位数),默认选取FPKM≥1作为基因是否表达的阈值,分析不同基因表达水平和甲基化水平的相关性,绘制折线图。基因的甲基化水平按照启动子(promoter)和基因体(gene body)两种方式进行统计,分析每个基因的启动子和基因体区甲基化水平和基因表达水平的相关性,绘制箱式图。将基因的甲基化水平值以20%为梯度,按照甲基化水平值大小(甲基化水平值>0)分为5个组别:第一组(1 st group)、第二组(2nd group)、第三组(3rd group)、第四组(4th group)及第五组(5th group)。其中,第一组(1 st group)代表甲基化水平最低,第五组(5th group)代表甲基化水平最高,基因没有发生(未)甲基化(unmethylated)代表甲基化水平为0单独归为一组。分析不同甲基化水平和基因表达水平的相关性,绘制折线图。将DMGs与DEGs进行整合,分析基因甲基化不同功能区域与基因表达的联系,筛选与肝脂质代谢相关的候选基因。
2 结果 2.1 鸡肝全基因组甲基化测序结果与鸡参考基因组序列比对分析产蛋前期和产蛋高峰期卢氏绿壳蛋鸡全基因组甲基化测序数据分析结果显示,平均每个样品产出过滤后数据36.39 Gb,平均每个样品得到95 572 532条唯一比对序列,平均唯一比对率为73.51%,平均BS转化率为99.84%,≥10×平均覆盖率为73.50%(表 1)。
|
|
表 1 测序结果与参考基因组比对情况 Table 1 The comparison of reads to the reference genome |
2.2.1 鸡肝全基因组甲基化水平分析 产蛋前期和产蛋高峰期两个生理时期卢氏鸡肝全基因组范围内约有4%的胞嘧啶(C)被甲基化(mC)。根据胞嘧啶(C)序列特征可以将其分为CG、CHH和CHG(H代表A、T或C碱基)3种序列环境,C位点甲基化主要发生在CG序列环境,其次是CHG和CHH(图 1A)。产蛋高峰期mCG百分比(71.40%)要高于产蛋前期(67.73%),在CHH和CHG序列环境下,C甲基化比例较低,产蛋前期mCHG和mCHH占比分别为9.39%、22.88%,产蛋高峰期mCHG、mCHH占比分别为7.98%、20.61%(图 1B)。
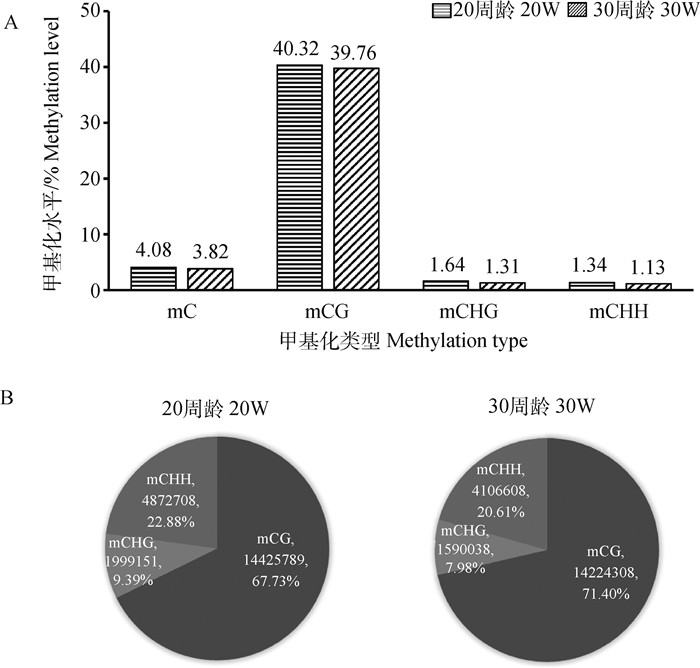
|
A.整个基因组中CG、CHG和CHH的平均甲基化水平;B.不同生理阶段的不同序列环境(CG、CHG和CHH)胞嘧啶甲基化百分比 A. Average methylation levels of CG, CHG and CHH in the whole genome; B. The proportion of mCG, mCHH and mCHG in different physiological stages 图 1 产蛋前期和产蛋高峰期鸡全基因组DNA甲基化模式 Fig. 1 Genome-wide DNA methylation patterns of hens at pre-laying and peak-laying stages |
2.2.2 鸡全基因组功能区域甲基化水平分析 为了更好地理解基因外显子、内含子、3′ UTR、5′ UTR和重复序列,将每个基因的各个功能区域分别等分成20个单位长度,然后对所有的功能元件区域相应单位长度C位点甲基化水平均值进行统计分析(图 2)。结果表明,两生理时期不同基因功能元件在CG、CHG和CHH 3种序列环境下的甲基化水平变化趋势一致。DNA甲基化水平在启动子区域周围呈现“V”型趋势,向基因体区域上升;同一生理时期不同基因功能元件的甲基化水平存在差异,外显子、内含子、3′ UTR和重复序列甲基化水平较高,启动子和5′ UTR的甲基化程度较低。

|
将每个基因的各个功能区域分别等分成20个单位长度,再计算每个单位长度的甲基化水平求平均值绘制成折线图。横坐标代表不同的基因组功能元件,纵坐标为不同序列环境下各个功能元件的甲基化水平 Each element of each gene was divided into 20 bins and the average of all bins in each gene element was obtained. The abscissa represent different gene elements, and the ordinate represent methylation levels of elements in different sequence contexts 图 2 甲基化水平在不同基因组功能元件上的分布 Fig. 2 Methylation levels of various functional regions of gene |
DMR分析显示,与产蛋前期相比,产蛋高峰期卢氏绿壳蛋鸡全基因组中有670个DMRs,其中,405个超甲基化区域,265个低甲基化区域(图 3A)。重复序列及内含子区域在DMRs占主导地位,超甲基化区域数量分别为300及174个,低甲基化区域数量分别为122及125个(图 3B)。通过对670个DMRs分析,共鉴定到356个有注释的差异甲基化基因(DMGs),其中超甲基化211个,低甲基化153个(同一基因不同功能元件可能发生不同的甲基化)(图 3C)。
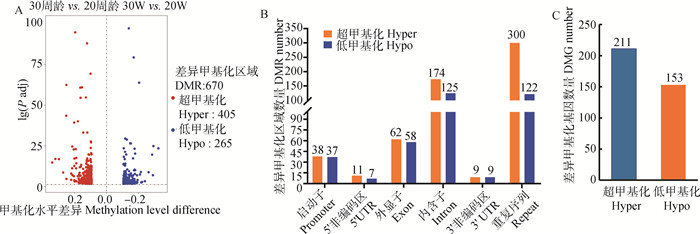
|
A.不同比较组DMR火山图:横坐标代表甲基化水平差异,纵坐标代表DMR差异显著性;B.DMR结构注释:横坐标代表不同功能区域,纵坐标代表处在不同功能区域DMR所占的数量;C.全基因组甲基化测序确定的差异甲基化基因(DMGs)的数量 A. DMR volcano plot for different comparison groups: The abscissa represent differences in methylation levels, and the ordinate represent significant differences in DMR; B. DMR structure notes: The abscissa represent different functional areas, and the ordinate represent the number of DMR in different functional areas; C. The number of differentially methylated genes (DMGs) as determined by WGBS 图 3 差异甲基化区域分析 Fig. 3 The analysis of differentially methylated regions |
DMGs的GO富集分析表明,超甲基化DMGs主要富集于发育的正向调控及细胞形态改变的调控等过程(P < 0.05),而低甲基化DMGs主要富集于胚胎消化道形态的发生、间充质细胞增殖的正向调控(P < 0.05, 图 4A、B)。KEGG通路分析表明,超甲基化DMGs主要参与VEGF信号、肌动蛋白细胞骨架的调控、黏着斑及间隙连接等生物学通路(P < 0.05),而低甲基化DMGs主要参与淀粉和蔗糖代谢及Wnt信号等生物学通路(P < 0.05,图 5A、B)。

|
纵坐标代表GO条目;横坐标代表基因数量。柱子长度代表通路的基因数,柱子越长,基因数越多。不同颜色代表P值,颜色越红,P值越小 The ordinate represent the GO terms, the abscissa represent the number of genes. The length of column represent the number of genes contained in the particular class, the longer the column indicate the more genes. Different colors represent P value, the redder the color, the smaller the P value 图 4 GO富集分类柱状图 Fig. 4 GO enrichment classification histogram |

|
纵坐标代表KEGG通路;横坐标代表富集因子。圈的大小代表通路的基因数,圈越大,基因数越多。不同颜色代表-lg (P-value),颜色越红,-lg (P-value) 越大 The ordinate represents the KEGG pathways and the abscissa represents the rich factor. The size of circles represent the number of genes contained in the particular class, the larger the circle indicate the more genes. The different colors represent the -lg (P-value), the redder the circles indicate the higher -lg (P-value) 图 5 KEGG富集气泡图 Fig. 5 KEGG enrichment bubble chart |
2.5.1 鸡肝全基因组甲基化与基因表达关联分析 为了研究DNA不同区域甲基化状态与基因表达之间的关系,将甲基化数据与本课题组先前发表的肝转录组数据进行整合分析[12]。
结果表明,两个时期鸡肝启动子区甲基化水平均与基因表达水平显著负相关(产蛋前期:r=-0.56,P=5.14×10-14;产蛋高峰期:r=-0.48,P=1.12×10-10),基因体(gene body)甲基化水平也与基因表达水平显著负相关(产蛋初期:r=-0.78,P=3.55×10-26;产蛋高峰期:r=-0.68,P =6.89×10-20);随着甲基化水平降低(5th group~ unmethylated),基因表达水平也会相应升高(图 6A、B)。进一步研究发现,CG序列环境下,甲基化水平越低基因表达水平越高,特别是甲基化发生在启动子、外显子及5′ UTR区域时,但在3′ UTR和内含子处甲基化水平与基因表达水平无明显关系。在CHG和CHH序列环境下也观察到相似结果(图 6C、D)。产蛋前期与产蛋高峰期结果一致。
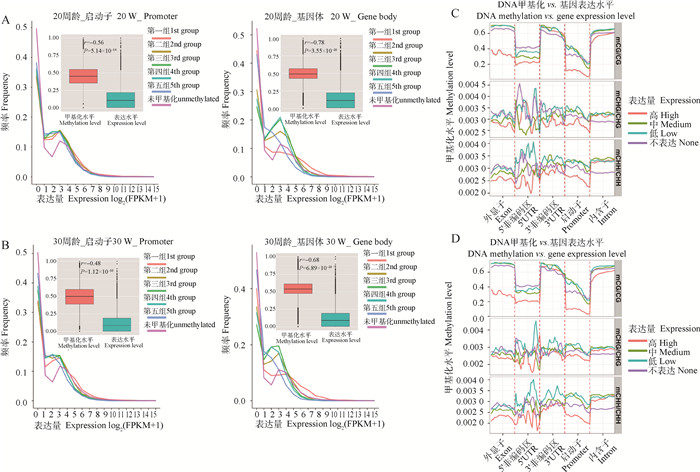
|
A.20周龄甲基化水平组别下的基因表达水平分布图,基因甲基化水平和表达水平分布盒形图;C.20周龄在不同区域的甲基化水平分布图,横坐标代表基因功能区域(gene body);纵坐标代表不同序列环境下C位点的甲基化水平,以不同颜色区分不同的表达水平分组;B、D.30周龄同A、C A. Distribution of gene expression levels under methylation level groups in 20 weeks, the box plots of gene methylation levels and expression level distribution; C. Methylation level distribution of whole genome in 20 weeks, the abscissa represents the functional region of the gene (gene body), the ordinate represent methylation levels of C sites in different sequence environments, with different colors to distinguish different expression level groups; B, D. The same as the A, C in 30 weeks, respectively 图 6 甲基化水平与基因表达水平分析 Fig. 6 Analysis of methylation levels and gene expression levels |
2.5.2 DMG与DEG共有基因筛选 前期对卢氏鸡产蛋前期和产蛋高峰期肝转录组的研究表明,与产蛋前期相比,产蛋高峰期有2 567个差异表达基因(DEGs),包括1 082个上调基因及1 485个下调基因[12]。
将全基因组甲基化数据中超甲基化基因及低甲基化基因分别与转录组数据中下调表达、上调表达基因进行整合分析发现,交集中共有8个超甲基化表达下调的DMGs(DEGs),9个低甲基化表达上调的DMGs(DEGs)(图 7A、B)。其中4个与肝脂质代谢相关基因(即RASD1、HAO1、UBE2O、MSRB3)的表达水平受到甲基化调控(表 2)。

|
图 7 肝DMGs和DEGs分析 Fig. 7 Analysis of DMGs and DEGs in liver |
|
|
表 2 鸡肝异甲基化基因与差异表达共有基因信息 Table 2 The genes information shared by DMGs and DEGs in chicken liver |
DNA甲基化作为重要的表观遗传修饰之一,在哺乳动物肝发育[18]及脂质代谢调控[19-20]等生理过程的研究较多,但在家禽,尤其是在蛋鸡不同生理时期肝全基因组甲基化方面的研究还未见报道。本研究利用WGBS对卢氏绿壳母鸡不同生理阶段肝全基因组甲基化进行检测,结合转录组分析,比较分析了产蛋前期与产蛋高峰期卢氏绿壳母鸡肝全基因组甲基化差异及其与转录之间的联系,进一步加深了对产蛋期鸡肝代谢的表观调控机制的理解。
总体而言,产蛋前期和产蛋高峰期全基因组胞嘧啶(C)甲基化(mC)率分别为4.08%和3.82%,两生理阶段甲基化水平差异不显著。在CG、CHH和CHG 3种序列环境中,CG序列环境中C甲基化率最高。同时,鸡基因组中甲基化水平在TSS之前急剧下降,而向基因体内区域上升,这与前人研究结果一致[21-22]。此外,本研究还发现,鸡基因组中>75%的DMR位于内含子及重复序列区域,而位于基因5′UTR、3′UTR和启动子区域的DMR较少,这与在人上的研究结果相似[23]。内含子区域及重复序列区域甲基化的生物学功能和生理意义还有待进一步研究,以阐明鸡肝代谢复杂的表观调控机制。
此外,机体可通过改变基因组甲基化状态调控基因的表达,以适应不同的生理和病理条件[24-25]。本研究中,虽然两个不同生理时期鸡基因组DMGs均富集在与生长发育及维持细胞基本功能有关的生物学过程,但超甲基化DMGs主要参与VEGF信号、肌动蛋白的细胞骨架调节、黏着斑及间隙连接等生物学通路,而低甲基化DMGs主要参与淀粉和蔗糖代谢及Wnt等生物学通路。而淀粉和蔗糖代谢与肝脂质代谢密切相关[26],R-spndin(RSPO)能够与LGR4/5跨膜受体相互作用激活Wnt/β-catenin通路信号转导,进而促进肝发育以及参与代谢过程[27]。可见,在不同生理阶段,可通过基因组中甲基化水平的变化调控肝中相关基因的功能,以满足生理需要。
众所周知,DNA甲基化的主要遗传效应是基因沉默,但甲基化位点在基因中的位置与其效应密切相关[28-29]。本研究通过对DNA甲基化图谱与RNA-seq数据之间的相关性进行分析,确定了功能区域相关DNA甲基化与基因表达的关系,启动子及基因体区域甲基化与基因表达显著负相关,这与已有的研究结果一致[30-31]。基因启动子区域的甲基化可抑制基因转录过程[32],基因体甲基化可能通过延缓RNA聚合酶Ⅱ的速度导致转录延伸的速率延缓来影响基因表达水平[33]。也有研究表明,人脑细胞[34]及山羊皮肤组织[35]中基因体甲基化与基因表达的变化无关,甲基化和表达的调控是一个复杂的过程,受多种因素影响[36]。本研究发现,产蛋高峰期与产蛋前期母鸡肝转录组相比,有2 567个DEGs,包括1 082个上调基因及1 485个下调基因,但只有17个DEGs受到甲基化水平变化的影响。可见,基因组甲基化在产蛋高峰期母鸡肝转录组显著变化过程中并不起主要作用。
本研究发现,4个与脂质代谢相关基因的表达受到基因组甲基化的调控。其中HAO1是肝特有的一种具有高脂肪酸氧化酶活性的过氧化物酶,该基因的异常表达会导致肝代谢紊乱,进而促使脂肪肝的形成[37]。而UBE2O基因敲除小鼠对胰岛素敏感性增加,血浆总胆固醇(TC)、游离脂肪酸(FFA)和低密度脂蛋白(LDL)水平降低[38];同时,该基因还参与调控细胞脂质的形成[39]。MSRB3参与多种生命活动的调节,其作用之一就是参与骨化和脂肪沉积,直接调节动物的生长和发育[40]。RASD1受雌激素调控参与各种细胞过程(如细胞生长、增殖和分化等),参与鸡生殖系统发育,对鸡生产性能有重要作用[41]。这些基因在卢氏鸡产蛋高峰期的甲基化水平均显著降低,且表达水平均显著上调,从而促进产蛋高峰期肝脂质代谢。
4 结论本研究利用WGBS测序技术构建了卢氏绿壳蛋鸡肝全基因组甲基化图谱,并结合转录组数据分析,通过比较产蛋前期(20周龄)及产蛋高峰期(30周龄)的DMRs,鉴定到356个差异甲基化基因(DMGs),阐述了DNA甲基化在基因表达方面的调控作用,筛选到4个受甲基化调控参与肝脂质代谢的基因。该研究结果有助于了解全基因组甲基化在蛋鸡不同生理阶段肝代谢的表观遗传机制。
| [1] |
FLANAGAN J M, WILD L. An epigenetic role for noncoding RNAs and intragenic DNA methylation[J]. Genome Biol, 2007, 8(6): 307. DOI:10.1186/gb-2007-8-6-307 |
| [2] |
ROBERTSON K D. DNA methylation and human disease[J]. Nat Rev Genet, 2005, 6(8): 597-610. |
| [3] |
LI S Z, TOLLEFSBOL T O. DNA methylation methods: global DNA methylation and methylomic analyses[J]. Methods, 2021, 187: 28-43. DOI:10.1016/j.ymeth.2020.10.002 |
| [4] |
YOON A R, TAMMEN S A, PARK S, et al. Genome-wide hepatic DNA methylation changes in high-fat diet-induced obese mice[J]. Nutr Res Pract, 2017, 11(2): 105-113. DOI:10.4162/nrp.2017.11.2.105 |
| [5] |
CORDERO P, GÓMEZ-ÚRIZ A M, CAMPION J, et al. Dietary supplementation with methyl donors reduces fatty liver and modifies the fatty acid synthase DNA methylation profile in rats fed an obesogenic diet[J]. Genes Nutr, 2013, 8(1): 105-113. DOI:10.1007/s12263-012-0300-z |
| [6] |
BRULPORT A, VAIMAN D, BOU-MAROUN E, et al. Hepatic transcriptome and DNA methylation patterns following perinatal and chronic BPS exposure in male mice[J]. BMC Genomics, 2020, 21(1): 881. DOI:10.1186/s12864-020-07294-3 |
| [7] |
STRAHL B D, ALLIS C D. The language of covalent histone modifications[J]. Nature, 2000, 403(6765): 41-45. DOI:10.1038/47412 |
| [8] |
GUTTMAN M, AMIT I, GARBER M, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals[J]. Nature, 2009, 458(7235): 223-227. DOI:10.1038/nature07672 |
| [9] |
COSMA M P, TANAKA T, NASMYTH K. Ordered recruitment of transcription and chromatin remodeling factors to a cell cycle-and developmentally regulated promoter[J]. Cell, 1999, 97(3): 299-311. DOI:10.1016/S0092-8674(00)80740-0 |
| [10] |
HERMIER D. Lipoprotein metabolism and fattening in poultry[J]. J Nutr, 1997, 127(5): 805S-808S. DOI:10.1093/jn/127.5.805S |
| [11] |
TIAN W H, WANG Z, YUE Y X, et al. miR-34a-5p increases hepatic triglycerides and total cholesterol levels by regulating ACSL1 protein expression in laying hens[J]. Int J Mol Sci, 2019, 20(18): 4420. |
| [12] |
LI H, WANG T A, XU C L, et al. Transcriptome profile of liver at different physiological stages reveals potential mode for lipid metabolism in laying hens[J]. BMC Genomics, 2015, 16: 763. DOI:10.1186/s12864-015-1943-0 |
| [13] |
TIAN W H, ZHENG H, YANG L Y, et al. Dynamic expression profile, regulatory mechanism and correlation with egg-laying performance of ACSF gene family in chicken (Gallus gallus)[J]. Sci Rep, 2018, 8(1): 8457. DOI:10.1038/s41598-018-26903-6 |
| [14] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: A flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [15] |
WANG Z X, WU X P, WU Z X, et al. Genome-wide DNA methylation comparison between Brassica napus genic male sterile line and restorer line[J]. Int J Mol Sci, 2018, 19(9): 2689. DOI:10.3390/ijms19092689 |
| [16] |
YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq: Accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI:10.1186/gb-2010-11-2-r14 |
| [17] |
MAO X Z, CAI T, OLYARCHUK J G, et al. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary[J]. Bioinformatics, 2005, 21(19): 3787-3793. DOI:10.1093/bioinformatics/bti430 |
| [18] |
WANG S, ZHANG C, HASSON D, et al. Epigenetic compensation promotes liver regeneration[J]. Dev Cell, 2019, 50(1): 43-56. DOI:10.1016/j.devcel.2019.05.034 |
| [19] |
GARCÍA-CALZÓN S, PERFILYEV A, DE MELLO V D, et al. Sex differences in the methylome and transcriptome of the human liver and circulating HDL-cholesterol levels[J]. J Clin Endocrinol Metab, 2018, 103(12): 4395-4408. DOI:10.1210/jc.2018-00423 |
| [20] |
CASTELLANO-CASTILLO D, MORENO-INDIAS I, SANCHEZ-ALCOHOLADO L, et al. Altered adipose tissue DNA methylation status in metabolic syndrome: Relationships between global DNA methylation and specific methylation at adipogenic, lipid metabolism and inflammatory candidate genes and metabolic variables[J]. J Clin Med, 2019, 8(1): 87. DOI:10.3390/jcm8010087 |
| [21] |
ZHANG M, LI D H, ZHAI Y H, et al. The landscape of DNA methylation associated with the transcriptomic network of intramuscular adipocytes generates insight into intramuscular fat deposition in chicken[J]. Front Cell Dev Biol, 2020, 8: 206. DOI:10.3389/fcell.2020.00206 |
| [22] |
LI Q H, LI N, HU X X, et al. Genome-wide mapping of DNA methylation in chicken[J]. PLoS One, 2011, 6(5): e19428. DOI:10.1371/journal.pone.0019428 |
| [23] |
GUO W L, CHUNG W Y, QIAN M P, et al. Characterizing the strand-specific distribution of non-CpG methylation in human pluripotent cells[J]. Nucleic Acids Res, 2014, 42(5): 3009-3016. DOI:10.1093/nar/gkt1306 |
| [24] |
ZHANG M, WANG Y L, WANG Y, et al. Integrative analysis of DNA methylation and gene expression to determine specific diagnostic biomarkers and prognostic biomarkers of breast cancer[J]. Front Cell Dev Biol, 2020, 8: 529386. DOI:10.3389/fcell.2020.529386 |
| [25] |
ZHANG X, SUN W J, HE L J, et al. Global DNA methylation pattern involved in the modulation of differentiation potential of adipogenic and myogenic precursors in skeletal muscle of pigs[J]. Stem Cell Res Ther, 2020, 11(1): 536. DOI:10.1186/s13287-020-02053-3 |
| [26] |
KRAWCZYŃSKA A, HERMAN A P, ANTUSHEVICH H, et al. Modifications of Western-type diet regarding protein, fat and sucrose levels as modulators of steroid metabolism and activity in liver[J]. J Steroid Biochem Mol Biol, 2017, 165: 331-341. DOI:10.1016/j.jsbmb.2016.07.012 |
| [27] |
BIRCHMEIER W. Orchestrating Wnt signalling for metabolic liver zonation[J]. Nat Cell Biol, 2016, 18(5): 463-465. DOI:10.1038/ncb3349 |
| [28] |
HYUN J, JUNG Y. DNA methylation in nonalcoholic fatty liver disease[J]. Int J Mol Sci, 2020, 21(21): 8138. DOI:10.3390/ijms21218138 |
| [29] |
GREENBERG M V C. Get out and stay out: New insights into DNA methylation reprogramming in mammals[J]. Front Cell Dev Biol, 2021, 8: 629068. DOI:10.3389/fcell.2020.629068 |
| [30] |
WANG G P, ZHONG Y Y, LIANG J C, et al. Upregulated expression of pyruvate kinase M2 mRNA predicts poor prognosis in lung adenocarcinoma[J]. PeerJ, 2020, 8: e8625. DOI:10.7717/peerj.8625 |
| [31] |
刘雨萌, 马艳艳, 姜海煦, 等. 鸡脂肪组织TCF21基因启动子区DNA甲基化与其表达的关系[J]. 畜牧兽医学报, 2021, 52(12): 3375-3389. LIU Y M, MA Y Y, JIANG H X, et al. The relationship between promoter region DNA methylation of TCF21 gene and its expression in chicken adipose tissue[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(12): 3375-3389. DOI:10.11843/j.issn.0366-6964.2021.012.006 (in Chinese) |
| [32] |
SUZUKI M M, BIRD A. DNA methylation landscapes: Provocative insights from epigenomics[J]. Nat Rev Genet, 2008, 9(6): 465-476. DOI:10.1038/nrg2341 |
| [33] |
ZILBERMAN D, GEHRING M, TRAN R K, et al. Genome-wide analysis of Arabidopsis thaliana DNA methylation uncovers an interdependence between methylation and transcription[J]. Nat Genet, 2007, 39(1): 61-69. DOI:10.1038/ng1929 |
| [34] |
ARAN D, TOPEROFF G, ROSENBERG M, et al. Replication timing-related and gene body-specific methylation of active human genes[J]. Hum Mol Genet, 2011, 20(4): 670-680. DOI:10.1093/hmg/ddq513 |
| [35] |
LI C, LI Y, ZHOU G X, et al. Whole-genome bisulfite sequencing of goat skins identifies signatures associated with hair cycling[J]. BMC Genomics, 2018, 19(1): 638. DOI:10.1186/s12864-018-5002-5 |
| [36] |
MOORE L D, LE T, FAN G P. DNA methylation and its basic function[J]. Neuropsychopharmacology, 2013, 38(1): 23-38. DOI:10.1038/npp.2012.112 |
| [37] |
TAN X D, LIU R R, XING S Y, et al. Genome-wide detection of key genes and epigenetic markers for chicken fatty liver[J]. Int J Mol Sci, 2020, 21(5): 1800. DOI:10.3390/ijms21051800 |
| [38] |
VILA I K, PARK M K, SETIJONO S R, et al. A muscle-specific UBE2O/AMPKα α 2 axis promotes insulin resistance and metabolic syndrome in obesity[J]. JCI Insight, 2019, 4(13): e128269. DOI:10.1172/jci.insight.128269 |
| [39] |
ZHANG X F, ZHANG J, BAUER A, et al. Fine-tuning BMP7 signalling in adipogenesis by UBE2O/E2-230K-mediated monoubiquitination of SMAD6[J]. EMBO J, 2013, 32(7): 996-1007. DOI:10.1038/emboj.2013.38 |
| [40] |
WU M L, LI S P, ZHANG G L, et al. Exploring insertions and deletions (indels) of MSRB3 gene and their association with growth traits in four Chinese indigenous cattle breeds[J]. Arch Anim Breed, 2019, 62(2): 465-475. DOI:10.5194/aab-62-465-2019 |
| [41] |
JEONG W, BAE H, LIM W, et al. RAS-related protein 1: An estrogen-responsive gene involved in development and molting-mediated regeneration of the female reproductive tract in chickens[J]. Animal, 2018, 12(8): 1594-1601. DOI:10.1017/S1751731117003226 |
(编辑 郭云雁)



