2. 扬州大学动物科学与技术学院,扬州 225001
2. College of Animal Science and Technology, Yangzhou University, Yangzhou 225001, China
染色体重排(chromosomal rearrangement)导致不同染色体间遗传物质的交换,造成生物体基因组内出现了大量的结构变异(structural variation, SV),这是产生遗传变异的主要原因之一。一般来说,SV是基因组中大于50个核苷酸的重排,且主要出现在基因组的非编码区域,通常不会改变成熟蛋白的结构[1]。尽管如此,SV的大小和位置可以决定转录调控元件的结合、mRNA剪接和加工、基因组折叠和高阶结构以及翻译的变化[1-2],从而影响某些物种的表型[3-4]。此外,SV与基因组的进化也有着密切的联系[5-6]。
早在上世纪二十年代,Bridges[7-8]就报道了果蝇(Drosophila melanogaster)的基因组中存在着大的染色体重排,且后续研究发现棒眼基因(Bar)的复制能够导致果蝇棒眼的形成。但是关于畜禽的SV研究直到上世纪五十年代才有所进展,当时Knudsen[9]在牛(Bos taurus)的基因组中检测到染色体易位,并发现其与公牛的生育能力降低有关。关于猪(Sus scrofa)基因组的SV,近年来基于全基因组扫描技术的进步已经开展了一些拷贝数变异(copy number variation, CNV)相关的研究[10-11]。Chen等[12-13]首次详细注解了猪基因组的RNA转座子,发现SINE转座子在猪基因组内贡献了大量的多态并开发了分子标记。Zhou等[14]则构建了猪基因组第一个综合数据库PigVar (http://www.biomedical.net/pigvar/),这个数据库汇集了来自287头猪及其亲属的SNP和211头猪的SV。
随着测序技术的发展和测序成本的降低以及SV检测方法不断的进步,SV在畜禽驯化、适应、生产、毛色、形态和疾病方面已被证实扮演了重要的角色。本文对SV的分类、形成机制、检测方法以及在猪的繁殖、肉质、生长、毛色、疾病、嗅觉和耳朵大小性状等方面的研究进行综述,并对未来的发展方向进行了展望和讨论。
1 SV分类一般来说,按照基因组中DNA量的变化进行分类,基因组中的SV可分为两种类型,其中一类是非平衡性的CNV,即基因组DNA发生量的改变,例如核苷酸的缺失(deletion, DEL)、插入(insertion, INS)、移动元素插入(mobile-element insertion, MEI)和重复(duplication, DUP);另一类是平衡性的重排,即基因组DNA仅发生顺序的改变,例如染色体的倒位(inversion, INV)和易位(translocation, TRA)[15](图 1)。目前,CNV作为SV的主要形式,更多的被定义为长度从一千碱基到数兆碱基不等的DNA片段[16-17]。

|
该示意图描述了与参考基因组比对时测试基因组中的缺失、插入、转座子插入、串联和散在的节段重复、倒位和易位。Ref.参考基因组 The schematic depicts deletions, novel sequence insertions, mobile-element insertions, tandem and interspersed segmental duplications, inversions and translocations in a test genome when compared with the reference genome. Ref. Reference genome 图 1 结构变异的类别[23] Fig. 1 Classification of structural variation[23] |
DEL是最常见的一种SV,指染色体上一段核苷酸序列的缺失,导致了碱基数目的减少。根据缺失位置的不同可分为中间DEL和末端DEL。
INS包括一般核苷酸片段的插入和MEI。一般的INS是指染色体中某一片段的插入,导致碱基数目的改变。通常来说,插入片段来源于基因组,同时会伴随着TRA的发生。MEI是指移动元素(即转座子)在基因组移动或转座的过程。转座子插入不同于一般核苷酸片段的插入,有研究已证实转座子插入不仅会造成DNA水平的结构变化,而且常常携带顺式调控元件的作用,例如启动子[18]、增强子[19]、隔离子[20]和沉默子[21]。
DUP一般包括串联重复(tandem duplications, TD) 和节段重复(segmental duplications, SD)两种类型,而基因区域发生重复被认为是基因组和系统进化的主要驱动力之一[22]。TD是指染色体中某一段核苷酸序列以前后相接的方式重复多次形成的重复序列,而SD是指某一段核苷酸序列通过染色体重排的方式增加多个拷贝,且拷贝散落在基因组的其它位置。
INV是指染色体上某一片段的序列发生180度的颠倒,导致碱基顺序的改变,但是该区段碱基的数目不会发生改变。根据发生的次数,又可分为只发生一次的简单INV和发生两次或以上的复杂INV。
TRA包括染色体内易位和染色体间易位,一般不会改变核苷酸序列的碱基数目。染色体内易位是指某段序列转移到同一染色体其它区域,能够改变染色体上的基因位置。染色体间易位是指染色体某一片段转移到另一条染色体的某一区域,包括两条染色体的序列之间互相易位和单向易位。
2 SV形成机制染色体重排主要由非等位基因同源重组(non-allelic homologous recombination, NAHR)、非同源末端连接(non-homologous end joining,NHEJ)、叉停顿和模板转换(fork stalling and template switching,FoSTeS)和转座子插入(mobile-element insertion, MEI)引起,以上几种机制可以解释基因组内大多数SV的形成(图 2)。

|
A.非等位基因同源重组(NAHR);B.复制叉延迟和模板转换(FoSTeS);C.非同源末端连接(NHEJ);D.转座子插入(MEI) A. Non-allelic homologous recombination(NAHR); B. Fork stalling and template switching(FoSTeS); C. Non-homologous end joining(NHEJ); D. Mobile-element insertion(MEI) 图 2 结构变异的主要形成机制[3] Fig. 2 The main formation mechanism of structural variation[3] |
NAHR通常发生在有丝分裂和减数分裂时期,是指两个非等位基因的染色体同源片段发生交叉重组, 可导致染色体片段的INS、DEL和INV等。有报道称,NAHR一般发生在低拷贝重复序列(low copy repeats, LCR)之间,并与DNA复制密切相关[24]。
2.2 FoSTeSFoSTeS通常发生在DNA复制的S期。当DNA复制受阻或暂停时,滞后链与聚合酶解离,导致其从模板上脱落,并与另一个区域结合重新开始合成DNA。由于复制起点的位置改变引起DEL或DUP的发生,可导致大片段的SV。需要注意的是,通过FoSTeS形成的SV很难与由微同源介导的断点诱导修复(microhomology-mediated break-induced replication, MMBIR) 生成的SV进行区分[25]。
2.3 NHEJNHEJ是一种DNA修复机制,经常参与DNA双链断裂(double-strand break,DSB)的修复[26],且在完成对DSB片段的末端修复后,在修复位点会留下了一个“疤痕”核苷酸序列[27]。NHEJ机制通常会导致DEL[28-29]和TRA[30]的发生。
2.4 MEI转座子的移动会引起染色体重排,导致大量的SV产生[31]。根据转座机制,转座子可以分为两大类:第I类转座子通过“复制-粘贴”的方式进行转座称为反转录转座子(retrotransposon)。反转录转座子又可以分成两大亚类:一类是具有长末端重复序列(long terminal repeat, LTR)的反转录转座子,主要是内源性逆转录病毒(endogenous retrovirus, ERV)。另一类是非LTR反转录转座子,包括长散在核元素(long interspersed nuclear element, LINE)和短散在核元素(short interspersed nuclear element, SINE);第II类转座子通过“剪切-粘贴”的方式进行转座,主要包括Tc1/mariner,hAT,PiggyBac,CACTA,P和PIF-Harbinger等家族。据报道,转座子占据哺乳动物基因组中的三分之一到一半左右[32],且在SV中扮演了极其重要的角色,比如人类基因组研究中超过40%的SV与转座子重叠[33]。
3 SV检测方法自本世纪起,随着高通量基因组扫描技术的进步,微阵列比较基因组杂交(array-based comparative genomic hybridization, aCGH)、单核苷酸多态性(single nucleotide polymorphisms, SNP)芯片和下一代测序(next-generation sequencing, NGS)成为了SV检测的主要手段。其中,aCGH则是最早应用于CNV检测的技术,几乎参与了所有畜禽的CNV研究。随着测序成本的降低,NGS则成为了检测SV的主要手段,且与aCGH相比具有分辨率高、可检测倒位和易位以及一些新序列插入的优势。利用NGS数据与参考基因组比较来调查表型调控背后的机制一直是SV研究的主要方式,但是由于较短的读取长度,基因组重复序列区域尚未得到充分研究,而且短读的SV检测灵敏度低(30%~70%)和错误发现率高(85%)的特点有一定的应用限制[34]。近年来,一些新兴基因组技术的出现,包括基于关联分子策略的Linked-Reads测序、Strand-seq测序和染色质构象捕捉技术(Hi-C)与基于单分子策略的PacBio单分子实时测序、Oxford Nanopore测序和光学图谱(BioNano)技术(图 3),显著提升了SV检测能力和整体敏感性[35]。使用三代测序(thirds-generation sequencing, TGS)生成的长reads可以弥补NGS技术的不足,因其具备足够跨越SV的长度,并且在重复区域中具有更高的可映射性[36]。在50~1 000 bp范围内,TGS可以检测到比NGS多3~4倍的SV[37-38]。目前,基于长reads的SV检测已开发了npInv[39]、svim[40]和pbsv[41]等多个工具。另一方面,将三代测序和多种基因组技术结合已经成为基因组组装的黄金标准[42],利用新的基因组技术获得高质量基因组进行SV的研究也已在多个领域开展[43-46]。
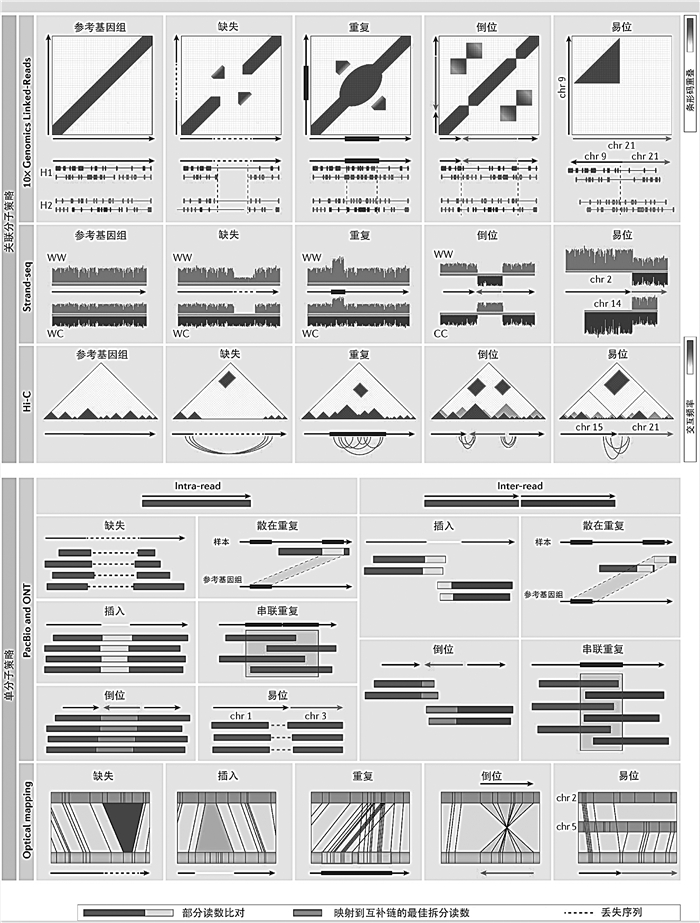
|
新兴技术在检测结构变异(SV) 的方式上各不相同:1) 10×Genomics Linked-Reads基于基因组位点之间的条形码重叠检测SV;2) Strand-seq根据读取深度或映射方向的突然变化来确定SV;3) 高通量染色体构象捕获(Hi-C) 通过寻找基因组位点之间异常高频的接触来检测SV;4) 单分子测序方法(PacBio、ONT和Optical mapping)基于可能涉及一个(读取内)或多个(读取间)读取的不一致映射特征推断SV。chr. 染色体; H1. 单倍型1; H2. 单倍型2; WW. 沃森-沃森;WC. 沃森-克里克;ONT. 牛津纳米孔技术; PacBio. 太平洋生物科学 Emerging technologies vary in how they detect structural variations (SVs): 1) 10×Genomics Linked-Reads detect SVs based on barcode overlap between genomic loci; 2) Strand-seq determines SVs based on read depth or sudden changes in mapping orientation; 3) High-throughput chromosome conformation capture (Hi-C) detects SVs by looking for unusually high-frequency contacts between genomic loci; 4) Single-molecule sequencing (PacBio, ONT and Optical mapping) methods infer SVs based on discordant mapping signatures that can involve one (intra-read) or many (inter-read) reads. chr. Chromosome; H1. Haplotype 1; H2. Haplotype 2; WW. Watson-Watson; WC. Watson-Crick; ONT. Oxford nanopore technologies; PacBio. Pacific Biosciences 图 3 单分子和关联分子策略中的结构变异特征[35] Fig. 3 Structural variation signatures in single-molecule and connected-molecule strategies[35] |
染色体重排导致的SV与一些繁殖性状息息相关,比如TRA是导致家猪生殖功能障碍的主要原因之一,据估计,50%的低产公猪是携带者[47]。另外,CNV已被证实可能在塑造繁殖性状方面发挥作用[48-49]。
在繁殖性能方面,梅山猪一直是世界高产猪的代表品种,对梅山猪的SV检测有望解释其高产背后的机制。Zhou等[45]使用NGS、Pacbio、GemCode Linked、BioNano和Hi-C技术,首次完成了梅山猪的高质量基因组组装。与杜洛克猪基因组比较发现,在BMPR1B的内含子区域检测到3个DEL和6个INS,并推测可能对繁殖性能有影响。另外,藏猪与梅山猪和大白猪相比,IGF2R的内含子发生了DEL,推测其影响了生长速度。Du等[50]发现,梅山猪基因组中IDO2基因中140 bp的DEL以及CYP2J2和PLA2G4A基因中的DUP与繁殖性能有关。Zhao等[51]对包括梅山猪在内的中国猪种和欧洲猪种进行了大规模的SV检测,构建了一个包括56 930个推定SV的单核苷酸分辨率图谱,并确定在X染色体上一个35 Mb区域中的35个SV相关基因对繁殖能力具有重要意义,从而使中国和欧洲起源品种的进化速度存在显著差异,研究也为评估系统发育关系提供了新的证据。产仔数可以最直观地反映繁殖能力的高低,Liu等[52]通过比较产仔数大(XL)的香猪和产仔数小(XS)的香猪基因组,总共鉴定了28 040个SV。其中4 637个SV仅存在于XL组,4 119个SV是XS组特有的,分别影响1 697和1 582个基因。通过分析发现,这些SV及其相关基因参与了多种分子功能,并与猪的繁殖性状相关。此外,还在香猪基因组中鉴定了14个与窝产仔数相关的候选基因。Ran等[53]使用SNP阵列发现了一个在香猪6号染色体上包含MTHFSD基因的496 kb CNVR,调查了其在545头猪中的分布,证实MTHFSD CNV与卵巢中MTHFSD基因转录水平呈正相关,并且改变了产仔数性状。Liu等[54]发现,SINE插入多态性存在于猪群中PDIA4基因的第一个内含子区域,与香猪的窝产仔数性状有关,其中SINE+/+基因型相对于SINE+/-基因型和SINE-/-基因型具有更大的窝产仔数,可作为香猪窝产仔数的候选DNA标记。此外,Zheng等[55]报道了中西方猪之间AHR基因中的差异CNV可能与总产仔数和产活仔数有关。Wang等[56]利用80K SNP芯片对大白猪进行CNV分析,结果表明CNVR61和CNVR283两个区域与窝产仔数相关。
4.2 SV与肉质和生长性状肌内脂肪含量(intramuscular fat, IMF)是评价猪肉品质的重要性状,Wang等[57-58]利用CNV进行GWAS分析,发现CNV可能通过其基因剂量影响NTN1基因的表达,而NTN1的表达可能通过AMPK途径影响肌肉细胞的增殖和分化,最终影响IMF含量。随后,Wang等[59]又发现,CNVR可能通过调节PELP1基因的可变剪接来改变PELP1蛋白的结构,从而影响猪的IMF。
在一些生长性状研究中。Yoshidomi等[60]证明,猪胰淀粉酶CNV多态对30~100 kg猪生长过程中的平均日采食量、总采食量和饲料转化率有显著影响。Revilla等[61]则报道了一个CNVR含有GPAT2,该基因在磷脂和三酰基甘油的生物合成中起关键作用,表明这种CNVR可能有助于脂肪酸组成和生长性状的遗传变异。Qiu等[62]证明,CNVR与杜洛克猪的平均日增重(ADG)、百公斤体重日龄(AGE)和背膘厚度(BFT)有关,表明这些CNVR可能在调节猪生长和脂肪沉积方面发挥多效作用。此外,通过组装高质量宁乡猪基因组并与杜洛克猪基因组比较,Ma等[63]鉴定了大量的SV,并在MYL4基因的第一个内含子鉴定了281 bp的DEL,其高的转录水平可能促进了皮下脂肪沉积。
4.3 SV与毛色和色素沉着毛色是区分不同驯化物种的重要特征之一,在猪的不断驯化和选择中,已经产生了大量不同品种和种群特征的毛色和图案[64]。关于InDel和错义突变引起的毛色变化已进行了不少研究,然而仅有KIT原癌基因酪氨酸激酶受体(kit proto-oncogene, receptor tyrosine kinase, KIT)是由大的结构变化引起。正常的KIT信号传导是神经嵴衍生的黑素细胞发育和存活所必需的[65]。在已报道的研究中,在KIT基因座中SV的不同形式会导致3种表型:显性白色(完全白色)、斑块状白色(部分白色)和带状白色(前肢白色)。相较于野生型猪种,显性白色(长白猪和大白猪) KIT基因座包含整个KIT基因座的450 kb DUP1和至少一个KIT拷贝中外显子跳跃的剪接突变,然后是位于KIT上游100 kb的4.3 kb DUP2和下游100 kb的23 kb DUP3以及DUP3内部的一个4.3 kb DUP4;斑块状白色(皮特兰猪)只包括相对于野生型KIT基因座的450 kb DUP1,而带状白色(汉普夏猪)只包括位于KIT上游100 kb的4.3 kb DUP2和下游100 kb的23 kb DUP3以及DUP3内部的一个4.3 kb DUP4 [66](图 4)。关于KIT基因座DUP的机制,之前有研究证实是由基因座两侧的LINE转座子的NAHR引起的[67]。随后,Wu等[68]利用杜洛克×(长白×大白)杂交猪(DLY)对KIT基因座进行研究发现了影响毛色变化的新等位基因,不仅加深了对猪颜色表型分子机制的认识,而且为筛选有色DLY的大白猪和长白猪的KIT I N/IN纯合子建立了一种简单而准确的方法。Qin等[69]则利用CRISPR/Cas9基因编辑技术成功的校正了KIT基因座450 kb的结构突变,为产生具有正常KIT基因座的基因编辑猪奠定了基础。上海的浦东白猪是除荣昌猪外唯一具有白毛色的中国本土猪,Huang等[70]发现浦东白猪携带与大白猪相同的KIT基因型,并在PARG-MARCHF8基因座与大白猪共享单倍型,这表明浦东白猪中的欧洲猪谱系起源于大白猪。
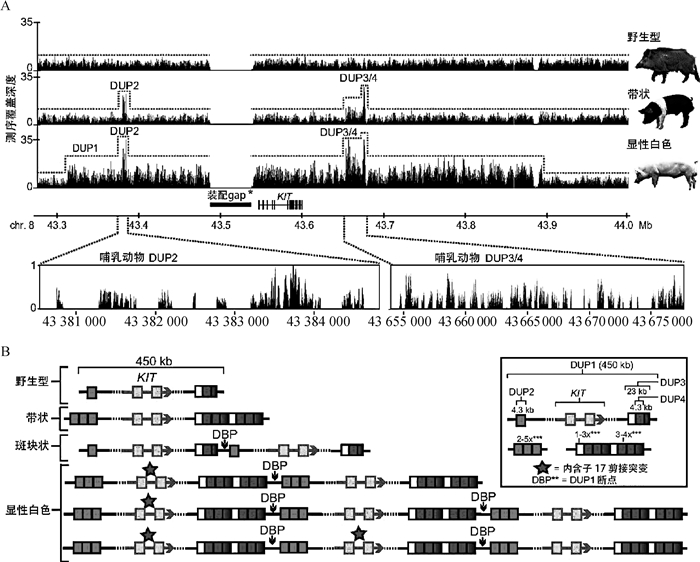
|
A. 野猪(野生型)、汉普夏猪(带状)和长白猪(显性白色)基因库的测序覆盖深度表明,除了前面描述的DUP1之外还存在3个重复; B. 猪KIT等位基因的示意图。4个重复的剪接位点突变和可变拷贝数共同创造了巨大的单倍型多样性 A. Sequencing depth of coverage for the wild boar(wild-type), Hampshire(belt) and Landrace(dominant white) pools demonstrating the presence of 3 duplications in addition to the previously described DUP1; B. Schematic of porcine KIT alleles. Together, the splice site mutation and the variable copy numbers of the 4 duplications create great haplotype diversity 图 4 猪KIT 基因座白斑等位基因的进化[67] Fig. 4 Evolution of white spotting alleles at the KIT locus in pigs[67] |
在其它研究中,Xu等[71]利用公共全基因组测序数据鉴定的SNP、InDel和CNV对57个品种的469头猪进行了全基因组关联分析(GWAS),确定了RAPGEF2、ADAMTS9、ATXN7、CSPG4、PEAK1和PDGFC为与毛色相关的候选基因。其中,CSPG4和PEAK1位于显著CNV附近的候选区域,而RAPGEF2、ADAMTS9、ATXN7和PDGFC基因位于显著InDel附近的候选区域。而据先前的报道,PDGFC基因与巴马猪两端黑色素沉积有关[72]。
4.4 SV与疾病SV的发生通常会导致基因的破坏和新基因的融合,引起一些疾病的发生,例如公猪TEX14基因2号外显子序列中51 bp的INS导致了公猪不育[73],母猪INHA基因283 bp的INS会引发更高的滤泡囊肿风险[74],还有仔猪的腭裂与TRA有关[75]。
巴马小型猪(BM)是巴马香猪(BX)的近交系,目前已成为研究人类疾病的重要模型动物。Zhang等[46]完成了第一个染色体水平巴马猪基因组组装,并通过与杜洛克猪基因组的比较发现ANHAK、ADGRF5/GPR116和ATP10D三个调节肥胖的基因外显子区域内出现了DEL和DUP,可能与巴马猪更难承受糖尿病的压力有关。Fowler等[76]使用上百分位数和下百分位数的动物的估计育种值数据进行GWAS分析,推定了几个CNVR,它们包含的基因(MCHR1、PPARα、SLC5A1和SLC5A4)可能与肥胖有关。Dong等[77]对3个本土小型猪种进行研究,利用SNP分型获得了一些与心血管疾病和阿尔茨海默病等疾病相关的人类直系同源基因的CNVR。Long等[78]则发现一些罕见的CNV与猪的脐疝有关。Stachowiak等[79]在SOX9基因下游(约500 kb)检测到一个新的CNVR多态性与睾丸性发育障碍表型相关。Hay等[80]发现,CNV可能与特定群体对猪繁殖与呼吸综合征(PRRS)的反应有关。此外,Wang等[81]在TLR6发现5个插入多态,表明192 bp ERV插入等位基因可能对免疫反应有益,可作为抗病育种的候选标记。
4.5 SV与其它性状猪具有最大的功能性嗅觉受体(olfactory receptors, ORs)基因库,这反映了嗅觉在这种动物中的重要性[82-83]。ORs显然是非常容易发生CNV的基因[46, 84-85]。据报道,猪基因组中ORs的获得和损失加剧是导致种群之间发生CNV的主要原因[86]。CNV在ORs的巨大进化潜力可以为猪提供必要的可塑性来进化新的觅食策略,为快速适应不同环境发挥了作用。另一项报道发现,ORs的CNV在猪的物种形成过程中维持生殖隔离方面可能也发挥了重要作用[87]。
耳朵的大小和类型是区分猪品种的重要特征,有研究报道了一个38.7 kb的CNV改变了miR-584-5p的表达,导致了MSRB3基因的mRNA翻译受到抑制,从而影响了猪耳朵的大小[88]。另一项研究发现,一个5号染色体的CNV在所有亚欧野生品种以及商业猪种中都是两个拷贝,但其在大多数亚欧家养品种中拷贝数大于3个。特别是八眉猪和民猪的所有个体中都有超过5个拷贝,这表明CNV的拷贝数增加可能导致了更大且不直立的耳朵[72]。
Liu等[89]报道了香猪一种全身皱纹、皮肤增厚和毛发稀疏的新皮肤表型。通过NGS的方法,将无皱褶香猪和欧洲猪比较确定了59个基因中的65个候选SV可能与该性状有关。
5 展望核苷酸长度大于50个碱基的SV广泛的分布在基因组中的内含子和非编码调控区内,它们产生的多态性可以解释大部分物种之间的表型差异。过往的研究已经证明,SV与一些动物经济性状、疾病和生物机制的调控密切相关。因此,准确的解析SV与表型变异之间的联系蕴藏着巨大的科研价值和经济效益。尽管近些年科学家在该领域取得了一定的进展,但是鉴于SV的类型和大小的多变性以及新基因组技术的检测偏差,SV的准确检测仍然是一个问题。继续开发新的检测技术并解决多平台间造成的差异,对每个物种构建较为一致的SV图谱可能是未来研究的趋势。另外,应用三代测序以及一些新的基因组技术组装高质量的基因组进行研究有望解决低质量的基因组组装导致的假阳性问题。利用SV进行分子标记开发也有巨大的潜力,尤其是转座子,因其具有的调控能力和广泛的分布非常适合作为分子标记的开发。而对于已开发标记的物种,解决大批量高效分型的方法,甚至基于SV开发芯片可能是另一个发展方向。总之,结合不断发展的新兴SV检测技术,并将其应用到动物育种领域研究中,不仅可以解析一些重要性状的调控机制,还可以提高基因组选择的准确性,提升育种效率和水平。
| [1] |
ROSES A D, AKKARI P A, CHIBA-FALEK O, et al. Structural variants can be more informative for disease diagnostics, prognostics and translation than current SNP mapping and exon sequencing[J]. Expert Opin Drug Metab Toxicol, 2016, 12(2): 135-147. DOI:10.1517/17425255.2016.1133586 |
| [2] |
CHIANG C, SCOTT A J, DAVIS J R, et al. The impact of structural variation on human gene expression[J]. Nat Genet, 2017, 49(5): 692-699. DOI:10.1038/ng.3834 |
| [3] |
BICKHART D M, LIU G E. The challenges and importance of structural variation detection in livestock[J]. Front Genet, 2014, 5: 37. |
| [4] |
ZHANG F, GU W L, HURLES M E, et al. Copy number variation in human health, disease, and evolution[J]. Annu Rev Genomics Hum Genet, 2009, 10: 451-481. DOI:10.1146/annurev.genom.9.081307.164217 |
| [5] |
PETROV D A. Mutational equilibrium model of genome size evolution[J]. Theor Popul Biol, 2002, 61(4): 531-544. DOI:10.1006/tpbi.2002.1605 |
| [6] |
OLSON M V. When less is more: Gene loss as an engine of evolutionary change[J]. Am J Hum Genet, 1999, 64(1): 18-23. DOI:10.1086/302219 |
| [7] |
BRIDGES C B. Triploid intersexes in drosophila melanogaster[J]. Science, 1921, 54(1394): 252-254. DOI:10.1126/science.54.1394.252 |
| [8] |
BRIDGES C B. The Bar "gene" a duplication[J]. Science, 1936, 83(2148): 210-211. DOI:10.1126/science.83.2148.210 |
| [9] |
KNUDSEN O. Studies on spermiocytogenesis in the bull[J]. Obstet Gynecol Surv, 1959, 14(3): 404-405. DOI:10.1097/00006254-195906000-00035 |
| [10] |
经珍珠, 秦盼盼, 陈冰洁, 等. 拷贝数变异在畜禽中的研究进展[J]. 中国畜牧兽医, 2021, 48(7): 2512-2522. JING Z Z, QIN P P, CHEN B J, et al. Research progress of copy number variation in livestock and poultry[J]. China Animal Husbandry & Veterinary Medicine, 2021, 48(7): 2512-2522. DOI:10.16431/j.cnki.1671-7236.2021.07.027 (in Chinese) |
| [11] |
金美林, 卢增奎, 李青, 等. 畜禽拷贝数变异研究进展[J]. 农业生物技术学报, 2019, 27(10): 1840-1848. JIN M L, LU Z K, LI Q, et al. Research progress on copy number variation of livestock and poultry[J]. Journal of Agricultural Biotechnology, 2019, 27(10): 1840-1848. (in Chinese) |
| [12] |
CHEN C, WANG W, WANG X Y, et al. Retrotransposons evolution and impact on lncRNA and protein coding genes in pigs[J]. Mob DNA, 2019, 10: 19. DOI:10.1186/s13100-019-0161-8 |
| [13] |
CHEN C, D'ALESSANDRO E, MURANI E, et al. SINE jumping contributes to large-scale polymorphisms in the pig genomes[J]. Mob DNA, 2021, 12(1): 17. DOI:10.1186/s13100-021-00246-y |
| [14] |
ZHOU Z Y, LI A M, OTECKO N O, et al. PigVar: A database of pig variations and positive selection signatures[J]. Database, 2017, 2017: bax048. |
| [15] |
FEUK L, CARSON A R, SCHERER S W. Structural variation in the human genome[J]. Nat Rev Genet, 2006, 7(2): 85-97. |
| [16] |
REDON R, ISHIKAWA S, FITCH K R, et al. Global variation in copy number in the human genome[J]. Nature, 2006, 444(7118): 444-454. DOI:10.1038/nature05329 |
| [17] |
STANKIEWICZ P, LUPSKI J R. Structural variation in the human genome and its role in disease[J]. Annu Rev Med, 2010, 61: 437-455. DOI:10.1146/annurev-med-100708-204735 |
| [18] |
LI J F, KANNAN M, TRIVETT A L, et al. An antisense promoter in mouse L1 retrotransposon open reading frame-1 initiates expression of diverse fusion transcripts and limits retrotransposition[J]. Nucleic Acids Res, 2014, 42(7): 4546-4562. DOI:10.1093/nar/gku091 |
| [19] |
DING M T, LIU Y H, LIAO X H, et al. Enhancer RNAs (eRNAs): new insights into gene transcription and disease treatment[J]. J Cancer, 2018, 9(13): 2334-2340. DOI:10.7150/jca.25829 |
| [20] |
ROMÁN A C, GONZÁLEZ-RICO F J, MOLTÓ E, et al. Dioxin receptor and SLUG transcription factors regulate the insulator activity of B1 SINE retrotransposons via an RNA polymerase switch[J]. Genome Res, 2011, 21(3): 422-432. DOI:10.1101/gr.111203.110 |
| [21] |
MASTRANGELO M F, WEINSTOCK K G, SHAFER B K, et al. Disruption of a silencer domain by a retrotransposon[J]. Genetics, 1992, 131(3): 519-529. DOI:10.1093/genetics/131.3.519 |
| [22] |
MOORE R C, PURUGGANAN M D. The early stages of duplicate gene evolution[J]. Proc Natl Acad Sci U S A, 2003, 100(26): 15682-15687. DOI:10.1073/pnas.2535513100 |
| [23] |
ALKAN C, COE B P, EICHLER E E. Genome structural variation discovery and genotyping[J]. Nat Rev Genet, 2011, 12(5): 363-376. DOI:10.1038/nrg2958 |
| [24] |
STANKIEWICZ P, LUPSKI J R. Molecular-evolutionary mechanisms for genomic disorders[J]. Curr Opin Genet Dev, 2002, 12(3): 312-319. DOI:10.1016/S0959-437X(02)00304-0 |
| [25] |
ZHANG F, KHAJAVI M, CONNOLLY A M, et al. The DNA replication FoSTeS/MMBIR mechanism can generate genomic, genic and exonic complex rearrangements in humans[J]. Nat Genet, 2009, 41(7): 849-853. DOI:10.1038/ng.399 |
| [26] |
VAN GENT D C, VAN DER BURG M. Non-homologous end-joining, a sticky affair[J]. Oncogene, 2007, 26(56): 7731-7740. DOI:10.1038/sj.onc.1210871 |
| [27] |
GU W L, ZHANG F, LUPSKI J R. Mechanisms for human genomic rearrangements[J]. Pathogenetics, 2008, 1(1): 4. DOI:10.1186/1755-8417-1-4 |
| [28] |
INOUE K, OSAKA H, THURSTON V C, et al. Genomic rearrangements resulting in PLP1 deletion occur by nonhomologous end joining and cause different dysmyelinating phenotypes in males and females[J]. Am J Hum Genet, 2002, 71(4): 838-853. DOI:10.1086/342728 |
| [29] |
SHAW C J, LUPSKI J R. Non-recurrent 17p11.2 deletions are generated by homologous and non-homologous mechanisms[J]. Hum Genet, 2005, 116(1-2): 1-7. DOI:10.1007/s00439-004-1204-9 |
| [30] |
LIEBER M R, GU J F, LU H H, et al. Nonhomologous DNA end joining (NHEJ) and chromosomal translocations in humans[M]//NASHEUER H P. Genome Stability and Human Diseases. Dordrecht: Springer, 2010: 279-296.
|
| [31] |
LÖNNIG W E, SAEDLER H. Chromosome rearrangements and transposable elements[J]. Annu Rev Genet, 2002, 36: 389-410. DOI:10.1146/annurev.genet.36.040202.092802 |
| [32] |
PLATT Ⅱ R N, VANDEWEGE M W, RAY D A. Mammalian transposable elements and their impacts on genome evolution[J]. Chromosome Res, 2018, 26(1-2): 25-43. DOI:10.1007/s10577-017-9570-z |
| [33] |
QUAN C, LI Y F, LIU X Y, et al. Characterization of structural variation in Tibetans reveals new evidence of high-altitude adaptation and introgression[J]. Genome Biol, 2021, 22(1): 159. DOI:10.1186/s13059-021-02382-3 |
| [34] |
SEDLAZECK F J, LEE H, DARBY C A, et al. Piercing the dark matter: bioinformatics of long-range sequencing and mapping[J]. Nat Rev Genet, 2018, 19(6): 329-346. DOI:10.1038/s41576-018-0003-4 |
| [35] |
HO S S, URBAN A E, MILLS R E. Structural variation in the sequencing era[J]. Nat Rev Genet, 2020, 21(3): 171-189. DOI:10.1038/s41576-019-0180-9 |
| [36] |
DE COSTER W, VAN BROECKHOVEN C. Newest methods for detecting structural variations[J]. Trends Biotechnol, 2019, 37(9): 973-982. DOI:10.1016/j.tibtech.2019.02.003 |
| [37] |
AUDANO P A, SULOVARI A, GRAVES-LINDSAY T A, et al. Characterizing the major structural variant alleles of the human genome[J]. Cell, 2019, 176(3): 663-675. DOI:10.1016/j.cell.2018.12.019 |
| [38] |
CHAISSON M J P, SANDERS A D, ZHAO X F, et al. Multi-platform discovery of haplotype-resolved structural variation in human genomes[J]. Nat Commun, 2019, 10(1): 1784. DOI:10.1038/s41467-018-08148-z |
| [39] |
SHAO H J, GANESAMOORTHY D, DUARTE T, et al. npInv: accurate detection and genotyping of inversions using long read sub-alignment[J]. BMC Bioinformatics, 2018, 19(1): 261. DOI:10.1186/s12859-018-2252-9 |
| [40] |
HELLER D, VINGRON M. SVIM: structural variant identification using mapped long reads[J]. Bioinformatics, 2019, 35(17): 2907-2915. DOI:10.1093/bioinformatics/btz041 |
| [41] |
PACIFIC BIOSCIENCES. Pbsv. Github, 2018.
|
| [42] |
SEO J S, RHIE A, KIM J, et al. De novo assembly and phasing of a Korean human genome[J]. Nature, 2016, 538(7624): 243-247. DOI:10.1038/nature20098 |
| [43] |
LOW W Y, TEARLE R, LIU R J, et al. Haplotype-resolved genomes provide insights into structural variation and gene content in Angus and Brahman cattle[J]. Nat Commun, 2020, 11(1): 2071. DOI:10.1038/s41467-020-15848-y |
| [44] |
ZHU F, YIN Z T, WANG Z, et al. Three chromosome-level duck genome assemblies provide insights into genomic variation during domestication[J]. Nat Commun, 2021, 12(1): 5932. DOI:10.1038/s41467-021-26272-1 |
| [45] |
ZHOU R, LI S T, YAO W Y, et al. The Meishan pig genome reveals structural variation-mediated gene expression and phenotypic divergence underlying Asian pig domestication[J]. Mol Ecol Resour, 2021, 21(6): 2077-2092. DOI:10.1111/1755-0998.13396 |
| [46] |
ZHANG L, HUANG Y M, WANG M, et al. Development and genome sequencing of a laboratory-inbred miniature pig facilitates study of human diabetic disease[J]. iScience, 2019, 19: 162-176. DOI:10.1016/j.isci.2019.07.025 |
| [47] |
GUSTAVSSON I. Chromosomes of the pig[J]. Adv Vet Sci Comp Med, 1990, 34: 73-107. |
| [48] |
KEEL B N, NONNEMAN D J, LINDHOLM-PERRY A K, et al. A survey of copy number variation in the porcine genome detected from whole-genome sequence[J]. Front Genet, 2019, 10: 737. DOI:10.3389/fgene.2019.00737 |
| [49] |
WU Q, ZHOU Y, WANG Y, et al. Whole-genome sequencing reveals breed-differential CNVs between Tongcheng and Large White pigs[J]. Anim Genet, 2020, 51(6): 940-944. DOI:10.1111/age.12993 |
| [50] |
DU H, ZHENG X R, ZHAO Q Q, et al. Analysis of structural variants reveal novel selective regions in the genome of Meishan pigs by whole genome sequencing[J]. Front Genet, 2021, 12: 550676. DOI:10.3389/fgene.2021.550676 |
| [51] |
ZHAO P J, LI J H, KANG H M, et al. Structural variant detection by large-scale sequencing reveals new evolutionary evidence on breed divergence between Chinese and European pigs[J]. Sci Rep, 2016, 6: 18501. DOI:10.1038/srep18501 |
| [52] |
LIU C, RAN X Q, YU C Y, et al. Whole-genome analysis of structural variations between Xiang pigs with larger litter sizes and those with smaller litter sizes[J]. Genomics, 2019, 111(3): 310-319. DOI:10.1016/j.ygeno.2018.02.005 |
| [53] |
RAN X Q, PAN H, HUANG S H, et al. Copy number variations of MTHFSD gene across pig breeds and its association with litter size traits in Chinese indigenous Xiang pig[J]. J Anim Physiol Anim Nutr (Berl), 2018, 102(5): 1320-1327. DOI:10.1111/jpn.12922 |
| [54] |
LIU C, RAN X Q, NIU X, et al. Insertion of 275-bp SINE into first intron of PDIA4 gene is associated with litter size in Xiang pigs[J]. Anim Reprod Sci, 2018, 195: 16-23. DOI:10.1016/j.anireprosci.2018.04.079 |
| [55] |
ZHENG X R, ZHAO P J, YANG K J, et al. CNV analysis of Meishan pig by next-generation sequencing and effects of AHR gene CNV on pig reproductive traits[J]. J Anim Sci Biotechnol, 2020, 11: 42. DOI:10.1186/s40104-020-00442-5 |
| [56] |
WANG Y, ZHANG T R, WANG C D. Detection and analysis of genome-wide copy number variation in the pig genome using an 80 K SNP Beadchip[J]. J Anim Breed Genet, 2020, 137(2): 166-176. DOI:10.1111/jbg.12435 |
| [57] |
WANG L G, XU L Y, LIU X, et al. Copy number variation-based genome wide association study reveals additional variants contributing to meat quality in Swine[J]. Sci Rep, 2015, 5: 12535. DOI:10.1038/srep12535 |
| [58] |
WANG L G, ZHAO L L, ZHANG L C, et al. NTN1 affects porcine intramuscular fat content by affecting the expression of myogenic regulatory factors[J]. Animals, 2019, 9(9): 609. DOI:10.3390/ani9090609 |
| [59] |
WANG L G, ZHANG T, LI N, et al. Copy number variations contribute to intramuscular fat content differences by affecting the expression of PELP1 alternative splices in pig[J]. Animals, 2022, 12. |
| [60] |
YOSHIDOMI T, TANAKA K, TAKIZAWA T, et al. Copy number variation of amylase alpha 2B gene is associated with feed efficiency traits in Large White pigs[J]. Czech J Anim Sci, 2021, 66: 495-503. |
| [61] |
REVILLA M, PUIG-OLIVERAS A, CASTELLÓ A, et al. A global analysis of CNVs in swine using whole genome sequence data and association analysis with fatty acid composition and growth traits[J]. PLoS One, 2017, 12(5): e0177014. DOI:10.1371/journal.pone.0177014 |
| [62] |
QIU Y B, DING R R, ZHUANG Z W, et al. Genome-wide detection of CNV regions and their potential association with growth and fatness traits in Duroc pigs[J]. BMC Genomics, 2021, 22(1): 332. DOI:10.1186/s12864-021-07654-7 |
| [63] |
MA H M, JIANG J, HE J, et al. Long-read assembly of the Chinese indigenous Ningxiang pig genome and identification of genetic variations in fat metabolism among different breeds[J]. Mol Ecol Resour, 2022, 22(4): 1508-1520. DOI:10.1111/1755-0998.13550 |
| [64] |
SANFORD E. Pigs: A handbook to the breeds of the world[J]. Can Vet J, 1994, 35(11): 719-720. |
| [65] |
CHABOT B, STEPHENSON D A, CHAPMAN V M, et al. The proto-oncogene c-kit encoding a transmembrane tyrosine kinase receptor maps to the mouse W locus[J]. Nature, 1988, 335(6185): 88-89. DOI:10.1038/335088a0 |
| [66] |
RUBIN C J, MEGENS H J, BARRIO A M, et al. Strong signatures of selection in the domestic pig genome[J]. Proc Natl Acad Sci U S A, 2012, 109(48): 19529-19536. DOI:10.1073/pnas.1217149109 |
| [67] |
GIUFFRA E, TÖRNSTEN A, MARKLUND S, et al. A large duplication associated with dominant white color in pigs originated by homologous recombination between LINE elements flanking KIT[J]. Mamm Genome, 2002, 13(10): 569-577. DOI:10.1007/s00335-002-2184-5 |
| [68] |
WU Z P, DENG Z, HUANG M, et al. Whole-genome resequencing identifies KIT new alleles that affect coat color phenotypes in pigs[J]. Front Genet, 2019, 10: 218. DOI:10.3389/fgene.2019.00218 |
| [69] |
QIN K, LIANG X Y, SUN G J, et al. Highly efficient correction of structural mutations of 450 kb KIT locus in kidney cells of Yorkshire pig by CRISPR/Cas9[J]. BMC Mol Cell Biol, 2019, 20(1): 4. DOI:10.1186/s12860-019-0184-5 |
| [70] |
HUANG M, ZHANG H, WU Z P, et al. Whole-genome resequencing reveals genetic structure and introgression in Pudong White pigs[J]. Animal, 2021, 15(10): 100354. DOI:10.1016/j.animal.2021.100354 |
| [71] |
XU J Y, FU Y H, HU Y, et al. Whole genome variants across 57 pig breeds enable comprehensive identification of genetic signatures that underlie breed features[J]. J Anim Sci Biotechnol, 2020, 11(1): 115. DOI:10.1186/s40104-020-00520-8 |
| [72] |
YANG Y, ADEOLA A C, XIE H B, et al. Genomic and transcriptomic analyses reveal selection of genes for puberty in Bama Xiang pigs[J]. Zool Res, 2018, 39(6): 424-430. DOI:10.24272/j.issn.2095-8137.2018.068 |
| [73] |
SIRONEN A, UIMARI P, VENHORANTA H, et al. An exonic insertion within Tex14 gene causes spermatogenic arrest in pigs[J]. BMC Genomics, 2011, 12: 591. DOI:10.1186/1471-2164-12-591 |
| [74] |
LI W H, CHEN S X, LI H J, et al. A new insertion/deletion fragment polymorphism of inhibin-α gene associated with follicular cysts in Large White sows[J]. J Vet Med Sci, 2016, 78(3): 473-476. DOI:10.1292/jvms.14-0489 |
| [75] |
GRAHOFER A, LETKO A, HÄFLIGER I M, et al. Chromosomal imbalance in pigs showing a syndromic form of cleft palate[J]. BMC Genomics, 2019, 20(1): 349. DOI:10.1186/s12864-019-5711-4 |
| [76] |
FOWLER K E, PONG-WONG R, BAUER J, et al. Genome wide analysis reveals single nucleotide polymorphisms associated with fatness and putative novel copy number variants in three pig breeds[J]. BMC Genomics, 2013, 14: 784. DOI:10.1186/1471-2164-14-784 |
| [77] |
DONG K, PU Y, YAO N, et al. Copy number variation detection using SNP genotyping arrays in three Chinese pig breeds[J]. Anim Genet, 2015, 46(2): 101-109. DOI:10.1111/age.12247 |
| [78] |
LONG Y, SU Y, AI H S, et al. A genome-wide association study of copy number variations with umbilical hernia in swine[J]. Anim Genet, 2016, 47(3): 298-305. DOI:10.1111/age.12402 |
| [79] |
STACHOWIAK M, SZCZERBAL I, NOWACKA-WOSZUK J, et al. Polymorphisms in the SOX9 region and testicular disorder of sex development (38, XX; SRY-negative) in pigs[J]. Livest Sci, 2017, 203: 48-53. DOI:10.1016/j.livsci.2017.07.002 |
| [80] |
HAY E H A, CHOI I, XU L Y, et al. CNV analysis of host responses to porcine reproductive and respiratory syndrome virus infection[J]. J Genomics, 2017, 5: 58-63. DOI:10.7150/jgen.20358 |
| [81] |
WANG X Y, CHEN Z X, MURANI E, et al. A 192 bp ERV fragment insertion in the first intron of porcine TLR6 may act as an enhancer associated with the increased expressions of TLR6 and TLR1[J]. Mob DNA, 2021, 12(1): 20. DOI:10.1186/s13100-021-00248-w |
| [82] |
NGUYEN D T, LEE K, CHOI H, et al. The complete swine olfactory subgenome: Expansion of the olfactory gene repertoire in the pig genome[J]. BMC Genomics, 2012, 13: 584. DOI:10.1186/1471-2164-13-584 |
| [83] |
GROENEN M A M, ARCHIBALD A L, UENISHI H, et al. Analyses of pig genomes provide insight into porcine demography and evolution[J]. Nature, 2012, 491(7424): 393-398. DOI:10.1038/nature11622 |
| [84] |
WANG C B, CHEN H, WANG X P, et al. Identification of copy number variations using high density whole-genome single nucleotide polymorphism markers in Chinese Dongxiang spotted pigs[J]. Asian-Australas J Anim Sci, 2019, 32(12): 1809-1815. DOI:10.5713/ajas.18.0696 |
| [85] |
ZHANG L, HUANG Y M, SI J L, et al. Comprehensive inbred variation discovery in Bama pigs using de novo assemblies[J]. Gene, 2018, 679: 81-89. DOI:10.1016/j.gene.2018.08.051 |
| [86] |
PAUDEL Y, MADSEN O, MEGENS H J, et al. Evolutionary dynamics of copy number variation in pig genomes in the context of adaptation and domestication[J]. BMC Genomics, 2013, 14: 449. DOI:10.1186/1471-2164-14-449 |
| [87] |
PAUDEL Y, MADSEN O, MEGENS H J, et al. Copy number variation in the speciation of pigs: A possible prominent role for olfactory receptors[J]. BMC Genomics, 2015, 16(1): 330. DOI:10.1186/s12864-015-1449-9 |
| [88] |
CHEN C Y, LIU C L, XIONG X W, et al. Copy number variation in the MSRB3 gene enlarges porcine ear size through a mechanism involving miR-584-5p[J]. Genet Sel Evol, 2018, 50(1): 72. DOI:10.1186/s12711-018-0442-6 |
| [89] |
LIU X L, HU F B, HUANG S H, et al. Detection of genomic structure variants associated with wrinkled skin in Xiang pig by next generation sequencing[J]. Aging, 2021, 13(22): 24710-24739. DOI:10.18632/aging.203711 |
(编辑 郭云雁)



