近几十年来,抗生素在畜牧业中的不合理使用已导致细菌耐药性产生和畜产品抗生素残留问题,这些耐药菌不仅造成了难以治疗的畜禽疾病,还能通过动物相关食品传播到人类,严重威胁人类健康[1],当下新型抗生素的研发速度相比于耐药性细菌产生和变异的速度已趋于缓慢。早在2006年, 欧盟就实行了“饲料全面禁抗”,而在2019年7月10日,中华人民共和国农业农村部发布了第194号公告,公布了“全面禁止促生长类药物饲料添加剂”的决议[2],因此,寻找绿色、安全、高效的抗生素替代品已经成为当前迫切需要解决的问题。抗菌肽(antimicrobial peptides,AMPs)是宿主先天性免疫防御系统产生的一类抵抗外界病原体感染的小分子多肽,具有广谱抗菌、抗病毒、抗癌活性以及免疫调节功能,还可促进上皮伤口愈合,用于人类疾病的治疗[3-4]。根据当前抗菌肽数据库(Antimicrobial Peptide Database,APD)的最新结果显示,目前,已经发现了超过3 000种具有多样序列和结构的天然AMPs,在微生物、植物、无脊椎动物、以及更复杂的两栖动物和哺乳动物等多物种中广泛存在[5],其主要的物理破膜杀菌机制使病原体不易对其产生耐药性突变,因此有望成为新一代抗生素替代物。
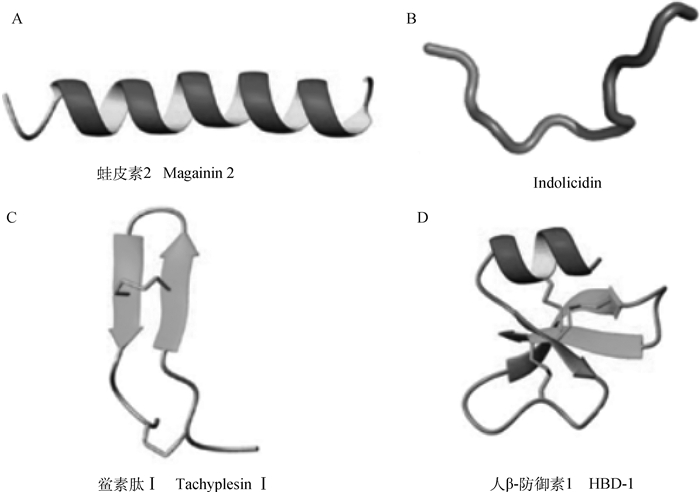
抗菌肽的分类依据多样,例如:根据物种来源、合成机制或分子靶标等[6],但较常见的是基于结构的差异将它们进行划分。如图 1所示,根据二级结构的类型,抗菌肽通常被分为4大类,分别是α-螺旋肽;不含α-螺旋和β-折叠成分但通常富含特定氨基酸(如Gly、Arg、Trp或Pro)的线性延伸结构;由一个或多个二硫键稳定的β-折叠肽;同时包含α-螺旋和β-折叠结构的肽[7]。α-螺旋目前是AMPs中研究的最为深入和广泛的一类二级结构,但已有研究表明某些α-螺旋肽提取工艺复杂、细胞毒性高以及抗菌效力参差不齐[8-9],限制了它们的应用。而β-折叠肽依靠保守的半胱氨酸残基之间的分子内二硫键维持三维结构,在相同条件下这类肽具有更加稳定的二级构象甚至更强的抗酶解能力[10]。有研究表明β-折叠肽与具有相同电荷和疏水性的α-螺旋肽相比,具有相近的抗菌活性和更优异的细胞选择性[11],而且,当稳定β-发卡或β-折叠结构抗菌肽中的二硫键被破坏时,所形成的线状多肽仍能保持其抗菌特性[12]。由于β-折叠AMPs所含的复杂二硫键合成困难且成本昂贵,其研究并不广泛,因此β-折叠抗菌肽的研发对于未来AMPs的应用转化十分关键。
|
A. α-螺旋结构;B.线性延伸结构;C. β-折叠结构;D. 同时包含α-螺旋和β-折叠元素 A. α-helical structure; B. Linear extension structure; C. β-sheet structure; D. Contain both α-helical and β-sheet elements 图 1 天然抗菌肽的常见结构类型[13-14] Fig. 1 Common structural classes of naturally occurring AMPs[13-14] |
本文介绍了β-折叠抗菌肽的天然来源、分子设计及主要抑菌机理,同时对其应用的局限性以及相应的策略进行了综述,全方位描述了β-折叠抗菌肽的研究进展,为该类肽的研发与利用提供参考。
1 天然源β-折叠抗菌肽抗菌肽来源广泛,作为非特异性天然防御系统的重要组成部分,是机体抵御病原微生物入侵的第一道防线。这些广泛来源的抗菌肽通常有着不同的结构,β-折叠就被包括其中。根据来源,可以将β-折叠抗菌肽分为哺乳动物源、昆虫源、植物源以及微生物源等大类,在表 1中列举了较为常见的几种天然β-折叠抗菌肽及其主要特征。
|
|
表 1 不同来源的几种常见β-折叠抗菌肽 Table 1 Several common β-sheet antimicrobial peptides from different sources |
基于半胱氨酸的含量和结构特征,可将β-折叠抗菌肽分为两类,分别是β-发卡抗菌肽和α-defensins肽[7]。β-发卡是反平行β-折叠结构的最简单形式,由两个β-链和单个二硫键组成的环状区域定义[15]。β-发卡抗菌肽尺寸小且生物活性广泛,在实际应用方面受到研究人员更多关注,但天然β-发卡抗菌肽并不丰富,已知的β-发卡型抗菌肽家族不仅在结构和生物学功能上有显著差异,在生物合成途径上也有显著差异[16]。Defensins肽跨越多个结构类别,α-defensins是一个亚组,包括三条反向平行的β-侧链,侧链以“trans”排列与二硫键相连(即β链末端的两个二硫键指向相反的方向,并连接两个不同的侧链)[17]。α-defensins被认为是通过基因复制而进化的,存在于大多数哺乳动物中,具有广谱抗细菌、真菌和病毒的活性[18]。这些肽含有保守的盐桥结构,有研究表明,盐桥对于多肽的正常折叠和蛋白水解抗性是必需的,但对抗菌活性而言不是必需的[19]。
1.1 哺乳动物源哺乳动物源抗菌肽已经广泛在动物皮肤、肠道、免疫系统、神经系统和泌尿系统等多种生理系统和上皮组织表面分离鉴定,主要包括Cathelicidins、Defensins和Histatins 3大家族[30],研究最广泛的为Defensins类抗菌肽。APD是一个集抗菌肽命名、分类、信息搜索、预测、设计和统计等多功能于一体的抗菌肽数据分析系统,截至目前APD数据库共收录有26种被明确鉴定为β-折叠结构的哺乳动物源AMPs,其中牛乳铁蛋白Lfcin B和猪源Protegrin 1的研究较为深入。
1.2 昆虫源昆虫免疫防御仅依赖于先天免疫,当微生物突破身体的第一道防线时,昆虫的先天免疫反应就会被激活,快速产生大量的AMPs,使昆虫迅速消灭入侵病原体[31]。昆虫源β-折叠抗菌肽具有分子量小、热稳定性强、水溶性好、无免疫原性等特点,不仅对细菌和真菌有广谱抗菌活性,对病毒、原虫及癌细胞也有杀伤作用[32]。目前, 研究较为深入的昆虫源β-折叠抗菌肽是拥有广谱抗菌活性的Thanatin[21],此外还有从长臂天牛体内分离出的拥有抗真菌功能的Alo-3[33]。昆虫作为世界上最大的生物种群,种类繁多,分布广泛,因此昆虫源β-折叠抗菌肽还具有巨大的发掘潜力。
1.3 植物源植物中存在多种具有药理作用的分子蛋白,β-折叠抗菌肽就是其中之一。当各种病原体入侵时,多种β-折叠抗菌肽(如从紫花苜蓿中分离出的NCR169)能够通过干扰细胞壁或膜脂的生物合成发挥抗菌活性[34]。此外,植物源β-折叠抗菌肽富含保守的半胱氨酸残基,拥有极高的稳定性且能通过改变非半胱氨酸残基的序列从而识别不同的靶点,预示着它们作为抗菌剂的巨大发展潜力[35]。
1.4 微生物源微生物源β-折叠抗菌肽是由微生物自身分泌的用来保护自己并杀灭或抑制其他细菌、真菌的小分子多肽,其主要来源于特定细菌,通常比抗生素稳定,可以承受高温和极端pH,但在自然和有机环境中的生物半衰期较短,主要通过作用于靶细胞膜来发挥作用[36]。迄今APD共收录了15种来自细菌的微生物源β-折叠抗菌肽,在食品保鲜和微生物感染治疗等领域有着广泛的应用前景。
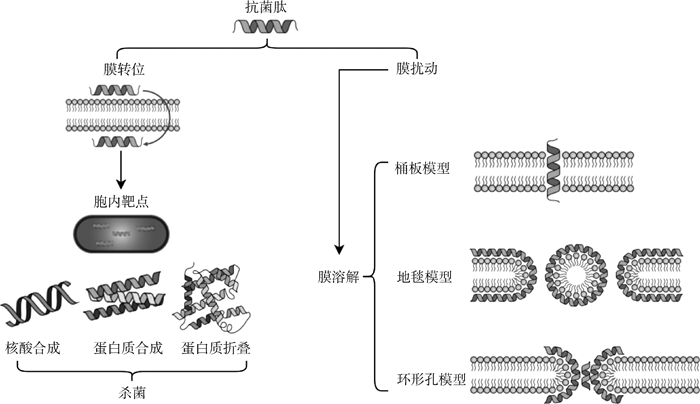
2 β-折叠抗菌肽抗细菌机理大部分β-折叠抗菌肽可以直接作用于细菌细胞膜,但有一些抗菌肽除了引起细胞膜损伤之外,还被证实可以通过细胞膜转运并结合到细胞内的目标来杀死细菌,产生一系列复杂的胞内活动[37]。如图 2所示,根据作用位点的不同,一般β-折叠抗菌肽的抑菌机理可以分为两类,分别是肽与细菌细胞膜相互作用以及肽与胞内靶点相互作用。
2.1 肽与细菌细胞膜相互作用β-折叠抗菌肽与细胞膜的作用过程,首先阳离子残基与靶细胞膜阴离子表面通过静电相互作用彼此靠近,然后肽的两亲结构域与磷脂双分子层之间产生疏水相互作用,抗菌肽插入细胞膜导致细胞内容物渗漏和死亡。目前,最为经典的抗菌肽膜作用模型有桶壁模型、地毯模型和环形孔模型,这几种作用机制模型并不是相互独立的,而在细菌细胞膜被破坏过程中可能同时存在[39-40]。由于β-折叠抗菌肽主要通过与细菌细胞膜相互作用,过程中不涉及特定的靶标,这也解释了细菌难以产生耐药性的原因。Bellamy等[41]研究发现,Lactoferricin B能够与细胞膜上带负电荷的磷脂和脂多糖结合并增加膜的通透性,且伴随着溶液中盐离子所带正电荷的增加抑菌效果显著降低;Sinha团队[42]通过将Thanatin与DPC-d38胶束相互结合阐述了Thanatin导致质膜结构扰动的机制;Mani等[43]使用19F和1H自旋扩散核磁共振技术确定了Protegrin 1(PG-1)在阴离子膜和含胆固醇中性膜中的低聚物结构,验证了PG-1能诱导细菌细胞膜快速通透从而产生杀菌活性。
2.2 肽与胞内靶点相互作用起初人们认为β-折叠抗菌肽的跨膜机制是其作用的唯一模式,但是越来越多的研究提供证据表明仅出现膜通透性不足以导致细胞死亡,并由此发现了部分β-折叠抗菌肽存在细胞内靶点。例如通过抑制核酸的生物合成和代谢或抑制蛋白合成与折叠,进而抑制和杀死病原菌,此外还有些能抑制细胞分裂和细胞壁的生物合成[44]。Leeuw等[45]研究发现α-防御素类多肽HNP1可以通过与细胞壁前体脂质Ⅱ相互作用抑制肽聚糖的合成,从而影响细胞壁合成以及膜功能导致细菌死亡;在海洋类动物Arenicola marina的体腔细胞中分离出来的β-折叠类抗菌肽Arenicin-1[46-47]可以诱导细胞内ROS的增加发挥抗真菌活性,介导细胞的凋亡过程;β-发卡肽Polyphemusin I通过作用于细胞内与DNA和RNA相关的蛋白质靶标从而造成细胞死亡[48]。部分β-折叠多肽还可以与细胞穿透肽结合从而进入细菌细胞内,两种作用模式之间会表现出协同关系使细菌对于此类杀伤机制更难产生突变抗性[49]。
3 β-折叠抗菌肽的分子设计 3.1 天然源β-折叠抗菌肽的改造天然β-折叠抗菌肽由于具有二硫键维持三维结构,稳定性相对会更高,但是一些二硫键较少的天然β-发卡抗菌肽仍然会受到高浓度酶的快速降解,从而失去抑菌活性。有部分天然β-折叠抗菌肽拥有足够的蛋白酶抗性,但缺少强效的抗菌活性,所以当面临这一系列的矛盾时,天然源抗菌肽的改造是一种有效手段。研发出具有良好抗菌活性和细胞选择性的β-折叠抗菌肽,需要利用现有的生物工程技术对这些AMPs的抗菌活性、生物安全性和结构关系进行系统探究解析,近年来,各种优化策略和分子设计技术的进步为肽性能的合理改良和优化提供了参考。目前,该方向的研究已经取得较大进展,常见的改造方法包括氨基酸替换、肽链修饰、肽链杂合等。
β-折叠抗菌肽的功能及活性往往是由结构所决定的,研究发现影响这些因素的主要结构参数包括净电荷数、疏水性、两亲性、序列长度、氨基酸组成等。总体带正电荷使β-折叠抗菌肽能与微生物膜的负电荷成分产生静电相互作用,疏水基团可以帮助其更深入地渗透细菌膜,从而决定β-折叠抗菌肽的抗菌效力,两亲性构象也是肽具有膜活性不可或缺的,然而研究表明这些参数体现在构效关系上时会有各自的阈值,它们之间纵横交错、相互影响[50-51]。因此在改造中不能盲目追求单个参数的最佳完美,而是要综合考虑各个参数的影响,以期达到一个最优的平衡点,使肽的抗菌活性和生物安全性趋向于更加合理。
3.1.1 氨基酸替代 在自然界中,许多生物体能产生天然β-折叠抗菌肽,利用这些肽作为起始模板对序列进行修改,可以达到提高肽的抗菌活性和细胞选择性的目的。氨基酸替代主要是对肽模板添加、删除、替换一个或几个氨基酸残基。Benfield等[52]设计了一种新型肽,首先对Gomesin(GM)骨架进行环化得到一种GM类似物cGM,当浓度高于2 μmol·L-1时cGM会产生毒性并破坏癌细胞的细胞膜,后来用D-Arg取代了原本在它第4、10和18位的Arg残基从而合成了[R/r]cGM,在保留了杀菌功能的同时蛋白酶抗性明显提高,并且解决了毒性过大的问题,可作为靶向和抗癌药物的输送系统而不损害健康细胞。另外,Dong等[53]将Protegrin 1(PG-1)的β转角序列添加上几个简单的氨基酸重复序列,设计了一系列末端对称肽(XR)nH(RX)n(n=1、2;X代表I、F、W和P;H代表CRRRFC),并利用治疗指数(therapeutic index,TI),即最小溶血浓度MHC与最小抑菌浓度MIC几何平均值的比值,评估多肽的细胞选择性,其中,序列为(IR)2H(RI)2的β-折叠结构IR2具有最高的TI,显示出比PG-1更高的抗微生物效力,溶血活性和细胞毒性显著低于PG-1。在改造过程中需要注意的是,β-折叠抗菌肽是一种结构复杂的分子,合理地去除非必须性质的部分肽区域可以获得较短的活性基序,降低大规模生产成本和衍生物的毒性或免疫原性,但此方法需要控制在合理范围,因为氨基酸组成的轻微改变可能会改变整个几何构型甚至物理化学性质,进而会影响这些肽杀灭微生物的能力和对宿主细胞的毒性。
3.1.2 肽链修饰 β-折叠抗菌肽常见的肽链修饰方法主要有环化、聚乙二醇修饰、胆固醇修饰、末端区域修饰(乙酰化或酰胺化)等,这些肽链修饰策略的可行性已被广泛证实,不仅能够用于提高肽的活性、稳定性和溶解性,提供更长的半衰期,也可以调节分子内相互作用和疏水性甚至改变多肽呈现的二级结构[54-55]。Lei等[56]将人α-defensin 5(HD5)的末端进行了十四烷酸(肉豆蔻酸)修饰,结果发现合成的肉豆蔻酰化HD5在体外具有显著增强的广谱杀菌活性,并且稳定性明显提高;Imura等[57]将聚乙二醇连接到β-折叠肽Tachyplesin I的N端,发现多肽聚乙二醇化后膜通透性并没有改变,而且抗菌活性显著减弱,但细胞毒性显著降低;Chan等[58]采用固相化学合成法合成了环状β-折叠抗菌肽Gomesin,与天然Gomesin相比,环化提高了Gomesin的体外稳定性,增强了其对癌细胞的杀伤作用以及抗疟疾活性。相比于其他改造方法,肽链修饰策略能够赋予β-折叠抗菌肽更多本身所不具备的性能,甚至定向赋予某项特性,更加可控与便捷,是一种性价比很高的肽设计方法。
3.1.3 肽链杂合 肽链杂合是将不同优势肽或优势肽段通过某种方式组合成新的嵌合肽的一类方法[59],这种方法通常将有效但毒性相对较大的肽序列与活性较低但毒性较低的肽序列融合,同样是设计新型β-折叠肽的一种简单有效策略,研究表明,嵌合肽活性的增加归因于每种肽组分同时作用于细菌的独特机制[60]。为了优化具有较强细胞特异性和抗炎活性的抗菌肽,Liu等[61]设计了基于Progetrin 1、Bovine lactoferricin的双发卡杂合肽,发现合成的杂合肽LB-PG不仅对多种微生物表现出较高的选择性,细胞毒性和溶血活性变低,还能抑制LPS诱导的促炎细胞因子和趋化因子的表达。虽然研究表明了杂合肽比亲本肽更有效,但杂合后导致肽链长度增加从而带来的合成成本增加的问题也是不容忽视的,因此需要综合考虑多种因素的影响。
3.2 全新设计在抗菌肽几十年的发展过程中,为进一步降低抗菌肽的生产成本,研发活性高、序列短且组成简单的β-折叠抗菌肽,抗菌肽的全新设计成为了研究的热门,常见的方法有模板确立法和数据库过滤筛选技术。
3.2.1 基于模板的设计 经过几十年的发展,抗菌肽模板的研究已经越来越全面。模板序列可以通过比较已知的天然肽序列并筛选出对功能重要的参数获得,与传统的序列修饰方法相比,该方法的优点是其减少了为获得理想结果而需要合成的肽的数量,从而在保持序列信息的同时降低研发成本[62]。Shao等[63]使用一种预测算法设计了最简单的反平行β-发卡抗菌肽模板RWYXYZZRWYXY-NH2,由两条含有阳离子(X=Arg、Lys、His)和疏水残基(Y=Val、Ile、Leu、Phe)的非共价键β-侧链,通过刚性转向区域(ZZ=Pro-Gly、D-Pro-Gly、Asn-Gly)连接而成,研发出的WKF-PG(RWFKFPGRWFKF-NH2)和WRF-NG(RWFRFNGRWFRF-NH2)可以通过直接的抗菌效果和抑制机体炎症来治疗细菌感染。Wu等[64]设计了基于(X1Y1X2Y2)n为模板的一系列肽,发现两个β-折叠结构的水溶性多肽(IKIK)2-NH2和(IRIK)2-NH2对包括真菌在内的多种微生物表现出强杀灭作用,同时保持低毒并具有中和内毒素的能力,是真菌性角膜炎药物的理想候选者。
3.2.2 数据库过滤筛选 数据库过滤筛选技术是一种利用数据库对具有某种特定功能多肽的关键参数进行多步筛选,从而最终设计出具有目标活性、性能更优的全新肽的方法(图 3)[65]。与传统的设计方法相比,该方法可以在最短的时间内筛选出最优序列,不仅节省大量成本, 还可以用来预测或进一步增强多肽的抗菌活性[66]。随着近年来大数据和人工智能技术的发展,基因组、蛋白质组和功能信息的数据库已逐渐成为抗菌药物研发不可或缺的工具。Cherkasov等[67]迭代构建了两个由9个氨基酸组成的随机大型肽库,通过Artificial Neural Networks(一种强大的机器学习技术)随机预测超过10万条虚拟肽的活性,筛选出的肽比目前最先进的临床候选抗菌肽更加有效,并且能有效杀灭超级耐药细菌;Fjell等[68]使用遗传算法的启发式搜索方法生成候选肽序列,它在识别新型高抗菌能力多肽方面有很大提高。尽管这种方法被认为是有效的,但目前仍没有明确的研究使用该方法设计全新β-折叠抗菌肽。
4 β-折叠抗菌肽的应用策略抗生素一直是人类抗击致病菌最重要的武器,但由于无节制的使用导致细菌耐药问题愈发严重,每年都有数以万计的人和动物因耐药菌感染而死亡,因此世界上许多国家对抗生素的使用提出了严格限制。由于优良的生物学活性、低毒性、丰富的化学多样性和天然来源的可得性,抗菌肽成为抗生素的理想替代品。经过几十年的发展,抗菌肽已逐渐应用到畜牧、食品、化学、医药等许多行业[70]。为使β-折叠抗菌肽得到更加合理的应用,切实有效的策略是必不可少的,以下论述针对目前的研究热点,从现实角度出发,介绍了β-折叠抗菌肽能够在未来应用转化的两个候选方向,分别是肽与纳米载体的结合、肽与抗生素的协同应用。
4.1 肽与纳米载体的结合无论是天然还是人工合成的β-折叠抗菌肽,大多数都具有广谱抗菌活性。传统观点通常认为抗菌肽所拥有的抗菌谱越广,其应用范围就会越大,其实不然。当一些活性较强的β-折叠抗菌肽应用到机体尤其是被动物采食进入胃肠道之后,除了起到杀灭病原微生物的功效以外,还可能会不分青红皂白地杀死体内益生菌,造成机体菌群失调,甚至破坏健康微生物区系和免疫系统之间的动态平衡[71]。此外,当β-折叠抗菌肽进入体内后,尽管分子内二硫键的存在会使它们的稳定性有所提高,但仍然需要面临唾液、胃黏膜、血液和组织中存在的盐离子和蛋白酶等影响,使其生物半衰期降低。而且,β-折叠抗菌肽在体内的扩散率也有限,运输效率相对较差,致使大多数给药方法都需要高剂量[72]。所以如果有一种能够更加稳定地将肽靶向运输到体内特定病原体附近而又不会杀灭益生菌的方法,将会极大缓解这些问题,无疑会更好地加速β-折叠抗菌肽的应用进展。
近年来,传递和释放生物分子的纳米生物技术得到快速发展。目前,已有的肽递送纳米载体材料提供了一种有效方法,这些纳米载体材料往往尺寸小、比表面积大、靶向能力强、毒性低,它们通过包裹、持续释放和延长活性来稳定以及更好地保护β-折叠抗菌肽不受酶降解的影响[73],利用载体固有的靶向特点将β-折叠抗菌肽运送至特定目标以防止对有益菌造成杀伤。因此β-折叠抗菌肽结合纳米材料后在体内可能会具有长效的抗菌活性、更低的毒性和更高的稳定性。一些纳米颗粒(如纳米纤维),它们有很大的接触面,可以携带几个单层或多层抗菌肽并将其引导到特定的目标,是β-折叠抗菌肽制剂理想的输送系统[74]。此外,一些无机纳米材料(如金属纳米颗粒、磁性纳米颗粒)以及一些有机纳米材料(如脂质体纳米材料、聚合物纳米材料等),均可作为β-折叠抗菌肽良好的递送载体[75]。直径<10 nm的颗粒会通过肾迅速代谢排出体外,>70 nm的颗粒在体内滞留时间较长,最终会到达脾被吞噬细胞清除[76],因此纳米颗粒的尺寸范围也极大地降低了这些颗粒从体内清除时所带来的风险。
新的纳米制剂策略的有效性仍受到生产技术、苛刻溶剂的使用以及肽的各种物理化学性质的限制。而且,用于生产这种纳米载体的材料要求必须无毒、生物兼容、在加工过程中稳定且适合与肽一同使用,因此,寻找用作纳米载体的新型材料也是一项需要克服的任务[77-78]。
4.2 肽与抗生素的协同应用很长一段时间来,研究人员对抗菌肽的研究主要集中在提纯和鉴定抗菌肽、阐明各种肽的作用机制、生产类似的合成肽并检测其生物物理特性以及对合成的抗菌肽进行修饰来获得具有更强杀菌性能的多肽等方面[79],但目前抗菌肽的商业化仍然存在许多障碍。在对抗菌肽研究中出现的一个新理念是抗菌肽与常规抗生素的协同作用。虽然许多肽本身具有显著的抗菌活性,但要使肽在体内发挥有效的活性往往需要更高的浓度。多肽与抗生素的混合物被发现能够极大地提高抗微生物效果,而且导致耐药性细菌出现的概率更低[80],这样就同时避免了肽的大量使用和抗生素的高剂量使用,从而降低了成本并减少了耐药性产生,因此肽与抗生素之间的协同作用备受关注。目前,这一策略在β-折叠抗菌肽领域已经有了成功的例子,Kim等[81]设计了一种序列为FKVKFKVKVK-NH2的β-折叠肽,他们将肽固定在聚乙二醇-聚苯乙烯树脂上合成了多肽-树脂结合物,当该结合物与万古霉素和四环素混合使用时,发现溶液对金黄色葡萄球菌和藤黄微球菌的杀灭效果显著增强;刘又铭等[82]将LfcinB分别与土霉素、庆大霉素和阿莫西林联合使用,验证了LfcinB和阿莫西林联合作用的协同效应最强,而与土霉素协同效应最差,同时发现LfcinB与抗生素的协同作用对于革兰阴性菌的抑制效果要优于革兰阳性菌。
目前,要完全放弃使用抗生素是不现实的,但可以将抗菌肽或拟肽类药物与抗生素的优势结合起来,相辅相成。在未来,β-折叠抗菌肽与抗生素优势的结合可能会成为对抗耐药细菌的有效手段,在减少细菌耐药性降低成本方面发挥更大的作用。
5 小结过去几十年,抗生素的过度和不当使用导致了药物残留和细菌耐药性问题出现,构成了严重的全球健康问题,随着各国政府一系列禁抗法令的颁布,给畜牧生产等行业造成极大冲击,迫切需要寻求新的抗生素替代物质。抗菌肽因对多种细菌、真菌、原生动物和包膜病毒具有广泛的活性,受到了越来越多的关注。尽管结构上的一些优势让β-折叠抗菌肽被认为是很有前途的饲用抗生素替代品,但是动物体内环境高浓度的蛋白酶、生理浓度盐离子等因素仍会造成一些短链β-发卡抗菌肽快速失活,此外β-折叠抗菌肽还面临着活性差异大、生产成本高等问题,阻碍其在生产中的广泛应用。目前的方法,一方面可以通过对天然β-折叠抗菌肽进行改造或者全新设计解决β-折叠抗菌肽活性低、毒性和成本高等问题,另一方面可通过与纳米载体的结合或与抗生素的协同来提高其应用潜力。这些研究进展对于促进β-折叠抗菌肽的应用起到了一定的推动作用,但关于β-折叠抗菌肽在动物体内的应用方法和应用效果均有待进一步探究。相信随着现代分子生物学技术的不断进步和各学科的交叉融合,在不远的将来,β-折叠抗菌肽会成为替代抗生素的新一代饲料添加剂,为畜牧业的可持续发展提供新的动力。
| [1] |
SCOTT H M, ACUFF G, BERGERON G, et al. Critically important antibiotics: criteria and approaches for measuring and reducing their use in food animal agriculture[J]. Ann N Y Acad Sci, 2019, 1441(1): 8-16. DOI:10.1111/nyas.14058 |
| [2] |
雷龙, 周颖, 李俊杰, 等. 从欧盟禁抗看国内无抗的趋势[J]. 广东饲料, 2020, 29(10): 11-14. LEI L, ZHOU Y, LI J J, et al. Looking at the trend of domestic non-resistance from the EU's anti-resistance ban[J]. Guangdong Feed, 2020, 29(10): 11-14. DOI:10.3969/j.issn.1005-8613.2020.10.003 (in Chinese) |
| [3] |
李静. 抗菌肽的生物学特性及在畜禽养殖中的应用[J]. 现代畜牧科技, 2021(9): 23-24. LI J. Biological functions of antimicrobial peptides and its application in livestock and poultry breeding[J]. Modern Animal Husbandry Science & Technology, 2021(9): 23-24. (in Chinese) |
| [4] |
MAHLAPUU M, BJÖRN C, EKBLOM J. Antimicrobial peptides as therapeutic agents: opportunities and challenges[J]. Crit Rev Biotechnol, 2020, 40(7): 978-992. DOI:10.1080/07388551.2020.1796576 |
| [5] |
SHAO C X, ZHU Y J, LAI Z H, et al. Antimicrobial peptides with protease stability: progress and perspective[J]. Future Med Chem, 2019, 11(16): 2047-2050. DOI:10.4155/fmc-2019-0167 |
| [6] |
WANG G S. Improved methods for classification, prediction, and design of antimicrobial peptides[M]//ZHOU P, HUANG J. Computational Peptidology. New York: Humana Press, 2015: 43-66.
|
| [7] |
KOEHBACH J, CRAIK D J. The vast structural diversity of antimicrobial peptides[J]. Trends Pharmacol Sci, 2019, 40(7): 517-528. DOI:10.1016/j.tips.2019.04.012 |
| [8] |
巫春旭, 卢雪梅, 金小宝, 等. 天蚕素类抗菌肽分子设计研究进展[J]. 中国生物工程杂志, 2016, 36(2): 96-100. WU C X, LU X M, JIN X B, et al. Advances in research on molecular design of cecropin-like peptides[J]. China Biotechnology, 2016, 36(2): 96-100. (in Chinese) |
| [9] |
AMOS S B T A, VERMEER L S, FERGUSON P M, et al. Antimicrobial peptide potency is facilitated by greater conformational flexibility when binding to gram-negative bacterial inner membranes[J]. Sci Rep, 2016, 6(1): 37639. DOI:10.1038/srep37639 |
| [10] |
WANG C K, CRAIK D J. Designing macrocyclic disulfide-rich peptides for biotechnological applications[J]. Nat Chem Biol, 2018, 14(5): 417-427. DOI:10.1038/s41589-018-0039-y |
| [11] |
WANG J J, DOU X J, SONG J, et al. Antimicrobial peptides: promising alternatives in the post feeding antibiotic era[J]. Med Res Rev, 2019, 39(3): 831-859. DOI:10.1002/med.21542 |
| [12] |
BASTOS P, TRINDADE F, DA COSTA J, et al. Human antimicrobial peptides in bodily fluids: current knowledge and therapeutic perspectives in the postantibiotic era[J]. Med Res Rev, 2018, 38(1): 101-146. DOI:10.1002/med.21435 |
| [13] |
TORRES A M, KUCHEL P W. The β-defensin-fold family of polypeptides[J]. Toxicon, 2004, 44(6): 581-588. DOI:10.1016/j.toxicon.2004.07.011 |
| [14] |
POWERS J P S, HANCOCK R E W. The relationship between peptide structure and antibacterial activity[J]. Peptides, 2003, 24(11): 1681-1691. DOI:10.1016/j.peptides.2003.08.023 |
| [15] |
LEE J H, SHIN S M. Understanding β-hairpin formation: computational studies for three different hairpins[J]. Bull Korean Chem Soc, 2008, 29(4): 741-748. DOI:10.5012/bkcs.2008.29.4.741 |
| [16] |
PANTELEEV P V, BALANDIN S V, IVANOV V T, et al. A therapeutic potential of animal β-hairpin antimicrobial peptides[J]. Curr Med Chem, 2017, 24(17): 1724-1746. |
| [17] |
SHAFEE T M A, LAY F T, PHAN T K, et al. Convergent evolution of defensin sequence, structure and function[J]. Cell Mol Life Sci, 2017, 74(4): 663-682. DOI:10.1007/s00018-016-2344-5 |
| [18] |
GAO X H, DING J Q, LIAO C B, et al. Defensins: the natural peptide antibiotic[J]. Adv Drug Deliv Rev, 2021, 179: 114008. DOI:10.1016/j.addr.2021.114008 |
| [19] |
ANDERSSON H S, FIGUEREDO S M, HAUGAARD-KEDSTRÖM L M, et al. The α-defensin salt-bridge induces backbone stability to facilitate folding and confer proteolytic resistance[J]. Amino Acids, 2012, 43(4): 1471-1483. DOI:10.1007/s00726-012-1220-3 |
| [20] |
HOSSAIN F, MOGHAL M R, ISLAM Z, et al. Membrane potential is vital for rapid permeabilization of plasma membranes and lipid bilayers by the antimicrobial peptide lactoferricin B Membrane potential is vital for AMP-induced permeabilization[J]. J Biol Chem, 2019, 294(27): 10449-10462. DOI:10.1074/jbc.RA119.007762 |
| [21] |
FEHLBAUM P, BULET P, CHERNYSH S, et al. Structure-activity analysis of thanatin, a 21-residue inducible insect defense peptide with sequence homology to frog skin antimicrobial peptides[J]. Proc Natl Acad Sci U S A, 1996, 93(3): 1221-1225. DOI:10.1073/pnas.93.3.1221 |
| [22] |
SELSTED M E, HARWIG S S, GANZ T, et al. Primary structures of three human neutrophil defensins[J]. J Clin Invest, 1985, 76(4): 1436-1439. DOI:10.1172/JCI112121 |
| [23] |
DE LEEUW E, BURKS S R, LI X Q, et al. Structure-dependent functional properties of human defensin 5[J]. FEBS Lett, 2007, 581(3): 515-520. DOI:10.1016/j.febslet.2006.12.036 |
| [24] |
SILVA P I JR, DAFFRE S, BULET P. Isolation and characterization of gomesin, an 18-residue cysteine-rich defense peptide from the spider Acanthoscurria gomesiana hemocytes with sequence similarities to horseshoe crab antimicrobial peptides of the tachyplesin family[J]. J Biol Chem, 2000, 275(43): 33464-33470. DOI:10.1074/jbc.M001491200 |
| [25] |
PARK C H, VALORE E V, WARING A J, et al. Hepcidin, a urinary antimicrobial peptide synthesized in the liver[J]. J Biol Chem, 2001, 276(11): 7806-7810. DOI:10.1074/jbc.M008922200 |
| [26] |
施益如, 李暄, 蔡明烩, 等. 抗菌肽Protegrin-1的研究进展[J]. 中国畜牧杂志, 2021, 57(6): 67-72. SHI Y R, LI X, CAI M H, et al. Research progress on antimicrobial peptide protegrin-1[J]. Chinese Journal of Animal Science, 2021, 57(6): 67-72. (in Chinese) |
| [27] |
NAKAMURA T, FURUNAKA H, MIYATA T, et al. Tachyplesin, a class of antimicrobial peptide from the hemocytes of the horseshoe crab (Tachypleus tridentatus). Isolation and chemical structure[J]. J Biol Chem, 1988, 263(32): 16709-16713. DOI:10.1016/S0021-9258(18)37448-9 |
| [28] |
HUBER A, GALGÓCZY L, VÁRADI G, et al. Two small, cysteine-rich and cationic antifungal proteins from Penicillium chrysogenum: a comparative study of PAF and PAFB[J]. Biochim Biophys Acta Biomembr, 2020, 1862(8): 183246. DOI:10.1016/j.bbamem.2020.183246 |
| [29] |
PAVITHRRA G, RAJASEKARAN R. Gramicidin peptide to combat antibiotic resistance: a review[J]. Int J Pept Res Ther, 2020, 26(1): 191-199. DOI:10.1007/s10989-019-09828-0 |
| [30] |
WANG G S. Human antimicrobial peptides and proteins[J]. Pharmaceuticals (Basel, Switzerland), 2014, 7(5): 545-594. DOI:10.3390/ph7050545 |
| [31] |
WANG Q, REN M J, LIU X Y, et al. Peptidoglycan recognition proteins in insect immunity[J]. Mol Immunol, 2019, 106: 69-76. DOI:10.1016/j.molimm.2018.12.021 |
| [32] |
WANG Y P, LAI R. Insect antimicrobial peptides: structures, properties and gene regulation[J]. Dongwuxue Yanjiu, 2010, 31(1): 27-34. |
| [33] |
BARBAULT F, LANDON C, GUENNEUGUES M, et al. Solution structure of Alo-3:a new knottin-type antifungal peptide from the insect Acrocinus longimanus[J]. Biochemistry, 2003, 42(49): 14434-14442. DOI:10.1021/bi035400o |
| [34] |
ISOZUMI N, MASUBUCHI Y, IMAMURA T, et al. Structure and antimicrobial activity of NCR169, a nodule-specific cysteine-rich peptide of Medicago truncatula[J]. Sci Rep, 2021, 11(1): 9923. DOI:10.1038/s41598-021-89485-w |
| [35] |
TAM J P, WANG S J, WONG K H, et al. Antimicrobial peptides from plants[J]. Pharmaceuticals (Basel, Switzerland), 2015, 8(4): 711-757. DOI:10.3390/ph8040711 |
| [36] |
陈鹏. 细菌素在动物饲料中的替抗应用[J]. 饲料研究, 2021, 44(18): 150-153. CHEN P. Antibiotic replacement application of bacteriocins in animal feed[J]. Feed Research, 2021, 44(18): 150-153. (in Chinese) |
| [37] |
BROGDEN K A. Antimicrobial peptides: pore formers or metabolic inhibitors in bacteria?[J]. Nat Rev Microbiol, 2005, 3(3): 238-250. DOI:10.1038/nrmicro1098 |
| [38] |
MOOKHERJEE N, ANDERSON M A, HAAGSMAN H P, et al. Antimicrobial host defence peptides: functions and clinical potential[J]. Nat Rev Drug Discov, 2020, 19(5): 311-332. DOI:10.1038/s41573-019-0058-8 |
| [39] |
LI S Q, WANG Y J, XUE Z H, et al. The structure-mechanism relationship and mode of actions of antimicrobial peptides: A review[J]. Trends Food Sci Technol, 2021, 109: 103-115. DOI:10.1016/j.tifs.2021.01.005 |
| [40] |
张溪, 弓磊. 抗菌肽抗菌机制及研究热点[J]. 中国组织工程研究, 2020, 24(10): 1634-1640. ZHANG X, GONG L. Antimicrobial mechanism of antimicrobial peptide and research progress[J]. Chinese Journal of Tissue Engineering Research, 2020, 24(10): 1634-1640. DOI:10.3969/j.issn.2095-4344.2202 (in Chinese) |
| [41] |
BELLAMY W, TAKASE M, WAKABAYASHI H, et al. Antibacterial spectrum of lactoferricin B, a potent bactericidal peptide derived from the N-terminal region of bovine lactoferrin[J]. J Appl Bacteriol, 1992, 73(6): 472-479. DOI:10.1111/j.1365-2672.1992.tb05007.x |
| [42] |
SINHA S, NG W J, BHATTACHARJYA S. NMR structure and localization of the host defense antimicrobial peptide thanatin in zwitterionic dodecylphosphocholine micelle: Implications in antimicrobial activity[J]. Biochim Biophys Acta Biomembr, 2020, 1862(11): 183432. DOI:10.1016/j.bbamem.2020.183432 |
| [43] |
MANI R, CADY S D, TANG M, et al. Membrane-dependent oligomeric structure and pore formation of a β-hairpin antimicrobial peptide in lipid bilayers from solid-state NMR[J]. Proc Natl Acad Sci U S A, 2006, 103(44): 16242-16247. DOI:10.1073/pnas.0605079103 |
| [44] |
JENSSEN H, HAMILL P, HANCOCK R E W. Peptide antimicrobial agents[J]. Clin Microbiol Rev, 2006, 19(3): 491-511. DOI:10.1128/CMR.00056-05 |
| [45] |
DE LEEUW E, LI C Q, ZENG P Y, et al. Functional interaction of human neutrophil peptide-1 with the cell wall precursor lipid Ⅱ[J]. FEBS Lett, 2010, 584(8): 1543-1548. DOI:10.1016/j.febslet.2010.03.004 |
| [46] |
CHO J, LEE D G. The antimicrobial peptide arenicin-1 promotes generation of reactive oxygen species and induction of apoptosis[J]. Biochim Biophys Acta Gen Subj, 2011, 1810(12): 1246-1251. DOI:10.1016/j.bbagen.2011.08.011 |
| [47] |
OVCHINNIKOVA T V, ALESHINA G M, BALANDIN S V, et al. Purification and primary structure of two isoforms of arenicin, a novel antimicrobial peptide from marine polychaeta Arenicola marina[J]. FEBS Lett, 2004, 577(1-2): 209-214. DOI:10.1016/j.febslet.2004.10.012 |
| [48] |
SHAH P, CHEN C S. Systematical screening of intracellular protein targets of polyphemusin-I using Escherichia coli proteome microarrays[J]. Int J Mol Sci, 2021, 22(17): 9158. DOI:10.3390/ijms22179158 |
| [49] |
ZHAN J, JIA H S, SEMCHENKO E A, et al. Self-derived structure-disrupting peptides targeting methionine aminopeptidase in pathogenic bacteria: a new strategy to generate antimicrobial peptides[J]. FASEB J, 2019, 33(2): 2095-2104. DOI:10.1096/fj.201700613RR |
| [50] |
SONG J, WANG J J, ZHAN N, et al. Therapeutic potential of trp-rich engineered amphiphiles by single hydrophobic amino acid end-tagging[J]. ACS Appl Mater Interfaces, 2019, 11(47): 43820-43834. DOI:10.1021/acsami.9b12706 |
| [51] |
TAKAHASHI D, SHUKLA S K, PRAKASH O, et al. Structural determinants of host defense peptides for antimicrobial activity and target cell selectivity[J]. Biochimie, 2010, 92(9): 1236-1241. DOI:10.1016/j.biochi.2010.02.023 |
| [52] |
BENFIELD A H, DEFAUS S, LAWRENCE N, et al. Cyclic gomesin, a stable redesigned spider peptide able to enter cancer cells[J]. Biochim Biophys Acta Biomembr, 2021, 1863(1): 183480. DOI:10.1016/j.bbamem.2020.183480 |
| [53] |
DONG N, ZHU X, CHOU S, et al. Antimicrobial potency and selectivity of simplified symmetric-end peptides[J]. Biomaterials, 2014, 35(27): 8028-8039. DOI:10.1016/j.biomaterials.2014.06.005 |
| [54] |
VINEETHKUMAR T V, ASHA R, SHYLA G, et al. Post-translationally modified frog skin-derived antimicrobial peptides are effective against Aeromonas sobria[J]. Microb Pathog, 2017, 104: 287-288. DOI:10.1016/j.micpath.2017.01.052 |
| [55] |
GOMES B, AUGUSTO M T, FELÍCIO M R, et al. Designing improved active peptides for therapeutic approaches against infectious diseases[J]. Biotechnol Adv, 2018, 36(2): 415-429. DOI:10.1016/j.biotechadv.2018.01.004 |
| [56] |
LEI R Y, HOU J C, CHEN Q X, et al. Self-assembling myristoylated human α-defensin 5 as a next-generation nanobiotics potentiates therapeutic efficacy in bacterial infection[J]. ACS Nano, 2018, 12(6): 5284-5296. DOI:10.1021/acsnano.7b09109 |
| [57] |
IMURA Y, NISHIDA M, OGAWA Y, et al. Action mechanism of tachyplesin I and effects of PEGylation[J]. Biochim Biophys Acta Biomembr, 2007, 1768(5): 1160-1169. DOI:10.1016/j.bbamem.2007.01.005 |
| [58] |
CHAN L Y, ZHANG V M, HUANG Y H, et al. Cyclization of the antimicrobial peptide gomesin with native chemical ligation: influences on stability and bioactivity[J]. ChemBioChem, 2013, 14(5): 617-624. DOI:10.1002/cbic.201300034 |
| [59] |
FOX M A, THWAITE J E, ULAETO D O, et al. Design and characterization of novel hybrid antimicrobial peptides based on cecropin A, LL-37 and magainin Ⅱ[J]. Peptides, 2012, 33(2): 197-205. DOI:10.1016/j.peptides.2012.01.013 |
| [60] |
BALABAN N, GOV Y, GIACOMETTI A, et al. A chimeric peptide composed of a dermaseptin derivative and an RNA Ⅲ-inhibiting peptide prevents graft-associated infections by antibiotic-resistant staphylococci[J]. Antimicrob Agents Chemother, 2004, 48(7): 2544-2550. DOI:10.1128/AAC.48.7.2544-2550.2004 |
| [61] |
LIU Y F, XIA X, XU L, et al. Design of hybrid β-hairpin peptides with enhanced cell specificity and potent anti-inflammatory activity[J]. Biomaterials, 2013, 34(1): 237-250. DOI:10.1016/j.biomaterials.2012.09.032 |
| [62] |
TORRES M D T, SOTHISELVAM S, LU T K, et al. Peptide design principles for antimicrobial applications[J]. J Mol Biol, 2019, 431(18): 3547-3567. DOI:10.1016/j.jmb.2018.12.015 |
| [63] |
SHAO C X, ZHU Y J, JIAN Q, et al. Cross-strand interaction, central bending, and sequence pattern act as biomodulators of simplified β-hairpin antimicrobial amphiphiles[J]. Small, 2021, 17(7): 2003899. DOI:10.1002/smll.202003899 |
| [64] |
WU H, ONG Z Y, LIU S Q, et al. Synthetic β-sheet forming peptide amphiphiles for treatment of fungal keratitis[J]. Biomaterials, 2015, 43: 44-49. DOI:10.1016/j.biomaterials.2014.11.052 |
| [65] |
CHOU S, WANG J J, SHANG L, et al. Short, symmetric-helical peptides have narrow-spectrum activity with low resistance potential and high selectivity[J]. Biomater Sci, 2019, 7(6): 2394-2409. DOI:10.1039/C9BM00044E |
| [66] |
HAMMAMI R, FLISS I. Current trends in antimicrobial agent research: chemo- and bioinformatics approaches[J]. Drug Discovery Today, 2010, 15(13-14): 540-546. DOI:10.1016/j.drudis.2010.05.002 |
| [67] |
CHERKASOV A, HILPERT K, JENSSEN H, et al. Use of artificial intelligence in the design of small peptide antibiotics effective against a broad spectrum of highly antibiotic-resistant superbugs[J]. ACS Chem Biol, 2009, 4(1): 65-74. DOI:10.1021/cb800240j |
| [68] |
FJELL C D, JENSSEN H, CHEUNG W A, et al. Optimization of antibacterial peptides by genetic algorithms and cheminformatics[J]. Chem Biol Drug Des, 2011, 77(1): 48-56. DOI:10.1111/j.1747-0285.2010.01044.x |
| [69] |
李丘轲, 李金泽, 吴华, 等. 靶向抗菌肽的设计策略与应用[J]. 畜牧兽医学报, 2020, 51(2): 243-251. LI Q K, LI J Z, WU H, et al. Design strategy and application on targeted antimicrobial peptides[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 243-251. (in Chinese) |
| [70] |
邓赣奇, 黄增颖, 梁耀文, 等. "减抗"、"替抗"背景下抗菌肽的应用和研究进展[J]. 家畜生态学报, 2020, 41(6): 1-7. DENG G Q, HUANG Z Y, LIANG Y W, et al. Application and research advance of antimicrobial peptide[J]. Acta Ecologiae Animalis Domastici, 2020, 41(6): 1-7. DOI:10.3969/j.issn.1673-1182.2020.06.001 (in Chinese) |
| [71] |
ECKERT R, QI F X, YARBROUGH D K, et al. Adding selectivity to antimicrobial peptides: rational design of a multidomain peptide against Pseudomonas spp[J]. Antimicrob Agents Chemother, 2006, 50(4): 1480-1488. DOI:10.1128/AAC.50.4.1480-1488.2006 |
| [72] |
URBAN P, VALLE-DELGADO J J, MOLES E, et al. Nanotools for the delivery of antimicrobial peptides[J]. Curr Drug Targets, 2012, 13(9): 1158-1172. DOI:10.2174/138945012802002302 |
| [73] |
PARDHI D M, KARAMAN D Ș, TIMONEN J, et al. Anti-bacterial activity of inorganic nanomaterials and their antimicrobial peptide conjugates against resistant and non-resistant pathogens[J]. Int J Pharm, 2020, 586: 119531. DOI:10.1016/j.ijpharm.2020.119531 |
| [74] |
SOUSA M G C, REZENDE T M B, FRANCO O L. Nanofibers as drug-delivery systems for antimicrobial peptides[J]. Drug Discov Today, 2021, 26(8): 2064-2074. DOI:10.1016/j.drudis.2021.03.008 |
| [75] |
康芷若, 王如霞, 陈梦涵, 等. 纳米材料作为抗菌肽递送载体的研究进展[J]. 动物医学进展, 2021, 42(6): 97-102. KANG Z R, WANG R X, CHEN M H, et al. Progress on nanomaterials as vehicles for delivery of antimicrobial peptides[J]. Progress in Veterinary Medicine, 2021, 42(6): 97-102. DOI:10.3969/j.issn.1007-5038.2021.06.018 (in Chinese) |
| [76] |
STOLNIK S, ILLUM L, DAVIS S S. Long circulating microparticulate drug carriers[J]. Adv Drug Deliv Rev, 2012, 64: 290-301. DOI:10.1016/j.addr.2012.09.029 |
| [77] |
GERA S, KANKURI E, KOGERMANN K. Antimicrobial peptides - unleashing their therapeutic potential using nanotechnology[J]. Pharmacol Ther, 2022, 232: 107990. DOI:10.1016/j.pharmthera.2021.107990 |
| [78] |
TEIXEIRA M C, CARBONE C, SOUSA M C, et al. Nanomedicines for the delivery of antimicrobial peptides (AMPs)[J]. Nanomaterials (Basel), 2020, 10(3): 560. DOI:10.3390/nano10030560 |
| [79] |
HANEY E F, HUNTER H N, MATSUZAKI K, et al. Solution NMR studies of amphibian antimicrobial peptides: linking structure to function?[J]. Biochim Biophys Acta Biomembr, 2009, 1788(8): 1639-1655. DOI:10.1016/j.bbamem.2009.01.002 |
| [80] |
LAZZARO B P, ZASLOFF M, ROLFF J. Antimicrobial peptides: application informed by evolution[J]. Science, 2020, 368(6490): eaau5480. DOI:10.1126/science.aau5480 |
| [81] |
KIM J M, JANG S J, YANG M H, et al. Characterization of antibacterial activity and synergistic effect of cationic antibacterial peptide-resin conjugates[J]. Bull Korean Chem Soc, 2011, 32(11): 3928-3932. |
| [82] |
刘又铭, 房鑫, 陈璐, 等. 牛乳源肽Lfcin B抗菌活性、稳定性及协同效应研究[J]. 饲料工业, 2021, 42(3): 49-53. LIU Y M, FANG X, CHEN L, et al. Study on antibacterial activity, stability and synergistic effect of Lactoferricin Bovine[J]. Feed Industry, 2021, 42(3): 49-53. (in Chinese) |
(编辑 白永平)