基因组选择(genomic selection, GS)[1]目前已广泛应用于畜禽遗传评估[2-3]。相对于基于系谱的最佳线性无偏预测(best linear unbiased prediction, BLUP),基因组选择能有效提高基于表型的预测准确性[4]、缩短世代间隔和加快遗传进展[5]。然而,由于基因分型成本较高,通常只测定育种群中关键个体的基因型,因此,经典基因组预测方法只能利用部分个体的基因型和表型信息。为解决这一问题,Legarra等[6]及Christensen和Lund[7]提出了基因组选择一步法(single-step genomic BLUP, SSGBLUP)。一步法通过有效整合群体所有个体(包括有基因型和没有基因型信息个体)的系谱、表型和基因型信息,提高了基因组估计育种值(genomic estimated breeding value, GEBV)的准确性。
基因组选择的准确性与参考群的大小密切相关[8-9],参考群规模越大,基因组选择效果越好。而我国由于很多核心育种场规模较小,基因组选择的效果不理想[10-11],对此我国制定了基因组联合育种方案[12-13]。使用SSGBLUP法进行基因组联合育种,由于多个群体之间的基因型频率不同[14],群体间没有系谱上的关联,不同群体个体间亲缘关系为0,群体间不能进行有效的关联,基因组关系矩阵G与系谱亲缘关系矩阵A难以兼容[15],造成基因组联合育种效果不理想[16-17]。
为解决G矩阵与A矩阵不兼容的问题,Legarra等[18]和Christensen[19]提出了元共祖(metafounder)的概念。元共祖是用来描述基础群内部关系的伪个体,可以理解为一个无限大小的配子池。基础群的个体就是由配子池随机抽取配对形成,各配子之间的共亲关系(coancestry relationship)为γ/2。多个元共祖之间的亲缘关系为祖先关系矩阵(ancestral relationships)Γ,基于Γ矩阵与A矩阵构建A(Γ)矩阵。含元共祖的一步法(single- step method with metafounders, MF-SSGBLUP)使用0.5为等位基因频率构建G05矩阵并与A(Γ)矩阵联合构建H(Γ)矩阵,可以自动兼容G矩阵与A矩阵[18-20]。
将MF-SSGBLUP应用于基因组联合育种不仅可以解决G矩阵和A矩阵的兼容性问题,也解决了多个群体基因型频率不同的问题[19],并且在A(Γ)矩阵构建过程中加入了祖先关系矩阵,可以在不同群体个体间建立亲缘关系,将多个群体产生关联[21]。
为了研究MF-SSGBLUP在基因组联合育种中的有效性,本研究基于多个模拟群体进行以下研究:1)比较MF-SSGBLUP法和SSGBLUP法构建的G矩阵与A矩阵的兼容性;2)比较MF-SSGBLUP、SSGBLUP和BLUP的遗传参数估计结果;3)对MF-SSGBLUP在基因组联合育种中的使用效果进行评估。
1 材料与方法 1.1 数据模拟本研究使用QMSim[22]模拟2个具有不同遗传力(0.1和0.3)的限性性状,表型方差设置为1,每个性状进行10次重复。历史群体起始规模为5 000头,经过1 000个世代,群体衰减为250头,再经过100个世代,群体扩增为1 000头,通过群体规模的波动达到与真实群体相似的连锁不平衡(linkage disequilibrium,LD)程度,并且建立了突变漂变平衡。整个历史群体公、母比例保持1∶1,个体间采取随机交配。模拟3个扩增群体(Line1、Line2、Cross),世代数为10,每个世代增长率为60%,个体间采取随机交配。扩增群体模拟策略为:1)在历史群体的最后一个世代中抽取真实育种值较高的10头公畜和100头母畜构成Line1的初始群体,抽取真实育种值较低的10头公畜与100头母畜构成Line2;2)从Line1的第10世代中抽取100头公畜,从Line2的第10世代中抽取100头母畜,构成Cross1的初始群体(图 1)。
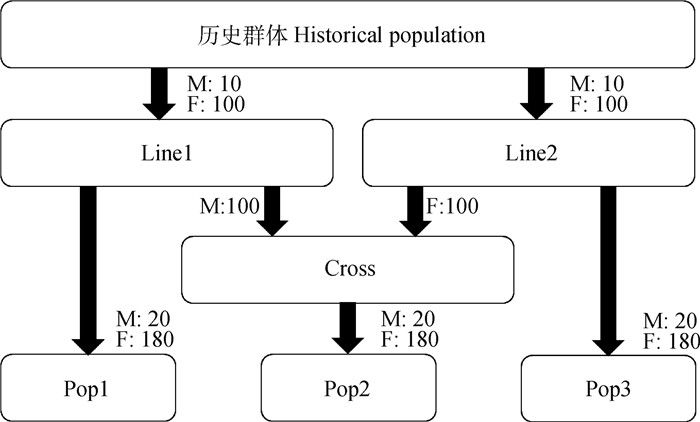
|
Line1、Line2和Cross代表不同的扩繁群体,Pop1、Pop2和Pop3代表不同的当代群体 Line1, Line2 and Cross correspond to different expansion populations, while Pop1, Pop2 and Pop3 correspond to different current populations 图 1 群体结构模拟策略 Fig. 1 Simulation strategy for population structures |
分别从3个扩增群体的第10世代中抽取EBV高的20头公畜和180头母畜构成3个当代群体Pop1、Pop2和Pop3(图 1),世代数均设为10代,设置3个元共祖对应3个群体。模拟中,每代公畜淘汰率为30%,增长率为10%;母畜淘汰率为20%,增长率为10%。每代公、母畜间随机交配。假定所研究畜种为单胎,每头母畜后裔数为1,后裔公、母各半,选择EBV高的个体留种。保留当代群体第1~9世代所有母畜的表型信息,将第1~9世代作为参考群,将第10个世代作为候选群。
本试验模拟奶牛的基因组,共设置29对染色体,全长为2 333 cM,性状受到1 000个随机分布在全基因组中的数量性状基因座(quantitative trait loci, QTL)影响。QTL效应值服从形状参数为0.4的伽马分布。模拟50 000个均匀分布于全基因组上的SNP标记。设置起始历史群体SNP和QTL等位基因频率均为0.5,每个位点突变率为10-5,标记位点的错误率为0.5%。选取群体后5代有后裔的公畜和最后2个世代的所有个体都进行基因分型。对模拟产生的基因型数据进行质量控制,删除次要等位基因频率低于5%的基因型。经过质量控制,对于h2为0.3的模拟数据,保留的SNP标记数为47 313,10次重复的方差为173.5;对于h2为0.1的模拟数据,保留的SNP标记数为47 289,10次重复的方差为168.7。
1.2 基因组预测模型
1.2.1 SSGBLUP
本研究使用的模型为:y=Xb+Zu+e,其中,y为表型值向量,b为固定效应向量,u为随机效应向量,
| $ \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{X}}\prime \mathit{\boldsymbol{X}}}&{\mathit{\boldsymbol{X}}\prime \mathit{\boldsymbol{Z}}}\\ {\mathit{\boldsymbol{Z}}\prime \mathit{\boldsymbol{X}}}&{\mathit{\boldsymbol{Z}}\prime \mathit{\boldsymbol{Z}} + \mathit{\boldsymbol{H}}{^{ - 1}}\lambda } \end{array}} \right]\left[ {\begin{array}{*{20}{c}} {\hat b}\\ {\hat u} \end{array}} \right] = \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{X}}\prime y}\\ {\mathit{\boldsymbol{Z}}\prime y} \end{array}} \right] $ | (1) |
其中,
| $ {\mathit{\boldsymbol{H}}^{ - 1}} = \mathit{\boldsymbol{A}}{^{ - 1}} + \left[ {\begin{array}{*{20}{c}} 0&0\\ 0&{{\mathit{\boldsymbol{G}}_w}^{ - 1} - {\mathit{\boldsymbol{A}}^{ - 1}}_{22}} \end{array}} \right] $ | (2) |
其中,Gw=(1-w)G*+wA22[23],w为加权因子(设为常数0.05),Gw矩阵能避免G矩阵无法求逆的问题,且通过混合A22矩阵解释了一些基因型标记未能解释的效应。考虑到G矩阵与A22矩阵兼容性的问题,使用矫正后的矩阵G*:G*=βG+α,其中,α和β通过以下方程组求解得出[23]:
| $ \left\{ {\begin{array}{*{20}{l}} {\frac{{tr(\mathit{\boldsymbol{G}})}}{m}\beta + \alpha = \frac{{tr\left( {{\mathit{\boldsymbol{A}}_{22}}} \right)}}{m}}\\ {\alpha + \beta \mathit{\boldsymbol{\bar G}} = \mathit{\boldsymbol{\bar A}}{_{22}}} \end{array}} \right. $ | (3) |
其中,m为基因分型个体数,tr(G)和tr(A22)为G矩阵与A22矩阵的迹,G和A22为G矩阵与A22矩阵非对角线元素的均值。MME的求解通过BLUPF90[24]软件完成。
1.2.2 MF-SSGBLUP MF-SSGBLUP法的MME为:
| $ \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{X}}\prime \mathit{\boldsymbol{X}}}&{\mathit{\boldsymbol{X}}\prime \mathit{\boldsymbol{Z}}}\\ {\mathit{\boldsymbol{Z}}\prime \mathit{\boldsymbol{X}}}&{\mathit{\boldsymbol{Z}}\prime \mathit{\boldsymbol{Z}} + \mathit{\boldsymbol{H}}{{(\mathit{\boldsymbol{ \boldsymbol{\varGamma} }})}^{ - 1}}\lambda } \end{array}} \right]\left[ {\begin{array}{*{20}{c}} {\hat b}\\ {\hat u} \end{array}} \right] = \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{X}}\prime y}\\ {\mathit{\boldsymbol{Z}}\prime y} \end{array}} \right] $ | (4) |
其中,H(Γ)-1为:
| $ \mathit{\boldsymbol{H}}{(\mathit{\boldsymbol{ \boldsymbol{\varGamma} }})^{ - 1}} = k\left( {\mathit{\boldsymbol{A}}{{\left( \mathit{\boldsymbol{ \boldsymbol{\varGamma} }} \right)}^{ - 1}} + \left[ {\begin{array}{*{20}{c}} 0&0\\ 0&{\mathit{\boldsymbol{G}}_{05}^{ - 1} - \mathit{\boldsymbol{A}}\left( \mathit{\boldsymbol{ \boldsymbol{\varGamma} }} \right)_{22}^{ - 1}} \end{array}} \right]} \right) $ | (5) |
通过Legarra等[18]的方法构建A(Γ)-1矩阵,用0.5为基础群体等位基因频率构建G05矩阵。k是一个标量,计算方式为:
祖先关系矩阵Γ构建方法为:Γ=8Ω=8Cov(P)[19],其中P是一个矩阵,行数为总标记数m,列数为群体数,P的元素pij代表第i个标记在第j个群体中的频率;Ω是维度等于群体数的一个方阵,其中对角线元素为该群体基因型频率的方差,非对角线元素为两个群体间基因型频率的协方差。本研究使用两种方法计算Γ矩阵,以对两种方法的性能做比较。一种方法是只使用基因型数据来估计的原始方法(naïve,NAI)[25]:
| $ {m_i} = \mathit{\boldsymbol{Q}}{\mu _i} + {e_i} $ | (6) |
| $ {{\hat \mu }_i} = {(\mathit{\boldsymbol{Q}}\prime \mathit{\boldsymbol{Q}})^{ - 1}}\mathit{\boldsymbol{Q}}\prime {m_i} $ | (7) |
其中,mi为基因分型个体第i个位点的基因型,由{0, 1, 2}组成;Q是一个将元共祖与后代个体联系起来的矩阵,每行元素之和为1;μi为各群体在第i个位点的基因型均值向量;ei为误差向量。所有位点的基因型均值为μ=[μ1μ2…μm]。由于,μi= 2pi,所以,Γ=2Var(μ)。
第二种是同时利用基因型与系谱信息计算Γ矩阵的广义最小二乘法(generalized least squares,GLS):
| $ {m_i} = \mathit{\boldsymbol{Q}}{\mu _i} + \mathit{\boldsymbol{W}}{u_i} + {e_i} $ | (8) |
其中,ui为基因分型个体间的偏差,
| $ \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{Q}}\prime \mathit{\boldsymbol{Q}}}&{\mathit{\boldsymbol{Q}}\prime \mathit{\boldsymbol{W}}}\\ {\mathit{\boldsymbol{W}}\prime \mathit{\boldsymbol{Q}}}&{\mathit{\boldsymbol{W}}\prime \mathit{\boldsymbol{W + A}}{\mathit{\boldsymbol{}}^{ - 1}}\lambda } \end{array}} \right]\left[ {\begin{array}{*{20}{c}} {{{\hat u}_i}}\\ {\hat u} \end{array}} \right] = \left[ {\begin{array}{*{20}{c}} {\mathit{\boldsymbol{Q}}\prime {m_i}}\\ {w\prime {m_i}} \end{array}} \right] $ | (9) |
该模型的BLUE值为
1.2.3 遗传参数估计
使用约束最大似然法(residual maximum likelihood, REML)[26]估计遗传方差
1.3.1基因组关系矩阵与系谱亲缘关系矩阵的兼容性
用对角线元素与非对角线元素之间的相关系数(r)、回归系数(b1)和回归截距(b0)3个指标来评价G矩阵与A22矩阵之间的兼容性。相关系数是G矩阵与A22矩阵对角线元素之间和非对角元素之间的皮尔森相关系数:
| $ \left\{ {\begin{array}{*{20}{c}} {{r_{diag}} = \frac{{co\upsilon \left( {diag\left( \mathit{\boldsymbol{G}} \right), diag\left( {{\mathit{\boldsymbol{A}}_{22}}} \right)} \right)}}{{\sqrt {\upsilon ar(diag\left( \mathit{\boldsymbol{G}} \right)\upsilon ar(diag(\mathit{\boldsymbol{A}}{_{22}}))} }}}\\ {{r_{offdiag}} = \frac{{co\upsilon \left( {offdiag\left( \mathit{\boldsymbol{G}} \right), offdiag\left( {\mathit{\boldsymbol{A}}{_{22}}} \right)} \right)}}{{\upsilon ar(offdiag\left( \mathit{\boldsymbol{G}} \right)\upsilon ar(offdiag(\mathit{\boldsymbol{A}}{_{22}}))}}{\rm{ }}} \end{array}} \right. $ | (10) |
其中,diag和offdiag分别对应对角线元素与非对角线元素,cov为协方差,var为方差,相关系数越高表示相关性越好。
回归系数与回归截距是通过构建G矩阵对角线元素(非对角线元素)对A22矩阵对角线元素(非对角线元素)的回归方程:
| $ \left\{ {\begin{array}{*{20}{l}} {diag\left( \mathit{\boldsymbol{G}} \right) = {b_{0(diag)}} + {b_{1(diag)}}diag({\mathit{\boldsymbol{A}}_{22}}) + e}\\ {offdiag\left( \mathit{\boldsymbol{G}} \right) = {b_{0(offdiag)}} + {b_{1(offdiag)}}offdiag\left( {{\mathit{\boldsymbol{A}}_{22}}} \right) + e} \end{array}} \right. $ | (11) |
回归系数越接近于1,回归截距越接近于0,表示无偏性越好。
1.3.2 模型评价 用准确性(accuracy)和无偏性(bias)来比较不同模型的性能。准确性通过EBV与TBV之间的相关系数衡量:
| $ r = \frac{{co\upsilon \left( {EBV, TBV} \right)}}{{\sqrt {\upsilon ar\left( {EBV} \right)\upsilon ar\left( {TBV} \right)} }} $ |
无偏性用GEBV对TBV的回归系数[29]来衡量:
| $ b = \frac{{co\upsilon \left( {EBV, TBV} \right)}}{{\upsilon ar\left( {EBV} \right)}} $ |
回归系数越接近于1越好,用bias=|1-b|衡量无偏性。
2 结果 2.1 群体模拟结果模拟的3个当代群体(Pop1、Pop2和Pop3),个体总数为8 430。其中,参考群和候选群个体分别为7 404和1 026个,基因分型个体为2 103个,具有表型的个体约为3 702个。
为了评估模拟产生的群体遗传结构,利用3个群体的基因组标记信息进行主成分分析(principal component analysis, PCA)。由于10次模拟的群体遗传结构相似,仅展示第1次模拟结果(图 2)。h2为0.1时,群体前两个主成分所解释的方差占总方差的百分比分别为9.906%和6.081%;h2为0.3时,分别为9.049%和7.479%。表明模拟的3个群体具有明显的遗传差异。
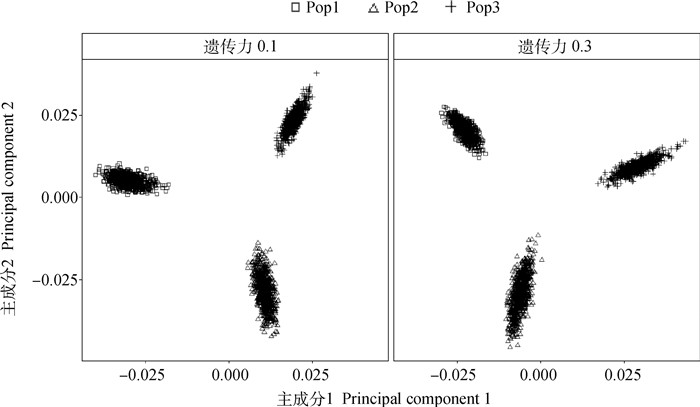
|
图 2 模拟的3个群体间的遗传差异 Fig. 2 The genetic differences among 3 simulated populations |
使用GLS和NAI法对Γ矩阵进行估计。h2为0.1时,10次重复的均值为:
| $ {\mathit{\boldsymbol{ \boldsymbol{\varGamma} }}_{GLS}} = \left[ {\begin{array}{*{20}{c}} {0.58}&{0.42}&{0.38}\\ {0.42}&{0.51}&{0.42}\\ {0.38}&{0.42}&{0.52} \end{array}} \right]; $ |
| $ {\mathit{\boldsymbol{ \boldsymbol{\varGamma} }}_{NAI}} = \left[ {\begin{array}{*{20}{c}} {0.65}&{0.42}&{0.38}\\ {0.42}&{0.57}&{0.42}\\ {0.38}&{0.42}&{0.57} \end{array}} \right]。$ |
h2为0.3时,10次重复的均值为:
| $ {\mathit{\boldsymbol{ \boldsymbol{\varGamma} }}_{GLS}} = \left[ {\begin{array}{*{20}{c}} {0.56}&{0.42}&{0.38}\\ {0.42}&{0.51}&{0.42}\\ {0.38}&{0.42}&{0.52} \end{array}} \right]; $ |
| $ {\mathit{\boldsymbol{ \boldsymbol{\varGamma} }}_{NAI}} = \left[ {\begin{array}{*{20}{c}} {0.62}&{0.42}&{0.38}\\ {0.42}&{0.59}&{0.42}\\ {0.38}&{0.42}&{0.58} \end{array}} \right]。$ |
Γ矩阵为元共祖之间的关系矩阵,元素的值反映了元共祖之间的亲缘关系。在不同h2下,Γ矩阵差异不大,说明不同h2下基础群个体间亲缘关系相似。Γ矩阵对角线元素的值普遍大于非对角线元素,表明在同一个基础群体内的个体亲缘关系高于不同基础群体个体间的亲缘关系。ΓGLS对角线元素略小于ΓNAI的对角线元素,非对角元素之间没有差异。
2.3 不同方法兼容性的比较兼容性通过亲缘关系矩阵与基因组关系矩阵的对角线(非对角线)元素的相关系数、回归系数和回归截距来说明,其中相关系数与回归系数越接近于1,回归截距越接近于0兼容性越好。通过ΓGLS矩阵和ΓNAI矩阵分别构建A22(ΓGLS)和A22(ΓNAI)矩阵,并计算与G05矩阵的兼容性,用常规SSGBLUP法构建的A22矩阵与G*进行对照,结果见表 1。在不同h2下,A22(ΓGLS)~G05和A22(ΓNAI)~G05对角线(非对角线)元素相关系数0.750~0.775(0.954~ 0.964)、回归系数0.859~0.992(0.639~0.812)和回归截距-0.013~0.135(0.071~0.098) 均显著(P<0.05)优于A22~G*相关系数0.508~ 0.572(0.723~0.738)、回归系数0.543~0.652 (0.683~0.745)和回归截距0.374~0.493(0.134~0.139),表明元共祖法构建的A矩阵与G矩阵间兼容性更好(表 1)。
|
|
表 1 3种方法构建的G和A22矩阵的兼容性比较 Table 1 Comparison of compatibility between G and A22 matrices constructed by 3 methods |
表 2为10次重复下MF-SSGBLUP(GLS)、MF-SSGBLUP(NAI)、SSGBLUP和传统BLUP对方差组分和h2估值的均值和方差与当代群体遗传参数的对比。在h2初值为0.1和0.3的群体中,4种方法对h2的估计值介于0.138~0.173和0.273~0.340,与当代群体遗传力0.107和0.296相符。此外,两种MF-SSGBLUP在不同h2下对方差组分的估值比SSGBLUP和BLUP法更接近于真值,表明MF-SSGBLUP法在遗传参数的估计上要优于SSGBLUP和传统BLUP法。
|
|
表 2 4种方法估计的方差组分和遗传力 Table 2 Estimated variance components and heritabilities by 4 methods |
图 3为4种方法估计育种值的准确性和无偏性。在h2为0.1情况下,两种MF-SSGBLUP的准确性均为0.888,MF-SSGBLUP(GLS)的无偏性均值为0.030,MF-SSGBLUP(NAI)的无偏性均值为0.032,SSGBLUP为0.863和0.066,传统BLUP为0.854和0.078;在h2为0.3情况下,两种MF-SSGBLUP的准确性和无偏性均值均为0.908和0.029,SSGBLUP为0.876和0.057,传统BLUP为0.871和0.067。不同h2下,两种MF-SSGBLUP的准确性与无偏性显著优于SSGBLUP与BLUP法(P<0.05),而MF-SSGBLUP的两种算法(GLS vs. NAI)间则无显著差异。以上结果表明,在基因组联合育种中,MF-SSGBLUP法有一定优势。
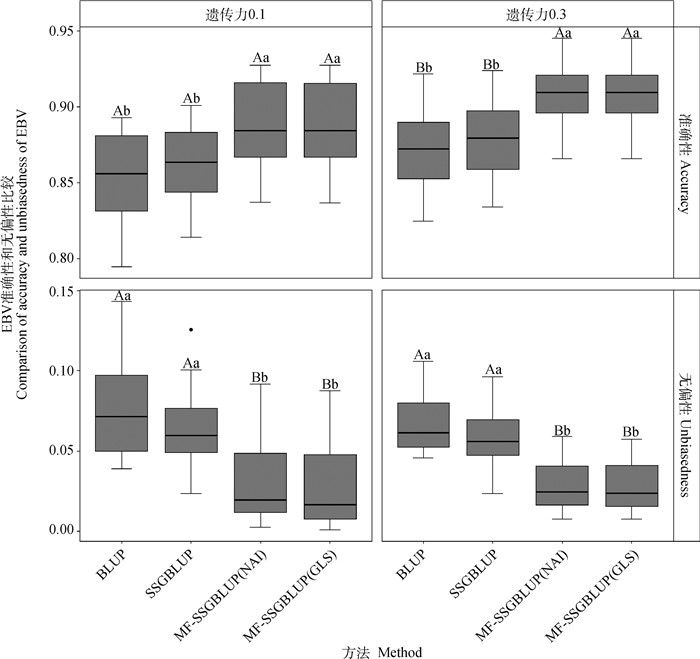
|
不同大写字母表示差异极显著(P<0.01),不同小写字母表示差异显著(P<0.05) Different capital and lowercase letters mean significant differences at P<0.01 and P<0.05, respectively 图 3 4种方法估计育种值的准确性与无偏性比较 Fig. 3 Comparison of accuracy and unbiasedness of estimating breeding values by 4 methods |
用MF-SSGBLUP法进行遗传评估[30-32]已有报道,并且均表现出一定的优势,但是在基因组联合育种中还未应用。本研究模拟了不同h2(0.1和0.3)的性状,使用传统BLUP法、SSGBLUP法和MF-SSBLUP法进行遗传评估。其中,用GLS和NAI两种算法估计祖先关系矩阵Γ。Bradford等[30]指出,Γ矩阵不依赖表型信息,只与基因型信息与系谱信息相关。本研究在不同h2下获得的Γ矩阵数值上差异不大,是因为不同h2下模拟的群体结构类似。不同h2情况下,GLS法和NAI法所得的Γ矩阵非对角线元素间差异不大,而对角线元素则有明显差异,即NAI法所得的元共祖近交系数高于GLS法[19-25]。Garcia-Baccino等[25]发现NAI法对Γ矩阵中的元素估值偏高,GLS法能有效获得Γ矩阵的无偏估值,与本研究结果相符。本试验中基因分型个体都是经过了多个世代的选择,部分基因型频率漂变到极端值,NAI法忽略了这一部分的影响[25],GLS法考虑了基因型在世代间的传递,将系谱信息纳入模型之中,因而更为准确。
对比不同方法构建的G矩阵与A22矩阵,发现A22(ΓGLS)~G05和A22 (ΓNAI)~G05兼容性要优于A22 ~G*,这与Kudinov等[33]在丹麦红牛上的研究成果相似。Christensen[19]指出基因分型个体与所有个体间育种值平均值的差异导致了G矩阵与A矩阵的不兼容。一些研究通过调整G矩阵使其与A矩阵相兼容,但是都没有从本质上解决问题。元共祖法与之相反,通过调整A矩阵,达到与基因型关系矩阵相兼容的目的。VanRaden[14]指出,A矩阵中的元素为亲缘相关的期望值,然而在基因组联合育种中由于多个群体在系谱上没有关联,不同群体个体间相关关系为0,这与基因组关系矩阵在不同群体个体间亲缘相关大于0的情况相悖。基于元共祖构建的亲缘关系矩阵,不同群体中个体间的相关关系通常不为0。A22 (ΓGLS)~G05在非对角线元素的回归系数与回归截距上要优于A22(ΓNAI)~G05,这可能是因为GLS法获得Γ矩阵的估计值比NAI法无偏性更好。
在Garcia-Baccino等[25]的研究中,传统BLUP估计的h2更接近于真值。然而,本研究显示,两种算法下,MF-SSGBLUP法遗传参数估值的无偏性较好,可能是由于3个模拟群体没有系谱关联导致了传统BLUP对遗传参数估计的准确性下降。
Garcia-Baccino等[25]的研究指出,在基因组选择中,MF-SSGBLUP比SSGBLUP法能获得更高的准确性与更小的偏差。Bradford等[30]设置了3个不同的元共祖,对于h2为0.3和0.1的性状,与SSGBLUP和BLUP相比,MF-SSGBLUP能获得更高的准确性。而且,Bradford等[30]在系谱缺失情况下,发现MF-SSGBLUP法所得结果的一致性最好,能有效减少由于系谱缺失造成的偏差。Xiang等[34]和Van Grevenhof等[31]分别在二元杂交系统与三元杂交系统中验证了元共祖方法的优越性。本研究中,两种算法的MF-SSGBLUP法的遗传评估准确性均显著高于SSGBLUP和BLUP法,且EBV的偏差更小,与前人研究结果相似。两种MF-SSGBLUP算法的准确性与无偏性没有明显差异,说明GLS和NAI两种算法估计的Γ矩阵对EBV的准确性没有较大影响。但是,GLS能获得更加无偏的Γ矩阵[25],且通过ΓGLS构建的A矩阵与G矩阵兼容性更好,所以,建议使用MF-SSGBLUP(GLS)进行基因组联合育种。
4 结论用MF-SSGBLUP对多个模拟群体进行基因组联合育种研究,发现MF-SSGBLUP通过估计多个系谱独立群体的元共祖间亲缘关系,优化多群体联合育种的关系矩阵,可有效提高遗传力和育种值的估计准确性和无偏性。因此,MF-SSGBLUP能有效解决多群体间系谱独立和基因频率差异的问题,在基因组联合育种中具有良好应用潜力。
| [1] |
MEUWISSEN T H E, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense markermaps[J]. Genetics, 2001, 157(4): 1819-1829. DOI:10.1093/genetics/157.4.1819 |
| [2] |
MEUWISSEN T, HAYES B, GODDARD M. Genomic selection: a paradigm shift in animal breeding[J]. Anim Front, 2016, 6(1): 6-14. DOI:10.2527/af.2016-0002 |
| [3] |
AGUILAR I, MISZTAL I, JOHNSON D L, et al. Hot topic: a unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final score[J]. J Dairy Sci, 2010, 93(2): 743-752. DOI:10.3168/jds.2009-2730 |
| [4] |
HAYES B J, VISSCHER P M, GODDARD M E. Increased accuracy of artificial selection by using the realized relationship matrix[J]. Genet Res, 2009, 91(1): 47-60. DOI:10.1017/S0016672308009981 |
| [5] |
HABIER D, TETENS J, SEEFRIED F R, et al. The impact of genetic relationship information on genomic breeding values in German Holstein cattle[J]. Genet Sel Evol, 2010, 42(1): 5. DOI:10.1186/1297-9686-42-5 |
| [6] |
LEGARRA A, AGUILAR I, MISZTAL I. A relationship matrix including full pedigree and genomic information[J]. J Dairy Sci, 2009, 92(9): 4656-4663. DOI:10.3168/jds.2009-2061 |
| [7] |
CHRISTENSEN O F, LUND M S. Genomic prediction when some animals are not genotyped[J]. Genet Sel Evol, 2010, 42(1): 2. DOI:10.1186/1297-9686-42-2 |
| [8] |
NILFOROOSHAN M A, ZUMBACH B, JAKOBSEN J, et al. Validation of national genomicevaluations[J]. Int Bull, 2010(42): 56-61. |
| [9] |
EGGEN A. The development and application of genomic selection as a new breeding paradigm[J]. Anim Front, 2012, 2(1): 10-15. DOI:10.2527/af.2011-0027 |
| [10] |
HAYES B J, BOWMAN P J, CHAMBERLAIN A J, et al. Invited review: genomic selection in dairy cattle: progress and challenges[J]. J Dairy Sci, 2009, 92(2): 433-443. DOI:10.3168/jds.2008-1646 |
| [11] |
VANRADEN P M, VAN TASSELL C P, WIGGANS G R, et al. Invited review: reliability of genomic predictions for North American Holstein bulls[J]. J Dairy Sci, 2009, 92(1): 16-24. DOI:10.3168/jds.2008-1514 |
| [12] |
全国生猪遗传改良计划(2009-2020)实施方案[J]. 中国牧业通讯, 2010(23): 25-28. The national pig improvement program (2009-2020)[J]. China Animal Husbandry Bulletin, 2010(23): 25-28. (in Chinese) |
| [13] |
张勤, 丁向东, 陈瑶生. 种猪遗传评估技术研发与评估系统应用[J]. 中国畜牧杂志, 2015, 51(8): 61-65, 84. ZHANG Q, DING X D, CHEN Y S. Development and application of swine genetic evaluation system in China[J]. Chinese Journal of Animal Science, 2015, 51(8): 61-65, 84. DOI:10.3969/j.issn.0258-7033.2015.08.012 (in Chinese) |
| [14] |
VANRADEN P M. Efficient methods to computegenomic predictions[J]. J Dairy Sci, 2008, 91(11): 4414-4423. DOI:10.3168/jds.2007-0980 |
| [15] |
LEGARRA A, CHRISTENSEN O F, AGUILAR I, et al. Single Step, a general approach for genomic selection[J]. Livest Sci, 2014, 166: 54-65. DOI:10.1016/j.livsci.2014.04.029 |
| [16] |
HIDALGO A M, BASTIAANSEN J W M, LOPES M S, et al. Accuracy ofpredicted genomic breeding values in purebred and crossbred pigs[J]. G3 Genes|Genom|Genet, 2015, 5(8): 1575-1583. |
| [17] |
余健, 杨文静, 王晔, 等. 多个场联合遗传评估提高基因组选择准确性[J]. 中国畜牧杂志, 2021, 57(S1): 25-28. YU J, YANG W J, WANG Y, et al. Combined genetic evaluation of multiple populations improves genomic selection accuracy[J]. Chinese Journal of Animal Science, 2021, 57(S1): 25-28. (in Chinese) |
| [18] |
LEGARRA A, CHRISTENSEN O F, VITEZICA Z G, et al. Ancestral relationships using metafounders: finite ancestral populations and across population relationships[J]. Genetics, 2015, 200(2): 455-468. DOI:10.1534/genetics.115.177014 |
| [19] |
CHRISTENSEN O F. Compatibility of pedigree-based and marker-based relationship matrices for single-step genetic evaluation[J]. Genet Sel Evol, 2012, 44(1): 37. DOI:10.1186/1297-9686-44-37 |
| [20] |
FU C K, OSTERSEN T, CHRISTENSEN O F, et al. Single-step genomic evaluation with metafounders for feed conversion ratio and average daily gain in Danish Landrace and Yorkshire pigs[J]. Genet Sel Evol, 2021, 53(1): 79. DOI:10.1186/s12711-021-00670-x |
| [21] |
付川珂, 赵书红, 李新云, 等. 基于元共祖的基因组选择一步法理论及研究进展[J]. 中国畜牧杂志, 2021, 57(5): 1-6. FU C K, ZHAO S H, LI X Y, et al. Advances in the theories and applications of single-step genomic evaluation with metafounders[J]. Chinese Journal of Animal Science, 2021, 57(5): 1-6. (in Chinese) |
| [22] |
SARGOLZAEI M, SCHENKEL F S. QMSim: a large-scale genome simulator for livestock[J]. Bioinformatics, 2009, 25(5): 680-681. DOI:10.1093/bioinformatics/btp045 |
| [23] |
CHRISTENSEN O F, MADSEN P, NIELSEN B, et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012, 6(10): 1565-1571. DOI:10.1017/S1751731112000742 |
| [24] |
LOURENCO D, LEGARRA A, TSURUTA S, et al. Single-step genomic evaluations from theory to practice: using SNP chips and sequence data in BLUPF90[J]. Genes (Basel), 2020, 11(7): 790. DOI:10.3390/genes11070790 |
| [25] |
GARCIA-BACCINO C A, LEGARRA A, CHRISTENSEN O F, et al. Metafounders are related to Fst fixation indices and reduce bias in single-step genomic evaluations[J]. Genet Sel Evol, 2017, 49(1): 34. DOI:10.1186/s12711-017-0309-2 |
| [26] |
PATTERSON H D, THOMPSONR. Recovery of inter-block information when block sizes are unequal[J]. Biometrika, 1971, 58(3): 545-554. DOI:10.1093/biomet/58.3.545 |
| [27] |
DEMPSTER A P, LAIRD N M, et al. Maximum likelihood from incomplete data via the EM algorithm[J]. J R Stat Soc Series B Stat Methodol, 1977, 39(1): 1-22. |
| [28] |
JENSEN J, MÄNTYSAARI E A, MADSEN P, et al. Residual maximum likelihood estimation of (Co) variance components in multivariate mixedlinear models using average information[J]. J Indian Soc Agric Stat, 1997, 49: 215-236. |
| [29] |
KANG H, ZHOU L, MRODE R, et al. Incorporating the single-step strategy into a random regression model to enhance genomic prediction of longitudinal traits[J]. Heredity (Edinb), 2017, 119(6): 459-467. DOI:10.1038/hdy.2016.91 |
| [30] |
BRADFORD H L, MASUDA Y, VANRADEN P M, et al. Modeling missing pedigree in single-step genomic BLUP[J]. J Dairy Sci, 2019, 102(3): 2336-2346. DOI:10.3168/jds.2018-15434 |
| [31] |
VAN GREVENHOF E M, VANDENPLAS J, CALUS M P L. Genomic prediction for crossbred performance usingmetafounders[J]. J Anim Sci, 2019, 97(2): 548-558. DOI:10.1093/jas/sky433 |
| [32] |
MACEDO F L, CHRISTENSEN O F, ASTRUC J M, et al. Bias and accuracy of dairy sheep evaluations using BLUP and SSGBLUP with metafounders and unknown parent groups[J]. Genet Sel Evol, 2020, 52(1): 47. DOI:10.1186/s12711-020-00567-1 |
| [33] |
KUDINOV A A, MÄNTYSAARI E A, AAMAND G P, et al. Metafounder approach for single-step genomic evaluations of Red Dairy cattle[J]. J Dairy Sci, 2020, 103(7): 6299-6310. DOI:10.3168/jds.2019-17483 |
| [34] |
XIANG T, CHRISTENSEN O F, LEGARRA A. Technical note: genomic evaluation for crossbred performance in a single-step approach with metafounders[J]. J Anim Sci, 2017, 95(4): 1472-1480. |
(编辑 郭云雁)



