2. 北京市畜牧总站,北京 100101
2. Beijing General Station of Animal Husbandry, Beijing 100101, China
玻璃化冷冻是一种在高浓度的冷冻保护剂的作用下,以极快的速度将细胞温度降至-196 ℃,防止冰晶的形成,从而减少毒性、渗透损伤和冷冻伤害的冷冻保存方法[1-2]。相较于常规冷冻方法而言,玻璃化冷冻因其冷冻效果更好,已成为卵母细胞冷冻保存的首选方法[3]。同时,哺乳动物卵母细胞玻璃化冷冻能够跨越时间和空间的限制,实现家畜胚胎体外生产和移植更大范围的推广应用,对于濒危动物和优秀品种种质资源保存有着重要意义[4]。卵母细胞玻璃化冷冻还可以为那些愿意推迟生育的夫妇或过早失去卵巢功能的女性扩大生育选择范围[5]。
但是,玻璃化冷冻卵母细胞的发育潜力问题仍待解决。大量研究证实,在配子成熟、合子基因组激活[6]以及胚胎着床前发育过程中,表观遗传[7]发挥着重要作用。DNA甲基化的动态变化可能会影响早期细胞命运的决定[8],同时DNA甲基化的异常重编程可能导致发育缺陷和胚胎停滞[9]。前人研究表明,玻璃化冷冻会对卵母细胞的表观遗传状态产生影响。玻璃化冷冻小鼠卵母细胞体外受精(in vitro fertilization,IVF) 囊胚中H19、Peg3和Snrpn的DNA甲基化水平降低[10],玻璃化冷冻牛MII期卵母细胞的整体DNA甲基化水平和H3K9me3水平降低、而H3K9ac水平增加[11]。在本课题组的前期研究中,对体内囊胚和新鲜、玻璃化冷冻卵母细胞IVF囊胚进行了全基因组甲基化测序,结果表明,与体内组和新鲜组相比,冷冻组中IGF2R基因的甲基化水平显著降低((18.76±2.77)%、(15.04± 6.64)% vs. (9.41±3.52)%,P<0.05)[12]。IGF2R是IGF2的受体,是一个印记基因,在调节早期胎儿正常生长[13]、IGF2循环水平和心发育中起着至关重要的作用[14]。绵羊体外生产胚胎IGF2R基因印迹丢失会直接导致其后代出现胎盘和胚胎过度生长的现象,即巨胎症(large offspring syndrome,LOS)[15]。鉴于IGF2R在胚胎发育过程中的重要作用,如何精确调控玻璃化冷冻卵母细胞IVF囊胚中IGF2R的甲基化水平,使之恢复正常,成为了当前需要解决的问题。
CRISPR/Cas9基因编辑技术自2012年诞生以来已经在基因精确敲除/敲入领域得到了广泛的应用[16]。随后,研究人员将Cas9蛋白结构域中引入突变,使得蛋白活性发生缺陷,生成了dCas9 (dead Cas9)蛋白[17],其可以通过融合不同蛋白产生多种衍生工具。DNMT3A是DNA从头甲基转移酶,其功能独立于复制并在未甲基化和/或半甲基化DNA的CpG位点启动甲基化[18]。2016年,研究人员将dCas9蛋白与DNMT3A相融合,实现了对靶基因位点甲基化水平的精准调控[19-21]。2017年,Huang等[22]利用与重复肽表位(SunTag) 融合的dCas9蛋白募集多份抗体融合的DNMT3A (dCas9-SunTag-DNMT3A),增加了局部DNMT3A浓度,并最终增强了目标位点的CpG甲基化。2019年,研究人员将dCas9-DNMT mRNA注射进入小鼠卵母细胞,成功提高了靶基因的DNA甲基化水平,并获得了后代[23]。
综上,本试验拟采用dCas9-SunTag-DNMT3A DNA甲基化调控技术,对玻璃化冷冻牛卵母细胞IVF囊胚中IGF2R的甲基化水平进行精准调增,使其甲基化水平恢复正常。本研究结果将为冷冻卵母细胞/胚胎的特定位点DNA甲基化的精确调控提供理论基础和技术支撑。
1 材料与方法 1.1 试剂和仪器若无特殊说明,文中所提到的试剂均采购自Sigma公司(美国)。
1.2 卵母细胞采集和体外成熟将屠宰场来源的牛卵巢在2 h内运送到实验室,并使用提前热好的生理盐水(含双抗)清洗2~3次去除表面杂物。选取卵巢上直径约为2~8 mm的卵泡,利用真空泵抽取卵泡液于15 mL离心管中。静置,吸取沉淀于90 mm的培养皿中,在显微镜下选择符合培养要求的卵丘卵母细胞复合体(COCs),在洗卵液中清洗2次,成熟液中清洗2~3次后,转入装有成熟液的4孔板中,在含有5% CO2,湿度为100%,温度为38.5 ℃的恒温培养箱中培养22~24 h。
1.3 卵母细胞玻璃化冷冻及解冻在Hou等[24]的玻璃化冷冻和解冻程序上略作修改。冷冻程序:保持室温在25 ℃左右,使试验用具和试剂平衡充分,并在恒温工作台(38~39 ℃)上进行试验。用OPS管将成熟好的卵母细胞移入预处理液(10% EG + 10% DMSO)中平衡30 s后,移入冷冻液中(EDFSF40)平衡25 s,然后将卵母细胞重新吸入到OPS管中,直接投入液氮中进行冷冻。
解冻程序:将OPS管从液氮中取出,立刻将卵母细胞吹入到0.25 mol·L-1蔗糖解冻液中平衡1 min,然后转移入0.15 mol·L-1蔗糖解冻液中平衡5 min,最后再将卵母细胞在IVM液中洗涤2次,选取符合要求的卵母细胞备用。
1.4 卵母细胞IVFIVF步骤借鉴Brackett和Oliphant[25]的方法,稍作修改。将冻精从液氮罐中取出后,在38 ℃的水浴锅中进行解冻,后移入超净台,将精液加入到含有7 mL洗精液的离心管中,离心(1 500 r·min-1, 5 min),去上清,重复上述操作一次。离心结束后,加入受精液,调整精液密度为5×106个·mL-1,随后取20 μL精液与80 μL受精液相混合成100 μL受精滴,使精子终密度为1×106·mL-1,放入培养箱(38.5 ℃、5%CO2、100%湿度)中平衡1.5 h。将新鲜、冷冻的卵母细胞移入受精滴中,在恒温培养箱中培养16~18 h进行体外受精。受精完成后,脱去卵丘细胞,放入胚胎前期培养液中培养48 h,计算卵裂率,随后将卵裂的胚胎移入胚胎后期液中培养,并每隔48 h进行半量换液,在受精后第7天统计囊胚率。
1.5 dCas9-SunTag-DNMT3A和gRNA制备1.5.1 构建dCas-GCN、scFv-DNMT3A基因真核表达质粒和体外转录质粒 通过PCR将dCas-GCN片段从pPlatTET-gRNA2 (addgene#82559)扩增下来,并通过胶回收纯化获得dCas-GCN片段。之后将dCas-GCN片段、质粒scFv-sfGFP-DNMT3A (addgene#102278)通过EcoR I/Not I双酶切链接在pcDNA3.1(+)上,成为pcDNA3.1-dCas-GCN和pcDNA3.1-scFv-DNMT3A。
1.5.2 体外转录dCas-GCN、scFv-DNMT3A mRNA 采用KOD plus-neo高保真扩增PCR体系,从第1步中的质粒上扩增下mRNA的DNA模板,随后采用NEB公司的T7 RNA聚合酶试剂盒(#E2040)。反应时需加入各1.5 mmol·L-1的ATP、UTP、GTP、CTP和带有T7启动子的模板DNA 1 μg,在37 ℃温育16 h。最后加入DNAase 1 μL,在37 ℃温育0.5 h。最后使用Trizol (Invitrogen)试剂抽提纯化。
1.5.3 设计牛IGF2R基因的启动子区sgRNA 从UCSC查找出牛IGF2R基因的启动子区序列,在网站设计sgRNA靶点,设计并合成引物。上游引物T7-gIGF2R F(5′-3′):ATGACTCAACTCTTCGCATG;下游引物gRNA-ovlap R(5′-3′): CTTGCTATTTCTAGCTCTAAAAC。
1.5.4 合成牛IGF2R基因启动子区gRNA的模板和体外转录 将上述上、下游引物100 μmol·L-1各3 μL,于Taq酶buffer中混匀(30 μL体系),在沸水中与水一起慢慢降温,实现退火。3 h后,加3 μL dNTP、1 μL Taq酶,于72 ℃水浴15 min。用康为Cleanup试剂盒纯化,测定浓度。体外转录gIGF2R,方法同上。
1.6 显微注射采用显微操作仪将dCas9- DNMT3A mRNA (20、40、60 ng·μL-1)和sgRNA(20、40、60 ng·μL-1) 注射到受精后的原核胚的胞质中。注射完成后,受精卵放入胚胎培养液中继续培养。48 h后,将卵裂的胚胎放到胚胎培养液+10% FBS中继续培养120 h,每隔48 h进行半量换液。最后,收集获得的囊胚进行DNMT3A甲基化模式分析,检验dCas9- DNMT3A的DNA甲基化水平调增效果。
1.7 甲基化检测1.7.1 基因组DNA的提取和亚硫酸氢盐处理 利用EZ DNA Mthylation-DirectTMKit试剂盒说明书提取胚胎DNA,随后进行DNA的亚硫酸氢盐修饰及纯化。
1.7.2 亚硫酸氢盐聚合酶链式反应(BSP-PCR)引物设计及合成 本试验是针对IGF2R基因的启动子区进行特定甲基化修饰,所以引物是根据启动子区的序列进行设计,第一步,获得IGF2R基因的启动子区序列。第二步,使用Methylation primer网站得到基因的甲基化序列,并设计甲基化引物。第三步,使用Snapgene找到引物对应的参考序列并合成。
1.7.3 亚硫酸氢盐聚合酶链式反应(BSP-PCR) 采用25 μL的PCR体系进行扩增,模板为1 μL经过亚硫酸氢盐修饰过后的DNA溶液,12.5 μL 2×EasyTaq PCR SuperMix (TRAN),表 1中上、下游引物各1 μL,补足水至总反应体积25 μL。反应条件为94 ℃预变性5 min;然后于94 ℃ 30 s,55 ℃退火30 s,72 ℃延伸30 s,此过程循环35次;最后于72 ℃延伸7 min。
|
|
表 1 BSP-PCR引物 Table 1 BSP-PCR primers |
将BSP-PCR产物与PMD-19T Vector载体连接。Solution1 5 μL, PMD-19T载体0.5 μL, 线性载体4.5 μL,16 ℃孵育4 h。将连接好的重组载体转化到DH5α感受态细胞中,筛选阳性菌进行测序。
1.9 测序分析在超净台中用无菌枪头挑取单克隆菌落作为模板,以通用引物进行扩增,随后利用琼脂糖凝胶电泳进行检测,确定阳性克隆后进行测序。对测序结果进行比对,检验dCas9-DNMT3A的DNA甲基化水平调增效果。
1.10 实时荧光定量PCR(RT-qPCR)采用BIO-RAD(美国)CFX96TM实时荧光定量PCR仪进行定量分析。采用Oligo7软件进行引物设计,引物序列见表 2。试验采用15 μL反应体系:上、下游引物各0.5 μL,cDNA模板2 μL,TB Green Premix Ex Taq Ⅱ 7.5 μL,RNase free ddH2O 4.5 μL。反应程序:先95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 30 s,共计39个循环。以牛GAPDH为内参基因。
|
|
表 2 实时荧光定量PCR引物序列 Table 2 Primer sequences for RT-qPCR |
所有试验组至少进行3次以上的生物学重复,采用EXCEL 2010软件对囊胚细胞中目的基因和内参基因mRNA表达量进行处理,采用2-△△Ct法计算基因的相对表达量。采用SAS软件对所得数据进行显著性分析,结果以“平均值±标准差”的形式表示,P<0.05表示差异显著。
2 结果 2.1 sgRNA和dCas9-SunTag-DNMT3A注射对卵母细胞发育的影响如表 3所示,相较于冷冻组((50.92±4.53)%、(13.25±1.19)%)而言,20、40、60 ng·μL-1注射组的卵裂率((54.62±4.62)%、(61.18±5.36)%、(55.41±3.18)%)和囊胚率((14.08±0.95)%、(22.58±2.12)%、(15.85±1.42)%)都有所提高。同时,40 ng·μL-1组发育能力最好,但仍显著低于新鲜组((84.80±7.32)%、(40.57±3.28)%,P<0.05)。
|
|
表 3 外源注射sgRNA和dCas9-SunTag-DNMT3A对卵母细胞发育的影响 Table 3 Effects of exogenous injection of sgRNA and dCas9-SunTag-DNMT3A on oocytes development |
如表 4所示,与冷冻组相比,显微注射40 ng·μL-1 sgRNA和dCas9-SunTag-DNMT3A显著提高IGF2R启动子区域的甲基化水平((14.33±1.19)% vs. (9.33±0.56)%,P<0.05),与新鲜组无显著差异((15.67±1.23)%,P>0.05)。
|
|
表 4 IGF2R启动子甲基化水平 Table 4 IGF2R promoter methylation level |
本试验利用荧光定量PCR对经过40 ng·μL-1组调控后的玻璃化冷冻牛卵母细胞IVF囊胚中IGF2R、IFN-tau、CTNNBL1、OCT-4基因的表达水平进行检测。由图 1可以看出,40 ng·μL-1组中IGF2R的mRNA表达量显著低于冷冻组(P<0.05),与新鲜组相似(P>0.05)。同时,40 ng·μL-1组CTNNBL1和OCT-4的表达量显著高于冷冻组和新鲜组(P<0.05),IFN-tau表达量显著高于冷冻组(P<0.05),与新鲜组相似(P>0.05)。
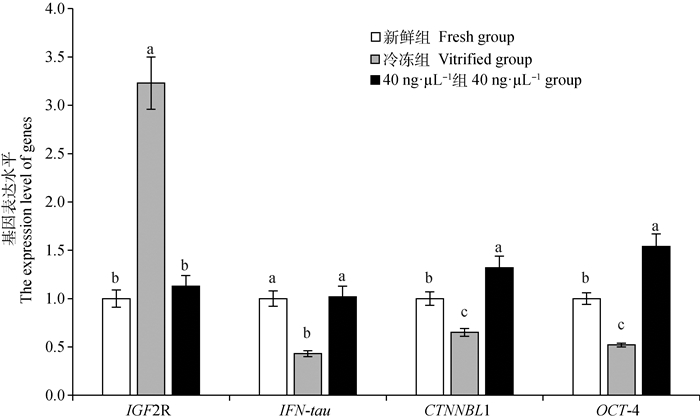
|
同组数据上标不同字母表示差异显著(P<0.05) Different letter superscripts in the same row means significant difference between the treatments (P < 0.05) 图 1 sgRNA和dCas9-SunTag-DNMT3A注射后IGF2R和发育相关基因的表达水平 Fig. 1 The expression level of IGF2R and development related genes after sgRNA and dCas9-SunTag-DNMT3A injection |
在哺乳动物基因组中,DNA甲基化是一种表观遗传机制,可以将甲基转移到胞嘧啶的C5位置以形成5-甲基胞嘧啶并通过招募参与基因抑制的蛋白质或通过抑制转录因子与DNA的结合来调节基因表达[26]。前期试验表明,玻璃化冷冻牛卵母细胞IVF囊胚中IGF2R基因的甲基化水平会异常降低[12]。查阅前人的试验也表明,卵母细胞玻璃化冷冻会导致胚胎的DNA甲基化水平降低[27],在单峰驼[28]、牛[11]和小鼠[29-30]卵母细胞中都得到过相似的结果。但在上述试验中,还未明确DNA甲基化水平异常降低是由冷冻损伤引起的还是由高浓度的抗冻剂化学毒性引起,故该部分内容还有待论证,需要进一步的研究。
有研究表明,受精卵显微注射不影响发育[31]。如表 3所示, 相较于冷冻对照组,显微注射dCas9- SunTag-DNMT3A和sgRNA均可有效提高玻璃化冷冻牛卵母细胞IVF胚胎的发育效果,其中40 ng·μL-1组的效果明显好于20和60 ng·μL-1组。本试验中,20 ng·μL-1组的注射浓度较低且没有达到显著的调控效果,这可能是由于编辑效率会随着注射剂量的增加而提高[32]。同时,60 ng·μL-1组调控效果也不理想,这可能是由于高浓度的dCas9 mRNA与gRNA显微共注射后具有一定的细胞毒性,会对细胞有一定的副作用,从而导致细胞的发育效果降低[33]。
至今,dCas9-SunTag-DNMT3A已被成功应用于甲基化精准调控中。2016年,Liu等[34]利用dCas9-SunTag-DNMT3A靶向两个CTCF结合位点,诱导了这些位点中CpG的从头甲基化,成功抑制了相关基因的表达,并干扰了相关蛋白质的功能。2017年,Huang等[22]证实,该系统具有甲基化效率高、脱靶效应等优点。本研究表明,40 ng·μL-1组中IGF2R启动子区域甲基化水平与新鲜组相似,显著高于冷冻组,这为冷冻卵母细胞IVF胚胎中基因甲基化的精准调控提供了有效参考。
DNA甲基化一般与基因表达水平抑制或沉默相关[34]。同时DNA甲基化在协调所有物种的IGF2R等位基因表达中起着至关重要的作用[35]。本研究表明,注射40 ng·μL-1的sgRNA和dCas9-SunTag-DNMT3A后,显著降低IGF2R基因的mRNA表达水平,这主要是由于调控系统升高了IGF2R基因启动子区域的甲基化水平。Hiura等[36]证实了母体印迹的建立与卵母细胞的生长有关。同时,IGF2R在动物胚胎发育、胎儿生长和疾病中发挥重要作用[37]。小鼠中IGF2R敲除导致胚胎过度生长[38]。IGF2R是IGF2的受体,IGF2基因编码一种在组织分化、胎儿生长中起关键作用的生长因子[39],综上所述,通过正向调节IGF2R基因的表达就能够对卵母细胞的生长发育起到积极的作用。前人研究表明,IFN-tau是衡量体外生产牛胚胎发育能力和质量的指标[40],CTNNBL1是囊胚滋养外胚层分化的重要标志物[41],OCT-4在建立和维持胚胎的多能性中发挥重要作用[42],三者的表达水平均能反映胚胎的发育情况和质量。本试验结果表明,40 ng·μL-1 sgRNA和dCas9-SunTag-DNMT3注射后囊胚中IFN-tau、CTNNBL1、OCT-4基因的表达水平上升,这可能是由于dCas9-SunTag-DNMT3A甲基化调控系统提高了IGF2R基因启动子的甲基化水平,降低了IGF2R基因的表达水平,使之恢复正常,从而提高了玻璃化冷冻牛卵母细胞IVF胚胎的发育能力,显著提高了体外生产胚胎的质量。
4 结论本研究发现,注射40 ng·μL-1 dCas9-SunTag-DNMT3A甲基化编辑系统后,成功提高了玻璃化冷冻牛卵母细胞IVF囊胚中IGF2R基因启动子甲基化水平(P<0.05)并降低了其mRNA表达水平(P<0.05),使得玻璃化冷冻卵母细胞IVF胚胎的发育能力得到显著提高。本研究结果将为玻璃化冷冻卵母细胞的广泛、安全应用奠定基础,具有十分重要的经济价值和社会效益。
| [1] |
RIENZI L, GRACIA C, MAGGIULLI R, et al. Oocyte, embryo and blastocyst cryopreservation in ART: systematic review and meta-analysis comparing slow-freezing versus vitrification to produce evidence for the development of global guidance[J]. Human Reprod Update, 2017, 23(2): 139-155. |
| [2] |
DUJČKOVÁ L, MAKAREVICH A V, OLEXIKOVÁ L, et al. Methodological approaches for vitrification of bovine oocytes[J]. Zygote, 2021, 29(1): 1-11. DOI:10.1017/S0967199420000465 |
| [3] |
LEVI-SETTI P E, PATRIZIO P, SCARAVELLI G. Evolution of human oocyte cryopreservation: slow freezing versus vitrification[J]. Curr Opin Endocrinol Diabetes Obes, 2016, 23(6): 445-450. DOI:10.1097/MED.0000000000000289 |
| [4] |
QUAAS A M, PENNINGS G. The current status of oocyte banks: domestic and international perspectives[J]. Fertil Steril, 2018, 110(7): 1203-1208. DOI:10.1016/j.fertnstert.2018.07.013 |
| [5] |
NAGY Z P, SHAPIRO D, CHANG C C. Vitrification of the human embryo: a more efficient and safer in vitro fertilization treatment[J]. Fertil Steril, 2020, 113(2): 241-247. DOI:10.1016/j.fertnstert.2019.12.009 |
| [6] |
ECKERSLEY-MASLIN M A, ALDA-CATALINAS C, REIK W. Dynamics of the epigenetic landscape during the maternal-to-zygotic transition[J]. Nat Rev Mol Cell Biol, 2018, 19(7): 436-450. DOI:10.1038/s41580-018-0008-z |
| [7] |
REIK W, KELSEY G. Epigenetics: cellular memory erased in human embryos[J]. Nature, 2014, 511(7511): 540-541. DOI:10.1038/nature13648 |
| [8] |
RULANDS S, LEE H J, CLARK S J, et al. Genome-scale oscillations in DNA methylation during exit from pluripotency[J]. Cell Syst, 2018, 7(1): 63-76.e12. DOI:10.1016/j.cels.2018.06.012 |
| [9] |
XU R M, LI C, LIU X Y, et al. Insights into epigenetic patterns in mammalian early embryos[J]. Protein Cell, 2021, 12(1): 7-28. DOI:10.1007/s13238-020-00757-z |
| [10] |
CHENG K R, FU X W, ZHANG R N, et al. Effect of oocyte vitrification on deoxyribonucleic acid methylation of H19, Peg3, and Snrpn differentially methylated regions in mouse blastocysts[J]. Fertil Steril, 2014, 102(4): 1183-1190.e3. DOI:10.1016/j.fertnstert.2014.06.037 |
| [11] |
CHEN H H, ZHANG L, DENG T F, et al. Effects of oocyte vitrification on epigenetic status in early bovine embryos[J]. Theriogenology, 2016, 86(3): 868-878. DOI:10.1016/j.theriogenology.2016.03.008 |
| [12] |
ZHAO Y H, WANG J J, ZHANG P P, et al. Oocyte IVM or vitrification significantly impairs DNA methylation patterns in blastocysts as analysed by single-cell whole-genome methylation sequencing[J]. Reprod Fertil Dev, 2020, 32(7): 676-689. DOI:10.1071/RD19234 |
| [13] |
LE F, WANG L Y, WANG N, et al. In vitro fertilization alters growth and expression of Igf2/H19 and their epigenetic mechanisms in the liver and skeletal muscle of newborn and elder mice[J]. Biol Reprod, 2013, 88(3): 75. |
| [14] |
LAU M M, STEWART C E, LIU Z, et al. Loss of the imprinted IGF2/cation-independent mannose 6-phosphate receptor results in fetal overgrowth and perinatal lethality[J]. Genes Dev, 1994, 8(24): 2953-2963. DOI:10.1101/gad.8.24.2953 |
| [15] |
YOUNG L E, FERNANDES K, MCEVOY T G, et al. Epigenetic change in IGF2R is associated with fetal overgrowth after sheep embryo culture[J]. Nat Genet, 2001, 27(2): 153-154. DOI:10.1038/84769 |
| [16] |
JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [17] |
QI L S, LARSON M H, GILBERT L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [18] |
OKANO M, BELL D W, HABER D A, et al. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development[J]. Cell, 1999, 99(3): 247-257. DOI:10.1016/S0092-8674(00)81656-6 |
| [19] |
VOJTA A, DOBRINI Ć P, TADI Ć V, et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation[J]. Nucleic Acids Res, 2016, 44(12): 5615-5628. DOI:10.1093/nar/gkw159 |
| [20] |
MCDONALD J I, CELIK H, ROIS L E, et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation[J]. Biol Open, 2016, 5(6): 866-874. DOI:10.1242/bio.019067 |
| [21] |
MARX N, GRVNWALD-GRUBER C, BYDLINSKI N, et al. CRISPR-based targeted epigenetic editing enables gene expression modulation of the silenced beta-galactoside alpha-2, 6-sialyltransferase 1 in CHO cells[J]. Biotechnol J, 2018, 13(10): e1700217. DOI:10.1002/biot.201700217 |
| [22] |
HUANG Y H, SU J Z, LEI Y, et al. DNA epigenome editing using CRISPR-cas suntag-directed DNMT3A[J]. Genome Biol, 2017, 18(1): 176. DOI:10.1186/s13059-017-1306-z |
| [23] |
WEI Y C, LANG J W, ZHANG Q, et al. DNA methylation analysis and editing in single mammalian oocytes[J]. Proc Natl Acad Sci U S A, 2019, 116(20): 9883-9892. DOI:10.1073/pnas.1817703116 |
| [24] |
HOU Y P, DAI Y P, ZHU S E, et al. Bovine oocytes vitrified by the open pulled straw method and used for somatic cell cloning supported development to term[J]. Theriogenology, 2005, 64(6): 1381-1391. DOI:10.1016/j.theriogenology.2005.03.012 |
| [25] |
BRACKETT B G, OLIPHANT G. Capacitation of rabbit spermatozoa in vitro[J]. Biol Reprod, 1975, 12(2): 260-274. DOI:10.1095/biolreprod12.2.260 |
| [26] |
MOORE L D, LE T, FAN G P. DNA methylation and its basic function[J]. Neuropsychopharmacology, 2013, 38(1): 23-38. DOI:10.1038/npp.2012.112 |
| [27] |
HU W H, MARCHESI D, QIAO J, et al. Effect of slow freeze versus vitrification on the oocyte: an animal model[J]. Fertil Steril, 2012, 98(3): 752-760.e3. DOI:10.1016/j.fertnstert.2012.05.037 |
| [28] |
MOULAVI F, SAADELDIN I M, SWELUM A A, et al. Oocyte vitrification induces loss of DNA methylation and histone acetylation in the resulting embryos derived using ICSI in dromedary camel[J]. Zygote, 2021, 29(5): 383-392. DOI:10.1017/S0967199421000150 |
| [29] |
FU L Z, CHANG H Y, WANG Z Q, et al. The effects of TETs on DNA methylation and hydroxymethylation of mouse oocytes after vitrification and warming[J]. Cryobiology, 2019, 90: 41-46. DOI:10.1016/j.cryobiol.2019.09.001 |
| [30] |
LIANG Y, FU X W, LI J J, et al. DNA methylation pattern in mouse oocytes and their in vitro fertilized early embryos: effect of oocyte vitrification[J]. Zygote, 2014, 22(2): 138-145. DOI:10.1017/S0967199412000512 |
| [31] |
CRISPO M, MULET A P, TESSON L, et al. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes[J]. PLoS One, 2015, 10(8): e0136690. DOI:10.1371/journal.pone.0136690 |
| [32] |
FOGARTY N M E, MCCARTHY A, SNIJDERS K E, et al. Genome editing reveals a role for OCT4 in human embryogenesis[J]. Nature, 2017, 550(7674): 67-73. DOI:10.1038/nature24033 |
| [33] |
WANG H Y, YANG H, SHIVALILA C S, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [34] |
LIU X S, WU H, JI X, et al. Editing DNA methylation in the mammalian genome[J]. Cell, 2016, 167(1): 233-247.e17. DOI:10.1016/j.cell.2016.08.056 |
| [35] |
VU T H, JIRTLE R L, HOFFMAN A R. Cross-species clues of an epigenetic imprinting regulatory code for the IGF2R gene[J]. Cytogenet Genome Res, 2006, 113(1-4): 202-208. DOI:10.1159/000090833 |
| [36] |
HIURA H, OBATA Y, KOMIYAMA J, et al. Oocyte growth-dependent progression of maternal imprinting in mice[J]. Genes Cells, 2006, 11(4): 353-361. DOI:10.1111/j.1365-2443.2006.00943.x |
| [37] |
ZHOU Q, MENG Q R, MENG T G, et al. Deletion of BAF250a affects oocyte epigenetic modifications and embryonic development[J]. Mol Reprod Dev, 2020, 87(5): 550-564. DOI:10.1002/mrd.23339 |
| [38] |
BRESSAN F F, DE BEM T H C, PERECIN F, et al. Unearthing the roles of Imprinted genes in the Placenta[J]. Placenta, 2009, 30(10): 823-834. DOI:10.1016/j.placenta.2009.07.007 |
| [39] |
CAMPAGNOLO K, ONGARATTO F L, DE FREITAS C R, et al. In vitro development of IVF-derived bovine embryos following cytoplasmic microinjection for the episomal expression of the IGF2 gene[J]. Reprod Domest Anim, 2020, 55(5): 574-583. DOI:10.1111/rda.13654 |
| [40] |
YAO N, WAN P C, HAO Z D, et al. Expression of interferon-tau mRNA in bovine embryos derived from different procedures[J]. Reprod Domest Anim, 2009, 44(1): 132-139. DOI:10.1111/j.1439-0531.2007.01009.x |
| [41] |
MAMO S, MEHTA J P, MCGETTIGAN P, et al. RNA sequencing reveals novel gene clusters in bovine conceptuses associated with maternal recognition of pregnancy and implantation[J]. Biol Reprod, 2011, 85(6): 1143-1151. DOI:10.1095/biolreprod.111.092643 |
| [42] |
PESCE M, SCHÖLER H R. Oct-4:gatekeeper in the beginnings of mammalian development[J]. Stem Cells, 2001, 19(4): 271-278. DOI:10.1634/stemcells.19-4-271 |
(编辑 郭云雁)



