2. 昆山市畜牧兽医站,苏州 215300;
3. 南京农业大学,南京 210095
2. Animal Husbandry and Veterinary Station of Kunshan City, Suzhou 215300, China;
3. Nanjing Agricultural University, Nanjing 210095, China
遗传资源是种业科技原始创新、现代种业持续发展的源泉以及国家发展的重要战略资源[1]。我国鸭遗传资源丰富,现有品种数量约占世界的48%左右[2]。其中娄门鸭是江苏省著名的地方品种,原产于昆山市,以肉质优良而闻名。苏州历史悠久的熟食鸭制品大多均选用苏州当地产的膘肥、体大、肉嫩、皮白的娄门鸭为原料。娄门鸭历史上饲养量较大,解放前年产量达到100多万只,远销四周县市。由于受到外来品种的冲击,自上世纪60年代开始饲养量逐年降低,从2008年开始昆山市农业农村局与江苏省家禽科学研究所合作进行娄门鸭抢救性保护工作[3]。从2013年开始新建娄门鸭保种场进行系统保种以来,尚未全面评估娄门鸭的保种效果。因此对娄门鸭的遗传多样性的评价工作迫在眉睫。
遗传多样性的评估可靠性依赖于遗传标记的质量。目前DNA分子遗传标记可以从DNA水平上直接反映个体之间的差异,稳定性高、多态性好[4],因此是目前最常用、应用范围最广的一类有效标记。其中代表性的DNA分子遗传标记有SSR(简单重复序列的微卫星标记)和SNP(单核苷酸多态性)。微卫星标记由于多态性较高[5],在遗传多样性的早期研究中得到了大量应用[6-10]。虽然单个SNP的多态性较微卫星低,通常为二等位标记,但全基因组范围内的SNPs数量多、分布广[11-13],检测稳定性和判定准确率都较高,易实现标准化和自动化检测[14-15],因此SNP检测方法在遗传多样性评估中越来越受到重视。随着二代高通量技术的发展,基于酶切的简化基因组技术[16-17]能够得到全基因组范围内足够数量的SNPs位点,且成本较基因组重测序大幅降低[18-19],因此成为分析群体遗传多样性和亲缘关系,评估种群近交系数的理想方法[20-21],并在不同物种上都得到了大量应用[22-26]。
本研究通过简化基因组测序技术检测娄门鸭保种群全基因组范围内的SNPs标记,系统分析基于SNPs标记的群体遗传多样性,鉴定保种群的群体结构,评估娄门鸭群体的保种效果,为后续娄门鸭这一宝贵遗传资源保护方案的修正提供科学合理的指导和建议。
1 材料与方法 1.1 试验动物及样品采集娄门鸭保种群来自江苏省昆山市娄门鸭原种场,所有个体饲养标准和环境条件一致。10周龄时随机选取同一世代163只鸭(39只公鸭,124只母鸭),翅静脉采血后血样冷冻保存。
1.2 试验方法1.2.1 基因组DNA的提取 采用酚-氯仿法提取血样基因组DNA。冻存后的每管血样解冻后吸取100 μL,加入900 μL SDS裂解液和20 μL蛋白酶K后摇匀,过夜消化后,加入900 μL酚、氯仿、异戊醇(25∶24∶1)混合液萃取基因组DNA,加入无水乙醇后沉淀基因组DNA,最后加入TE缓冲液溶解基因组DNA,-20 ℃保存备用。
1.2.2 基因组DNA的质量鉴定 通过核酸蛋白浓度测定仪测量基因组DNA的浓度和OD值,采用琼脂糖凝胶电泳检测基因组DNA的完整性。质量合格后进行下一步简化基因组测序。
1.2.3 简化基因组测序 所有基因组DNA样品均委托广州基迪奥生物科技有限公司进行简化基因组测序。具体试验流程为:选择EcoR Ⅰ和Nla Ⅲ限制性内切酶对基因组DNA酶切后加上带barcode的测序接头; 混合样本后,构建小片段文库(250~550 bp),进行PE125双末端测序。
1.3 数据处理1.3.1 数据质控与基因组比对 为了保证测序数据的质量,首先对下机的clean reads进行过滤:1)去除含adapter的reads; 2)去除含N比例大于10%的reads; 3)去除低质量reads(质量值Q≤10的碱基数占整条read的50%以上)。使用bwa软件(v0.7.12)[27]中的mem算法将过滤后的reads比对到鸭参考基因组IASCAAS_PekingDuck_PBH1.5(https://www.ncbi.nlm.nih.gov/assembly/GCA_003850225.1)上,比对参数为-k 32 -M。比对完成之后利用plink软件(v1.9)[28]对SNPs位点质控,筛除不合格的样品和SNPs位点。质控标准如下:1)只使用常染色体上的位点; 2)最小等位基因频率(MAF)>0.05;3)哈代-温伯格平衡检验的P值>10-6; 4)每个样品的SNPs检出率>90%。
1.3.2 群体遗传多样性分析 根据基因组上不同SNPs位点的分型结果,利用R软件[29]计算群体遗传多样性不同指标,包括多态信息含量(polymorphism information content,PIC)、固定指数(Fix-index)、基因多样性指数(Nei)、有效等位基因数(Ne)以及香农信息指数(Shannon-Weiner index,SHI)。4种遗传多样性指标的计算公式:
| $ P I C=1-p^{2}-q^{2}-2 p^{2} q^{2} $ | (公式1[30]) |
| $ Fix \text { -index }=\frac{H e-H o}{H e} $ | (公式2[31]) |
| $ N e i=\frac{n \times H e}{n-1} $ | (公式3[32]) |
| $ N e=\frac{1}{p^{2}+q^{2}} $ | (公式4[33]) |
| $ S H I=-(p \ln p+q \ln q) $ | (公式5[34]) |
其中,p和q分别表示某个SNP位点两个等位基因的频率; He表示期望杂合度,Ho表示观测杂合度; n表示样品个体数。
对于二等位基因的单个SNP标记而言,当p=q=0.5时,PIC值最大,其值为0.375。定义单个SNP标记的相对PIC(rPIC)为以0.375为基准的PIC值,即:
| $ r P I C=P I C / 0.375 $ | (公式6) |
1.3.3 群体结构分析 利用plink软件(v1.9)和admixture软件(v1.3)[35]分析群体遗传结构。利用筛选后得到的SNPs标记,假设样品的分群数(K值)为1~9,分别聚类分析。根据交叉验证错误率(cross-validation error,CV error)确定最优分群数。使用R包pophelper[36]将每个样本在各个亚群的遗传组成绘制成柱形图。使用gcta软件(v1.92.2)[37]分析群体主成分以及个体间的亲缘关系,获得PCA结果后使用R软件绘制前2个主成分的散点图。
1.3.4 ROH分布及近交系数评估 利用plink软件(v1.9)检测基因组上不同滑动窗口的ROHs,并统计检测到的ROH长度及其不同染色体上的密度。
基于ROH评估近交系数是利用全基因组分子信息评价保种效果的一种方法,即利用ROH计算基因组近交系数FROH,计算方法:
| $ F_{{ROH}}=\sum \frac{L_{{ROH}}}{L_{{AUTO}}} $ | (公式7) |
其中,当计算个体FROH时,LRHO为基因组上ROHs的总长度,LAUTO为基因组的总长度; 当计算染色体FROH时,LROH为单一染色体ROHs的总长度,LAUTO为该染色体的总长度。
2 结果 2.1 基因组DNA检测使用酚-氯仿法对娄门鸭血样提取基因组DNA后,检测所有DNA样品的浓度和OD值。结果显示,每份DNA样品浓度均在50 ng·μL-1,OD260 nm/OD280 nm均在1.80以上,表明DNA样品的浓度和纯度均符合要求。对基因组DNA样品进行琼脂糖凝胶电泳,结果显示DNA条带清晰,无拖尾现象,亮度均一,表明基因组DNA样品质量较好,无蛋白质污染以及降解现象(图 1),可用于后续基因组测序。
|
M. DNA相对分子质量标准; 1~8. 基因组DNA M. DL2000 marker; 1-8. Genomic DNA 图 1 基因组DNA的琼脂糖凝胶电泳图 Fig. 1 The agarose gel electrophoresis of genomic DNA |
对测序下机数据进行clean reads质控后,公鸭群体平均每个个体得到3.59 M高质量reads,母鸭群体平均得到4.38 M高质量reads,与鸭基因组比对后,公、母鸭高质量reads的比对率均达到97%以上(表 1),数据质量达到后续分析的要求。
|
|
表 1 测序后的reads在鸭基因组中的比对情况 Table 1 Mapping quality of reads after sequencing to duck genome |
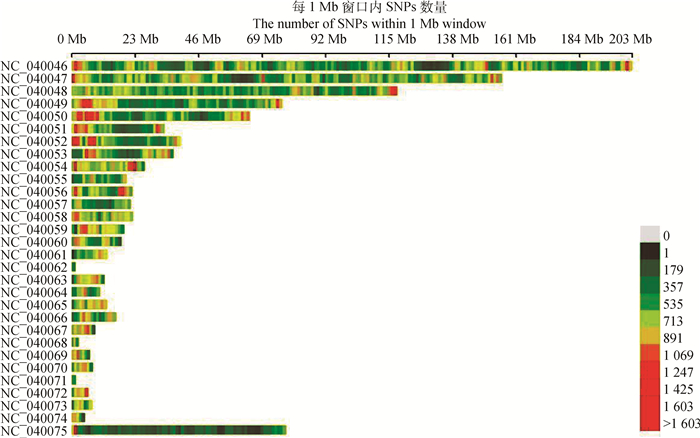
将高质量reads与鸭基因组比对后,共得到622 205个SNPs位点。使用plink软件对获得的SNPs进行质控,最终获得374 455个高质量的SNPs位点。这些SNPs位点在不同染色体上的分布呈现明显差异,其中在NC_040046、NC_040047、NC_040048和NC_040049四个染色体上的分布比例达到50.70%(图 2)。
|
图 2 SNPs在不同染色体上的密度分布图 Fig. 2 Density distribution of SNPs on different chromosomes |
基于SNPs位点的测序分型数据,计算不同SNPs位点对应等位基因频率,并统计不同染色体的遗传多样性指标分布情况。多态信息含量PIC在整个基因组上的分布范围在0.001 5~0.375 0之间,其中34.08% SNPs位点的PIC大于0.25,为中度多态性位点。不同染色体上PIC的分布规律相似,均呈现“两端分布集中,中间分布稀少”的状态(图 3A)。基因多样性指数Nei(图 3C)、有效等位基因数Ne(图 3D)、香农信息指数SHI(图 3E)的分布规律与PIC相似,从这几个指标之间的相关性(表 2)也可以看出,它们的皮尔逊相关系数都在0.97以上,甚至达到0.99。固定指数(Fix-index)与PIC等4个指标的分布规律明显不同,与其他4个指标的皮尔逊相关系数只有0.20左右。具体到各个染色体,Fix-index除了在NC_040075染色体上呈现两端集中分布外,其他29个染色体均偏向左侧分布(图 3B),这也导致NC_040075染色体上的Fix-index值大于其他染色体。

|
图 3 不同遗传多样性指标在染色体上的分布规律 Fig. 3 Distribution of different genetic diversity indexes in chromosomes |
|
|
表 2 不同遗传多样性指标之间的相关性 Table 2 Correlations between different genetic diversity indexes |
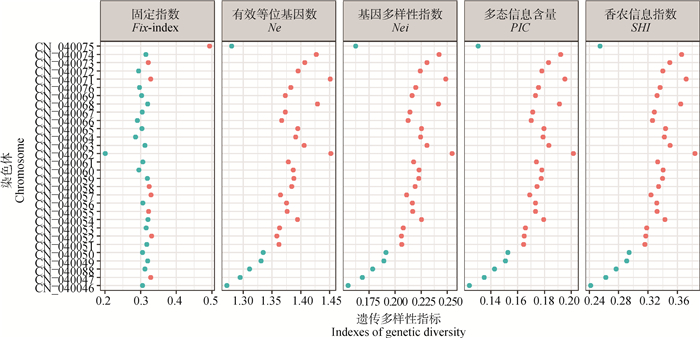
PIC、Nei、Ne、SHI 4个遗传多样性指标在整个基因组上的均值分别为0.154 1、0.192 9、1.336、0.296 9,且不同指标在各染色体上的均值变化规律一致(图 4)。NC_040046、NC_040047、NC_040048、NC_040049、NC_040050、NC_040075等6个染色体上PIC、Nei、Ne、SHI 4个指标均小于各自的基因组均值,且NC_040046染色体上的4个遗传多样性指标均最小,分别为0.124 0、0.155 1、1.271 0、0.242 0。NC_040062染色体上的4个遗传多样性指标均最大,分别为0.201 6、0.254 6、1.452 1、0.383 7。Fix-index与其他4个指标有较大差异,其基因组上的总体均值为0.320 8,除了Fix-index值最小(0.201 4)的NC_040062染色体以及值最大(0.492 6)的NC_040075染色体外,其他28个染色体的Fix-index值较为接近,均在基因组总体均值上下波动。
|
图中红色点表示该染色体的遗传多样性指标高于基因组均值,绿色表示低于基因组均值 The red dots in the figure indicate that the genetic diversity indexes of the chromosome is higher than the genomic mean, while the green indicates lower 图 4 不同染色体上的遗传多样性指标比较 Fig. 4 Comparison of genetic diversity indexes on different chromosomes |
根据PIC公式(公式1),SNP标记作为二等位基因,其最大PIC值为0.375。以0.375为基准值,计算SNP标记的rPIC,统计娄门鸭保种群rPIC的分布特征。结果显示,娄门鸭保种群相对PIC值在0.004 0~1.000 0之间,整个基因组上平均PIC为0.410 9。根据PIC指标划分低度(小于0.25)、中度(0.25~0.50)、高度多态性位点(大于0.50)的标准,娄门鸭保种群9.30%的SNPs位点属于中度多态性位点,38.80%的SNPs位点属于高度多态性位点。
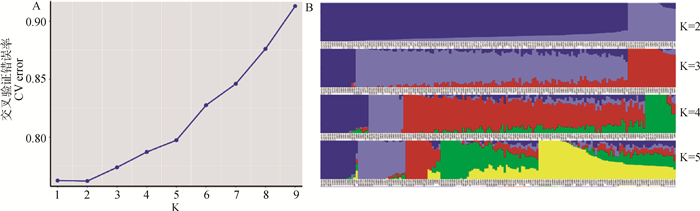
2.4 娄门鸭保种群的群体结构分析使用admixture软件分析163只鸭群体的遗传结构,根据Cross-validation error最小化原则确定最佳K值为2(图 5A)。当K=2时,娄门鸭群体分为两个相对独立的亚群,分别有141个个体和22个个体; 当K=3时,娄门鸭群体分为3个相对分明的亚群,分别由16个个体、125个个体和22个个体组成(图 5B)。当K=4、5或之后更大数值时,娄门鸭亚群划分更为复杂,且离K=2的最佳K值较远,群体划分与实际结果有较大偏差。
|
图 5 娄门鸭保种群体的admixture遗传结构图 Fig. 5 Genetic structure of Loumen duck conservation population by admixture |
使用GCTA软件分析群体的主成分,结果表明,使用主成分1和主成分2可以将163只娄门鸭个体明显分为3个聚类簇(图 6)。其中左上角A亚群包含22个个体(2公, 20母),右上角B亚群包含125个个体(35公, 90母),右下角C亚群包含16个个体(2公, 14母)。该3个亚群的个体组成与admixture分析中K=3时的亚群分组结果完全一致。
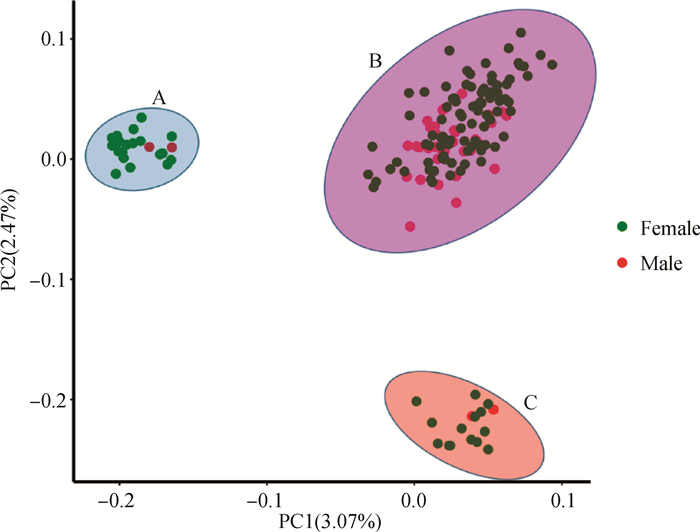
|
图 6 基于SNPs标记位点的娄门鸭保种群主成分分析图 Fig. 6 PCA graph of Loumen duck conservation population based on SNPs loci |
基于过滤后的标记,使用GCTA软件分析个体间的亲缘关系,获得两两样本间的亲缘关系矩阵。基于亲缘关系矩阵分析发现,娄门鸭保种群大部分个体之间亲缘关系系数较低(图 7A)。根据亲缘关系热图(图 7B),可将娄门鸭保种群分为3个亚群,分别为左上角22个个体组成的亚群、右下角16个个体组成的亚群以及剩余125个个体组成的亚群。该亚群分组结果与admixture、PCA两种方法得到的结果一致。
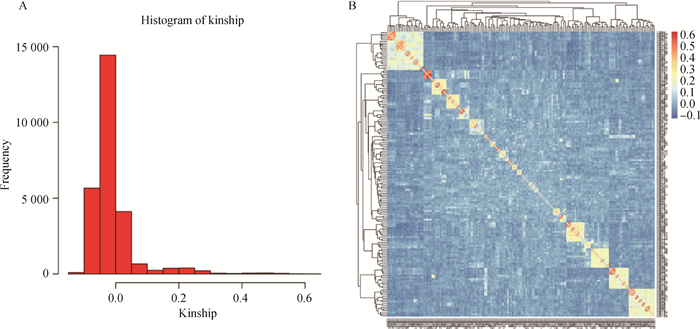
|
图 7 娄门鸭保种群个体间亲缘关系分布(A)及热图(B) Fig. 7 Distribution (A) and heat map (B) of genetic relationships among individuals from Loumen duck conservation population |
在163只娄门鸭保种群中共发现2 966条ROHs片段,其中39只公鸭493条(平均每只12.64条),124只母鸭2 473条(平均每只19.94条)。公鸭和母鸭ROHs片段长度主要集中在0~2 Mb区间,占比分别达到94.93%和92.00%(表 3)。
|
|
表 3 娄门鸭群体中不同ROHs长度的分布 Table 3 The distribution of different length of ROHs of Loumen ducks population |
基于ROH计算每个个体不同染色体的基因组近交系数FROH,分析娄门鸭保种群不同染色体近交系数的分布规律。NC_040068、NC_040074染色体的FROH分布较为分散,且FROH值分别达到0.352 4和0.319 3;其他染色体的FROH分布主要集中于左侧,其中NC_040046染色体的FROH值最小,仅为0.006 0(图 8A)。对于个体而言,娄门鸭保种群公鸭FROH为0.027 5,母鸭FROH为0.043 3(图 8B),差异没有统计显著性(P=0.137 9)。
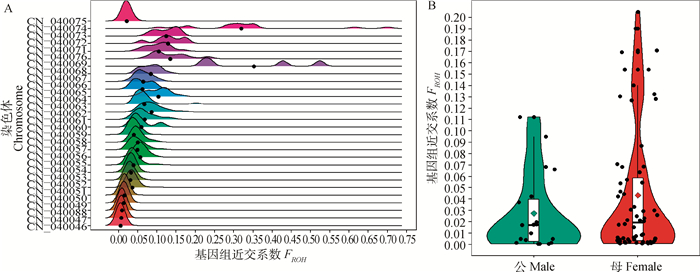
|
A图中每个黑点表示相应染色体的FROH均值,B图中绿点和红点分别表示公鸭、母鸭群体FROH的均值 Each black dot in figure A indicates the mean FROH value of the corresponding chromosome, while green and red dots in figure B indicate the mean FROH values of the male and female duck populations, respectively 图 8 娄门鸭保种群不同个体及染色体基因组近交系数FROH分布 Fig. 8 The distribution of genomic inbreeding coefficient FROH of different individuals and chromosomes |
经常评估保种群体的遗传多样性及近交系数是保种工作的一项重要内容。在物种资源保护中,尤其在家禽上一般不记录详细的系谱,因此群体内个体间的亲缘关系及近交系数等的评估结果往往与真实值之间存在较大的差距[38-39]。基因组中SNPs标记不受环境影响,且在基因组中分布广泛和易于检测[13],是评估种群遗传多样性的理想载体。随着高通量技术的发展,SNP芯片[40-42]和基因组二代测序[43-45]方法成为基因组范围内大量SNPs标记批量检测的重要技术。目前,鸭上尚没有一款能够实用的SNP芯片,而简化基因组技术在鸭上的成功开发[46],促使其成为鸭遗传多样性评估中的常用方法。因此,本课题组利用简化基因组测序技术系统研究了娄门鸭保种群体的遗传多样性,并分析了娄门鸭的群体结构。
群体遗传多样性有多种度量方法,各有优缺点和适用范围。本课题组比较了多态信息含量PIC、固定指数Fix-index、有效等位基因数Ne、基因多样性指数Nei和香农信息指数SHI等5个代表性的遗传多样性指标,发现除了Fix-index之外的其他4个指标之间具有较高的相关性,其在染色体上的分布规律也较为一致,说明这4个遗传多样性指标所蕴含的信息是冗余的,因此选择其中一种即可说明保种群体的遗传多样性情况。
PIC是衡量多样性高低的较好指标,尤其适用于分子标记,因此文献报道中对PIC的研究较为系统,当PIC≥0.5时,该座位为高度多态性位点; 当0.25≤PIC<0.5时为中度多态性位点; 当PIC<0.25时为低度多态性位点[30]。该判别标准适合于微卫星等复等位基因的分子标记,这些分子标记的特点是单个位点的等位基因数量不固定,当无限多个等位基因数时,最大PIC值为1。SNP标记属于二等位基因,其最大PIC为0.375,因此直接以PIC为0.25和0.50的划分标准来判定娄门鸭保种群结果有失偏颇。为了继续使用该判定标准,课题组以SNP位点PIC最大值0.375为基准,创新性地使用相对PIC(rPIC)这一指标来判定SNP位点的多态性高低。本研究结果显示,娄门鸭保种群相对PIC值在0.004 0~1.000 0之间,整个基因组上平均相对PIC为0.410 9,中度多态性位点占比9.30%,高度多态性位点达到38.80%,结果表明该群体遗传多样性较为丰富,目前娄门鸭保种方案比较合理。
对娄门鸭保种群每个单SNP位点遗传多样性指标分别分析,并考察这些指标在不同染色体上的分布情况。总体而言,PIC、Nei、Ne、SHI四个遗传多样性指标之间的分布规律相似,而且每个指标在不同染色体上的分布规律也相似; 而固定指数Fix-index与其他指标在染色体上的分布规律有较大差异,且Fix-index在不同染色体上的分布规律也不尽相同。该结果进一步表明,PIC、Nei、Ne和SHI四个遗传多样性指标中,可以选择其中一种,比如PIC来说明群体的遗传多样性情况,而Fix-index可作为其他多样性指标信息的有效补充。
固定指数Fix-index是群体中杂合子频率差异的估计值,能够通过杂合子频率偏差反映群体在某个座位的遗传分化程度。当一个群体分化成多个亚群时,整个群体中的杂合子必然会减少,大量位点的Fix-index高于0,表明出现了近亲杂交,群体有分化倾向。娄门鸭保种群整个基因组的Fix-index均值为0.320 8,说明娄门鸭群体可能有多个亚群出现。这从群体结构分析结果中也可以看出。群体结构指遗传变异在物种或群体中的一种非随机分布。处于同一亚群内的不同个体亲缘关系较高或SNPs分布状态接近,而亚群与亚群之间则亲缘关系稍远或SNPs分布状态差异较大。本研究从遗传结构、PCA主成分以及分子亲缘关系三个角度分析娄门鸭保种群的群体结构,均将娄门鸭群体分为3个亚群,其中一个较大的亚群(35公、90母)和两个较小的亚群(分别为2公、20母和2公、14母)。Fix-index和群体结构分析结果都说明娄门鸭保种群出现了一定的分化现象。群体的分化首先从基因组局部区域的遗传多样性变化开始,逐步扩展到基因组大部分区域,分化程度更高时甚至出现外观、性状表型的变化。本研究中, 娄门鸭分化成3个亚群,根据Fix-index结果可知其主要表现在染色体局部区域,分化群体间尚没有表现出外观、性状表型的明显差异。群体分化的出现可能由保种群留种时的抽样误差导致的遗传漂变造成,后续保种过程中可以将群体结构分析得到的3个分化的亚群之间的个体进行适当的非随机交配,消除目前的群体分化现象。
利用SNPs位点信息估计分子近交系数的方法主要有基于单个位点纯合性以及长纯合片段(runs of homozygosity,ROH)[47-48]两种。与传统的基于系谱的近交系数以及SNPs位点杂合度的近交系数相比,基于ROH的基因组近交系数FROH更加接近真实的近交系数[49-50]。刘家鑫等[51]利用绵羊全基因组ROH比较了10个绵羊群体的近交系数FROH,认为基于ROH评估的近交系数可为绵羊种群的保种提供参考依据。史良玉等[52]利用ROH对不同来源的大白猪产仔数近交衰退进行评估,发现法系群体中只有FROH检测到近交衰退现象,说明基于FROH的方法敏感性更高。曹愉夏等[53]利用简化基因组测序数据计算基于ROH的基因组近交系数,有效校正了狼山鸡由于部分系谱缺失导致的近交系数失真。本研究利用简化基因组测序得到了全基因组水平的SNPs标记信息,分析ROH片段在整个基因组以及不同染色体上的分布情况,并评估基因组近交系数。对于个体而言,娄门鸭公鸭近交系数FROH均值为0.027 5,母鸭为0.043 3,说明该种群近交系数较低,这也与该群体遗传多样性较为丰富有关。对于染色体而言,不同染色体的近交系数差异较大,其中NC_040068、NC_040074的FROH达到0.30以上,远高于其他染色体均值,这可能与娄门鸭在保种过程中这两个染色体上部分区域受到纯化选择有关。后续保种过程中应根据ROH片段鉴定并重点监测这两个染色体上近交系数较高的基因组区域,防止过度近交,避免个别染色体近交系数上升过快。
4 结论本研究利用不同遗传多样性指标分析娄门鸭保种群的遗传多样性,显示娄门鸭保种群的遗传多样性较丰富,群体基因组近交系数较低,但个别染色体的近交系数较高,且群体出现了部分分化。后续保种中可以将遗传结构分析得到的3个分化亚群个体之间进行适当的非随机交配,消除目前的群体分化现象,并重点监测染色体近交系数较高的基因组区域,避免个别染色体近交系数上升过快。
| [1] |
NOTTER D R. The importance of genetic diversity in livestock populations of the future[J]. J Anim Sci, 1999, 77(1): 61-69. DOI:10.2527/1999.77161x |
| [2] |
邹剑敏, 李慧芳, 陈宽维, 等. 我国家鸭品种资源遗传多样性保护等级分析[J]. 畜牧兽医学报, 2011, 42(1): 25-32. ZOU J M, LI H F, CHEN K W, et al. Conservation priorities of genetic diversity in Chinese indigenous duck breeds[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(1): 25-32. (in Chinese) |
| [3] |
章双杰, 杨建生, 王靖, 等. 娄门鸭种质特性分析[J]. 中国家禽, 2017, 39(16): 50-52. ZHANG S J, YANG J S, WANG J, et al. Analysis of germplasm characteristics of Loumen duck[J]. China Poultry, 2017, 39(16): 50-52. (in Chinese) |
| [4] |
陈旭, 张元跃. 分子标记及其在标记辅助选择中的应用[J]. 畜牧与饲料科学, 2006, 27(4): 29-32. CHEN X, ZHANG Y Y. The application of molecular marker in livestock marker-assisted selection[J]. Animal Husbandry and Feed Science, 2006, 27(4): 29-32. DOI:10.3969/j.issn.1672-5190.2006.04.015 (in Chinese) |
| [5] |
张云武, 张亚平, RYDER O A. 微卫星及其应用[J]. 动物学研究, 2001, 22(4): 315-320. ZHANG Y W, ZHANG Y P, RYDER O A. Microsatellites and its application[J]. Zoological Research, 2001, 22(4): 315-320. DOI:10.3321/j.issn:0254-5853.2001.04.012 (in Chinese) |
| [6] |
李慧芳, 李碧春, 陈宽维, 等. 中国地方鸭品种资源的分子遗传多样性[J]. 畜牧兽医学报, 2006, 37(11): 1107-1113. LI H F, LI B C, CHEN K W, et al. Study on molecular genetic diversity of native duck breeds in China[J]. Acta Veterinaria et Zootechnica Sinica, 2006, 37(11): 1107-1113. DOI:10.3321/j.issn:0366-6964.2006.11.003 (in Chinese) |
| [7] |
李慧芳, 高玉时, 苏一军, 等. 中国地方鸡种资源不同保种方法的分子检测[J]. 云南农业大学学报, 2006, 21(2): 228-230, 245. LI H F, GAO Y S, SU Y J, et al. Molecular evaluation of different conservation measures of Chinese indigenous chicken breeds[J]. Journal of Yunnan Agricultural University, 2006, 21(2): 228-230, 245. DOI:10.3969/j.issn.1004-390X.2006.02.020 (in Chinese) |
| [8] |
秦豪荣, 李晓鸣, 董飚, 等. 基于STR技术的4个地方鹅种遗传多样性分析[J]. 西北农业学报, 2016, 25(2): 182-187. QIN H R, LI X M, DONG B, et al. Analysis of genetic diversity of 4 Chinese indigenous domestic geese based on STR technology[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2016, 25(2): 182-187. (in Chinese) |
| [9] |
王世强, 王立儒, 刘帅, 等. 基于SSR标记的黄精品种(系)DNA指纹图谱库构建[J]. 分子植物育种, 2018, 16(6): 1878-1887. WANG S Q, WANG L R, LIU S, et al. Construction of DNA fingerprint database based on SSR marker for Polygonatum varieties (lines)[J]. Molecular Plant Breeding, 2018, 16(6): 1878-1887. (in Chinese) |
| [10] |
王统苗, 常国斌, 白皓, 等. 利用16个微卫星标记分析6个鸭品种的遗传多样性[J]. 四川农业大学学报, 2019, 37(2): 221-226. WANG T M, CHANG G B, BAI H, et al. Analysis of genetic diversity of 6 duck populations using 16 microsatellite markers[J]. Journal of Sichuan Agricultural University, 2019, 37(2): 221-226. (in Chinese) |
| [11] |
VIGNAL A, MILAN D, SANCRISTOBAL M, et al. A review on SNP and other types of molecular markers and their use in animal genetics[J]. Genet Sel Evol, 2002, 34(3): 275-305. DOI:10.1186/1297-9686-34-3-275 |
| [12] |
邹喻苹, 葛颂. 新一代分子标记——SNPs及其应用[J]. 生物多样性, 2003, 11(5): 370-382. ZOU Y P, GE S. A novel molecular marker — SNPs and its application[J]. Biodiversity Science, 2003, 11(5): 370-382. DOI:10.3321/j.issn:1005-0094.2003.05.003 (in Chinese) |
| [13] |
朱涛, 谷洪昌, 张泽宾, 等. 鸭不同品种特异性分子标记SNP的筛选与鉴定[J]. 中国家禽, 2018, 40(15): 7-10. ZHU T, GU H C, ZHANG Z B, et al. SNP identification for specific molecular markers of different duck breeds[J]. China Poultry, 2018, 40(15): 7-10. (in Chinese) |
| [14] |
罗怀容, 施鹏, 张亚平. 单核苷酸多态性的研究技术[J]. 遗传, 2001, 23(5): 471-476. LUO H R, SHI P, ZHANG Y P. Detection for single nucleotide polymorphisms[J]. Hereditas (Beijing), 2001, 23(5): 471-476. DOI:10.3321/j.issn:0253-9772.2001.05.021 (in Chinese) |
| [15] |
李慧芳, 束婧婷, 张莹, 等. 基于连接酶检测反应的并行分型系统检测鸡肉风味相关候选基因多态性[J]. 畜牧兽医学报, 2010, 41(3): 262-267. LI H F, SHU J T, ZHANG Y, et al. A typing system based on ligase detection reaction for chicken meat flavor related candidate genes polymorphism[J]. Acta Veterinaria et Zootechnica Sinica, 2010, 41(3): 262-267. (in Chinese) |
| [16] |
CHEN Q, MA Y F, YANG Y M, et al. Genotyping by genome reducing and sequencing for outbred animals[J]. PLoS One, 2013, 8(7): e67500. DOI:10.1371/journal.pone.0067500 |
| [17] |
WANG S, MEYER E, MCKAY J K, et al. 2b-RAD: a simple and flexible method for genome-wide genotyping[J]. Nat Methods, 2012, 9(8): 808-810. DOI:10.1038/nmeth.2023 |
| [18] |
PAN Y Z, WANG X Q, SUN G L, et al. Application of RAD sequencing for evaluating the genetic diversity of domesticated Panax notoginseng (Araliaceae)[J]. PloS One, 2016, 11(11): e0166419. DOI:10.1371/journal.pone.0166419 |
| [19] |
VALDISSER P A M R, PAPPAS G J Jr, DE MENEZES I P P, et al. SNP discovery in common bean by restriction-associated DNA (RAD) sequencing for genetic diversity and population structure analysis[J]. Mol Genet Genomics, 2016, 291(3): 1277-1291. DOI:10.1007/s00438-016-1182-3 |
| [20] |
刘宏祥, 朱满兴, 秦豪荣, 等. 基于简化基因组测序评价太湖鹅不同保种场的保种效果[J]. 华北农学报, 2018, 33(S1): 94-99. LIU H X, ZHU M X, QIN H R, et al. Assessment of conservation effects of different Taihu goose conservation farms based on simplified genome sequencing[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(S1): 94-99. DOI:10.7668/hbnxb.2018.S1.016 (in Chinese) |
| [21] |
刘宏祥, 宋卫涛, 薛敏开, 等. 简化基因组测序评价高邮鸭保种效果的研究[J]. 中国家禽, 2018, 40(22): 11-15. LIU H X, SONG W T, XUE M K, et al. Assessment of conservation effect of Gaoyou duck based on simplified genome sequencing[J]. China Poultry, 2018, 40(22): 11-15. (in Chinese) |
| [22] |
XIAO Q, ZHANG Z, SUN H, et al. Genetic variation and genetic structure of five Chinese indigenous pig populations in Jiangsu Province revealed by sequencing data[J]. Anim Genet, 2017, 48(5): 596-599. DOI:10.1111/age.12560 |
| [23] |
WU F, SUN H, LU S X, et al. Genetic diversity and selection signatures within Diannan small-ear pigs revealed by next-generation sequencing[J]. Front Genet, 2020, 11: 733. DOI:10.3389/fgene.2020.00733 |
| [24] |
王凤娇, 孟宪红, 傅强, 等. 基于简化基因组测序的中国明对虾3个选育世代遗传多样性分析[J]. 渔业科学进展, 2020, 41(4): 68-76. WANG F J, MENG X H, FU Q, et al. Analysis of genetic diversity in three generations of breeding populations of Fenneropenaeus chinensis based on reduced-representation genome sequencing[J]. Progress in Fishery Sciences, 2020, 41(4): 68-76. (in Chinese) |
| [25] |
韩威, 朱云芬, 殷建玫, 等. 基于RAD-seq简化基因组测序的19个地方鸡种遗传进化研究[J]. 畜牧兽医学报, 2020, 51(4): 670-678. HAN W, ZHU Y F, YIN J M, et al. Study on genetic evolution of 19 indigenous chicken breeds based on RAD-seq[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(4): 670-678. (in Chinese) |
| [26] |
FOSTER Y, DUTOIT L, GROSSER S, et al. Genomic signatures of inbreeding in a critically endangered parrot, the Kākāpō[J]. G3 (Bethesda), 2021, 11(11). |
| [27] |
LI H, DURBIN R. Fast and accurate long-read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2010, 26(5): 589-595. DOI:10.1093/bioinformatics/btp698 |
| [28] |
CHANG C C, CHOW C C, TELLIER L C, et al. Second-generation PLINK: rising to the challenge of larger and richer datasets[J]. Giga Science, 2015, 4: 7. DOI:10.1186/s13742-015-0047-8 |
| [29] |
The R Core Team.R: a language and environment for statistical computing[EB/OL]. (2015). https://www.R-project.org/.
|
| [30] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314-331. |
| [31] |
NEI M. Definition and estimation of fixation indices[J]. Evolution, 1986, 40(3): 643-645. DOI:10.1111/j.1558-5646.1986.tb00516.x |
| [32] |
NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proc Natl Acad Sci U S A, 1979, 76(10): 5269-5273. DOI:10.1073/pnas.76.10.5269 |
| [33] |
常洪. 动物遗传资源学[M]. 北京: 科学出版社, 2009. CHANG H. Animal genetic resources[M]. Beijing: Science Press, 2009. (in Chinese) |
| [34] |
袁志发. 群体遗传学、进化与熵[M]. 北京: 科学出版社, 2011. YUAN Z F. Population genetics, evolution and entropy[M]. Beijing: Science Press, 2011. (in Chinese) |
| [35] |
ALEXANDER D H, NOVEMBRE J, LANGE K. Fast model-based estimation of ancestry in unrelated individuals[J]. Genome Res, 2009, 19(9): 1655-1664. DOI:10.1101/gr.094052.109 |
| [36] |
FRANCIS R M. POPHELPER: an R package and web app to analyse and visualize population structure[J]. Mol Ecol Resour, 2017, 17(1): 27-32. DOI:10.1111/1755-0998.12509 |
| [37] |
YANG J, LEE S H, GODDARD M E, et al. GCTA: a tool for genome-wide complex trait analysis[J]. Am J Hum Genet, 2011, 88(1): 76-82. DOI:10.1016/j.ajhg.2010.11.011 |
| [38] |
OLIEHOEK P A, BIJMA P. Effects of pedigree errors on the efficiency of conservation decisions[J]. Genet Sel Evol, 2009, 41(1): 9. DOI:10.1186/1297-9686-41-9 |
| [39] |
HAMMERLY S C, MORROW M E, JOHNSON J A. A comparison of pedigree- and DNA-based measures for identifying inbreeding depression in the critically endangered Attwater's Prairie-Chicken[J]. Mol Ecol, 2013, 22(21): 5313-5328. DOI:10.1111/mec.12482 |
| [40] |
师睿, 张毅, 王雅春, 等. 利用SNP芯片信息评估新疆近交牛基因组纯合度[J]. 遗传, 2020, 42(5): 493-505. SHI R, ZHANG Y, WANG Y C, et al. The evaluation of genomic homozygosity for Xinjiang inbred population by SNP panels[J]. Hereditas (Beijing), 2020, 42(5): 493-505. (in Chinese) |
| [41] |
王宏浩, 任小康, 张毅, 等. 基因芯片技术在晋南牛种公牛选育中的应用[J]. 畜牧兽医学报, 2021, 52(10): 2803-2813. WANG H H, REN X K, ZHANG Y, et al. Application of gene chip technology in Jinnan bull breeding[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2803-2813. DOI:10.11843/j.issn.0366-6964.2021.010.011 (in Chinese) |
| [42] |
蔡春波, 张雪莲, 张万峰, 等. 运用SNP芯片评估马身猪保种群体的遗传结构[J]. 畜牧兽医学报, 2021, 52(4): 920-931. CAI C B, ZHANG X L, ZHANG W F, et al. Evaluation of genetic structure in Mashen pigs conserved population based on SNP chip[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(4): 920-931. (in Chinese) |
| [43] |
BERIHULAY H, LI Y F, GEBREKIDAN B, et al. Whole genome resequencing reveals selection signatures associated with important traits in Ethiopian indigenous goat populations[J]. Front Genet, 2019, 10: 1190. DOI:10.3389/fgene.2019.01190 |
| [44] |
魏强, 奥岩, 杨漫漫, 等. 利用全基因组重测序技术鉴定五指山猪GHR突变体转基因插入位点[J]. 遗传, 2021, 43(12): 1149-1158. WEI Q, AO Y, YANG M M, et al. Identification of genomic insertion of dominant-negative GHR mutation transgenes in Wuzhishan pig using whole genome sequencing method[J]. Hereditas (Beijing), 2021, 43(12): 1149-1158. (in Chinese) |
| [45] |
王海龙, 王巧, 邢思远, 等. 基于表型和基因组信息评价北京油鸡保种群保种情况[J]. 畜牧兽医学报, 2021, 52(9): 2406-2415. WANG H L, WANG Q, XING S Y, et al. Evaluating Beijing You chickens conservation status by phenotype and genome information[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(9): 2406-2415. (in Chinese) |
| [46] |
ZHU F, CUI Q Q, HOU Z C. SNP discovery and genotyping using genotyping-by-sequencing in Pekin ducks[J]. Sci Rep, 2016, 6: 36223. DOI:10.1038/srep36223 |
| [47] |
PERIPOLLI E, MUNARI D P, SILVA M V G B, et al. Runs of homozygosity: current knowledge and applications in livestock[J]. Anim Genet, 2017, 48(3): 255-271. DOI:10.1111/age.12526 |
| [48] |
MASTRANGELO S, TOLONE M, DI GERLANDO R, et al. Genomic inbreeding estimation in small populations: evaluation of runs of homozygosity in three local dairy cattle breeds[J]. Animal, 2016, 10(5): 746-754. DOI:10.1017/S1751731115002943 |
| [49] |
KELLER M C, VISSCHER P M, GODDARD M E. Quantification of inbreeding due to distant ancestors and its detection using dense single nucleotide polymorphism data[J]. Genetics, 2011, 189(1): 237-249. DOI:10.1534/genetics.111.130922 |
| [50] |
FORUTAN M, ANSARI MAHYARI S, BAES C, et al. Inbreeding and runs of homozygosity before and after genomic selection in North American Holstein cattle[J]. BMC Genomics, 2018, 19(1): 98. DOI:10.1186/s12864-018-4453-z |
| [51] |
刘家鑫, 魏霞, 邓天宇, 等. 绵羊全基因组ROH检测及候选基因鉴定[J]. 畜牧兽医学报, 2019, 50(8): 1554-1566. LIU J X, WEI X, DENG T Y, et al. Genome-wide scan for run of homozygosity and identification of corresponding candidate genes in sheep populations[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(8): 1554-1566. (in Chinese) |
| [52] |
史良玉, 王立刚, 张鹏飞, 等. 不同来源大白猪总产仔数近交衰退评估[J]. 畜牧兽医学报, 2021, 52(10): 2772-2782. SHI L Y, WANG L G, ZHANG P F, et al. Evaluation of inbreeding depression on the total numbers of piglets born in different groups of large white pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2772-2782. DOI:10.11843/j.issn.0366-6964.2021.010.008 (in Chinese) |
| [53] |
曹愉夏, 韩威, 李国辉, 等. 基于RAD-seq简化基因组测序估算狼山鸡分子亲缘系数和近交系数[J]. 中国家禽, 2019, 41(10): 14-19. CAO Y X, HAN W, LI G H, et al. Estimation of genomic kinship coefficients and inbreeding coefficients based on RAD-seq simplified genome sequencing data in Langshan chicken[J]. China Poultry, 2019, 41(10): 14-19. (in Chinese) |
(编辑 郭云雁)



