2. 北方民族大学生物科学与工程学院,银川 750021;
3. 国家民委发酵酿造工程生物技术重点实验室,银川 750021
2. College of Biological Science and Engineering, Beifang University of Nationalities, Yinchuan 750021, China;
3. Key Laboratory of Fermentation Brewing Engineering and Biotechnology State Nationalities Affairs Commission, Yinchuan 750021, China
疯草是豆科棘豆属(Oxytropis)和黄芪属(Astragalus)有毒植物的统称[1-2],动物过量采食疯草会发生以神经系统机能紊乱为特征的慢性中毒病[3-4]。苦马豆素(swainsonine, SW)是疯草的主要毒性成分,由疯草中的链格孢属波状芽管孢组(Alternaria Section Undifilum spp)内生真菌产生[5-7],该类内生真菌包括棘豆链格孢菌(Alternaria oxytropis)、A. fulvum和A. cinereum等3个种。目前,疯草内生真菌合成SW的机制仍未阐明,该机制的阐释可为从控制内生真菌角度预防动物疯草中毒病提供新思路。据报道,酵母氨酸还原酶、酵母氨酸脱氢酶、酵母氨酸氧化酶、吡咯-5-羧酸还原酶、聚酮合酶和细胞色素氧化酶P450等基因可能是棘豆链格孢菌中参与SW合成的关键基因[8-10],但上述基因的具体功能及其在棘豆链格孢菌SW合成中的作用机制仍不清楚。
研究表明,植物病原真菌豆类丝核菌(Slafractonia leguminicola)、昆虫病原真菌金龟子绿僵菌(Metarhizium robertsii)、番薯属植物表生菌(Chaetothyriaceae spp)、人和动物皮肤病原真菌Trichophyton spp和Arthroderma spp等也可产生SW[11-13]。比较基因组学研究表明,上述真菌中均存在一类高度同源、结构相似的I型聚酮合酶(type I polyketides synthetase, T1-PKS)基因簇,T1-PKS基因簇广泛存在于细菌、真菌、植物中,负责次生代谢产物的合成[11]。Cook等[11]将绿僵菌中该基因簇的swnK基因敲除后,敲除菌株不再产生SW,确定该基因簇在绿僵菌SW生物合成中发挥关键作用,并将产SW真菌中的这一类基因簇统一命名为SW合成基因簇(swainsonine biosynthesis gene cluster,SWN)。SWN基因簇主要由swnR、swnN、swnH1、swnH2、swnK等基因组成,在棘豆链格孢菌SWN基因簇5′端还存在一个氨基酸转运样蛋白基因(amino-acid transporter-like,AATL)。swnK是SWN基因簇中最主要的基因,由具有腺苷酰化作用区域(adenylylation,A)、2个磷酸泛酰巯基乙胺结合作用区域(phosphopantetheine-binding/thiolation,T)、β-聚酮合酶(β-ketoacyl synthase,KS),酰基转移酶(acyltransferase,AT),β-酮酯酰基还原酶(β-ketoacyl reductase,SDR)和硫酯还原酶(thioester reductase,SDRe1)等多个功能区组成(图 1)。功能预测分析表明,swnK可能编码从L-哌可酸到1羟基吲哚里西啶形成之前6步酶促反应所需的多功能复合酶。而swnR、swnN、swnH1、swnH2等基因可能分别编码从1羟基吲哚里西啶到SW的3步酶促反应所需的酶[11-12]。但迄今为止,棘豆链格孢菌SWN基因簇中各基因的功能仍未经过试验验证,其功能和SW合成途径是否与预测结果一致还有待进一步研究。
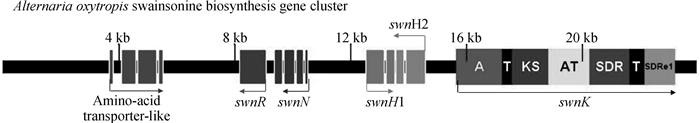
|
图 1 棘豆链格孢菌的SWN基因簇 Fig. 1 Swainsonine biosynthesis gene cluster Alternaria oxytropis |
从不同种疯草中分离到不同的棘豆链格孢菌菌株,虽然这些菌株生长特性和显微形态相近,且在遗传进化上高度同源,但不同菌株在SW合成、分生孢子产生等方面存在显著差异[14-16]。近年来,一些未被归类到疯草的豆科植物中也分离到链格孢属波状芽管孢组真菌,如Alternaria bornmuelleri和甘肃链格孢菌(Alternaria gansuense),但它们均为植物病原真菌,仅能产生极微量的SW。进一步的研究表明,这两种病原真菌基因组中也存在与棘豆链格孢菌高度同源的SWN基因簇[17-19]。因此,需要对棘豆链格孢菌等疯草内生真菌和甘肃链格孢菌等病原真菌SWN基因簇各基因的功能及其在SW合成的分子调控机制进行深入研究。
目前,棘豆链格孢菌SWN基因簇各基因表达水平的变化是否与真菌的SW合成相关仍未见报道。本研究拟对棘豆链格孢菌野生型菌株UA003进行EMS诱变处理,测定各菌株菌丝中SW的含量,筛选SW产率显著变化的诱变菌株。然后对棘豆链格孢菌野生型菌株、SW产率发生显著变化的EMS诱变菌株及仅能产生微量SW的甘肃链格孢菌SWN基因簇各基因表达模式和swnK基因突变位点进行分析,探究其基因表达变化的原因。通过上述研究,初步了解棘豆链格孢菌SWN基因簇各基因的表达模式及其与SW合成的关系,以期为进一步阐明棘豆链格孢菌SWN基因簇中各基因的功能奠定基础。
1 材料与方法 1.1 材料1.1.1 供试菌株 棘豆链格孢菌Alternaria Section Undifilum Oxytropis(=Alternaria Oxytropis)UA003菌株,分离自变异黄芪[20];甘肃链格孢菌Alternaria Section Undifilum gansuense(=Alternaria gansuense)EA菌株分离自直立黄芪[19]。
1.1.2 仪器与试剂 Simpli Nano超微量分光光度计,美国GE公司;qTOWER 2.2荧光定量PCR仪,德国Jena公司;总RNA提取试剂(TaKaRa RNAiso Plus)、primeScript RT reagent kit with a gDNA eraser和B GreenTM Premix Ex TaqTM Ⅱ购自宝生物工程有限公司;蜗牛酶、溶壁酶、纤维素酶、甲基磺酸乙酯(EMS)购于Sigma公司;无水乙醇、三氯甲烷和异丙醇购自上海国药集团化学试剂有限公司;焦碳酸二乙酯(DEPC)原液购自北京天根生化有限公司。相关引物(表 1、表 2)由上海生工生物技术有限公司合成。
|
|
表 1 Real-time PCR引物序列 Table 1 Primer sequences for real-time PCR |
|
|
表 2 swnK基因扩增引物序列 Table 2 Primer sequences for swnK genes PCR amplification |
1.2.1 棘豆链格孢菌原生质体制备 将棘豆链格孢菌UA003菌株接种于PDA培养基,25 ℃培养20~30 d。将培养的真菌菌落制成6 mm直径的菌饼,接种于装有100 mL PDB培养基的三角瓶中,25 ℃恒温振荡培养15 d。用神奇滤布过滤菌液,并用0.6 mol·L-1 MgSO4溶液洗涤菌丝。称量100 mg菌丝,按质量体积比1:10加入纤维素酶、溶壁酶和蜗牛酶质量分数比为1:1:1.2的酶解液,室温90 r·min-1孵育2 h。用神奇滤布过滤酶解消化的菌丝,获得原生质体。将原生质体转入50 mL离心管中室温4 000 r·min-1离心。去上清后,在离心管中加入10 mL STC buffer重悬细胞,4 000 r·min-1离心。弃去上清,在离心管中加入1 mL STC buffer重悬细胞,取少量悬液滴入血球计数板中,置显微镜下进行细胞计数。然后加入适量STC buffer调节原生质体浓度至1×105~1×106个·mL-1备用。
1.2.2 原生质体EMS诱变处理 取100 μL原生质体悬液,加入不同体积10 mol·L-1EMS原液,混匀,使EMS工作浓度分别为0.02、0.04、0.06、0.08 mol·L-1。分别置恒温摇床200 r·min-1室温孵育20、30 min,反应结束后10 000 r·min-1离心6 min,弃上清。向离心管中加入100 μL STC buffer重悬细胞,将混悬液均匀涂布于再生培养基上,20 ℃恒温培养。每个浓度做3组平行处理,每个处理重复3次。同时设空白对照组,对照组不加EMS。
1.2.3 诱变菌株筛选 随机挑取经EMS诱变处理和未经EMS处理的再生菌落,接种于PDA固体培养基上培养30 d。分别收集PDA培养基培养的各菌株新鲜菌丝0.5~1.0 g,液氮冷冻研磨成粉末并干燥。将粉末置于装有5 mL甲醇的10 mL离心管中,30 ℃超声提取30 min。每个菌株同时设置3个平行重复,每个样品提取3次,合并提取液,然后减压挥干溶剂。粗提物用适量甲醇溶解并用0.22 μm孔径滤膜过滤,定容至5 mL,制成待检液。采用张蕾蕾等[21]报道的α-甘露糖苷酶抑制法对菌丝中SW含量进行测定。比较经EMS诱变处理的诱变菌株和未经EMS处理的野生型菌株菌丝中的SW含量,筛选SW含量发生显著变化的诱变菌株。将对照菌株和筛选所得的SW含量发生显著变化的诱变菌株分别接种于PDA培养基上,连续传代培养3代,分别测定每代真菌菌丝中的SW含量,进一步筛选与对照菌株SW合成差异显著、且始终稳定的EMS诱变菌株。
1.2.4 真菌总RNA提取 收集PDA培养基上培养30 d的棘豆链格孢菌野生型菌株UA003、SW含量发生显著变化的EMS诱变菌株和甘肃链格孢菌EA菌株的真菌菌丝50~100 mg,用液氮将其研磨成粉末。将粉末转移至2 mL离心管中,按照试剂盒说明书提取各样品总RNA,测定总RNA浓度,并用电泳对RNA的完整性进行评估。
1.2.5 cDNA合成 以真菌总RNA为模板,按照试剂盒操作说明将总RNA反转录为cDNA。去基因组DNA反应体系为5×gDNA Eraser Buffer 2 μL,gDNA Eraser 1 μL,RNA 90 ng,添加RNase Free ddH2O到10 μL。反应参数为42 ℃ 2 min,然后4 ℃保存。反转录反应体系为去基因组DNA后的反应液10 μL,PrimeScript RT Enzyme Mix I 1 μL,RT Primer Mix 1 μL,5× PrimeScript Buffer 4 μL,RNase Free ddH2O 4 μL。反转录条件为37 ℃ 15 min,85 ℃ 5 s,4 ℃保存。
1.2.6 EMS诱变菌株与野生型菌株中SWN基因簇各基因表达分析 根据Cook等[11]报道的棘豆链格孢菌SWN基因簇中AATL、swnR、swnN、swnH1、swnH2、swnK(A、AT、KS、SDRe1、SDR)各基因的序列信息,利用NCBI中的BLAST-Primer软件设计引物(表 1)。使用TaKaRa SYBR Green I Real-time PCR试剂盒进行Real-Time PCR扩增反应,分别以β-actin和GAPDH为内参基因,以dd H2O为阴性对照,反转录所得的cDNA为模板按下列组分配制PCR反应液,每个样品设置3个重复。反应体系为TB Green Premix Ex Taq Ⅱ(RNaseH Plus)10 μL,PCR Forward Primer 0.8 μL,PCR Reverse Primer 0.8 μL,ROX Reference Dye 0.4 μL,cDNA模板2 μL,RNase Free dd H2O 6 μL。反应条件为95 ℃预变性30 s; 95 ℃变性5 s,60 ℃退火20 s,72 ℃延伸1 min,40个循环。PCR扩增产物经凝胶回收后送至上海生工生物技术公司进行测序验证。将各组所得Ct值分别以β-actin和GAPDH基因为内参校正,采用2-ΔΔCt法计算各菌株不同基因的表达量,ΔΔCt=[Ct(处理组目的基因)-Ct(处理组内参基因)]-[Ct(对照组目的基因)-Ct(对照组内参基因)]。
1.2.7 swnK基因的扩增与突变位点分析 收集棘豆链格孢菌UA003、SW含量发生显著变化的EMS诱变菌株和甘肃链格孢菌EA菌株真菌菌丝100 mg,液氮研磨至粉末,并转移至2 mL离心管中。然后按照试剂盒说明书提取DNA,测定各样品的DNA浓度。使用表 2中的引物对swnK基因的编码区和非编码区进行PCR扩增,每个片段同时设置3个平行重复。扩增体系为rTaq酶0.5 μL,10× PCR Buffer 5 μL,dNTP 4 μL,F-Primer 0.5 μL,R-Primer 0.5 μL,DNA模板0.5 μL,加ddH2O至50 μL。反应条件为94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸45 s,共30个循环;最后72 ℃延伸10 min。
将每个基因片段的3个平行重复的PCR扩增产物经凝胶回收后,均送至上海生工生物技术公司进行双向测序,应用DNAStar软件中的SeqMan程序对双向测序片段进行拼接,使用DNAMAN 8软件中的Clustal W程序对拼接好的swnK基因进行序列比对分析。
1.2.8 数据处理与分析 使用IBM SPSS Statistics 20和GraphPad Prism 7统计软件中的单因素方差法对各组真菌的SW含量及各基因的表达量进行差异分析。
2 结果 2.1 EMS诱变菌株的筛选随着EMS工作浓度的增加,菌株致死率也随之增高。当EMS工作浓度为0.06和0.08 mol·L-1时,真菌的死亡率为100%,但当EMS工作浓度为0.02和0.04 mol·L-1时,再生培养基上均有菌株存活。本研究随机挑取了34株经EMS诱变处理且能够在再生培养基生长的菌株,并分别对上述菌株和对照菌株真菌菌丝的SW含量进行了测定。结果表明,当EMS工作浓度为0.02 mol·L-1,处理30 min,再生后随机挑选的所有菌株,其SW含量均高于对照菌株。经初步筛选,共有12株EMS诱变处理菌株的SW含量与对照菌株相比发生了显著改变。本研究对初步筛选所得的12株菌株进行了连续3代的传代培养和SW检测,其中诱变菌株的SW含量经连续3代培养后始终与对照组差异显著且高于对照菌株的有E02、E23和E25等。SW测定结果表明,对照菌株UA003中SW含量为(0.087±0.004)mg·g-1菌丝干重,EMS诱变菌株E02、E23、E25的SW含量均显著高于对照菌株UA003(P < 0.05或P < 0.01),其中E02菌株的SW含量最高,为(0.202±0.006)mg·g-1,极显著高于对照菌株UA003(P < 0.01)(图 2)。此外本研究也测定了甘肃链格孢菌EA菌株的SW含量,其SW含量仅为(0.004±0.001)mg·g-1菌丝干重,极显著低于其他菌株(P < 0.01)。
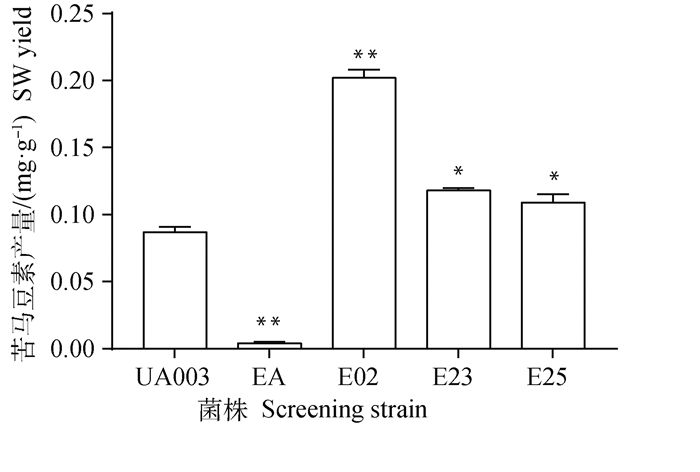
|
与对照组(UA003)比较,*.差异显著(P < 0.05);**.差异极显著(P < 0.01) Compared with the control (UA003), *. significant difference (P < 0.05); **. Very significant diffevence (P < 0.01) 图 2 各菌株菌丝中SW的含量 Fig. 2 Contents of SW in mycelia of each test strain |
如表 3所示,当以β-actin为内参基因时,与UA003菌株相比,AATL、swnR、swnN、swnH1及swnH2在EA、E02、E23和E25菌株中表达均显著下调(P < 0.05或P < 0.01)。A、KS、SDR和SDRe1基因在EA、E02、E23和E25菌株中表达均显著上调(P < 0.05或P < 0.01),而AT基因在E23菌株中表达极显著上调(P < 0.01),在EA、E02和E25菌株中表达则极显著下调(P < 0.01)。
|
|
表 3 SWN基因簇各基因的相对表达量(x±s) Table 3 Relative expression of each gene in SWN gene cluster(x±s) |
由表 3所示,当以GAPDH为内参基因时,与UA003菌株相比,AATL、swnR、swnN、swnH1及swnH2在EA、E02、E23和E25菌株中表达均显著下调(P < 0.05或P < 0.01)。A、KS、SDR和SDRe1基因在EA、E02、E23和E25菌株中表达均显著上调(P < 0.01或P < 0.05),但AT基因仅在E23菌株中表达上调(P < 0.01),在EA、E02和E25菌株中的表达下调(P < 0.01)。
2.3 swnK基因突变位点分析测序结果表明,UA003、E02、E23、E25和EA等5株真菌的swnK基因序列与已公开的棘豆链格孢菌的swnK基因(GenBank登录号为KY365741.1)均具有较高一致性。
UA003、E23、E25等3菌株swnK基因序列完全一致,与已公布的棘豆链格孢菌swnK基因序列(KY365741.1)一致性为99.97%,仅有2处碱基存在差异,均在SDR区域,但其编码的氨基酸序列与KY365741.1完全一致。E02菌株swnK基因序列与KY365741.1的序列一致性为99.80%,与UA003的swnK基因序列一致性为99.83%。与UA003相比,E02共有13个位点的碱基发生了突变,其中,转换突变有11个位点(G16756A、G16871A、G16939A、T16993C、T17189C、A17305G、C17429T、T17446C、G17502A、C17505T、A17307G),颠换突变有2个位点(G16997A、T17280C),分别位于swnK基因KS区域上游的A、T区域(图 3A),其编码的氨基酸序列共有6个位点发生了突变(D403N、L445V、S509P、G613D、A614V、S615G)(图 3B)。

|
A. E02菌株swnK基因核苷酸序列突变位点的示意图;B. E02菌株swnK基因编码产物氨基酸序列突变位点的示意图;C. EA菌株swnK基因编码产物氨基酸序列突变位点的示意图 A. The schematic diagram of the nucleotide sequence mutation site of swnK gene in strain E02;B. The schematic diagram of swnK gene encoding product mutation sites of E02; C. The schematic diagram of swnK gene encoding product mutation sites of EA 图 3 真菌swnK基因及其编码的氨基酸序列比对结果 Fig. 3 Alignment results of swnK gene and its encoded amino acid sequence |
甘肃链格孢菌EA菌株的swnK基因与KY365741.1序列一致性为97.55%,与UA003 swnK基因序列一致性为97.80%。与KY365741.1相比,EA菌株共有163个位点的碱基不同,分布于swnK基因的非编码区和编码区的各个区域,其编码的氨基酸序列有67个位点不同,位于swnK基因的各个区域。其转录产物的起始段缺失了一段由15个氨基酸组成的肽链(MLTPAVSLKNLTKPK),在转录产物的1 135与1 143处(KY365741.1中对应的位置)插入了一条由7个氨基酸组成的短肽(TEVDGVP)(图 3C)。
3 讨论棘豆链格孢菌SW合成能力及其在宿主植物组织中的生物量与宿主中SW的含量紧密相关[15]。据报道,棘豆链格孢菌SW合成能力受底物、环境、菌株等因素的影响[22-23]。棘豆链格孢菌SWN基因簇相关基因可能是真菌SW生物合成的关键基因[11],但相关基因表达水平与SW合成存在何种联系仍有待进一步研究。目前,人们从不同种疯草中分离到不同的棘豆链格孢菌菌株,由于环境和遗传等因素的影响,直接分析不同来源的菌株SWN基因簇各基因的表达水平,并不能真实的反映各菌株SW合成能力与SWN基因簇各基因表达水平的相关性。因此,本研究对棘豆链格孢菌野生型菌株UA003进行了EMS诱变处理,对经EMS诱变处理并能再生的诱变菌株进行了筛选,筛选出与野生型菌株SW合成能力差异显著的突变菌株,以此为研究模型来分析SWN基因簇各基因表达水平的变化与SW合成的关系。在本研究中,突变菌株E02、E23和E25由野生型菌株UA003诱变而来,其遗传背景与UA003相同,比较分析其与野生型菌株UA003菌株SWN基因簇各基因的表达模式,从理论上来说能够更为真实地反映真菌SW合成与SWN基因簇各基因之间的关系。
在本研究中,E02、E23和E25菌株经EMS诱变处理后,SW生物合成能力显著升高。与UA003相比,3株诱变菌株SWN基因簇各基因表达水平也显著改变。其中swnK中A、KS、SDR、SDRel等基因的表达均显著上调,其表达模式与SW合成呈正相关。而AATL、swnR、swnN、swnH1及swnH2基因表达均显著下调,其表达模式与SW产率呈负相关。而swnK中的AT基因在E02、E25菌株中表达显著下调,呈现出与swnK中A、KS、SDR、SDRel等基因相反的表达模式。在金龟子绿僵菌的研究中,swnH2基因的表达水平与SW合成呈负相关,这与本研究结果相一致,但swnR基因表达水平与SW合成呈正相关,而swnH1、swnK、swnN等基因表达与SW合成未表现出相关性[24],该研究也表明,金龟子绿僵菌中SWN基因簇各基因的表达模式可能不同于棘豆链格孢菌。在本研究中,E02、E23和E25菌株来源于野生型菌株UA003,但各菌株的SW产率和SWN基因簇各基因表达模式发生了显著变化,应该是由于EMS诱导野生型菌株基因组中的碱基突变,导致了相关基因结构和功能的改变,抑制了AATL、swnR、swnN、swnH1、swnH2等基因的表达,增强了swnK基因的表达,但swnK基因表达的增强是否是导致诱变菌株SW合成增加的直接原因还有待进一步研究。
EMS是一种可改变DNA结构的高效烷化剂,能在基因组中引发多个位点的突变[25-28]。EMS通过不同的方式引发转换型突变、颠换型突变或造成染色体片段的缺失[29]。本研究中,突变菌株E02 swnK基因编码区13个位点的碱基发生了突变,分别位于A、T区域,其中11个位点为转换型突变,2个位点为颠换型突变,而swnK基因5′端的上游非编码区序列未发生突变。E02菌株swnK基因的突变使其编码产物中6个氨基酸位点发生错义突变,可能会导致swnK基因表达水平和功能的改变。尽管E23、E25菌株SWN基因簇中各基因的表达模式与E02相似,然而这两株突变菌株swnK基因编码区及其5′端上游非编码区序列未发生突变。在真菌基因表达调控中,转录水平的调控发挥着至关重要的作用。转录水平的调节由位于基因转录起始位点上游的非编码区顺式作用元件,以及可与非编码区或编码区互作的反式作用因子、RNA聚合酶等共同参与。启动子、增强子、抑制子等顺式作用元件,通过引发基因转录起始来调控转录频率。而转录因子等反式作用因子,则能与基因5`端上游非编码区特定序列专一性结合,通过调节基因的转录从而保证目的基因以特定的强度在特定的时间与空间的表达[30-31]。在本研究中,E02、E23和E25等3株突变菌株swnK基因的表达水平与野生型菌株相比均发生了显著变化,但swnK基因5′端上游非编码区序列(顺式作用元件)并未发生突变。表明3株突变菌株swnK基因表达水平变化与启动子等顺式作用元件无关,其在转录水平的调控可能是由转录因子等反式作用因子或RNA聚合酶等其他元件来调节。目前仍不清楚与swnK基因互作的反式作用因子有哪些,上述诱变菌株中相关转录因子编码基因是否也发生了突变仍不清楚。由于棘豆链格孢菌SWN基因簇全长约23 kb,受研究成本限制,本研究并未对AATL、swnR、swnN、swnH1及swnH2等基因及其非编码区突变位点进行分析,这些基因核苷酸序列是否发生了突变,并导致转录产物或基因表达水平的变化仍不清楚。进一步的研究应结合全基因组测序、SNP等分析技术,对诱变菌株突变位点进行全面分析,这将为进一步揭示SWN基因簇各基因的分子调控机制奠定基础。
在本研究中,甘肃链格孢菌EA菌株分离自直立黄芪,同属于链格孢属波状芽管孢组真菌,其基因组中同样存在与棘豆链格孢菌高度同源的SWN基因簇,但该菌仅能产生极微量的SW。在以UA003为对照时,EA菌株呈现出与E02、E23和E25等菌株相同的表达模式,尽管培养条件一致,但EA菌株与UA003、E02、E23和E25等菌株的遗传背景和来源不同。突变位点分析结果表明,与棘豆链格孢菌相比,EA菌株swnK基因序列共有163个位点不同,分布于swnK基因的非编码区和编码区的各个区域,其编码的氨基酸序列也有67个位点不同,其转录起始位点也不同,表达产物缺失了一段由15个氨基酸组成的肽链,在AT区域还插入了一段由7个氨基酸组成的短肽,上述位点的差异表明,甘肃链格孢菌EA菌株swnK基因的结构和功能必然与同属的棘豆链格孢菌等疯草内生真菌不同。因此,以UA003为对照菌株,并不能客观地反映EA菌株SWN基因簇各基因的表达模式及其与SW产率的关系。进一步的研究需要对甘肃链格孢菌和棘豆链格孢菌SWN基因簇各基因的结构和功能进行比较分析,这将为阐明棘豆链格孢菌等疯草内生真菌SWN基因簇各基因的功能及其转录调控机制奠定基础。
4 结论EMS能显著改变棘豆链格孢菌的苦马豆素合成能力和SWN基因簇各基因的表达水平。棘豆链格孢菌SWN基因簇中AATL、swnR、swnN、swnH1、swnH2等基因的表达模式与苦马豆素合成呈现负相关趋势,A、KS、SDR和SDRe1等基因的表达模式与苦马豆素合成呈现负相关趋势,EMS突变菌株苦马豆素的合成的变化可能与SWN基因簇相关基因结构和功能的变化有关。本研究可为进一步阐明棘豆链格孢菌等疯草内生真菌SWN基因簇中各基因的功能奠定基础。
| [1] |
MOLYNEUX R J, JAMES L F. Loco intoxication indolizidine alkaloids of spotted locoweed (Astragalus lentiginosus)[J]. Science, 1982, 216(4542): 190-191. DOI:10.1126/science.6801763 |
| [2] |
WU C C, WANG W L, LIU X X, et al. Pathogenesis and preventive treatment for animal disease due to locoweed poisoning[J]. Environ Toxicol Pharmacol, 2014, 37(1): 336-347. DOI:10.1016/j.etap.2013.11.013 |
| [3] |
GARDNER D R, MOLYNEUX R J, RALPHS M H. Analysis of swainsonine: extraction methods, detection, and measurement in populations of locoweeds (Oxytropis spp.)[J]. J Agric Food Chem, 2001, 49(10): 4573-4580. DOI:10.1021/jf010596p |
| [4] |
COOK D, RALPHS M H, WELCH K D, et al. Locoweed poisoning in livestock[J]. Rangelands, 2009, 31(1): 16-21. DOI:10.2111/1551-501X-31.1.16 |
| [5] |
BRAUN K, ROMERO J, LIDDELL C, et al. Production of swainsonine by fungal endophytes of locoweed[J]. Mycol Res, 2003, 107(8): 980-988. DOI:10.1017/S095375620300813X |
| [6] |
PRYOR B M, CREAMER R, SHOEMAKER R A, et al. Undifilum, a new genus for endophytic Embellisia oxytropis and parasitic Helminthosporium bornmuelleri on legumes[J]. Botany, 2009, 87(2): 178-194. DOI:10.1139/B08-130 |
| [7] |
COOK D, GARDNER D R, GRUM D, et al. Swainsonine and endophyte relationships in Astragalus mollissimus and Astragalus lentiginosus[J]. J Agric Food Chem, 2011, 59(4): 1281-1287. DOI:10.1021/jf103551t |
| [8] |
MUKHERJEE S, DAWE A L, CREAMER R. Development of a transformation system in the swainsonine producing, slow growing endophytic fungus, Undifilum oxytropis[J]. J Microbiol Methods, 2010, 81(2): 160-165. DOI:10.1016/j.mimet.2010.02.015 |
| [9] |
LI X, LU P. Transcriptome profiles of Alternaria oxytropis provides insights into swainsonine biosynthesis[J]. Sci Rep, 2019, 9(1): 6021. DOI:10.1038/s41598-019-42173-2 |
| [10] |
LU H, QUAN H Y, REN Z H, et al. The genome of Undifilum oxytropis provides insights into swainsonine biosynthesis and locoism[J]. Sci Rep, 2016, 6: 30760. DOI:10.1038/srep30760 |
| [11] |
COOK D, DONZELLI B G G, CREAMER R, et al. Swainsonine biosynthesis genes in diverse symbiotic and pathogenic fungi[J]. G3 (Bethesda), 2017, 7(6): 1791-1797. DOI:10.1534/g3.117.041384 |
| [12] |
TAN X M, CHEN A J, WU B, et al. Advance of swainsonine biosynthesis[J]. Chin Chem Lett, 2018, 29(3): 417-422. DOI:10.1016/j.cclet.2017.08.040 |
| [13] |
LUO F F, HONG S, CHEN B, et al. Unveiling of swainsonine biosynthesis via a multibranched pathway in fungi[J]. ACS Chem Biol, 2020, 15(9): 2476-2484. DOI:10.1021/acschembio.0c00466 |
| [14] |
YU Y T, ZHAO Q M, WANG J N, et al. Swainsonine-producing fungal endophytes from major locoweed species in China[J]. Toxicon, 2010, 56(3): 330-338. DOI:10.1016/j.toxicon.2010.03.020 |
| [15] |
COOK D, GRUM D S, GARDNER D R, et al. Influence of endophyte genotype on swainsonine concentrations in Oxytropis sericea[J]. Toxicon, 2013, 61: 105-111. DOI:10.1016/j.toxicon.2012.10.018 |
| [16] |
GRUM D, COOK D, GARDNER D R, et al. Influence of seed endophyte amounts on swainsonine concentrations in Astragalus and Oxytropis locoweeds[J]. J Agric Food Chem, 2011, 60(33): 8083-8089. |
| [17] |
LI Y Z, NAN Z B. A new species, Embellisia astragali sp. nov., causing standing milk-vetch disease in China[J]. Mycologia, 2007, 99(3): 406-411. DOI:10.1080/15572536.2007.11832565 |
| [18] |
NOOR A I, NEYAZ M, COOK D, et al. Molecular characterization of a fungal ketide synthase gene among swainsonine-producing Alternaria species in the USA[J]. Curr Microbiol, 2020, 77(9): 2554-2563. DOI:10.1007/s00284-020-02111-2 |
| [19] |
余永涛, 李金荣, 赵清梅, 等. 直立黄芪中产苦马豆素真菌的分离与鉴定[J]. 畜牧兽医学报, 2018, 49(8): 1770-1780. YU Y T, LI J R, ZHAO Q M, et al. Isolation and identification of swainsonine-producing fungi from Astragalus adsurgens pall[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(8): 1770-1780. (in Chinese) |
| [20] |
白晓南, 余永涛, 赵清梅, 等. 变异黄芪中产苦马豆素内生真菌的分离与鉴定[J]. 畜牧与兽医, 2017, 49(10): 95-102. BAI X N, YU Y T, ZHAO Q M, et al. Isolation and identification of swainsonine-producing fungal endophyte from Astragalus variabills bunge[J]. Animal Husbandry & Veterinary Medicine, 2017, 49(10): 95-102. (in Chinese) |
| [21] |
张蕾蕾, 余永涛, 何生虎, 等. 不同因素对疯草内生真菌合成苦马豆素的影响[J]. 畜牧兽医学报, 2015, 46(1): 163-173. ZHANG L L, YU Y T, HE S H, et al. Influence of different factors on swainsonine production in fungal endophyte from locoweed[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(1): 163-173. (in Chinese) |
| [22] |
BELFON R, CREAMER R. RAPD-PCR analysis of fungal endophytes of locoweed[J]. Phytopathology, 2003, 93: S7. |
| [23] |
OLDRUP E, MCLAIN-ROMERO J, PADILLA A, et al. Localization of endophytic Undifilum fungi in locoweed seed and influence of environmental parameters on a locoweed in vitro culture system[J]. Botany, 2010, 88(5): 512-521. DOI:10.1139/B10-026 |
| [24] |
张雨, 朱燕丽, 李博, 等. 金龟子绿僵菌发酵液中苦马豆素含量及催化酶基因mRNA表达分析[J]. 畜牧兽医学报, 2020, 51(4): 881-887. ZHANG Y, ZHU Y L, LI B, et al. mRNA expression analysis of catalytic enzyme gene and content of swainsonine in the fermentation broth of Metarhizium anisopliae[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(4): 881-887. (in Chinese) |
| [25] |
SHAFIQUE S, BAJWA R, SHAFIQUE S. Mutagenesis and genetic characterisation of amylolytic Aspergillus niger[J]. Nat Prod Res, 2010, 24(12): 1104-1114. DOI:10.1080/14786410802617474 |
| [26] |
KUNZ B A, GABRIEL M, KANG X L, et al. DNA repair modifies the site and strand specificity of ethyl methanesulfonate mutagenesis in yeast[J]. Mutagenesis, 1992, 7(6): 461-469. DOI:10.1093/mutage/7.6.461 |
| [27] |
LI Y X, WANG M N, SEE D R, et al. Ethyl-methanesulfonate mutagenesis generated diverse isolates of Puccinia striiformis f. sp. tritici, the wheat stripe rust pathogen[J]. World J Microbiol Biotechnol, 2019, 35: 28. DOI:10.1007/s11274-019-2600-6 |
| [28] |
SHAHID M G, NADEEM M, GULZAR A, et al. Novel ergot alkaloids production from Penicillium citrinum employing response surface methodology technique[J]. Toxins (Basel), 2020, 12(7): 427. DOI:10.3390/toxins12070427 |
| [29] |
SEGA G A. A review of the genetic effects of ethyl methanesulfonate[J]. Mutat Res, 1984, 134(2-3): 113-142. |
| [30] |
WU V W, THIEME N, HUBERMAN L B, et al. The regulatory and transcriptional landscape associated with carbon utilization in a filamentous fungus[J]. Proc Natl Acad Sci U S A, 2020, 117(11): 6003-6013. DOI:10.1073/pnas.1915611117 |
| [31] |
MOJZITA D, RANTASALO A, JÄNTTI J. Gene expression engineering in fungi[J]. Curr Opin Biotechnol, 2019, 59: 141-149. DOI:10.1016/j.copbio.2019.04.007 |
(编辑 白永平)



