2. 杨凌职业技术学院动物工程分院,杨凌 712100
2. Department of Animal Engineering, Yangling Vocational & Technical College, Yangling 712100, China
萨能奶山羊是世界著名的乳用山羊品种,对世界乳用山羊品种的改良和羊乳生产具有重要影响[1]。萨能奶山羊性情温顺,环境适应能力强,产奶量高达600~1 200 kg,乳脂率为3.8%~4.2%,现已遍布世界各国[2]。羊奶不仅营养丰富且酪蛋白结构与人奶相似,脂肪球小使其更易被人体吸收,被欧美国家视为营养精品[3-4]。经过对该品种的不断改良我国已培育出高产品系的西农萨能奶山羊[5-6]。从生产角度考虑,繁殖更多的母羊对需要羊奶的饲养者有利,所以进行奶山羊性别控制的研究可以提高奶山羊的饲养效益,促进奶山羊母羊群体的快速增长,也可以促进奶山羊产业的快速发展。
哺乳动物中第一个发现的促雄性发育基因是位于Y染色体短臂上的锌指蛋白Zfy基因,也被认为是最重要的性别决定基因之一[7-8]。Zfy基因高度保守,是锌指同系物基因。Zfy基因包含11个外显子和一个随机重复区域的13个锌指结构,分别由2个半胱氨酸和组氨酸残基络合1个Zn2+而形成稳定存在的手指状结构[9-11]。该锌指结构域是酸性活化结构域且具有修饰的反式激活潜力,能与靶DNA特异性结合,并通过其核定位信号引导靶基因穿过核膜,精准定位于精子细胞核内DNA上,从而调控其他基因表达,在精子早期发育过程中发挥重要作用[12-13]。随着对性别决定机制的深入研究,2013年Yamauchi等[14]通过敲除小鼠Y染色体上的全部性别决定基因,发现仅需要Y染色体上的睾丸决定因子Sry和精原细胞增殖因子Eif2 s3y即可建立转基因雄性小鼠。研究最初发现,当只有Sry基因时可见胚胎发育睾丸但不能产生生精细胞,加入Eif2 s3y基因后的雄性小鼠虽可产生圆形精子但不能继续发育为成熟精子而导致无法受精[15],研究者推测,Y染色体上负责将精细胞转化为精子的关键基因或许是Zfy2,随后通过转基因技术导入Zfy2后结果表明,仅携带Y染色体上的这3个基因就可使转基因小鼠睾丸产生成熟精子,虽无法正常排精但从睾丸收集精子也可成功体外受精,且胚胎移植后雄性幼鼠携带正常的Y染色体[16]。Nakasuji等[17]利用Cas9技术敲除小鼠Zfy1/2基因导致其精子内的PRSS21、PLCZ1、HTT、PLCD4蛋白表达下调,进而导致精子形态严重异常。此外,Peng等[18]使用RNAi技术沉默Zfy基因,从而获得了显著高于对照组的雌鼠比例(72.3%>50.6%)。由此可见,Zfy基因是与性别控制有关的一个重要基因。
目前,NCBI网站上已有预测的山羊Zfy基因序列,但是山羊的Zfy基因全长克隆未见报道。为探究山羊Zfy基因在性别控制中的作用,本研究克隆了西农萨能奶山羊Zfy基因CDS区全长序列,并对其进行生物信息学分析和同源性比对,同时利用CRISPR/Cas9系统构建稳定的Zfy基因敲除的GFFs细胞株,为山羊Zfy基因的功能、对后代性别比例的影响以及控制性别比例的研究提供理论支撑。
1 材料与方法 1.1 主要材料本研究所用实验动物来自杨凌科元克隆股份有限公司萨能奶山羊种羊场;睾丸采自3只3月龄健康公山羊;3只山羊胎儿分别采自3只成年妊娠45 d的母山羊,胎龄为45 d左右,胎儿组织经特异性性别基因PCR鉴定为两只雄性,一只雌性;胎儿成纤维细胞经采集的山羊胎儿组织块贴壁法分离培养而获得。所有采样过程均遵守实验动物管理条例以及伦理要求,得到本学院实验动物伦理委员会的批准。
1.2 主要试剂PX459/Cas9载体为Addgene公司产品;pMD 19T Simple载体、PrimeSTARⓇ HS DNA Polymerase、反转录试剂盒和Solution Ⅰ DNA Ligation试剂盒为TaKaRa公司产品;T4 DNA连接酶为NEB公司产品;Bbs Ⅰ酶为Thermo公司产品;T7E1酶为Biolabs公司产品;质粒小提试剂盒及胶回收试剂盒为AXYGEN公司产品;DNA提取试剂盒和DH5α感受态由北京天根生化科技有限公司提供;胎牛血清为Gibco公司产品;DF12、Opti-MEM培养液及Lipofectamine 3000为Invitrogen公司产品。
1.3 山羊Zfy基因CDS区序列克隆参照NCBI数据库中预测的山羊Zfy基因mRNA序列信息(登录号:XM_018044893),设计3对特异性引物(表 1)。提取山羊睾丸组织的总RNA,反转录合成cDNA,用表 1中的引物分段扩增Zfy基因的CDS区全序列。PCR反应体系:5×PrimeSTAR Buffer 5 μL,dNTP Mixture 4 μL,cDNA模板1 μL,上、下游引物各0.5 μL,PrimeSTARⓇ HS DNA Polymerase 0.5 μL,ddH2O补足至25 μL。PCR反应程序为:98 ℃预变性2 min;98 ℃变性10 s,59 ℃退火10 s,72 ℃延伸1 min,35个循环;72 ℃ 10 min,4 ℃保存。将扩增产物连接到pMD 19T Simple载体上,随机挑选6个阳性菌液PCR鉴定后送至杨凌奥科生物科技有限公司完成测序,并利用DNAMAN软件进行序列拼接。
|
|
表 1 山羊Zfy基因CDS区的扩增引物 Table 1 The primer sequence for amplifying Zfy CDS region of goat |
本研究根据测序软件DNAMAN拼接得到的山羊Zfy基因CDS区序列进行同源性比对并构建系统进化树,对其蛋白质理化性质、二级结构、三级结构、磷酸化位点以及信号肽等进行生物信息学分析,以便预测山羊Zfy基因的结构和功能。本试验中用到的主要生物信息学工具如表 2所示。
|
|
表 2 生物信息学软件及网站 Table 2 The tools of bioinformatics analysis |
本研究利用打靶网站https://chopchop.cbu.uib.no/针对山羊Zfy基因的CDS区设计sgRNA序列,其中sgRNA序列设计以NGG的PAM原则为基准(NGG为Cas9基因定位的PAM序列),并利用网络工具(http://www.rgenome.net/cas-offinder/) 进行脱靶位点分析,优选4个sgRNA靶位点(表 3),并在sgRNA正义链5′端加上序列CACCG接头,反义链5′端加上序列AAAC接头,可与Bbs Ⅰ酶切后形成的黏性末端互补。将上述引物退火后连接到经Bbs Ⅰ酶切过的PX459/Cas9载体上,挑选阳性菌液提取质粒,用60 mm皿培养GFFs(山羊胎儿成纤维细胞),当细胞汇合度达到80%时用Lipofectamine Ⓡ 3000试剂转染阳性重组载体,12 h后加1 μg·mL-1的嘌呤霉素筛选,48 h后将含嘌呤霉素的培养液换掉,继续培养至GFFs细胞长满时提取细胞基因组。设计针对靶序列的特异性扩增引物(表 4),以筛选到的细胞基因组为模板,对4个打靶载体对应的4个靶位点进行特异性扩增。将PCR产物重新变性、退火,加入T7E1酶进行酶切试验,应用Image软件对切割条带进行灰度分析,计算打靶位点的切割效率。
|
|
表 3 sgRNA克隆引物序列 Table 3 Primer sequence for sgRNA cloning |
|
|
表 4 扩增sgRNA靶点的引物序列 Table 4 Primer sequences for amplification of sgRNA targets |
选择sgRNA切割效率最高的重组打靶载体转染GFFs细胞,转染12 h后加入1 μg·mL-1嘌呤霉素筛选,在药物筛选48 h后,将未转染成功的成纤维细胞去除。每天在显微镜下观察细胞长势,待单克隆细胞长出时将其挑取到48孔板传代培养并保存。提取单克隆细胞基因组,对靶序列进行PCR扩增,连接T载体送测序,根据质粒检测结果分析鉴定Zfy基因是否成功敲除。
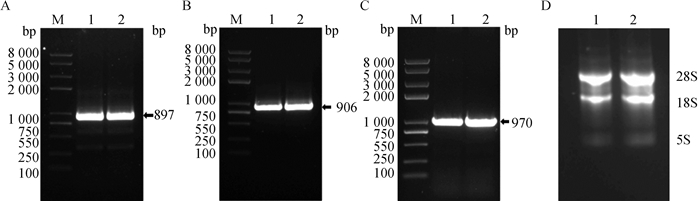
2 结果 2.1 山羊Zfy基因CDS区克隆总RNA经凝胶电泳检测可以清晰地看到28S、18S、5S三条带,证明本试验提取的山羊睾丸组织总RNA完整性良好。利用以总RNA反转录的cDNA第一链为模板对山羊Zfy基因CDS区全长序列进行分段克隆,结果得到了3条特异性且与预期结果一致的条带(图 1)。PCR扩增产物经TA克隆测序拼接比对,成功得到了2 406 bp的西农萨能奶山羊Zfy基因的CDS区全长序列,与NCBI上预测的山羊Zfy基因序列一致(覆盖率100%)。
|
A.Zfy-CDS1 PCR产物;B. Zfy-CDS2 PCR产物;C. Zfy-CDS3 PCR产物;D. 睾丸总RNA琼脂糖凝胶电泳图。M. DNA相对分子质量标准(DL8000);1、2. 两个重复样本 A. PCR products of Zfy-CDS1; B. PCR products of Zfy-CDS2; C. PCR products of Zfy-CDS3; D. Agarose gel electrophoresis of testicular total RNA. M. Trans2K Plus Ⅱ DNA Marker(DL8000); 1, 2. Two replicate samples 图 1 Zfy CDS区分段扩增产物 Fig. 1 Segmented amplification products of Zfy CDS region |
根据NCBI上公布的其他物种的Zfy基因信息,应用MegAlign软件对山羊、绵羊、牛、马鹿、驴、家猫、家犬、人、黑猩猩、小鼠10个物种的Zfy基因进行核苷酸序列及氨基酸序列同源性比对。结果如图 2A和2B所示,山羊与绵羊、牛、马鹿、驴、家猫、家犬、人、黑猩猩、小鼠的Zfy基因CDS区核苷酸序列相似性分别为94.7%、94.3%、97.2%、93.6%、92.7%、92.6%、91.8%、91.4%、83.0%;编码的氨基酸序列相似性分别为98.1%、98.5%、98.9%、97.0%、97.0%、97.3%、95.1%、94.7%、80.7%。应用Mega7.0软件构建Zfy基因系统进化树(图 2C),生物进化树的结果与同源性序列比对结果基本相符,可以发现不同物种之间Zfy基因均比较保守,山羊与马鹿、牛、绵羊亲缘关系较近,与家猫、家犬、驴亲缘关系稍远,与人、黑猩猩、小鼠亲缘关系最远,其中小鼠与其他物种的亲缘性均较低。

|
A.山羊Zfy基因CDS区序列与9个物种的同源性比对;B. 山羊Zfy氨基酸序列与9个物种的同源性比对;C. Zfy基因的系统进化树 A. Homology alignment of CDS region of Zfy gene between goat and 9 kinds of species; B. Homology alignment of amino acid sequence of Zfy between goat and 9 kinds of species; C. The phylogenetic tree of Zfy gene 图 2 山羊Zfy基因的序列分析 Fig. 2 Sequence analysis of Zfy gene of goat |
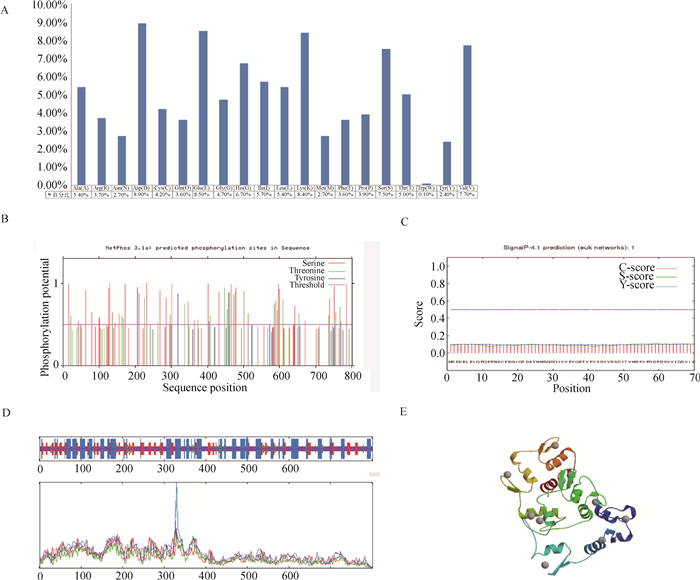
通过ProtParam tool在线软件对山羊Zfy氨基酸序列及蛋白的理化性质进行分析。结果显示,山羊Zfy基因CDS区共编码801个氨基酸,由20种氨基酸组成(各种氨基酸占比如图 3A所示),其中天门冬氨酸(Asp)和谷氨酸(Glu)占比最多;预测其蛋白分子式为C3890H6140N1118O1250S56,原子总数为12 454,分子质量为90.37 ku;理论等电点(pI)为5.66,说明该蛋白是酸性蛋白质;带负电的残基总数(Asp+Glu)为139,带正电荷的残基总数(Arg+Lys)为97;脂溶指数为71.15,蛋白亲水性系数为-0.633,属于亲水性蛋白;不稳定系数为49.52,说明该蛋白不稳定,在哺乳动物网织红细胞内的预计半衰期为30 h;消光系数(M-1 cm-1γ=280 nm)为35 935。
|
A.山羊Zfy蛋白氨基酸比例;B.山羊Zfy蛋白预测磷酸化位点图;C.山羊Zfy蛋白信号肽预测结果;D.山羊Zfy蛋白二级结构;E.山羊Zfy蛋白三级结构模型 A.Amino acid ratio of goat Zfy protein; B.The predicted phosphorylation sites of goat Zfy protein; C.The prediction results of signal peptide in goat Zfy protein; D.The secondary structure of goat Zfy protein; E.The tertiary structure model of Zfy protein in goat 图 3 山羊Zfy蛋白结构分析结果 Fig. 3 Results of structural analysis of goat Zfy protein |
本研究应用软件Net Phos 3.1对山羊Zfy蛋白的磷酸化位点进行预测。预测结果如图 3B所示,山羊Zfy蛋白可能的磷酸化位点有66个,其中Ser(丝氨酸)37个、Thr(苏氨酸)22个、Tyr(酪氨酸)9个。应用软件SignalP 4.1对山羊Zfy蛋白的信号肽进行预测,如图 3C所示。氨基酸残基C-score为0.108,S-score为0.110,Y-score为0.104,3个分值均小于0.5,据计算结果可以预测山羊Zfy蛋白不存在信号肽,说明该蛋白是非分泌蛋白。
利用SOPMA软件预测Zfy蛋白的二级结构,如图 3D所示,在山羊Zfy蛋白的二级结构中,α螺旋占31.21%,且分布比较均匀,β折叠占16.73%,β转角较少,占4.74%,无规则卷曲(random coil部分)占47.32%。山羊Zfy蛋白的二级结构比较复杂,尤其是前端和中间部分。应用在线软件SWISS-MODEL对山羊Zfy基因编码的氨基酸序列进行同源性建模,得到了与二级结构预测结果基本一致的山羊Zfy蛋白三级结构空间模型,如图 3E所示。
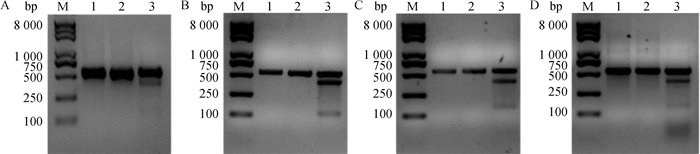
2.4 山羊Zfy基因sgRNA筛选及切割效率检测应用在线软件针对山羊Zfy基因的CDS区预测到8个评分较高的潜在sgRNA靶位点,对其进行脱靶分析,排除可能与Zfx基因结合且碱基错配数≥1的靶位点,最终筛选出4个理想的靶位点。为了获得对Zfy基因打靶效率较高的sgRNA,首先将表 3中合成的4对引物退火形成双链后分别连接到经Bbs Ⅰ酶切后的PX459/cas9载体上构建重组质粒并送测序正确,再将构建好的PX459/cas9-sgRNA1、PX459/cas9-sgRNA2、PX459/cas9-sgRNA3、PX459/cas9-sgRNA4表达载体分别转染GFFs,经嘌呤霉素筛选48 h后提取细胞基因组,用表 4中的引物对4个靶位点进行特异性扩增,均跑出特异性扩增条带。最后将回收的PCR产物通过T7E1酶切后用琼脂糖凝胶电泳检测,发现4个sgRNA均有效剪切了相应的靶序列(图 4)。经Image J灰度分析,计算得出其切割效率大小依次为sgRNA2 (40.86%)>sgRNA3 (31.52%)>sgRNA4 (26.37%)>sgRNA1 (12.31%),其中sgRNA2的切割效率最高达到40.86%。
|
A.sgRNA1切割效率;B.sgRNA2切割效率;C.sgRNA3切割效率;D.sgRNA4切割效率。M.DNA相对分子质量标准(DL8000);1.野生型;2.未加T7E1酶;3.加T7E1酶 A.Cutting efficiency of sgRNA1; B.Cutting efficiency of sgRNA2; C.Cutting efficiency of sgRNA3; D.Cutting efficiency of sgRNA4. M. Trans 2K Plus Ⅱ DNA Marker (DL8000); 1. Wild; 2. No T7E1 enzyme added; 3. Adding T7E1 enzyme 图 4 PX459/Cas9-sgRNA载体切割效率检测 Fig. 4 Cutting efficiency detection of PX459/Cas9-sgRNA vector |
选择sgRNA切割效率最高的PX459/cas9-sgRNA2重组打靶载体转染GFFs细胞,转染12 h后经过嘌呤霉素筛选,并对筛选到的46株单克隆细胞株进行PCR扩增,得到537 bp的特异性条带(图 5A)。将PCR产物胶回收纯化后,连接T载体,通过测序鉴定细胞株中Zfy基因是否发生突变,结果显示在转染PX459/cas9-sgRNA2载体的46株单克隆细胞中,发生Zfy基因突变的有11株,突变率为23.91%,如图 5B所示。

|
A. sgRNA2部分单克隆鉴定;B. PX459/cas9-sgRNA2在目标位点产生indels的DNA测序结果,右侧为对应的插入或缺失比率 A. Partial monoclonal identification of sgRNA2;B. DNA sequencing results of PX459/cas9-sgRNA2 generating indels at the target site, the right side is the corresponding insertion or deletion ratio 图 5 敲除Zfy基因的单克隆GFFs细胞株的鉴定结果 Fig. 5 Identification of monoclonal GFFs cell lines with knockout of Zfy gene |
1983年Mani等[19]首次在非洲爪蟾卵母细胞的转录因子ⅢA中发现了与各种DNA特异性结合的锌指蛋白(ZFP),绝大多数锌指蛋白与基因表达和转录因子有关[20-23]。Page等[24]1987年在基因型为XY的女性Y染色体上检测到唯一丢失的基因片段Zfy编码与DNA结合的锌指蛋白,并认为Zfy基因是睾丸决定因子(TDF),掀起了研究胚胎中Zfy基因表达的热潮,直到1990年Berta等[25]证实SRY才是TDF,之后对Zfy基因的研究极少且仅限于性别鉴定[26-29]。2012年,Decarpentrie等[30]的研究表明,小鼠Zfy1和Zfy2基因在第一次减数分裂粗线期之前高表达后沉默,在圆形精子细胞中Zfy2再次被强烈激活。随后,Vernet等[13, 31]进一步研究发现,Zfy2基因是小鼠次级精母细胞有效完成第二次减数分裂(产生单倍体圆形精子)的必要条件,对于精子头部重组及尾部发育具有重要的作用[32]。这些研究成果为Zfy基因在精子发生过程中的关键作用提供了有力依据,但目前国内外主要以小鼠为模型来研究Zfy基因在早期精子发育中的功能。小鼠具有Zfy1和Zfy2两种Y连锁基因,虽然其高度同源,但在减数分裂期间两种基因的功能仍然不同,而人类、牛、羊等哺乳动物的Y染色体上只有一种Zfy基因,那么其在哺乳动物精子生成及性别控制中究竟起什么作用值得深入探究。
最近的研究报道也有克隆驴、马鹿、猪、牛、犬、绵羊、牦牛[33-37]等动物的Zfy基因,并对Zfy基因功能及进化关系进行简单的预测分析,而关于奶山羊Zfy基因的研究鲜有报道。本研究克隆了全长为2 406 bp的西农萨能奶山羊Zfy基因的CDS区序列,编码801个氨基酸,与GenBank上传的预测序列比对结果一致,说明在克隆目的基因时采用分段扩增的方法能够有效避免产生碱基缺失和突变。山羊Zfy基因碱基序列与除小鼠外的其余8个物种相似性在91.4%~97.2%之间,氨基酸相似性在94.7%~98.9%之间,遗传进化分析结果显示,山羊与马鹿亲缘关系最近,其次是牛和绵羊,这与魏丽敏等[33]分析的马鹿与家牛、野牛、绵羊亲缘关系最近相一致,说明Zfy基因氨基酸同源性高于碱基,总体可信度较高,是较保守的基因序列。而山羊与小鼠Zfy基因碱基和氨基酸相似性分别为83.0%和80.7%,且进化树分析显示两者亲缘关系最远,表明在遗传进化过程中两者的基因组成可能存在差异。生物信息学分析Zfy基因编码的蛋白,结果显示蛋白分子质量为90.37 ku,理论等电点(pI)为5.66,说明该蛋白是酸性蛋白质,推测与其酸性结构域有关。山羊Zfy蛋白不存在信号肽,表明不是分泌蛋白,可能的磷酸化位点有66个,推测其可能通过磷酸化调控与其互作的蛋白从而发挥生理生化功能。二级结构主要由31.21%的α螺旋,16.73%的β折叠和47.32%的无规则卷曲组成,Zfy蛋白中大约占一半的无规则卷曲氨基酸是造成该蛋白不稳定的重要原因,这与理化性质分析其不稳定系数高达49.52相一致,但也有研究者认为半衰期越长的蛋白质稳定性越高[38],而山羊Zfy蛋白的半衰期较长但却是不稳定蛋白,本研究与营瑞文等[35]分析驴Zfy为不稳定蛋白一致,该蛋白的这种特性是否与其在减数分裂时间歇性表达有关值得进一步研究。
性别决定和分化是由众多基因共同调控的复杂过程,其中任何一个基因功能丧失或表达失控都有可能导致相应的性别发育异常。随着Zfy基因在早期Y精子发育中的功能被逐渐阐明,目前最主流的方法是利用RNAi技术沉默Zfy基因mRNA的表达来破坏单倍体Y精子的发育从而达到性别控制,且已经在多种动物上陆续实现[18, 39-41]。但Zhang等[27]研究报道,在绵羊上使用RNAi技术沉默Zfy基因,其后代雌雄比例与对照组无显著差异,分析原因可能是设计RNAi序列时参照了小鼠Zfy基因, 干扰Zfy基因的同时也干扰了Zfx基因的表达。本研究在筛选sgRNA时完全避开了与Zfx匹配的靶位点且以山羊Zfy基因设计sgRNA。相比于RNAi技术,基因编辑技术更有利于详细研究Zfy基因的功能,因为其可以实现对整个基因或某个功能域甚至是定点删除或缺失,而RNAi技术是干扰整条mRNA的表达,这种方式有时难以满足对基因功能深入研究的需要。
4 结论本研究通过分段克隆及测序获得了长为2 406 bp的西农萨能奶山羊Zfy基因CDS区全序列,共编码801个氨基酸。Zfy蛋白亲水性较高,是酸性蛋白,无信号肽,且结构不稳定。同时利用CRISPR/Cas9技术构建了4个敲除奶山羊Zfy基因的打靶载体,通过转染切割效率最高的打靶载体筛选到了能稳定敲除Zfy基因的GFFs细胞株,可为奶山羊Zfy基因的功能研究以及性别控制技术的研发奠定基础。
| [1] |
ARNAL M, LARROQUE H, LECLERC H, et al. Genetic parameters for first lactation dairy traits in the Alpine and Saanen goat breeds using a random regression test-day model[J]. Genet Sel Evol, 2019, 51(1): 43. DOI:10.1186/s12711-019-0485-3 |
| [2] |
常卫华, 王娟红, 崔子龙. 萨能奶山羊胎儿成纤维细胞miRNA文库的构建及生物信息学分析[J]. 中国畜牧兽医, 2020, 47(11): 3436-3444. CHANG W H, WANG J H, CUI Z L. Construction of miRNA library and bioinformatics analysis about fetal fibroblasts of Saanen Dairy Goat[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(11): 3436-3444. (in Chinese) |
| [3] |
耿亚楠, 翁玉楠, 罗军, 等. 西农萨能奶山羊泌乳期产奶量、乳成分及血液生理生化指标变化规律研究[J]. 畜牧兽医学报, 2021, 52(1): 28-41. GENG Y N, WENG Y N, LUO J, et al. Study on change regularity of milk yield, milk composition and blood physiological and biochemical indexes in Xinong Saanen dairy goat during lactation period[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(1): 28-41. (in Chinese) |
| [4] |
BIFFANI S, TIEZZI F, FRESI P, et al. Genetic parameters of weeping teats in Italian Saanen and Alpine dairy goats and their relationship with milk production and somatic cell score[J]. J Dairy Sci, 2020, 103(10): 9167-9176. DOI:10.3168/jds.2020-18175 |
| [5] |
SUN Y X, WANG C N, SUN X M, et al. Characterization of the milk fat globule membrane proteome in colostrum and mature milk of Xinong Saanen goats[J]. J Dairy Sci, 2020, 103(4): 3017-3024. DOI:10.3168/jds.2019-17739 |
| [6] |
SUN Y X, WANG C N, SUN X M, et al. Proteomic analysis of whey proteins in the colostrum and mature milk of Xinong Saanen goats[J]. J Dairy Sci, 2020, 103(2): 1164-1174. DOI:10.3168/jds.2019-17159 |
| [7] |
SEOK S H, KANG S Y, IM Y B, et al. Sex identification using ZFX and ZFY genes in leopard cats (Prionailurus bengalensis euptilurus) in Korea[J]. J Vet Med Sci, 2019, 81(5): 793-798. DOI:10.1292/jvms.18-0693 |
| [8] |
WATERS P D, RUIZ-HERRERA A. Meiotic executioner genes protect the Y from extinction[J]. Trends Genet, 2020, 36(10): 728-738. DOI:10.1016/j.tig.2020.06.008 |
| [9] |
KAM J, GRESSHOFF P M, SHORTER R, et al. The Q-type C2H2 zinc finger subfamily of transcription factors in Triticum aestivum is predominantly expressed in roots and enriched with members containing an EAR repressor motif and responsive to drought stress[J]. Plant Mol Biol, 2008, 67(3): 305-322. DOI:10.1007/s11103-008-9319-3 |
| [10] |
GRANT J, FLANAGAN E, YEE A, et al. Characterization of the DNA binding activity of the ZFY zinc finger domain[J]. Biochemistry, 2010, 49(4): 679-686. DOI:10.1021/bi9018626 |
| [11] |
MARDON G, LUOH S W, SIMPSON E M, et al. Mouse Zfx protein is similar to Zfy-2:each contains an acidic activating domain and 13 zinc fingers[J]. Mol Cell Biol, 1990, 10(2): 681-688. |
| [12] |
MARDON G, PAGE D C. The sex-determining region of the mouse Y chromosome encodes a protein with a highly acidic domain and 13 zinc fingers[J]. Cell, 1989, 56(5): 765-770. DOI:10.1016/0092-8674(89)90680-6 |
| [13] |
VERNET N, MAHADEVAIAH S K, YAMAUCHI Y, et al. Mouse Y-linked Zfy1 and Zfy2 are expressed during the male-specific interphase between meiosis I and meiosis Ⅱ and promote the 2nd meiotic division[J]. PLoS Genet, 2014, 10(6): e1004444. DOI:10.1371/journal.pgen.1004444 |
| [14] |
YAMAUCHI Y, RIEL J M, STOYTCHEVA Z, et al. Two Y genes can replace the entire Y chromosome for assisted reproduction in the mouse[J]. Science, 2013, 343(6166): 69-72. |
| [15] |
MATSUBARA Y, KATO T, KASHIMADA K, et al. TALEN-mediated gene disruption on Y chromosome reveals critical role of EIF2S3Y in mouse spermatogenesis[J]. Stem Cells Dev, 2015, 24(10): 1164-1170. DOI:10.1089/scd.2014.0466 |
| [16] |
YAMAUCHI Y, RIEL J M, RUTHIG V, et al. Mouse Y-encoded transcription factor Zfy2 is essential for sperm formation and function in assisted fertilization[J]. PLoS Genet, 2015, 11(12): e1005476. DOI:10.1371/journal.pgen.1005476 |
| [17] |
NAKASUJI T, OGONUKI N, CHIBA T, et al. Complementary critical functions of Zfy1 and Zfy2 in mouse spermatogenesis and reproduction[J]. PLoS Genet, 2017, 13(1): e1006578. DOI:10.1371/journal.pgen.1006578 |
| [18] |
PENG Q, LI R Y, JIA B, et al. Sex control by Zfy siRNA in the mouse[J]. Theriogenology, 2011, 76(3): 507-511. DOI:10.1016/j.theriogenology.2011.03.002 |
| [19] |
MANI M, KANDAVELOU K, DY F J, et al. Design, engineering, and characterization of zinc finger nucleases[J]. Biochem Biophys Res Commun, 2005, 335(2): 447-457. DOI:10.1016/j.bbrc.2005.07.089 |
| [20] |
KONG N R, BASSAL M A, TAN H K, et al. Zinc finger protein SALL4 functions through an AT-Rich motif to regulate gene expression[J]. Cell Rep, 2021, 34(1): 108574. DOI:10.1016/j.celrep.2020.108574 |
| [21] |
NOMAN A, AQEEL M, KHALID N, et al. Zinc finger protein transcription factors: integrated line of action for plant antimicrobial activity[J]. Microb Pathog, 2019, 132: 141-149. DOI:10.1016/j.micpath.2019.04.042 |
| [22] |
SU J, MIAO X, ARCHAMBAULT D, et al. ZC3H4-a novel Cys-Cys-Cys-His-type zinc finger protein-is essential for early embryogenesis in mice[J]. Biol Reprod, 2021, 104(2): 325-335. DOI:10.1093/biolre/ioaa215 |
| [23] |
HUNING L, KUNKEL G R. The ubiquitous transcriptional protein ZNF143 activates a diversity of genes while assisting to organize chromatin structure[J]. Gene, 2021, 769: 145205. DOI:10.1016/j.gene.2020.145205 |
| [24] |
PAGE D C, MOSHER R, SIMPSON E M, et al. The sex-determining region of the human Y chromosome encodes a finger protein[J]. Cell, 1987, 51(6): 1091-1104. DOI:10.1016/0092-8674(87)90595-2 |
| [25] |
BERTA P, HAWKINS J R, SINCLAIR A H, et al. Genetic evidence equating SRY and the testis-determining factor[J]. Nature, 1990, 348(6300): 448-450. DOI:10.1038/348448a0 |
| [26] |
AASEN E, MEDRANO J F. Amplification of the ZFY and ZFX genes for sex identification in humans, cattle, sheep and goats[J]. Bio/Technology, 1990, 8(12): 1279-1281. |
| [27] |
ZHANG Y S, DU Y C, SUN L R, et al. A genetic method for sex determination in Ovis spp. by interruption of the zinc finger protein, Y-linked (ZFY) gene on the Y chromosome[J]. Reprod Fertil Dev, 2018, 30(9): 1161-1168. DOI:10.1071/RD17339 |
| [28] |
SEOK S H, KANG S Y, IM Y B, et al. Sex identification using ZFX and ZFY genes in leopard cats (Prionailurus bengalensis euptilurus) in Korea[J]. J Vet Med Sci, 2019, 81(5): 793-798. DOI:10.1292/jvms.18-0693 |
| [29] |
席继锋, 袁立岗, 杨楠, 等. 奶牛性别控制试验的效果验证[J]. 江苏农业科学, 2019, 47(23): 195-197. XI J F, YUAN L G, YANG N, et al. Verification on effect of sex control experiment for dairy cattle[J]. Jiangsu Agricultural Sciences, 2019, 47(23): 195-197. (in Chinese) |
| [30] |
DECARPENTRIE F, VERNET N, MAHADEVAIAH S K, et al. Human and mouse ZFY genes produce a conserved testis-specific transcript encoding a zinc finger protein with a short acidic domain and modified transactivation potential[J]. Hum Mol Genet, 2012, 21(12): 2631-2645. DOI:10.1093/hmg/dds088 |
| [31] |
VERNET N, SZOT M, MAHADEVAIAH S K, et al. The expression of Y-linked Zfy2 in XY mouse oocytes leads to frequent meiosis 2 defects, a high incidence of subsequent early cleavage stage arrest and infertility[J]. Development, 2014, 141(4): 855-866. DOI:10.1242/dev.091165 |
| [32] |
VERNET N, MAHADEVAIAH S K, DECARPENTRIE F, et al. Mouse Y-encoded transcription factor Zfy2 is essential for sperm head remodelling and sperm tail development[J]. PLoS One, 2016, 11(1): e0145398. DOI:10.1371/journal.pone.0145398 |
| [33] |
魏丽敏, 孙丽荣, 张永生, 等. 马鹿Zfx/Zfy基因的克隆及序列分析[J]. 中国畜牧兽医, 2016, 43(9): 2272-2278. WEI L M, SUN L R, ZHANG Y S, et al. Cloning and sequence analysis of Zfx/Zfy gene in Cervus elaphus[J]. China Animal Husbandry & Veterinary Medicine, 2016, 43(9): 2272-2278. (in Chinese) |
| [34] |
季文博, 王会, 柴志欣, 等. 西藏牦牛mtDNA-Cytb及ZFY基因遗传多样性与系统进化分析[J]. 家畜生态学报, 2019, 40(11): 12-17. JI W B, WANG H, CHAI Z X, et al. Analysis on genetic diversity and phyletic evolution of mtDNA Cytb & ZFY gene in Tibetan yak[J]. Acta Ecologae Animalis Domastici, 2019, 40(11): 12-17. DOI:10.3969/j.issn.1673-1182.2019.11.003 (in Chinese) |
| [35] |
营瑞文, 张永生, 夏欢, 等. 驴Zfy基因cDNA克隆及生物信息学分析[J]. 中国畜牧兽医, 2018, 45(1): 39-46. YING R W, ZHANG Y S, XIA H, et al. cDNA cloning and bioinformatics analysis of Zfy gene in Equus asinus[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(1): 39-46. (in Chinese) |
| [36] |
熊勇, 徐亚欧. 藏系绵羊ZFX/ZFY基因片段进化分析[J]. 生物技术, 2010, 20(6): 1-5. XIONG Y, XU Y O. Evolution analysis the tibetan sheep ZFXY/ZFY gene fragment[J]. Biotechnology, 2010, 20(6): 1-5. DOI:10.3969/j.issn.1004-311X.2010.06.178 (in Chinese) |
| [37] |
唐榕, 马匆匆, 毛裕民, 等. 奶牛ZFY、ZFX基因片段的克隆及测序[J]. 生物化学与生物物理进展, 1995, 22(1): 60-63. TANG R, MA C C, MAO Y M, et al. Cloning and sequencing of ZFY and ZFX gene from cow[J]. Progress in Biochemistry and Biophysics, 1995, 22(1): 60-63. DOI:10.3321/j.issn:1000-3282.1995.01.003 (in Chinese) |
| [38] |
贾浩, 张小白, 宋晓峰. 人类胞内蛋白半衰期与其亚细胞定位的相关性研究[J]. 计算机与应用化学, 2011, 28(4): 411-414. JIA H, ZHANG X B, SONG X F. Relationship between intracellular protein half-life and subcellular localization in human cells[J]. Computers and Applied Chemistry, 2011, 28(4): 411-414. DOI:10.3969/j.issn.1001-4160.2011.04.007 (in Chinese) |
| [39] |
XI J F, WANG X Z, ZHANG Y S, et al. Sex control by Zfy siRNA in the dairy cattle[J]. Anim Reprod Sci, 2019, 200: 1-6. DOI:10.1016/j.anireprosci.2018.05.015 |
| [40] |
袁立岗, 席继峰, 柳炜, 等. Zfy干扰基因对荷斯坦牛性别控制的效果[J]. 中国奶牛, 2019(3): 18-21. YUAN L G, XI J F, LIU W, et al. Effects of Zfy interference genes on sex control in Holstein cows[J]. China Dairy Cattle, 2019(3): 18-21. (in Chinese) |
| [41] |
彭强, 班谦, 贾斌, 等. siRNA介导小鼠Zfy基因干扰的定量分析[J]. 石河子大学学报: 自然科学版, 2011, 29(4): 448-451. PENG Q, BAN Q, JIA B, et al. Quantitative analysis siRNA mediated silencing Zfy gene of Mus[J]. Journal of Shihezi University: Natural Science, 2011, 29(4): 448-451. DOI:10.3969/j.issn.1007-7383.2011.04.010 (in Chinese) |
(编辑 郭云雁)



