由于我国各地经济文化和地理条件的不同,形成了许多各具特色的地方鸡品种,这些地方鸡资源有丰富的遗传多样性和很大的遗传选择潜力,为地方鸡生产及品种改良提供了素材。江西及周边地区地方鸡品种丰富,江西地方鸡品种有崇仁麻鸡、安义瓦灰鸡、泰和乌鸡等8个地方鸡品种;广东省的地方鸡有惠阳胡须鸡、清远麻鸡等,浙江省的仙居鸡、江苏省的狼山鸡等,是我国地方鸡品种资源的重要组成部分。这些地方鸡品种各具特色,如泰和乌骨鸡的药用性、东乡绿壳蛋鸡所产天然绿壳鸡蛋、安义瓦灰鸡有类似于乌骨鸡的药用价值等。地方鸡品种在早熟性、肉质风味和适应性等方面有突出的特点,但是地方鸡大多生长或繁殖性能较低,所以经济效益较差。为适应市场和经济需求,生产和研究人员引进了经济价值较高的外来品种,外来品种的利用和杂交改良提高了经济效益,但也导致了许多特色基因资源丧失、地方鸡品种群体数量下降以及多样性降低等。因此,开展地方鸡的遗传多样性和系统关系的研究尤为迫切,通过提高资源研究的水平和完整性,可以加强地方鸡遗传资源的保护,为后续资源开发利用提供基础和依据[1-5]。利用多种分子标记对地方鸡的遗传多样性和系统进化的研究已有很多[6-9]。近年来,全基因组SNP芯片被应用到农业的各个方面,包括分子检测、育种以及保种等[10-12],刘彬等[13]利用SNP芯片分析青峪猪保种群体的遗传结构表明, 保种群体在闭锁的继代繁育过程中存在群体遗传多样性损失, 需要加强选配或导入外血以确保青峪猪遗传资源的长期保存。蔡春波等[14]运用SNP芯片评估马身猪保种群体的遗传结构,结果表明马身猪保种群体的遗传多样性较丰富, 但近交程度高, 家系少需扩大保种群体数量, 降低近交系数。许多学者利用鸡的SNP 60K芯片开展遗传多样性分析[15-16]、繁殖、肉质和生长等性状的研究[17-18]。SNP芯片工作原理是利用固定的标记DNA序列与待测基因组发生碱基互补配对,从而精准鉴定遗传变异信息。全基因组SNP芯片的出现为遗传多样性、群体遗传结构以及遗传进化等研究提供了有效的途径和方法,国内外开展了很多基于全基因组的遗传多样性和系统关系的研究[19-24]。
本研究将就江西及周边省份地方鸡品种进行全基因组SNP检测,分析地方鸡品种多样性、群体结构、网络关系及系统进化等。通过对检测结果不同变异位点分析,了解品种的遗传多样性,确定品种间的遗传关系,为地方鸡品种资源的保护和开发利用提供参考。
1 材料与方法 1.1 材料8个地方鸡种共125个血样分别采自各个地方鸡保种场、养殖场和保种中心。各品种样本数量及产地见表 1。每只鸡翅静脉采血,采用DNA提取试剂盒提取血液DNA, 利用DNA浓度仪检测提取的DNA浓度,取部分样稀释至100 ng·mL-1, -20 ℃保存备用。
|
|
表 1 地方鸡品种采样情况 Table 1 Sampling situation of local chicken breeds |
质检合格的DNA样品采用鸡的Illumina 60K芯片进行SNP分型,然后按照以下标准进行质控:先去除性染色体上的位点,再选择SNP检出率大于0.9和最小等位基因频率大于0.01的位点进行后续分析。
1.3 数据处理利用PLINK v1.07软件计算各品种多态性位点比例、SNP位点期望杂合度(He)以及个体间遗传距离,MEGA7.0软件用于碱基变异位点、系统进化分析[25]。GCTA v1.02 [26]软件用于主成分分析。分子差异等级分析(analysis of molecular variance,AMOVA)在ARLEQUIN 3.0统计软件计算获得[27]。使用powermarkerV3.25对样本数据进行杂合度及遗传距离分析。
2 结果 2.1 鸡的60K芯片多态性分析对样本数据进行统计处理, 选择检出率大于90%的样本, 并且MAF(minor allele frequency)大于5%的位点进入后期分析,最终得到45 647个位点上的数据用于群体结构分析, 为消除LD的影响,使用plinkv1.07的IBS聚类法对样本进行筛选,以25个SNP为一个窗口, 5个SNPs为步长,对常染色体上的SNP标记进行筛选,基予得到的独立12 045个SNPs计算样本之间的IBS距离,并进行MDS(多维度)分析,见图 1。不同地区的地方鸡没有明显的地域差异,崇仁麻鸡与其它品种距离较远,可能是因为选择了崇仁麻鸡的选育品种,经过长时间的选育,与其它未选育的地方鸡距离越来越远。
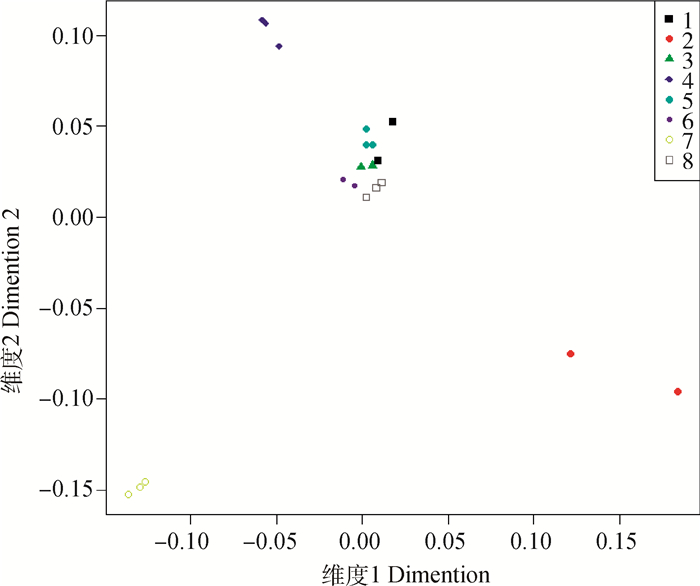
|
1.安义瓦灰鸡;2.崇仁麻鸡;3.惠阳胡须鸡;4.狼山鸡;5.泰和乌鸡;6.仙居鸡;7.东乡绿壳蛋鸡;8.修水黄羽乌鸡 1. Anyi gray chicken; 2. Chongren partridge chicken; 3. Huiyang bearded chicken; 4. Langshan chicken; 5. Taihe black-bone silky chicken; 6. Xianju chicken; 7. Dongxiang green-eggshell chicken; 8. Xiushui yellow feather black chicken 图 1 多维尺度分析群体结构图 Fig. 1 Population structure of multidimensional scaling(MDS) |
选择MAF>0.2的位点,共31 943个。使用powermarkerV3.25对样本数据进行杂合度及遗传距离分析。杂合度是从一个基因库中随机抽出的一个位点的两个拷贝为非纯合基因的概率。遗传杂合度的大小可近似反映出遗传结构变异的高低,遗传杂合度越大,表示品种变异程度越大。本研究的各位点的平均基因多样度0.442 1和平均遗传杂合度0.382 1。多态信息含量值是衡量基因变异程度高低、反映遗传信息的多少的指标。多态信息含量是等位基因频率和等位基因数的变化函数。当位点PIC>0.5时,该位点为高度多态位点;当位点0.25 < PIC < 0.5时为中度多态;当PIC < 0.25时为低度多态位点。根据表 2统计结果,在45 647位点中,表现为中度多态居多。统计了各个位点的等位基因频率(表 3),用于各品种多样性分析。不同品种的等位基因表明8个品种在这些位点上有较丰富的遗传多样性。
|
|
表 2 部分位点杂合度及PIC统计 Table 2 Heterozygosity and PIC statistics of partial loci |
|
|
表 3 部分位点等位基因统计 Table 3 Allele frequency statistics of partial loci |
本研究利用PLINK软件计算所有两两个体间的遗传距离,8个地方鸡种个体间的遗传距离范围在0.000~0.415。品种内部的个体的遗传距离要小于2个品种间的个体的遗传距离,同一品种的所有个体能够聚在一起。品种间的遗传距离见表 4,品种间的遗传距离在0.254 9~0.360 9,修水黄羽乌鸡和泰和乌鸡的遗传距离0.254 9,东乡绿壳蛋鸡和崇仁麻鸡的遗传距离0.306 9。安义瓦灰鸡与其它品种的遗传距离在0.274 7和0.340 8之间,其中和修水黄羽乌鸡遗传距离最近,和崇仁麻鸡遗传距离最远。修水黄羽乌鸡与其它品种有相对较近的遗传距离。
|
|
表 4 8个品种间的遗传距离 Table 4 Genetic distance between 8 breeds |
江西省及周边地区地方鸡遗传资源丰富,是遗传多样性的重要组成部分, 蕴藏着许多独特的性状。遗传多样性越高,种群选择潜力就越大。贾晓旭等[28]对5个江苏省地方鸡品种进行遗传多样性和群体结构分析,结果显示有些群体遗传多样性较低,品种间分化不显著,推测可能受到了外来品种的侵入。该研究结果为江苏地方鸡遗传资源的保护和开发利用提供了遗传背景信息。王珍珍等[29]通过对浙江省境内6个鸡品种的遗传多样性研究,分析了品种间的遗传距离,结果显示6个群体遗传多样性均较高,其中遗传多样性最高是白耳黄鸡,通过多样性研究完善了浙江省主要鸡品种的遗传信息,为地方鸡遗传资源的保护及开发利用提供了基础资料。黄玲玲[30]对浙江省地方鸡品种进行了系统进化研究,结果表明7个群体分为两支, 第1分支为白耳黄鸡和鹊山鸡,第2分支为仙居鸡与龙游麻鸡聚合后, 再与宫廷黄鸡、丝羽乌骨和鸡萧山鸡聚为一起。7个地方鸡品种群体分化程度较低,群体间基因交流水平较高,推测浙江品种鸡群体之间基因流动频繁,从而抑制群体遗传分化。唐修君等[31]利用Cytb基因全序列分析江西4个地方鸡品种多态性和遗传起源,结果显示白耳黄鸡和安义瓦灰鸡的遗传多样性较为丰富,泰和乌鸡群体相对较为稳定。李国辉等[32]基于RAD-seq技术分析边鸡保种群体的遗传进化,结果表明, 边鸡与大骨鸡、安义瓦灰鸡及狼山鸡聚为一类, 亲缘关系较近。基因功能分析的结果有助于了解地方鸡的特性,为我国地方鸡种的遗传进化、种质特性评价及品种保护提供理论依据。本研究利用60 KSNP芯片分析江西及部分周边省份的8个地方鸡品种的遗传多样性,多态信息含量大于0.25,属于中度多态,表明地方鸡有丰富的遗传多样性,泰和乌鸡和修水黄羽乌鸡这两个乌鸡品种相较于其它品种有较近的遗传距离, 与王海龙等[33]研究结果一致,其利用线粒体DNA分析了四川3个地方鸡品种的系统进化关系,发现这3个品种有较高遗传多样性,并且2个乌骨鸡品种聚在一起。
3.2 地方鸡的保种和利用现在地方鸡品种资源主要根据地理位置、表型性状及分布特点来确定, 对地方鸡的起源、遗传多样性信息及遗传关系并没有进行充分研究。随着分子生物技术的迅速发展,研究生物遗传信息的手段有了极大的丰富, 通过遗传背景的研究可以帮助研究人员准确地掌握和科学地分析生物遗传特点和特性,为更好地研究生物本质提供了依据。利用分子生物技术分析种质资源遗传信息, 为有效地保护生物多样性提供科学的依据和手段。Guo等[34]基于表型和基因组信息评价北京油鸡保种群保种情况,结果显示,1979—2019年以来, 北京油鸡保种群体凤冠、胫羽、五趾等典型的外貌特征明显且百分比稳定; 近交系数增长缓慢, 国家级北京油鸡保种场随机交配保种群体的保种工作是十分有效的。Liu等[35]开发了一个55K基因芯片,适用于地方鸡和商业配套系,该芯片可以有广泛的应用,如基因组选择育种,特定性状选育以及鸡种的多样性研究等。将芯片用于地方鸡的保种和利用已有报道,李凯航等[36]采用“京芯一号”SNP芯片对现有60个家系的浦东鸡群体进行了群体结构分析。结果显示,浦东鸡群体遗传多样性较低,选育程度较高,通过聚类分析划分家系,提出适合的配种方案。Li等[37]利用全基因组SNP相互作用分析鸡的体重生长规律,结果表明,鸡体重遗传网络具有动态性和时间依赖性。韩威等[38]利用全基因组SNP标记分析9个地方鸡种的遗传进化,研究结果在全基因组水平上揭示了部分地方鸡种遗传结构和选择驯化,也为进一步发掘重要功能基因奠定了基础。Mao等[39]利用全基因组SNP数据研究江西地方鸡种黑羽选择的群体结构和特征,发现了黑色羽毛的候选基因,为进一步鉴定鸡黑色羽毛突变提供了依据。莫国东等[40]利用微卫星和线粒体DNA分子标记分析了广西三黄鸡的遗传多样性并对其保种效果进行了分析, 结果表明,广西三黄鸡具有较为丰富的遗传多样性, 有些保种场需进一步提纯保种。本研究利用60K SNP芯片分析了8个地方鸡品种的遗传多样性以及品种的遗传距离等,分析各个鸡品种多态位点和特异单倍型,通过对各个鸡品种遗传信息的分析,探讨可能的来源和遗传关系,为地方鸡品种的保种以及专门化品系的培育提供参考。修水黄羽乌鸡是新发现的品种,还未经选育的传统原始品种,遗传多样性高,在保护好特异性品种资源的同时,可以进一步完善良种繁育体系,加强品系选育。泰和乌鸡与修水黄羽乌鸡和安义瓦灰鸡有较近的遗传关系,在后续的研究中,可以针对乌骨鸡进行专门的研究,进一步了解乌骨鸡的起源进化,为乌骨鸡的保种和开发利用提高理论依据。
4 结论通过对8个地方鸡品种的全基因组SNP分析,发现各个品种在各位点的基因多样度和遗传杂合度较高,多态信息含量为中度多态,表明8个地方鸡品种有较丰富的遗传多样性, 可为以后品种开发利用提供遗传素材;MDS(多维度)分析(图 1)发现,不同地区的地方鸡没有明显的地域差异,可能存在不同程度的基因交流;遗传距离分析表明两个乌鸡品种(泰和乌鸡和修水黄羽乌鸡)和安义瓦灰鸡品种有相对较近的遗传关系,为进一步研究乌鸡的起源进化提供了理论基础。
| [1] |
CHEN L, WANG X, CHENG D, et al. Population genetic analyses of seven Chinese indigenous chicken breeds in a context of global breeds[J]. Anim Genet, 2019, 50(1): 82-86. DOI:10.1111/age.12732 |
| [2] |
AZIMU W, MANATBAY B, LI Y, et al. Genetic diversity and population structure analysis of eight local chicken breeds of Southern Xinjiang[J]. Br Poult Sci, 2018, 59(6): 629-635. DOI:10.1080/00071668.2018.1523537 |
| [3] |
WANG M S, THAKUR M, PENG M S, et al. 863 genomes reveal the origin and domestication of chicken[J]. Cell Res, 2020, 30(8): 693-701. DOI:10.1038/s41422-020-0349-y |
| [4] |
LI Q Q, ZHANG P, LI M M, et al. Genetic diversity and relationship of Dulong chickens using mitochondrial DNA control region[J]. Mitochondrial DNA B Resour, 2020, 5(1): 275-280. DOI:10.1080/23802359.2019.1700837 |
| [5] |
武艳平, 霍俊宏, 刘林秀, 等. 江西地方鸡的系统进化及遗传多样性研究[J]. 江西农业大学学报, 2011, 33(6): 1160-1163. WU Y P, HUO J H, LIU L X, et al. Diversity and phylogenetic relationships of Jiangxi chicken breeds[J]. Acta Agriculturae Universitatis Jiangxiensis, 2011, 33(6): 1160-1163. DOI:10.3969/j.issn.1000-2286.2011.06.024 (in Chinese) |
| [6] |
NUNOME M, KINOSHITA K, ISHISHITA S, et al. Genetic diversity of 21 experimental chicken lines with diverse origins and genetic backgrounds[J]. Exp Anim, 2019, 68(2): 177-193. DOI:10.1538/expanim.18-0139 |
| [7] |
ISLAM M A, OSMAN S A M, NISHIBORI M. Genetic diversity of Bangladeshi native chickens based on complete sequence of mitochondrial DNA D-loop region[J]. Br Poult Sci, 2019, 60(6): 628-637. DOI:10.1080/00071668.2019.1655708 |
| [8] |
RASHID M A, MANJULA P, FARUQUE S, et al. Genetic diversity and population structure of indigenous chicken of Bangladesh using microsatellite markers[J]. Asian-Australas J Anim Sci, 2020, 33(11): 1732-1740. DOI:10.5713/ajas.20.0189 |
| [9] |
HABIMANA R, OKENO T O, NGENO K, et al. Genetic diversity and population structure of indigenous chicken in Rwanda using microsatellite markers[J]. PLoS One, 2020, 15(4): e0225084. DOI:10.1371/journal.pone.0225084 |
| [10] |
JIANG B, WANG M, TANG Z, et al. Genome-wide association study of bone mineral density trait among three pig breeds[J]. Animal, 2020, 14(12): 2443-2451. DOI:10.1017/S1751731120001111 |
| [11] |
ISLAM R, LIU X X, GEBRESELASSIE G, et al. Genome-wide association analysis reveals the genetic locus for high reproduction trait in Chinese Arbas Cashmere goat[J]. Genes Genomics, 2020, 42(8): 893-899. DOI:10.1007/s13258-020-00937-5 |
| [12] |
SCHOLTENS M, JIANG A, SMITH A, et al. Genome-wide association studies of lactation yields of milk, fat, protein and somatic cell score in New Zealand dairy goats[J]. J Anim Sci Biotechnol, 2020, 11: 55. DOI:10.1186/s40104-020-00453-2 |
| [13] |
刘彬, 沈林園, 陈映, 等. 基于SNP芯片分析青峪猪保种群体的遗传结构[J]. 畜牧兽医学报, 2020, 51(2): 260-269. LIU B, SHEN L Y, CHEN Y, et al. Analysis of genetic structure of conservation population in Qingyu pig based on SNP chip[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 260-269. (in Chinese) |
| [14] |
蔡春波, 张雪莲, 张万峰, 等. 运用SNP芯片评估马身猪保种群体的遗传结构[J]. 畜牧兽医学报, 2021, 52(4): 920-931. CAI C B, ZHANG X L, ZHANG W F, et al. Evaluation of genetic structure in Mashen pigs conserved population based on SNP chip[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(4): 920-931. (in Chinese) |
| [15] |
ZHANG M M, HAN W, TANG H, et al. Genomic diversity dynamics in conserved chicken populations are revealed by genome-wide SNPs[J]. BMC Genomics, 2018, 19(1): 598. DOI:10.1186/s12864-018-4973-6 |
| [16] |
MANJULA P, BED'HOM B, HOQUE M R, et al. Genetic diversity of MHC-B in 12 chicken populations in Korea revealed by single-nucleotide polymorphisms[J]. Immunogenetics, 2020, 72(6-7): 367-379. DOI:10.1007/s00251-020-01176-4 |
| [17] |
LIU X J, LIU L, WANG J, et al. Genome-wide association study of muscle glycogen in Jingxing Yellow Chicken[J]. Genes (Basel), 2020, 11(5): 497. DOI:10.3390/genes11050497 |
| [18] |
MASTRANGELO S, CENDRON F, SOTTILE G, et al. Genome-wide analyses identifies known and new markers responsible of chicken plumage color[J]. Animals (Basel), 2020, 10(3): 493. |
| [19] |
YIN H D, LI D Y, WANG Y, et al. Whole-genome resequencing analysis of Pengxian Yellow Chicken to identify genome-wide SNPs and signatures of selection[J]. 3 Biotech, 2019, 9(11): 383. DOI:10.1007/s13205-019-1902-6 |
| [20] |
WANG Y, DING X, TAN Z, et al. Genome-wide association study for reproductive traits in a Large White pig population[J]. Anim Genet, 2018, 49(2): 127-131. DOI:10.1111/age.12638 |
| [21] |
刘一帆, 骆科印, 屠云洁, 等. 基于SNP芯片分析贵州鸡资源的遗传多样性和群体遗传结构[C]//中国畜牧兽医学会2018年学术年会禽病学分会第十九次学术研讨会论文集. 南宁, 2018. LIU Y F, LUO K Y, TU Y J, et al. Analysis of genetic diversity and population genetic structure of Guizhou chicken resources based on SNP microarray[C]//Society of Avian Diseases, 2018 annual meeting of China Animal Husbandry and Veterinary Medical Association. Nanning, 2018. (in Chinese) |
| [22] |
MAO H, WANG X, FAN Y, et al. Whole-genome SNP data unravel population structure and signatures of selection for black plumage of indigenous chicken breeds from Jiangxi province, China[J]. Anim Genet, 2019, 50(5): 475-483. DOI:10.1111/age.12827 |
| [23] |
JOHANSSON A M, NELSON R M. Characterization of genetic diversity and gene mapping in two Swedish local chicken breeds[J]. Front Genet, 2015, 6: 44. |
| [24] |
ZHANG C Y, LIN D, WANG Y Z, et al. Widespread introgression in Chinese indigenous chicken breeds from commercial broiler[J]. Evol Appl, 2019, 12(3): 610-621. DOI:10.1111/eva.12742 |
| [25] |
KUMAR S, STECHER G, TAMURA K. MEGA7:Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [26] |
YANG J, LEE S H, GODDARD M E, et al. GCTA: a tool for genome-wide complex trait analysis[J]. Am J Human Genet, 2011, 88(1): 76-82. DOI:10.1016/j.ajhg.2010.11.011 |
| [27] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver3.5:A new series of programs to perform population genetics analyses under Linux and Windows[J]. Mol Ecol Resour, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [28] |
贾晓旭, 唐修君, 樊艳凤, 等. 江苏省地方鸡品种线粒体DNA遗传多样性分析[J]. 农业生物技术学报, 2018, 26(10): 1754-1761. JIA X X, TANG X J, FAN Y F, et al. Genetic diversity analysis of mitochondrial DNA in Jiangsu native chicken (Gallus gallus domesticus) breeds[J]. Journal of Agricultural Biotechnology, 2018, 26(10): 1754-1761. (in Chinese) |
| [29] |
王珍珍, 黄玲玲, 田勇, 等. 浙江省主要地方鸡品种遗传多样性分析[J]. 农业生物技术学报, 2019, 27(11): 2023-2032. WANG Z Z, HUANG L L, TIAN Y, et al. Genetic diversity analysis of major indigenous chicken (Gallus gallus) breeds in Zhejiang province[J]. Journal of Agricultural Biotechnology, 2019, 27(11): 2023-2032. (in Chinese) |
| [30] |
黄玲玲. 浙江地方品种鸡种质调查与遗传结构研究[D]. 福州: 福建农林大学, 2018. HUANG L L. Study on germplasm investigation and genetic structure of Zhejiang indigenous chicken breeds[D]. Fuzhou: Fujian Agriculture and Forestry University, 2018. (in Chinese) |
| [31] |
唐修君, 贾晓旭, 樊艳凤, 等. 基于Cytb基因全序列4个鸡品种遗传多态性和起源[J]. 农业生物技术学报, 2020, 28(6): 1030-1038. TANG X J, JIA X X, FAN Y F, et al. Genetic diversity and origins of four chicken (Gallus domesticus) breeds based on full sequence of Cytb gene[J]. Journal of Agricultural Biotechnology, 2020, 28(6): 1030-1038. (in Chinese) |
| [32] |
李国辉, 魏清宇, 张会永, 等. 基于RAD-seq技术分析边鸡保种群体的遗传进化[J]. 农业生物技术学报, 2019, 27(12): 2198-2206. LI G H, WEI Q Y, ZHANG H Y, et al. The analysis on genetic evolution of bian chicken (Gallus gallus) population based on RAD-seq technology[J]. Journal of Agricultural Biotechnology, 2019, 27(12): 2198-2206. (in Chinese) |
| [33] |
王海龙, 王巧, 邢思远, 等. 基于表型和基因组信息评价北京油鸡保种群保种情况[J]. 畜牧兽医学报, 2021, 52(9): 2406-2415. WANG H L, WANG Q, XING S Y, et al. Evaluating Beijing You chickens conservation status by phenotype and genome information[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(9): 2406-2415. (in Chinese) |
| [34] |
GUO H W, LI C, WANG X N, et al. Genetic diversity of mtDNA D-loop sequences in four native Chinese chicken breeds[J]. Br Poult Sci, 2017, 58(5): 490-497. DOI:10.1080/00071668.2017.1332403 |
| [35] |
LIU R R, XING S Y, WANG J, et al. A new chicken 55K SNP genotyping array[J]. BMC Genomics, 2019, 20(1): 410. DOI:10.1186/s12864-019-5736-8 |
| [36] |
李凯航, 赵乐乐, 陆雪林, 等. 基于SNP芯片的浦东鸡保种分析[J]. 中国家禽, 2020, 42(6): 31-36. LI K H, ZHAO L L, LU X L, et al. Analysis of conservation effect in Pudong Chicken based on SNP chip[J]. China Poultry, 2020, 42(6): 31-36. (in Chinese) |
| [37] |
LI F G, LI H. A time-dependent genome-wide SNP-SNP interaction analysis of chicken body weight[J]. BMC Genomics, 2019, 20(1): 771. DOI:10.1186/s12864-019-6132-0 |
| [38] |
韩威, 李国辉, 王洪志, 等. 基于全基因组SNP标记的鸡遗传进化分析[C]//江苏省遗传学会2016年学术年会. 无锡, 2016. HAN W, LI G H, WANG H Z, et al. Genetic and evolutionary analysis of chicken based on whole genome SNP markers[C]//2016 Annual Meeting of Jiangsu Genetics Society. Wuxi, 2016. (in Chinese) |
| [39] |
MAO H, WANG X, FAN Y, et al. Whole-genome SNP data unravel population structure and signatures of selection for black plumage of indigenous chicken breeds from Jiangxi province, China[J]. Anim Genet, 2019, 50(5): 475-483. DOI:10.1111/age.12827 |
| [40] |
莫国东, 黄英飞, 吴强, 等. 广西三黄鸡的遗传多样性及保种效果分析[J]. 基因组学与应用生物学, 2020, 39(5): 2021-2027. MO G D, HUANG Y F, WU Q, et al. Analysis on genetic diversity and conservation effect of Guangxi Three Yellow Chicken[J]. Genomics and Applied Biology, 2020, 39(5): 2021-2027. (in Chinese) |
(编辑 郭云雁)



